
Stata®︎を使ったメタ解析
今回はStata®︎を使ったメタ解析(メタ分析; meta-analysis)について、一通りの解説noteを記載しました。
システマティックレビューとメタ解析の個々の記事は、上のリンクに細かく記載しています。このnoteでは、主に解析の仕方を中心に、Stata®︎のcodeを書いていきます。基本的にコピー&ペーストで解析ができるようにしています。
データの入力
まずはデータの入力から説明します。
Stataのデータを入力する方法ですが、Excel®︎やCSVファイルをインポートする方法と、直接手入力する方法があります。
今回は手入力について説明しようと思います。手入力する場合は、"input"と"end"というコマンドを利用して、以下のようなコードを組みます。
clear
input trial str24 trialnam str24 authors startyr latitude cases1 tot1 cases0 tot0
1 "Canada" "Ferguson & Simes" 1933 55 6 306 29 303
2 "Northern USA" "Aronson" 1935 52 4 123 11 139
3 "Northern USA" "Stein & Aronson" 1935 52 180 1541 372 1451
4 "Chicago" "Rosenthal et al" 1937 42 17 1716 65 1665
5 "Chicago" "Rosenthal et al" 1941 42 3 231 11 220
6 "Georgia (School)" "Comstock & Webster" 1947 33 5 2498 3 2341
7 "Puerto Rico" "Comstock et al" 1949 18 186 50634 141 27338
8 "UK" "Hart & Sutherland" 1950 53 62 13598 248 12867
9 "Madanapalle" "Frimont-Moller et al." 1950 13 33 5069 47 5808
10 "Georgia (Community)" "Comstock et al." 1950 33 27 16913 29 17854
11 "Haiti" "Vandeviere et al" 1965 18 8 2545 10 629
12 "South Africa" "Coetzee & Berjak" 1965 27 29 7499 45 7277
13 "Madras" "TB Prevention Trial" 1968 13 505 88391 499 88391
endポイントは、inputの後に変数名を入れます。ここでは、
・trial : 研究の順番
・trialnam: 研究の場所
・authors: 著者の名前
・startyr: 年
・latitude: 緯度
・cases1: 治療グループで、アウトカム発症した人数
・tot1: 治療グループの人数
・cases0: コントロールグループで、アウトカム発症した人数
・tot0: コントロールグループの人数
となります。
"str24"は、trialnamやauthorsという変数が、文字変数であることを教えるコマンドです。また、文字変数は"Canada"のように、" "で囲う必要があります。
データの入力が終わったら、endで終了であることを示します。
入力 or インポートしたデータの確認
入力 or インポートしたデータの確認をする場合、以下のコマンドを使用します。
list
データ入力に成功した場合、こちらのようなアウトプットが出てくるでしょう。
アウトカム発症なしのデータも追加
Stata®︎のメタ解析では、「治療あり/なし X アウトカム あり/なし」の2 x 2 = 4通りで解析するため、アウトカムなしのデータも追加する必要があります。
単純に母数からアウトカム発症の人数を引けばようにで、以下のようなコマンドになります:
gen non1=tot1-cases1
gen non0=tot0-cases0メタ解析のパッケージをインストールする
メタ解析をする前に、メタ解析のパッケージをインストールする必要があります。私がよく使用しているのは、metan, metareg, metabias, metacumあたりです。
以下の"findit"のコマンドで探してインストールされると良いでしょう:
*Install packages
findit metan
findit metareg
findit metabias
findit metacum例えば、"findit metacum"をコマンド入力すると、インターネットが繋がった環境であれば、以下の表示が出ます:

sbe22_1を押して次に進みます:
(click here to install)を押していただければ、インストール完了です。
治療効果のメタ解析について
メタ解析によって治療効果を統合して推定する場合、いくつかの方法がありますが、メジャーなものは以下でした:
・固定効果 + Mantel-Haenszel法
・ランダム効果 + Mantel-Haenszel法
・固定効果 + Inverse-Variance法
・ランダム効果 + Inverse-Variance法
それぞれの解析方法をみていきましょう。
固定効果 + Mantel-Haenszel法
固定効果とMantel-Haenszel法の組み合わせでメタ解析をする場合は、以下のコマンドを入力します:
metan cases1 non1 cases0 non0, rr xlab(.1, 1, 10) label (namevar=authors)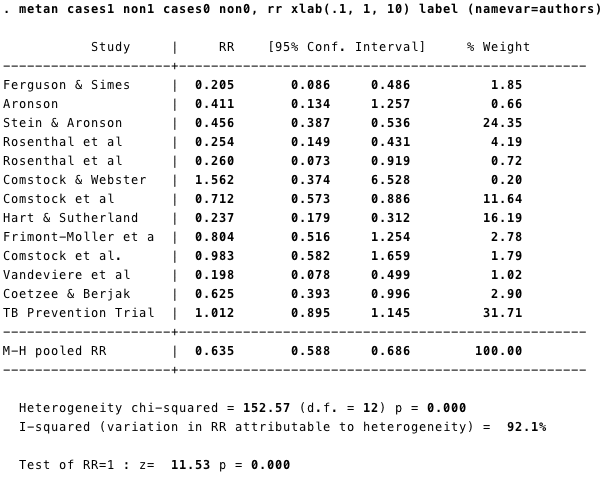
コマンドをきちんと入力すれば、上記のようなアウトプットが出てきます。
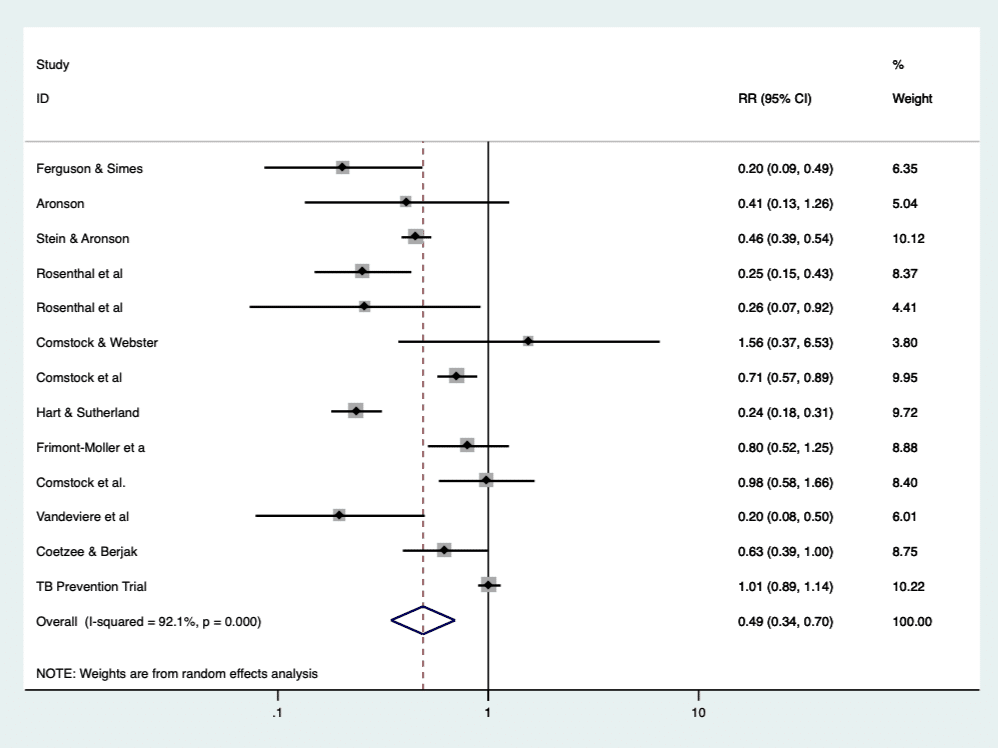
さらに、以下のようなグラフも自動で作成されます:

たった一行のコマンドで、ここまで仕上げてくれます。
ランダム効果 + Mantel-Haenszel法
ランダム効果モデルを使用して、Mantel-Haenszel法でメタ解析する場合は以下の通りです。optionのところに"random"と入れるだけです:
metan cases1 non1 cases0 non0, rr random xlab(.1, 1, 10) label (namevar=authors)以下のようなアウトプットが出てくると思います:

また、以下のような図が自動的に作成されます:

固定効果 + Inverse-Variance法
次に、固定効果 + Inverse-variance法です。この場合はoptionに"fixedi"を入れるだけでOKです:
metan cases1 non1 cases0 non0, fixedi rr xlab(.1, 1, 10) label (namevar=authors)
さらに以下のような図が作成されます。

ランダム効果 + Inverse-Variance法
この場合は"randomi"というコマンドをオプションに入れます:
metan cases1 non1 cases0 non0, randomi rr xlab(.1, 1, 10) label (namevar=authors)
異質性を対処する
こちらの研究では、行われた国や地域の「緯度(latitude)」が異質性の原因としてありそうでした。緯度を低い順に並べてメタ解析する場合は、最初にsortを使ってデータの順番を決め、その後にmetanでメタ解析をします:
sort latitude
metan cases1 non1 cases0 non0, rr xlab(.1, 1, 10) label(namevar=authors)先ほどと順番が変わっているのがわかると思います:

緯度が低いほど(図の上の方)RRは1に近く、緯度が高いほど(図の下の方)RRは小さくなっているようにも見えます。
緯度を「> 35度 vs < 35度」に分けてサブグループにすることで、異質性の対処をしてみましょう。これは "if 〜"のコマンドを入れることで可能となります:
metan cases1 non1 cases0 non0 if latitude > 35, random rr xlab(.1, 1, 10) label(namevar=authors) textsize(200)
metan cases1 non1 cases0 non0 if latitude < 35, random rr xlab(.1, 1, 10) label(namevar=authors) textsize(200)*textsizeで文字の大きさを修正しています
35度以上はこちら:

35度未満はこちら:

累積メタ解析
累積メタ解析やメタ回帰分析を行う場合、対数化したRisk RatioとStandard errorが必要になります。それはこちらのコマンドで計算できます:
gen logrr=log((cases1/tot1)/(cases0/tot0))
gen selogrr=sqrt((non1/(cases1*tot1)) + (non0/(cases0*tot0)))最初の研究、最初 + 2番目の研究、1〜3番目の研究、と研究を年代順に積み重ねていくのを累積メタ解析と言いましたが、そちらのコマンドは以下で行えます:
sort startyr
metacum logrr selogrr, eform label(namevar=authors)最初に"sort startye"で、研究の開始年で並び替えをして、その後に"metacum〜"で、累積メタ解析を行います:

メタ回帰分析
まずは、「35度以上 vs. 以下」で2分した場合の、メタ回帰分析をしてみましょう:
gen higherlat = latitude>=35
metareg logrr higherlat, wsse(selogrr)
緯度が高い方が、log(RR)は、-0.92 [-1.42〜-0.41]低い傾向にあるのが分かります。
緯度が1度上がるとlog(RR)がどのくらい変わるのかは、以下のコマンドで分かります:
metareg logrr latitude, wsse(selogrr)
1度上がる度とに、log(RR)は-0.0245ずつ下がるのが分かります。10度下がる場合はどうでしょうか:
lincom latitude*10
10度上がると、log(RR)は-0.245下がります。
log(RR)ではなく、Risk Ratioで知りたい方は、"eform"をオプションに入れましょう:
metareg logrr latitude, eform wsse(selogrr)
緯度(latitude)や研究が行われた年(startyr)を同時にメタ回帰分析をすることも可能です:
metareg logrr latitude startyr, wsse(selogrr)
以下のようなアウトプットが出てきます。log(RR)でなく、Risk ratioにしたい場合は、eformをオプションに入れます:
metareg logrr latitude startyr, eform wsse(selogrr)
出版バイアス1:Begg test
Begg testは "metabias"のコマンドで可能です:
metabias logrr selogrr, begg
出版バイアス2:Egger test
Egger testのコマンドはこちらになります:
metabias logrr selogrr, egger
出版バイアス3:Funnel plotをみる
これは"metafunnel"を使えば可能です:
metafunnel logrr selogrr
おおよそ左右対称のようにも見えますし、logrrが低い方に偏っているようにも見えますね。どちらかというと後者の意見の方が多そうです。
小技集
ここからはStata®︎の小技を載せていきます。
小技1:Risk DifferenceやOdds ratioを計算する方法
メタ解析をする際、Risk Ratioだけでなく、Risk Differenceも一緒に報告した方が私は好ましいと考えています。というのも、RR = 2であったとしても、RD = 0.1%ということもあり得るからです。
Risk differenceに変換する際には、rdをオプションに入れると行えます:
metan cases1 non1 cases0 non0, rd xlab(-0.05, 0, 0.05) label (namevar=authors)
アウトカムが稀なので、Risk differenceにすると、わずかな予防効果となります。

Risk Ratioを眺めた時とは、だいぶ景色が異なるのではないでしょうか。
metan cases1 non1 cases0 non0, or xlab(.1, 1, 10) label (namevar=authors)"or"を入れて、Odds ratioにしてみましょう:

アウトカムが稀なので、OR≒RRとなります。

予想通り、RRとORのForest plotは似ていますね。
小技2:背景を白にする
Stata®︎に慣れていない方は、背景の色がこの微妙な薄青色のまま論文を提出したりしています。それ自体は問題ないのですが、読んでいると「この人、Stata®︎に慣れていないのかな?」という素人っぽさになってしまいます。
これもoptionにコマンドを加えるだけで解決します:
metan cases1 non1 cases0 non0, rr xlab(.1, 1, 10) label (namevar=authors) ///
graphregion( color(white) ) plotregion( fcolor(white) ) 
単に、背景の色を指定するだけで素人っぽさがかなり消失します。
背景は白い方が、図は綺麗ですよね。
参考文献
メタ解析の勉強は、以下の書籍がオススメです:
こちらの書籍は、Matthias EggerやGeorge-Davey Smithなど、疫学研究者には超有名人の方々が書いた本で、バイブル的な存在です。
良質な記事をお届けするため、サポートしていただけると嬉しいです!英語論文などの資料取り寄せの費用として使用します。
