速報:ケビン・マッカーナン氏の第182回 米FDA ワクチンと関連生物製剤のアドバイサリー・コミッティーにおける発表内容
第182回 米FDA ワクチンと関連生物製剤のアドバイサリー・コミッティー

ケビン・マッカーナン氏の発表
自己紹介
公開するような対立はない。私はゲノムの分野で25年の経験を積んできました。 ホワイトヘッドやMITのヒトゲノム計画で研究開発のチームリーダーとして働いてきました。この分野の論文は57,000件以上引用され、PCR、シークエンスやその他にも複数の特許を有しています。

マッカーナン氏の宣言
COVID-19に関連するビジネスに関与していない。
農業用のqPCRキットを製造販売している。
プレプリント「mRNAワクチンにdsDNAが混入している」を提出している。

COVID-19 mRNAワクチンをイルミナ・シークエンシングとRT-qPCRとqPCRを行ったプレプリントを紹介する。
実際には2023年2月にRNA配列決定ライブラリーのスパイクコントロールとして、mRNAワクチンを使用しました。そしてショックなことに、そのワクチンの発現ベクターがまだワクチンの注射用バイアルに残っていたのです。 10数本のバイアルで実験しましたが、どうやらこの発現ベクターはEMAのガイドラインとFDAのガイドラインを上回っています。このことはここに記載されているプレプリントで確認することができます。

この論文に関するマッカーナン氏のブログを訳した記事へのリンク
COVID-19 mRNAワクチンの工場製造過程にはProcess 1 (IVT) の治験用 と Process 2 (E.Coli) の 市販用がある。このBMJの論文では2種類のプロセスが議論されています。
臨床試験を行ったのは1つ目のプロセスです。in vitroで合成したものをmRNAに転写する方法です。しかし、商業生産するためにスケールアップが必要になりました。どこで大腸菌とベクタープラスミドを使って増幅するプロセス2に製造方法を切り替えました。私たちは、このプラスミドが今もバイアル瓶に残っているのことを発見しました。繰り返しますが、このプラスミドベクターは臨床試験にはなかったものです。

下図は製造工程の模式図です。治験用 Process 1 (in Vitro Transcription: IVT) と、商用 Process 2 (E.Coli) を説明しています。
工場で大腸菌からプラスミドを取り出すのは大変なことのようで、時には残留してしまうこともあ流、ということでしょう。その結果、ワクチン内にプラスミドが残ってしまうことになった。

左はファイザー社のワクチンで発見された混入ベクター配列の模式図です。
ファイザーのワクチンに混入するベクター配列には、344塩基のプロモーターで核局在化シグナルを持つSV40プロモーターが存在しました。モデルナ社製ワクチンにも混入ベクターが存在しましたが、少し構造が違っていました。
FDAに提出された発現ベクターマップでは開示されていませんでした。

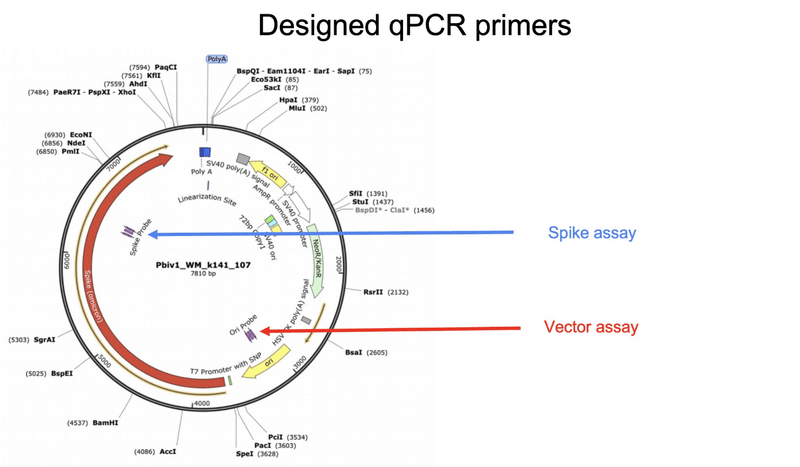
マッカーナン氏らは上図の混入DNAベクターからqPCRプライマーを設計した。
このスパイク配列をターゲットにした定量PCRアッセイをデザインして検証してみました。また、ベクター配列をターゲットにした定量PCRアッセイを開発しました。
混入dsDNAの量を推定しました。
Agilent Tape Station で7から11ng/μl
Qubit で 1から3ng/μl
qPCR/RT-qPCR で 40-80pg/μl
この研究では、qPCRで、ワクチンを100分の1 に希釈してCT 22が得られることが証明されました。ベクターに含まれるDNA配列は商用ワクチンのロットの中にあるべきものではありません。しかし実際には調査したバイアルに含まれていました。調査は 8本の商用バイアルを対象にして、3回に分けて行いました。その結果、EMAとFDAの制限を超える量が含まれていました。

RNAと混入dsDNAの比率を求めた。
EMA は 承認時に混入するdsDNA の上限を 330ng/mg DNA/RNA と決めた。
マッカーマン氏のデータから計算される dsDNA/RNA 量比 から、混入dsDNA量はEMA の上限を18から70倍超えていた。
EMAでは、RNAとDNAの比率を見る比率測定法の制限があり、測定することができます。スパイクとベクターの間に11.5CTのオフセットを期待する必要があります。しかし、5~7CTの差しかなく、18~70倍以上の差があることになります。EMAが推奨する1ミリグラムあたり330ナノグラムの限界値を超えています。


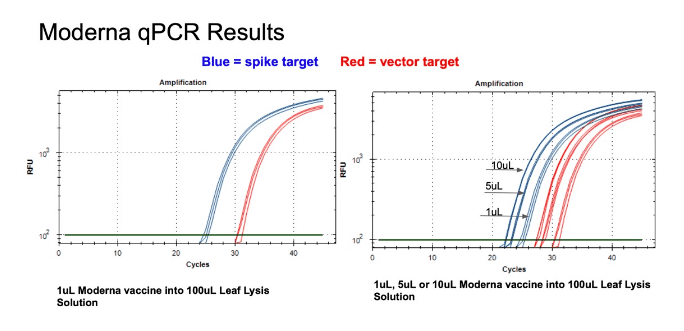
ワクチン5μlを直接qPCRアッセイにかけた。
結果は、CT値 17.5
これはもう世界中のどの研究室でも容易に測定できることでしょう。このワクチンを直接定量PCRにかけると、17という低いCT値を得ることができます。これは非常に重要なことで、COVIDはCTが40以下と診断されましたがこれは注射による汚染は100万倍以上高いのです。COVID-19感染者の鼻腔ぬぐい液に含まれるスパイク蛋白の100万倍以上の汚染物質がワクチン接種による体内に注入されているのです。

マッカーナン氏の調査限界は、調べたワクチンのロットは期限切れであった。
このバイアルは匿名で郵送されてきたもので、コールドチェーン がないことが分かっています。しかし、電気泳動装置 (テープステーション) でRNAの完全性を測定することは可能です。結果は、過去に発表されたものと比べてRNAの完全性に大きな違いはありませんでした。

マッカーナン氏の調査結果を確認した人たちを紹介する。
荒川先生のブログや藤川氏のツイートが紹介されています。
EMAの資料では、バッチごとに 815倍もの差があることが指摘されています。さらに、10ロットの二本鎖DNAの混入がEMAのプロセスで記録されていました。

dsDNAが混入していると血栓症を起こす。

qPCR実験条件です。参考にしてご使用の上、出典を明記してください。
なお、高い有害事象を示したSchmeling et al.にあるような悪いロットは測定していません。私たちは正常と思われるロットを測定していました

皆様へ
マッカーナン氏の発表に含まれるプレプリントの内容等、本日中にこのnote記事に情報追加できると思います
有志研究者の会のかたがたへ
異論がある場合にはプレプリント・サーバーにご投稿ください

下↓のツイッター内の動画をクリックするとマッカーナン氏のプレゼンテーションが始まります
🚩 Kevin McKernan Presents Evidence of COVID-19 Vaccine Contamination to the FDA VRBPAC Advisory Committee
— Chief Nerd (@TheChiefNerd) June 15, 2023
"The Pfizer vaccines specifically had this SV40 promotor which was not disclosed in the expression vector map that was given to the EMA"@Kevin_McKernan pic.twitter.com/HWuz6RSjuo
参照1: Sequencing of bivalent Moderna and Pfizer mRNA vaccines reveals nanogram to microgram quantities of expression vector dsDNA per dose
概要:モデルナとファイザーの二価 mRNA ワクチンの期限切れの 4 本のバイアルの核酸組成を評価するために、いくつかの方法が展開されました。各ベンダーから2本ずつ、イルミナシーケンス、qPCR、RT-qPCR、Qubit™ 3蛍光測定、Agilent Tape Station電気泳動で評価しました。各ベンダーからの 2 つのバイアルが評価されました
複数のアッセイで、欧州医薬品庁(EMA)の330ng/mg要件およびFDAの10ng/投与要件を超えるDNA混入が確認されました。ワクチンmRNAを標的としたRT-qPCRアッセイは、RNaseまたはDNaseヌクレアーゼ処理なしではDNAとRNAを識別できないため、これらのデータは母乳または血漿中のワクチンmRNAの監視に影響するかもしれません。同様に、LINE-1とワクチンmRNAの逆転写酵素活性を評価する研究では、ワクチンに含まれる高レベルのDNA汚染を考慮する必要があります。線状の断片化したDNAと無傷の円形プラスミドDNAの正確な比率は、まだ調査中である。DNA汚染を追跡するために使用される定量的PCRアッセイについて説明する。
はじめに
いくつかの研究で、母乳や血漿中にワクチンのmRNAが長期間存在することが指摘されています(Bansalら2021年、Hannaら2022年、Castruitaら2023年)。これは、ワクチンのmRNAにおけるN1-メチルシュードウリジン(m1Ψ)の安定性の結果であると考えられる。Nanceらは、ワクチンmRNAのインビトロT7ポリメラーゼ合成の前に大腸菌で最初に増幅されるdsDNAプラスミドを利用するワクチンRNA合成法を描いている(Nance and Meier 2021)。このDNAの除去に失敗すると、修飾RNAよりも安定なスパイクコード化核酸が注入される可能性がある。EMAは、RNAに対するDNAの330ng/mgでの制限を明言している(Josephson 2020-11-19)。FDAは、ワクチンにおける10ng/dose未満のガイダンスを発表している(Sheng-Fowler et al.2009)。注入された DNA が残留すると、I 型インターフェロン反応が起こり、DNA の統合の可能性が高まる可能性がある(Ulrich-Lewis et al.、2022)。
結果
ワクチンの核酸組成を評価するために、2 つの異なる方法を使用してワクチン DNA を詳細に配列決定しました。 最初の方法では、市販の New England Biolabs RNA-seq メソッドを使用しました。このメソッドは、RNA の配列決定に有利でありながら、予期しない DNA ベクターを 500 倍以上カバーしました (図 1 および 2)。


RNA-seq アセンブリは、Nance によって記載された構築物と比較して、ポリ A トラクトが短縮されていました。2 番目の方法では、RNase A 処理で RNA を除去し、Watchmaker Genomics フラグメント ライブラリ キットを使用して DNA のみを配列決定しました。 DNA に焦点を当てたアセンブリは、より完全なポリ A トラクトを備えたベクター アセンブリを提供しました (図 3)。

これらのアセンブリは、ワクチン mRNA と DNA ベクターの両方に存在するスパイク配列を標的とし、同時に DNA ベクターにのみ存在する複製起点配列も標的とするマルチプレックス qPCR および RT-qPCR アッセイを設計するために利用されました (図 3)。 ファイザー バイアル 1 のアセンブリには、ファイザー バイアル 2 のアセンブリには存在しない 72 bp の挿入が含まれています。このインデルは知られていない、または SV40 プロモーターおよびその核局在化シグナルに対するその増強です (Dean et al. 1999) (Moreau et al. 1981)
2 つのファイザー バイアルは同じロット番号を共有しているため、2 つのバイアル間でヘテロ接合コピー数の変化が見つかることは予想外です。 ヘテロプラズミックのコピー数変化の出現は、シーケンスライブラリーの挿入サイズが短すぎる (105bp) ため、実際には 72bp 配列の 2 コピーであるものを Megahit アセンブラーが 1 コピーに折りたたんだ結果であるという仮説が立てられました。
ライブラリー内のより長いペアエンドリードにより 72bp のタンデムリピートが解決されることは注目に値します。 参照に 72 bp のリピートのコピーが 1 つあり、サンプルにリピートのコピーが 2 つある場合、図 5A に示すように、リードは単一コピーの 72 bp 遺伝子座のカバレッジの 2 倍に達するはずです。 この仮説を検証するために、2 番目の 72 bp 配列を短いプラスミド アセンブリに追加し、アーティファクトやヘテロプラスミーの証拠がなくリード マップが観察されたことを観察しました (図 5B)。



これらのデータは、すべてのファイザー ベクターが、より強力な発現と核局在化に関連するホモプラスチック 2 コピー 72bp SV40 エンハンサーを含むと結論付けています。 最初のヘテロプラスチック インデルは、Megahit アセンブラーと短いインサート ライブラリーの成果物でした。
DNA のサイズを推定するために、DNA (ゲノム DNA スクリーン テープ) および RNA ベース (高感度 RNA テープ) 電気泳動テープを使用して、精製ワクチンを Agilent Tape Station™ で評価しました。
Agilent Tape Station™ 電気泳動では、各 300 μl サンプルで 23.7 ~ 55.9 ng/μl の mRNA が検出されるのに対し、7.5 ~ 11.3 ng/μl の dsDNA が検出されます。 Qubit™ 3 蛍光分析では、DNA が 1 ~ 2.8 ng/μl、RNA が 21.8 ng ~ 52.8 ng/μl と推定されました。 DNA 電気泳動では、より高度な断片化が見られます。 総 RNA レベルは、予想される 30 ug (100 ng/μl) および 100 ug (200 ng/μl) の用量よりも少なく、DNA および RNA の単離における収量の低下、製造上のばらつき、または期限切れロットによる RNA の劣化を示唆しています。

定量的 PCR アッセイは、Moderna と Pfizer のスパイク配列間で同一であるスパイクタンパク質内の領域とベクターの複製起点の共有配列をターゲットとする IDT Primer Quest ソフトウェアを使用して設計されました。 これにより、ワクチンの qPCR および RT-qPCR 評価が可能になりました。 qPCR は DNA のみを増幅しますが、RT-qPCR は DNA と RNA の両方を増幅します。 グラジエント qPCR を利用して、両方のターゲットが両方の RT-qPCR の同じサイクル条件下で実行される条件を調査しました (グラジエント PCR データは示されていません)。







この研究は、8 つの未開封のファイザー一価ワクチンを qPCR と RT-qPCR の両方でテストすることによってさらに検証されました。




議論
複数の方法により、一価ワクチンと二価ワクチンの両方で高レベルの DNA 汚染が明らかになりました。 Qubit™ 3 と Agilent Tape Station™ は絶対定量の点で異なりますが、両方の方法とも、EMA の制限である 330 ng DNA/1 mg RNA よりも桁違いに高いことが実証されています。 qPCR および RT-qPCR は、相対的な RNA と DNA の比率を確認します。1:3030 の汚染限界 (2^11.6 = 3100) を表すために、スパイク RT-qPCR シグナルとベクター RT-qPCR シグナルの間に 11 ~ 12 CT のオフセットが見られるはずです。
代わりに、これらのワクチンの qPCR および RT-qPCR データを確認すると、はるかに小さい CT オフセット (5 ~ 7 CT) が観察されます。
Qubit™ 3 および Agilent のメソッドは溶液中のすべての DNA を染色しますが、qPCR はプライマー間の DNase I 切断部位を持たずに増幅可能な分子のみを測定することに注意してください。
qPCR プライマーの間隔を離すほど、増幅する Qubit™ 3 および Agilent の検出可能な分子が少なくなります。
この研究で使用したプライマーは 106 bp と 114 bp 離れているため、この長さ未満で切断された DNase I の分子は、Qubit™ 3 または Agilent Tape Station™ によるより一般的な dsDNA 測定と比較して、qPCR メソッドではアンダーカウントされます。
これは、100% インタクトな合成 DNA 標準を使用した qPCR 標準曲線がより効率的に増幅するため、消化された DNA 汚染の合計が過小評価されることも意味します。 たとえば、106 ~ 114 bp の合成テンプレートを使用した標準曲線は、ピコグラム範囲 (低ナノグラム範囲ではない) で 20 未満の CT を示し、ライブラリの大部分が最小増幅可能なサイズより小さいことを示唆しています。 純粋な標準品には高濃度の物質も含まれていません。
同一配列を持つ修飾された mRNA は、競合プライマーシンクまたは qPCR 法の阻害剤として機能する可能性があります。
あるいは、Qubit™ 3 および Agilent Tape Station™ は、N1-メチルプソイドウリジン RNA とのインターカレーション色素クロストークにより DNA 定量を増大させている可能性があります。 このため、qPCR および RT-qPCR の各テンプレート タイプに特異的なポリメラーゼを使用してこれらの分子をより綿密に調査したときに観察された比率が、より関連性のある指標であると考えられます。 EMA メトリクスもそのような比率として記載されています。
これらの EMA 制限が DNA 汚染物質の性質を考慮している場合、このことにも焦点が当てられます。 複製能力のある DNA にはおそらくより厳しい制限があるはずです。 哺乳動物のプロモーターまたは抗生物質耐性遺伝子を含む DNA も、プラスミド調製物からの単なるランダムなバックグラウンド大腸菌ゲノム DNA よりも重要である可能性があります (Sheng-Fowler et al. 2009)。 バックグラウンド大腸菌 DNA は qPCR で測定され、CT は 35 以上でした。SAR-CoV-2 がヒトゲノムに組み込まれる能力については健全な議論が行われてきました (Zhang et al. 2021)。 この研究は、mRNA ワクチンのゲノム統合能力に関する疑問を引き起こしました。 このようなイベントには、Alden et al. が記載しているように、LINE-1 による mRNA の DNA への逆転写が必要となります。 (Alden et al. 2022)。 スパイクタンパク質をコードする配列に dsDNA が混入していれば、逆転写に LINE-1 は必要なく、ファイザーのワクチンベクターに SV40 核局在化シグナルが存在すると、組み込みの可能性がさらに高まるでしょう。 この研究はゲノム統合の証拠を示していないが、これらのワクチンの dsDNA レベルを考慮すると LINE-1 活性が必要ないことを強調している。 これらのベクターの核局在も検証する必要があります。
Jeongらによる一価ワクチンの以前の配列決定では、コンセンサス配列のみが公表されていた(Dae-Eun Jeong 2021)。 このプロジェクトの生のリードは利用できないため、ベクター シーケンスの存在について精査する必要があります。
これらのワクチンが Qubit™ 3 および Agilent のデータで EMA の制限 (330 ng/mg DNA/RNA) を超えており、これらのデータがより保守的な qPCR 標準曲線でも FDA の制限 (10 ng/用量) を超えていることを考えると、リポ多糖 ( LPS)レベルを再検討する必要があります。 E.coli 調製物からのプラスミドの汚染は、LPS と同時汚染されることがよくあります。 エンドトキシンの汚染は、注射時にアナフィラキシーを引き起こす可能性があります(Zheng et al. 2021)。
この研究の限界は、研究中のワクチンバイアルの出所が不明であることである。 これらのバイアルは保冷剤なしで匿名で郵送で送られてきました。 RNA は DNA よりも早く分解することが知られており、保存状態が悪いと DNA よりも RNA の分解が早くなる可能性があります。 分子としての RNA は非常に安定していますが、金属や熱、またはバックグラウンドの遍在 RNase の存在下では、非常に急速に分解する可能性があります。 この研究のワクチンはすべて、バイアルに記載されている有効期限を過ぎており、新鮮なロットの DNA と RNA の比率を理解するにはさらなる研究が必要であることが示唆されています。 これらの qPCR プライマーの公開は、より管理されたサプライ チェーンを備えた追加ロットの調査に役立つ可能性があります。 この配列はワクチンに固有であり、mRNA よりも長く持続する可能性があるため、母乳または血漿中でのワクチンの寿命を評価する研究では、ベクター DNA 監視の恩恵を受ける可能性があります。
配列決定ではプラスミド骨格が完全にカバーされていますが、DNase I で断片化されたライブラリーからプラスミドを組み立てるのが一般的です。 これらの方法では、バイアル内の直鎖状 DNA と環状 DNA の比率は識別されていません。 プラスミド DNA はより適切で安定していますが、直鎖状 DNA はゲノム組み込みのリスクがより高い可能性があります。
Qubit™ 3 および Agilent システムで使用されるインターカレーション色素は、DNA および RNA との蛍光クロストークが低いことが知られていますが、N1-メチルプソイドウリジンがこれらのインターカレーション色素の特異性をどの程度変化させるかは不明です。 結果として、我々は、EMA レシオメトリック規制の最良の相対評価として、RT-qPCR とベクターとスパイク シーケンスを使用した qPCR の間の CT オフセットに依存しました。 これらの qPCR および RT-qPCR 試薬は、将来、ワクチン、血液バンク、または患者組織内のこれらの汚染物質を追跡するのに役立つ可能性があります。
方法
LNP からの mRNA の精製
LiD/SPRI の精製
各バイアルから 100μl をサンプリングしました (用量の 1/3 ~ 1/5)
・100μlのワクチンに5μlの2% LiDを加えてLNPを溶解
・100%イソプロパノール 100μl
・Ampure(Beckman Genomics) 233μl
・25mM MgCl2 25μl (New England Biolabs)
サンプルを 10 回チップミキシングし、磁気ビーズ結合のために 5 分間インキュベートした。 磁性ビーズを96ウェル磁石プレート上で10分間分離し、200μlの80% EtOHで2回洗浄した。
ビーズを3分間風乾させ、100μlのddH2Oで溶出した。 2μl の溶出サンプルを Agilent Tape Station™ で分析した。
ワクチンの CTAB/クロロホルム/SPRI 精製
私達のワクチンの LiD/SPRI 精製方法では、qPCR パフォーマンスに多少のばらつきが認められました。 これにより一部のサンプルが不透明になり、精製時に残留した LNP が存在する可能性があります。 CTAB/クロロホルム/SPRI 分離はこれに対処するために最適化され、さらなる qPCR および Agilent 電気泳動に使用されました。 簡単に説明すると、300μlのワクチンを500μlのCTAB(SenSATIVAx MIP精製キットのMGC溶液A、#420004)に添加した。 次にサンプルをボルテックスし、37℃で5分間加熱しました。 800μlのクロロホルムを加え、ボルテックスし、19,000rpmで3分間回転させた。 水相の上部250μlを収集し、250μlの溶液Bおよび1mlの磁気結合緩衝液に添加した。 サンプルをボルテックスして 5 分間インキュベートし、磁気的に分離しました。 上清を除去し、ビーズを70%エタノールで2回洗浄した。 サンプルは最終的に 300μl の MGC 溶出バッファーで溶出されました。
ワクチン qPCR を評価するための簡単なボイル調製
この煮沸準備プロセスでは、1 ~ 10μl のワクチンを採取し、それを PCR に希釈します。
互換性のある葉溶解バッファーを使用し加熱する (Medicinal Genomics 部品番号 420208)。
・65℃ 6分
・95℃ 2分間
シーケンスのためのライブラリ構築
NEB NEBNext UltraII Directional RNA library Kit for Illumina (NEB#E7760S) を使用して、各 100 μl サンプルのうち 50 μl を Illumina シーケンシング用の RNA-Seq ライブラリーに変換しました。
より長いインサートライブラリーを濃縮するために、プロトコールの長いインサートの推奨に従い、断片化時間を 15 分から 10 分に短縮し、ファーストストランド合成時間を 42℃ で 50 分に延長しました。
ライブラリー内のすべてのフラグメントの最も公平な評価を提供するために、Ribo 枯渇または PolyA 濃縮は実行されませんでした。 製造業者のプロトコールに従って、ライブラリーを 16 サイクル増幅しました。 方向性ライブラリ構築法を使用して、mRNA の一本鎖の性質を評価しました。 dsRNA (>0.5%) は自然免疫応答を誘導する可能性があるため、これは EMA および TGA 開示文書における重要な品質指標です。 dsRNA 含有量は、ELISA を使用して推定されることがよくあります。 方向性 DNA シーケンスは、より包括的な情報を提供します。
この推定方法は以前に測定されており、Jeong et al. では 99.99% でした。
これがロットごとに、または新しい二価ワクチンの新しい製造プロセス内でどのように変化するかは不明です。
ワクチンのRNase A処理
RNase A はウラシルとシトシンの両方を切断する。 N1-メチルプソイドウリジンは RNAse-L 耐性があることが知られているが、RNase A は mRNA にまだ存在するシトシンを切断する。 これにより、主に配列決定用の DNA が残る。 以前に配列決定され、ここで議論されたワクチン mRNA を、NEB の 20 ユニット/μl Monarch RNase A 10μl で 37℃ で 30 分間処理した。 RNase 反応は、1.5 倍の SenSATIVAx (Medicinal Genomics #420001 ) を使用して精製された。DNA精製後、サンプルを20μlのddH2Oで溶出した。 15μlをDNA配列決定に使用した。
ワクチンのDNase処理
50μlのCTAB精製ワクチンを2μlのDNase Iおよび6μlのDNase Iバッファー(Grim reefer MGC#420143)で37℃で30分間処理した。 2.5μlのLiDs Lysisバッファーを添加してDNase反応を停止させた。 反応物は、60μlの100%イソプロパノール、140μlのAmpure、15μlのMgCl2を使用して精製した。 磁気ビーズを 10 回チップミキシングし、5 分間放置してインキュベートし、磁気的に分離し、80% EtOH で 2 回洗浄した。
RNase ワクチンの全ゲノムショットガン。
Watchmakers Genomics WGS ライブラリー構築キットを使用して、15μl の DNA を配列準備の整ったライブラリーに変換した。 このキットは DNA をさらに小さいサイズに断片化するため、ワクチン内の断片長の予測が困難になる。
Qubit™ 3 蛍光分析
Qubit™ 3 蛍光測定は、Biotum AccuBlue RNA Broad Range キット (#31073) および Biotum AccuGreen High Sensitivity dsDNA Quantitation Kit (#31066) を製造元の指示に従って使用して実行した。
大腸菌qPCR
Medicinal Genomics PathoSEEK™ E.coli Detection アッセイ (#420102) を製造業者の指示に従って利用した。
qPCR and RT-qPCR Spike Assay
・MedGen-Moderna_Pfizer_Janssen_Vax-Spike_Forward
・>AGATGGCCTACCGGTTCA
・MedGen-Moderna_Pfizer_Janssen_Vax-Spike_Reverse
・>TCAGGCTGTCCTGGATCTT
・MedGen-Moderna_Pfizer_Janssen_Vax-Spike_Probe
・>/56-FAM/CGAGAACCA/ZEN/GAAGCTGATCGCCAA/3IABkFQ/
qPCR and RT-qPCR Vector Origin Assay
・MedGen_Vax-vector_Ori_Forward
・>CTACATACCTCGCTCTGCTAATC
・MedGen_Vax-vector_Ori_Reverse
・GCGCCTTATCCGGTAACTATC
・MedGen_Vax-vector_Ori_Probe
・/5HEX/AAGACACGA/ZEN/CTTATCGCCACTGGC/3IABkFQ/
IDTの指示に従い、プライマーを100uMに溶出させます。
Make 50X primer-probe mix.
1. 25μl 100uM Forward Primer
2. 25μl 100uM Reverse Primer
3. 12.5μl 100uM Probe
4. 37.5μl nuclease free ddH20.
Use 15μl of this mixture in the qPCR master mix setup seen below. (0.5μl primer/probe per reaction)
Use 10μl of this mixture in the RT-qPCR master mix setup seen below.
Medicinal Genomics Master Mix kits used
1. https://store.medicinalgenomics.com/qPCR-Master-Kit-v3-200-rxns
2. https://store.medicinalgenomics.com/pathoseek-rt-qpcr-master-kit
Reaction setup for 30 reactions of qPCR
・114μl Enzyme Mix (green tube)
・24μl Reaction Buffer (blue tube)
・246μl nuclease free ddH20
・15μl of Primer-Probe set Spike
・15μl of Primer-Probe set Ori
Use 13.8μl of above MasterMix and 5μl of purified sample (1μl Vax DNA/RNA + 4μl ddH20 if CT <15)
Reaction setup for 34 reactions of RT-qPCR
・200μl Enzyme mix
・96μl nuclease free ddH20
・20μl RNase Inhibitor (purple tube)
・4μl DTT (green tube)
・10μl Primer-Probe set Spike
・10μl Primer-Probe set Ori
10μl of MasterMix and 1μl of Vax DNA/RNA
Medicinal Genomics MIP DNA Purification Kit used
1. https://store.medicinalgenomics.com/SenSATIVAx-DNA-Extraction-Kit-200-reactions_2
The CTAB/Chloroform/SPRI based DNA/RNA isolation methods are described above.
サイクリング条件
これらの条件は、qPCR と RT-qPCR の両方に機能します。 注: qPCR では 50°C RT ステップを省略できます。 使用される MGC qPCR MasterMix キットには、この 50°C ステップの影響を受けないホットスタート酵素が含まれています。 RNA と DNA の比較を制御するために、qPCR アッセイと RT-qPCR アッセイを同じプレートに配置し、すべてのサンプルに RT ステップを含めて以下のプログラムを実行しました。
Cycling Conditions used for qPCR and RT-qPCR

Sequences of amplicons for gBlock Positive Controls. Ori = 106bp, Spike = 114bp.

Sequencing Data
Raw Illumina Reads RNA-seq
・Pfizer Bivalent Vial 1 Forward reads
・Pfizer Bivalent Vial 1 Reverse reads
・Pfizer Bivalent Vial 2 Forward reads
・Pfizer Bivalent Vial 2 Reverse reads
・Moderna Vial 1 Forward reads
・Moderna Vial 1 Reverse reads
・Moderna Vial 2 Forward reads
・Moderna Vial 2 Reverse reads
読み取られたファイルは sha256 (ハッシュおよびスタッシュ) を通じて実行され、DASH ブロックチェーンにエッチングされる。 読み取られたファイルの sha256 ハッシュは、不変台帳の OP_RETURN に使用される。 ファイルのハッシュがこれらのトランザクションのハッシュと一致しない場合、ファイルは改ざんされています。
・Pfizer Vial 1 Forward hash
・Pfizer Vial 1 Reverse hash
・Pfizer Vial 2 Forward hash
・Pfizer Vial 2 Reverse hash
Megahit Assemblies
・Pfizer Vial 1
・Pfizer Vial 2
・Moderna Vial 1
・Moderna Vial 2
参照2: Effect of mRNA Vaccine Manufacturing Processes on Efficacy and Safety Still an Open Question
mRNAワクチンの製造プロセスが有効性と安全性に及ぼす影響は依然として未解決の疑問である
この論文の記事はこちら

いつも励ましてくれる大切な人たち
↓ ありがとうございます
この記事が気に入ったらサポートをしてみませんか?
