Nタンパク質の特性解析により、C末端ドメインの複数の機能的影響が明らかに
Characterization of SARS-CoV-2 nucleocapsid protein reveals multiple functional consequences of the C-terminal domain
SARS-CoV-2 ヌクレオカプシドタンパク質の特性解析により、C末端ドメインの複数の機能的影響が明らかになった
Highlights
・Domain-specific contributions of SARS-CoV-2 N to oligomerization and RNA binding are characterized
SARS-CoV-2 N のオリゴマー化と RNA 結合に対するドメイン固有の寄与が特徴付けられる
・Phosphorylation of N modulates RNA-binding affinity and N-RNA solution properties
N のリン酸化は RNA 結合親和性と N-RNA 溶液特性を調節
・HDX-MS results probe RNA binding interface of SARS-CoV-2 N
HDX-MS の結果は SARS-CoV-2 N の RNA 結合インターフェースを調査
・CTD may be a more sensitive and specific serological marker of infection
CTD は感染のより感度が高く特異的な血清学的マーカーである可能性
Summary
Nucleocapsid (N) encoded by severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) plays key roles in the replication cycle and is a critical serological marker.
重症急性呼吸器症候群コロナウイルス 2 (SARS-CoV-2) によってコード化されたヌクレオカプシド (N) は、複製サイクルで重要な役割を果たしており、また重要な血清学的マーカーです。
Here, we characterize essential biochemical properties of N and describe the utility of these insights in serological studies.
ここでは、N の重要な生化学的特性を特徴付け、血清学的研究におけるこれらの洞察の有用性について説明します。
We define N domains important for oligomerization and RNA binding and show that N oligomerization provides a high-affinity RNA-binding platform.
オリゴマー化と RNA 結合に重要な N ドメインを定義し、N オリゴマー化が高親和性の RNA 結合プラットフォームを提供することを示します。
We also map the RNA-binding interface, showing protection in the N-terminal domain and linker region.
また、RNA 結合インターフェイスをマッピングし、N 末端ドメインとリンカー領域での保護を示します。
In addition, phosphorylation causes reduction of RNA binding and redistribution of N from liquid droplets to loose coils, showing how N-RNA accessibility and assembly may be regulated by phosphorylation.
さらに、リン酸化により RNA 結合が減少し、液滴から緩いコイルへの N の再分配が起こり、リン酸化によって N-RNA のアクセス性とアセンブリがどのように制御されるかを示します。
Finally, we find that the C-terminal domain of N is the most immunogenic, based on antibody binding to patient samples.
最後に、患者サンプルへの抗体結合に基づいて、N の C 末端ドメインが最も免疫原性が高いことがわかりました。
Together, we provide a biochemical description of SARS-CoV-2 N and highlight the value of using N domains as highly specific and sensitive diagnostic markers.
これらを組み合わせることで、SARS-CoV-2 N の生化学的説明を提供し、N ドメインを非常に特異性が高く感度の高い診断マーカーとして使用する価値を強調します。
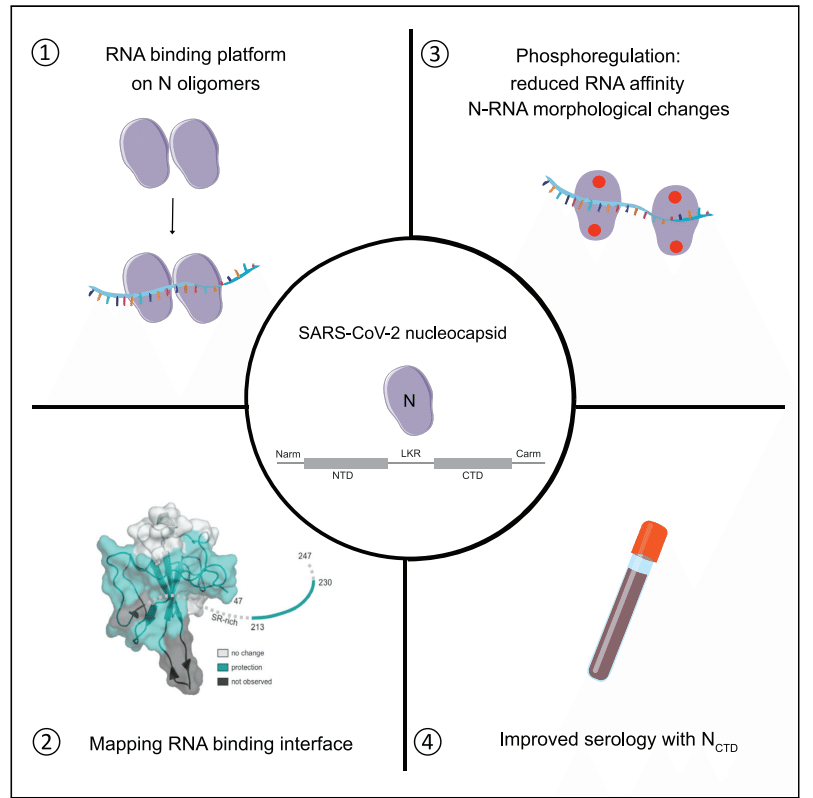
Introduction
Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) is a novel coronavirus and the causative agent of coronavirus disease 2019 (COVID-19).
重症急性呼吸器症候群コロナウイルス 2 (SARS-CoV-2) は、新型コロナウイルスであり、新型コロナウイルス感染症 (COVID-19) の原因物質です。
The coronavirus has a single-stranded, positive-sense RNA genome encoding for four major structural proteins: spike (S), envelope (E), membrane (M), and nucleocapsid (N).
コロナウイルスは、スパイク(S)、エンベロープ(E)、膜(M)、ヌクレオカプシド(N)という4つの主要な構造タンパク質をコードする一本鎖プラスセンスRNAゲノムを持っています。
The N protein is the second-most proximal to the 3′ end of the genome and is one of the most abundantly expressed viral proteins given the multifunctional roles of N during viral replication and assembly.
N タンパク質はゲノムの 3' 末端から 2 番目に近い位置にあり、ウイルスの複製と組み立てにおける N の多機能的役割を考えると、最も豊富に発現しているウイルスタンパク質の 1 つです。
It is estimated that 1,000 copies of N are incorporated into each virion compared with only 100 copies of S.
各ウイルス粒子には N が 1,000 コピー組み込まれているのに対し、S はわずか 100 コピーしか組み込まれていないと推定されています。
N exists mostly in a phosphorylated state in the cytoplasm, whereas it is predominantly dephosphorylated in mature virions, suggesting that N function is regulated by phosphorylation.
N は細胞質内では主にリン酸化状態で存在しますが、成熟したウイルス粒子では主に脱リン酸化されているため、N の機能はリン酸化によって制御されていると考えられます。
A major function of N is to encapsidate the ssRNA viral genome to evade immune detection and to protect the viral RNA from degradation by host factors.
N の主な機能は、ssRNA ウイルスゲノムをカプシド化して免疫検出を回避し、ウイルス RNA を宿主因子による分解から保護することです。
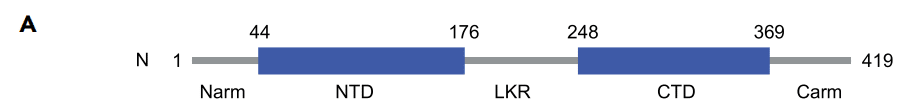
N has two structural domains (Figure 1A): an N-terminal domain (NTD; amino acid residues 44–176) and a C-terminal domain (CTD; amino acid residues 248–369).
N には 2 つの構造ドメインがあります (図 1A)。N 末端ドメイン (NTD; アミノ酸残基 44~176) と C 末端ドメイン (CTD; アミノ酸残基 248~369) です。
NTD is generally referred to as the RNA-binding domain, although regions outside of NTD are implicated in RNA binding as well.
NTD は一般に RNA 結合ドメインと呼ばれますが、NTD 以外の領域も RNA 結合に関与しています。
N-CTD exists as a dimer in solution and can also bind RNA.
N-CTD は溶液中で二量体として存在し、RNA にも結合します。
A conserved serine/arginine-rich linker region (LKR) connects the NTD and CTD (Figures 1A and S1A).
保存されたセリン/アルギニンに富むリンカー領域 (LKR) が NTD と CTD を接続します (図 1A および S1A)。
Phosphorylation of residues in the serine-arginine of the LKR is believed to regulate discontinuous transcription, particularly for shorter subgenomic mRNA closer to the 3′ end during early stages of replication.
LKR のセリン-アルギニン残基のリン酸化は、複製の初期段階で、特に 3' 末端に近い短いサブゲノム mRNA の不連続転写を制御すると考えられています。
The LKR along with residues at the extreme N and C termini (Narm: amino acid residues 1–43 and Carm: amino acid residues 370–419) are intrinsically disordered.
LKR は、N 末端および C 末端の残基 (Narm: アミノ酸残基 1~43、Carm: アミノ酸残基 370~419) とともに本質的に無秩序です。
However, relative to the N-arm and C-arm, the LKR is more conserved (Figures 1B and S1B).
ただし、N-arm および C-arm と比較すると、LKR はより保存されています (図 1B および S1B)。
Given its abundant expression and conservation within the genome, N has been used as an antigen for serology tests.
N は、ゲノム内での豊富な発現と保存性から、血清学的検査の抗原として使用されています。
Previous studies showed that N-specific antibodies dominate the overall antibody response.
これまでの研究では、N 特異的抗体が抗体反応全体を支配していることが示されています。
Furthermore, the T cell responses directed toward N are highly immunodominant in SARS-CoV and SARS-CoV-2 infection, with N-specific memory T cell responses evident 17 years after the initial SARS-CoV infection.
さらに、N に対する T 細胞反応は SARS-CoV および SARS-CoV-2 感染において非常に免疫優位であり、最初の SARS-CoV 感染から 17 年後に N 特異的メモリー T 細胞反応が明らかになっています。
Thus, N protein stability, RNA-binding characteristics, abundance, and conservation are likely to impact T and B cell immunity.
したがって、N タンパク質の安定性、RNA 結合特性、存在量、および保存は、T 細胞および B 細胞の免疫に影響を与える可能性があります。
Previous studies, including our own, revealed shared and unique functions among viral nucleocapsid proteins.
私たち自身の研究を含む以前の研究では、ウイルスのヌクレオカプシドタンパク質に共通する機能と固有の機能が明らかになりました。
These insights include oligomerization, RNA binding, and N-dependent functions such as RNA synthesis and immune evasion.
これらの洞察には、オリゴマー化、RNA 結合、RNA 合成や免疫回避などの N 依存機能が含まれます。
Here, we use a series of biochemical and biophysical assays to dissect different regions in N to determine how oligomerization, RNA binding, and phosphorylation are coupled to functions of N.
ここでは、一連の生化学的および生物物理学的アッセイを使用して、N のさまざまな領域を分析し、オリゴマー化、RNA 結合、およびリン酸化が N の機能とどのように結びついているかを判断します。
Our results reveal that oligomeric N provides a continuous platform for binding RNA with high affinity.
結果から、オリゴマー N は RNA を高い親和性で結合するための継続的なプラットフォームを提供することがわかりました。
Our hydrogen-deuterium exchange mass spectrometry (HDX-MS) data identified a novel RNA-binding region within the LKR in addition to a positively charged patch within the NTD.
水素重水素交換質量分析(HDX-MS)データにより、NTD 内の正電荷パッチに加えて、LKR 内の新しい RNA 結合領域が特定されました。
We also show that phosphorylation modulates N-RNA interactions and solution properties, including phase-separated droplets.
また、リン酸化が N-RNA 相互作用と溶液特性(相分離液滴を含む)を調節することも示しています。
Finally, we find that the CTD contributes to oligomerization and RNA binding, and is efficient at suppressing immune signaling.
最後に、CTD がオリゴマー化と RNA 結合に寄与し、免疫シグナル伝達を抑制するのに効果的であることがわかりました。
In SARS-CoV-2-infected patient plasma samples, the truncated CTD of N provides a highly sensitive serological marker.
SARS-CoV-2 に感染した患者の血漿サンプルでは、N の短縮 CTD が非常に感度の高い血清学的マーカーとなります。
Our studies add to the wealth of information on SARS-CoV-2 N protein and expand upon our knowledge into N-domain-specific functions.
私たちの研究は、SARS-CoV-2 N タンパク質に関する豊富な情報を追加し、N ドメイン固有の機能に関する知識を拡大します。
Results
Multiple regions within N contribute to oligomerization
N内の複数の領域がオリゴマー化に寄与する
While several studies on SARS coronavirus N proteins report that N proteins oligomerize and that N oligomers are important for the assembly of viral particles, the contribution of each N domain to oligomerization remains to be defined.
SARSコロナウイルスのNタンパク質に関するいくつかの研究では、Nタンパク質がオリゴマー化し、Nオリゴマーがウイルス粒子の組み立てに重要であることが報告されていますが、各Nドメインがオリゴマー化にどのように寄与するかはまだ明らかにされていません。
To better understand the regions within SARS-CoV-2 N important for promoting N-N interactions, we first used dynamic light scattering (DLS) to determine the hydrodynamic properties of isolated, RNA-free N domains and combinations of domains.
SARS-CoV-2 N内のN-N相互作用を促進するために重要な領域をよりよく理解するために、まず動的光散乱(DLS)を使用して、単離されたRNAフリーのNドメインとドメインの組み合わせの流体力学的特性を決定しました。
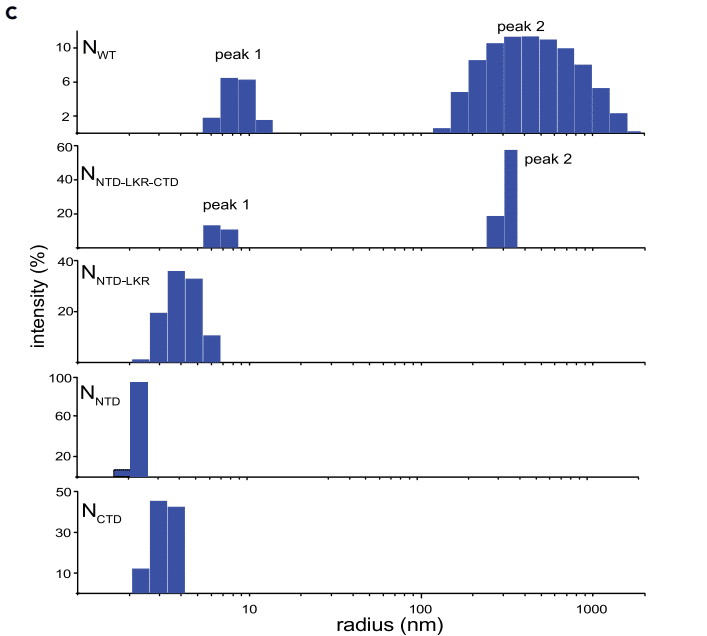
Analysis of DLS results show that there are two major oligomeric species for full-length N (N-WT; 46 kDa), with hydrodynamic radii (Rh) of 8.9 nm and 450 nm (Figures 1C and 1D), suggesting the presence of different oligomeric forms of the protein.
DLS 結果の分析では、全長 N (N-WT; 46 kDa) には 2 つの主要なオリゴマー種があり、その流体力学的半径 (Rh) はそれぞれ 8.9 nm と 450 nm であることが示されており (図 1C と 1D)、タンパク質の異なるオリゴマー形態が存在することが示唆されています。
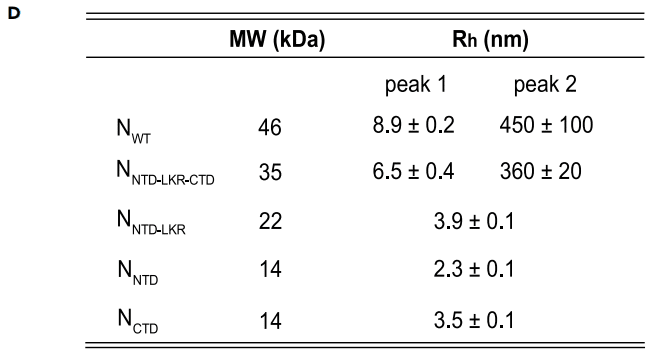
For comparison, the Rh values for maltose binding protein (44 kDa) and BSA (66 kDa) are 2.9 nm and 3.7 nm, respectively.
比較のため、マルトース結合タンパク質 (44 kDa) と BSA (66 kDa) の Rh 値はそれぞれ 2.9 nm と 3.7 nm です。
Removal of the Narm and Carm (NNTD-LKR-CTD) results in two major species that are similar to NWT.
Narm と Carm を除去すると (NNTD-LKR-CTD)、NWT に類似した 2 つの主要種ができます。
However, both NNTD-LKR-CTD populations display reduced polydispersity (narrower peak width, Figure S2A), suggesting that both Narm and Carm contribute to N oligomerization.
ただし、NNTD-LKR-CTD 集団は両方とも多分散性が低下し (ピーク幅が狭くなる、図 S2A)、Narm と Carm の両方が N オリゴマー化に寄与していることが示唆されます。
Further removal of the CTD (NNTD-LKR) results in a single peak representing a dimeric species (Rh = 3.9 nm), but with considerable polydispersity.
CTD をさらに除去すると (NNTD-LKR)、二量体種を表す単一のピーク (Rh = 3.9 nm) になりますが、多分散性はかなり高くなります。
NNTD and NCTD alone form stable domains; NNTD is a monomer (Rh = 2.3 nm), whereas NCTD is a dimer in solution (Rh = 3.5 nm), consistent with previous studies.
NNTD と NCTD のみで安定したドメインが形成されます;NNTD は単量体 (Rh = 2.3 nm) ですが、NCTD は溶液中で二量体 (Rh = 3.5 nm) であり、これは以前の研究と一致しています。
Exact mass measurement by denaturing mass spectrometry yields values corresponding to the mass expected from the amino acid sequence (±1 Da) and supports the identity of the constructs used here (Figures S2B–S2E).
変性質量分析による正確な質量測定では、アミノ酸配列から予想される質量 (±1 Da) に対応する値が得られ、ここで使用した構成の同一性が裏付けられます (図 S2B~S2E)。
Oligomeric N provides a platform for high-affinity RNA binding
オリゴマーNは高親和性RNA結合のプラットフォームを提供
A critical function for N is to bind and encapsidate viral genomic ssRNA.
N の重要な機能は、ウイルスゲノム ssRNA に結合してカプシド化することです。
This interaction is sequence independent and as a result of N-RNA interactions, newly synthesized viral RNA often avoids immune detection by cellular pattern recognition receptors.
この相互作用は配列に依存せず、N-RNA 相互作用の結果として、新しく合成されたウイルス RNA は、細胞パターン認識受容体による免疫検出を回避することがよくあります。
Previous results suggest that regions beyond the NTD are involved in RNA binding, but were limited by experimental detection and only select regions were characterized.
以前の結果では、NTD を超える領域が RNA 結合に関与していることが示唆されていますが、実験的検出によって制限されており、選択された領域のみが特徴付けられています。
To address this limitation and to gain insight into how each domain of SARS-CoV-2 N contributes to RNA binding, we developed a sensitive fluorescence polarization assay to measure binding of a fluorescein isothiocyanate (FITC)-labeled 20-nt ssRNA (sequence: UUUCACCUCCCUUUCAGUUU) (Figure 2A).
この制限に対処し、SARS-CoV-2 N の各ドメインが RNA 結合にどのように寄与するかについての洞察を得るために、フルオレセインイソチオシアネート (FITC) で標識された 20 nt ssRNA (配列: UUUCACCUCCCUUUCAGUUU) の結合を測定するための高感度蛍光偏光アッセイを開発しました (図 2A)。
From this assay, we find that NWT binds the 20-nt ssRNA with high affinity (KD = 0.007 ± 0.001 μM).
このアッセイから、NWTは20-nt ssRNAに高い親和性で結合することがわかりました(KD = 0.007 ± 0.001 μM)。
Removal of the Narm and Carm do not impact ssRNA binding (KD = 0.006 ± 0.002 and 0.006 ± 0.002 μM for NNTD-LKR-CTD-Carm and NNTD-LKR-CTD, respectively) (Figure 2B).
Narm と Carm を除去しても ssRNA 結合には影響しません (NNTD-LKR-CTD-Carm と NNTD-LKR-CTD ではそれぞれ KD = 0.006 ± 0.002 と 0.006 ± 0.002 μM) (図 2B)。
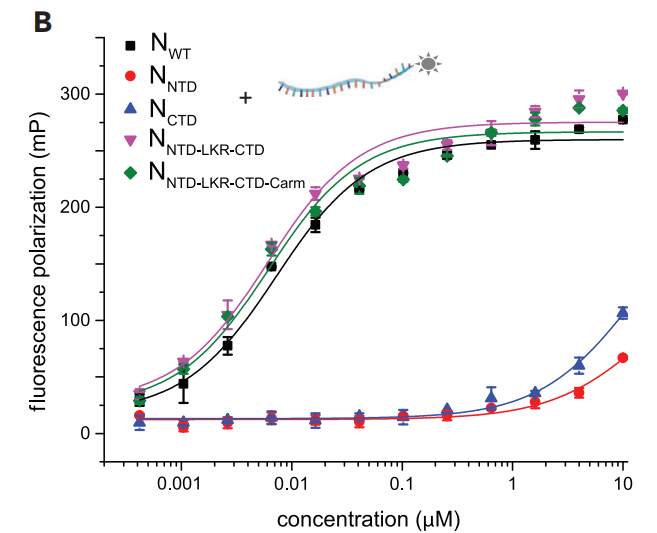
In contrast, the isolated NNTD and NCTD have low-affinity binding (KD = 20 ± 10 and 13 ± 5 μM, respectively).
対照的に、単離された NNTD と NCTD は低親和性結合を示します (それぞれ KD = 20 ± 10 と 13 ± 5 μM)。
However, inclusion of the LKR region increased RNA binding affinity significantly (0.50 ± 0.08 and 0.35 ± 0.04 μM for NNTD-LKR and NLKR-CTD, respectively) (Figures 2B–2D).
ただし、LKR 領域を含めると RNA 結合親和性が大幅に増加します (NNTD-LKR と NLKR-CTD ではそれぞれ 0.50 ± 0.08 と 0.35 ± 0.04 μM) (図 2B~2D)。
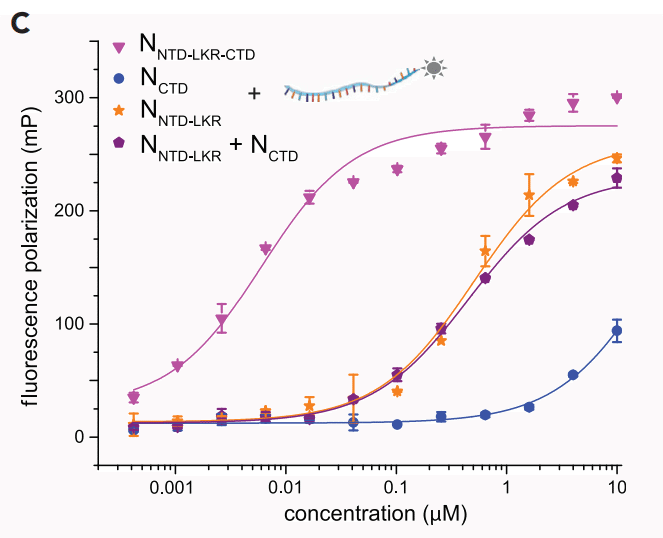
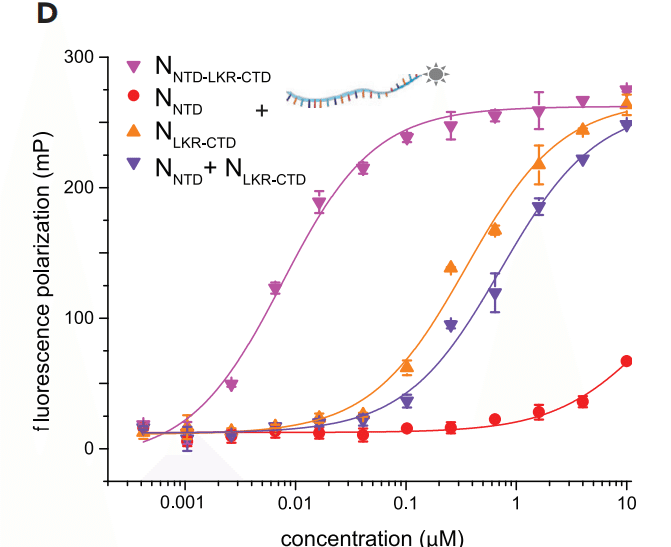
Addition of CTD onto NTD-LKR in cis increases the binding affinity to the single-digit nM range (0.006 ± 0.002 μM) (Figure 2C) but not in trans (compare N-NTD-LKR + N-CTD with N-NTD-LKR-CTD).
CTD をシスで NTD-LKR に追加すると、結合親和性が 1 桁の nM 範囲 (0.006 ± 0.002 μM) に増加します (図 2C) が、トランスでは増加しません (NNTD-LKR + NCTD と NNTD-LKR-CTD を比較)。
The increase in binding affinity also occurs when NTD is added to LKR-CTD in cis (Figure 2D, compare NLKR-CTD + NNTD with NNTD-LKR-CTD).
結合親和性の増加は、NTD をシスで LKR-CTD に加えた場合にも発生します (図 2D、NLKR-CTD + NNTD と NNTD-LKR-CTD を比較)。
Similar binding curves and KD values were obtained when fluorescence polarization values were converted to anisotropy (Figure S3A).
蛍光偏光値を異方性に変換した場合も、同様の結合曲線と KD 値が得られました (図 S3A)。
Collectively, our data quantitatively show that the NTD, CTD, and LKR each contribute to ssRNA binding and that the presence of these three domains in tandem confers N with high-affinity RNA binding, suggesting a more extensive and coupled RNA binding interface on N than is encompassed within a single domain.
全体として、私たちのデータは、NTD、CTD、および LKR がそれぞれ ssRNA 結合に寄与し、これら 3 つのドメインが直列に存在することで N に高親和性の RNA 結合が付与されることを定量的に示しています。 これは、N 上の RNA 結合インターフェースが単一ドメイン内に含まれるものよりも広範囲かつ連結していることを示唆しています。
N is proposed to disrupt dsRNA structures formed by transcription regulatory sequences during discontinuous transcription and impact viral RNA synthesis.
N は、不連続転写中に転写調節配列によって形成された dsRNA 構造を破壊し、ウイルス RNA 合成に影響を与えると考えられています。
To evaluate the impact of N binding to dsRNA structures, we used an RNA that forms a stable stem-loop structure (slRNA) (sequence: GGAAGAUUAAUAAUUUUCC) (Figure 2E).
N が dsRNA 構造に結合することの影響を評価するために、安定したステムループ構造 (slRNA) を形成する RNA (配列: GGAAGAUUAAUAAUUUUCC) を使用しました (図 2E)。

We find that NWT binds this slRNA with relative high affinity (KD = 0.051 ± 0.004 μM), whereas both NNTD and NCTD alone have significantly weaker binding affinities (KD = 120 ± 80 and 60 ± 40 μM, respectively).
NWT は比較的高い親和性 (KD = 0.051 ± 0.004 μM) でこの slRNA に結合するのに対し、NNTD と NCTD の両方を単独で使用した場合の結合親和性は大幅に低下します (それぞれ KD = 120 ± 80 および 60 ± 40 μM)。
Addition of LKR significantly improves binding, consistent with results observed for ssRNA.
LKR を追加すると結合が大幅に改善され、ssRNA で観察された結果と一致しています。
Overall, slRNA binding to N appears to be at an order of magnitude lower than to ssRNA binding (Figure S3B).
全体的に、N への slRNA の結合は、ssRNA の結合よりも 1 桁低いようです (図 S3B)。
This is potentially because, in part, of the energetic penalty of unfolding the stem-loop structure.
これは、ステムループ構造を展開することによるエネルギーペナルティが一部原因である可能性があります。
Furthermore, the Narm and Carm may contribute more to slRNA binding than ssRNA because the impact on N binding is more pronounced after removal of the Narm or Carm (Figure S3C).
さらに、Narm と Carm は、N 結合への影響が Narm または Carm の除去後に顕著になるため、ssRNA よりも slRNA 結合に大きく寄与する可能性があります (図 S3C)。
HDX-MS defines an extended RNA-binding interface in N
HDX-MSはNにおける拡張RNA結合インターフェースを定義
X-ray crystal and NMR structures are available for the SARS-CoV-2 N-NTD and N-CTD as well as hybrid models for RNA-bound N-NTD.
SARS-CoV-2 N-NTD および N-CTD の X 線結晶構造と NMR 構造、および RNA 結合 N-NTD のハイブリッド モデルが利用可能です。
While these static structures provide a wealth of information, a critical gap remains in our knowledge of N-RNA interactions.
これらの静的構造は豊富な情報を提供しますが、N-RNA 相互作用に関する知識には重大なギャップが残っています。
Our studies here show that other N domains in cis contribute significantly to RNA binding.
ここでの私たちの研究は、シス内の他の N ドメインが RNA 結合に大きく貢献していることを示しています。
To further define the RNA-binding interface on N, we performed HDX-MS to locate regions that become protected upon RNA binding.
N 上の RNA 結合界面をさらに定義するために、RNA 結合時に保護される領域を特定するために HDX-MS を実行しました。
We first tested the NNTD-LKR-CTD protein but observed aggregation on RNA binding that caused a 100-fold loss in peptide abundances for the bound state, possibly attributable to aggregation-induced poor digestion.
最初に NNTD-LKR-CTD タンパク質をテストしましたが、RNA 結合時に凝集が観察され、結合状態のペプチド存在量が 100 倍減少しました。これは、凝集によって引き起こされた消化不良に起因する可能性があります。
To circumvent RNA-binding-induced protein aggregation, we reasoned that shorter constructs, such as NNTD-LKR, could be used based on our binding studies.
RNA 結合によるタンパク質凝集を回避するために、結合研究に基づいて、N-NTD-LKR などのより短いコンストラクトを使用できると推論しました。
However, even for this shorter construct, we noticed that the sample became cloudy after mixing with RNA.
ただし、このより短いコンストラクトでも、RNA と混合するとサンプルが濁ることがわかりました。
Next, given phosphorylation of N is a crucial regulatory mechanism in the coronavirus life cycle, we hypothesized that N phosphorylation likely impacts N-RNA solution properties and stability.
次に、N のリン酸化はコロナウイルスのライフサイクルにおける重要な調節メカニズムであるため、N のリン酸化は N-RNA 溶液の特性と安定性に影響を与える可能性が高いという仮説を立てました。
Therefore, we examined phosphomimics of N for improved sample stability and solubility that are compatible with HDX-MS studies.
そのため、サンプルの安定性と溶解性を改善し、HDX-MS 研究に適合する N のリン酸化模倣体を検討しました。
For SARS-CoV, glycogen synthase kinase (GSK)-3 was shown to phosphorylate N at Ser177 (corresponding to Ser176 in SARS-CoV-2 N).
SARS-CoV の場合、グリコーゲン合成酵素キナーゼ (GSK)-3 は、N を Ser177 (SARS-CoV-2 N の Ser176 に相当) でリン酸化することが示されました。
Phosphorylation of Ser177 is preceded by phosphorylation of Ser189 and Ser207 (Ser188 and 206 in SARS-CoV-2 N) by priming kinases.
Ser177 のリン酸化の前に、プライミングキナーゼによる Ser189 と Ser207 (SARS-CoV-2 N の Ser188 と 206) のリン酸化が行われます。
While more phosphorylation events may occur, we focused on these three better-known positions to evaluate if introduction of S176D/S188D/S206D mutations into NNTD-LKR (NNTD-LKR S176D/S188D/S206D) will resolve N protein aggregation at the concentrations of interest.
リン酸化イベントは他にも発生する可能性がありますが、NNTD-LKR (NNTD-LKR S176D/S188D/S206D) への S176D/S188D/S206D 変異の導入により、対象濃度での N タンパク質凝集が解消されるかどうかを評価するために、これら 3 つのよく知られている位置に焦点を当てました。
With the introduction of the mutations on a shorter construct, NNTD-LKR S176D/S188D/S206D, we were able to use sequential FXIII and pepsin digestion to recover 152 peptides, resulting in 93.3% sequence coverage (Figure S4), which enabled us to further examine the system by HDX-MS.
より短いコンストラクトであるNNTD-LKR S176D/S188D/S206Dに変異を導入することで、FXIIIとペプシンによる連続消化を使用して152個のペプチドを回収することができ、93.3%の配列カバレッジが得られました(図S4)。これにより、HDX-MSでシステムをさらに調べることができました。
HDX-MS analysis of NNTD-LKR S176D/S188D/S206D shows clear protection in four distinct regions on RNA binding (amino acid residues 41–63, 105–108, and 146–171 in NTD, and 213–230 in LTR) (Figures 3A and 3B and Mendeley data set https://doi.org/10.17632/sv8r6phkzt.1).
NNTD-LKR S176D/S188D/S206DのHDX-MS分析では、RNA結合の4つの異なる領域(NTDではアミノ酸残基41~63、105~108、146~171、LTRでは213~230)で明確な保護が示されています(図3Aと3B)。
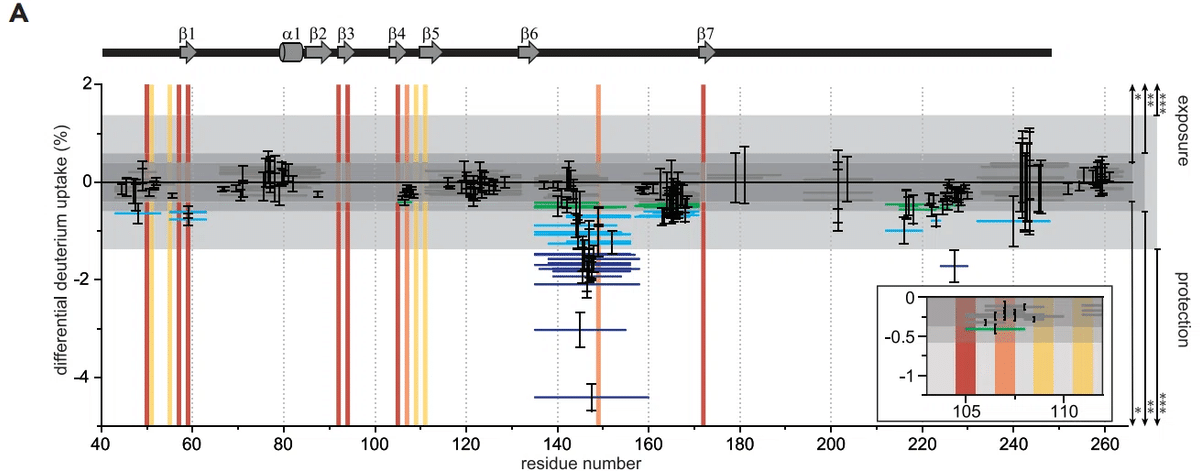
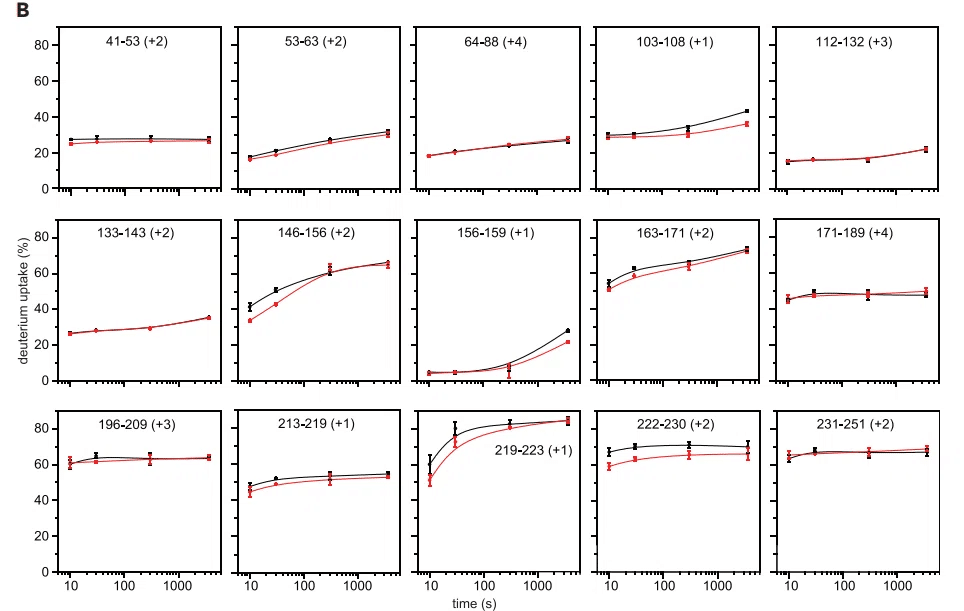
Residues 133–143 are not perturbed by RNA binding, but peptides covering residues 146–171 show clear protection.
残基 133~143 は RNA 結合によって乱されませんが、残基 146~171 をカバーするペプチドは明確な保護を示します。
The largest differences in HDX are observed where 50–80% of the residues of unbound peptides undergo a burst phase of HDX in the first 10 s (amino acid residues 146–156, 163–171, and 213–230), where the peptides cover regions of little hydrogen bonding in the unbound state.
HDX の最大の違いは、結合していないペプチドの残基の 50~80% が最初の 10 秒間に HDX のバースト フェーズを経る部分 (アミノ酸残基 146~156、163~171、および 213~230) で観察されます。この部分では、ペプチドは結合していない状態で水素結合がほとんどない領域をカバーします。
When bound to RNA, the fraction of residues participating in the burst phase decreases, resulting in observed protection.
RNA に結合すると、バースト フェーズに参加する残基の割合が減少し、保護が観察されます。
Then, HDX either converges over time (amino acid residues 146–156, 163–171, and 219–223) consistent with protein conformation or RNA binding dynamics or the HDX never converges in the timescale of the experiment (amino acid residues 222–230), consistent with relatively static binding. Interestingly, peptides covering amino acid residues 103–108 and 156–159 undergo very little HDX throughout the experiment, consistent with either a hydrophobic pocket or secondary or tertiary structure hydrogen bonding.
その後、HDX は、タンパク質の立体構造または RNA 結合ダイナミクスと一致して時間の経過とともに収束するか (アミノ酸残基 146~156、163~171、および 219~223)、または HDX が実験の時間スケールで収束しないか (アミノ酸残基 222~230)、どちらかになります (比較的静的な結合と一致します)。興味深いことに、アミノ酸残基 103~108 および 156~159 をカバーするペプチドは、実験全体を通じて HDX をほとんど受けません。これは、疎水性ポケットまたは二次または三次構造の水素結合と一致します。
Of note, HDX decreases for the bound state in these peptides only after 1 h.
注目すべきは、これらのペプチドの結合状態の HDX が減少するのは 1 時間後のみであるということです。
The low initial HDX limits the dynamic range of binding-induced protection from HDX, but statistically significant protection is still observed.
初期の HDX が低いため、結合によって誘発される HDX からの保護のダイナミック レンジが制限されますが、統計的に有意な保護が依然として観察されています。
Overall, HDX analysis revealed that the protected regions (Figure 3C) overlap with a basic patch groove in the NNTD structure (Figure 3D) that is also observed in recently published NMR titration studies; however, a region (amino acid residues 213–230) within the LKR domain, C-terminal to the SR-motif, also shows statistically significant HDX protection.
全体的に、HDX 分析により、保護された領域 (図 3C) が、最近発表された NMR 滴定研究でも観察されている NNTD 構造 (図 3D) の基本パッチ溝と重なっていることが明らかになりました。ただし、SR モチーフの C 末端にある LKR ドメイン内の領域 (アミノ酸残基 213~230) も、統計的に有意な HDX 保護を示しています。
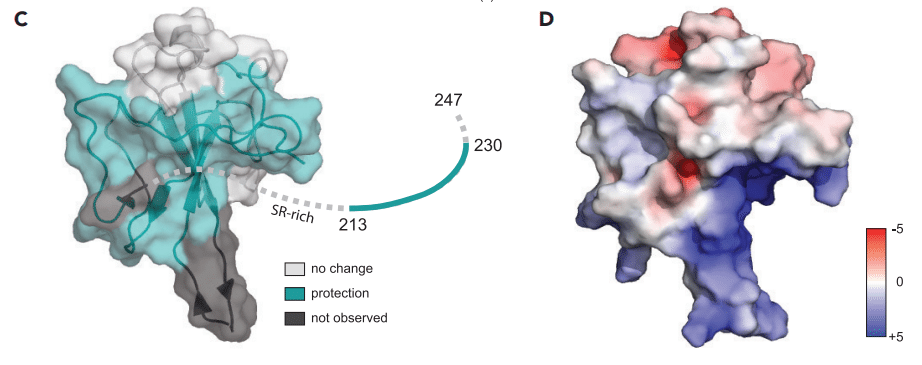
This observation is consistent with the C-terminal end of the LKR domain, a region truncated in other RNA-binding studies of similar N proteins, being perturbed by RNA binding; furthermore, the parallel HDX kinetics plots indicate relatively static binding interactions within this region.
この観察結果は、同様の N タンパク質の他の RNA 結合研究で切り詰められた領域である LKR ドメインの C 末端が RNA 結合によって乱されていることと一致しています。さらに、平行 HDX 速度論プロットは、この領域内の結合相互作用が比較的静的であることを示しています。
Interestingly, we did not detect HDX protection in the SR-motif, which was proposed to bind RNA.
興味深いことに、RNA に結合すると提案された SR モチーフでは HDX 保護が検出されませんでした。
This may be due to the Ser-to-Asp mutations introduced into this region, changing the RNA binding patterns.
これは、この領域に導入された Ser から Asp への変異によって RNA 結合パターンが変化することが原因である可能性があります。
Altogether, HDX results along with our biochemical data define an RNA-binding interface within the NTD and LKR regions of N.
全体として、HDX の結果と生化学データにより、N の NTD 領域と LKR 領域内の RNA 結合インターフェイスが定義されます。
N-RNA liquid droplet formation is impacted by N domains
N-RNA液滴形成はNドメインの影響を受ける
The aforementioned oligomerization and RNA-binding results were obtained using RNA-free protein.
前述のオリゴマー化と RNA 結合の結果は、RNA を含まないタンパク質を使用して得られました。
However, oligomerization is an intrinsic property of N and is complicated by RNA binding during copurification due to the high affinity of N proteins for RNA.
ただし、オリゴマー化は N の固有の特性であり、RNA に対する N タンパク質の親和性が高いため、共精製中に RNA が結合すると複雑になります。
We found that bacterial RNA copurified even with increased ionic strength in purification buffer.
精製バッファーのイオン強度を高めても、微生 RNA が共精製されることが分かりました。
Size-exclusion chromatography revealed three populations of N, including two RNA-bound states (p1 and p2) and an RNA-free state (p3) (Figure 4A).
サイズ排除クロマトグラフィーにより、2 つの RNA 結合状態 (p1 と p2) と 1 つの RNA フリー状態 (p3) を含む 3 つの N 集団が明らかになりました (図 4A)。
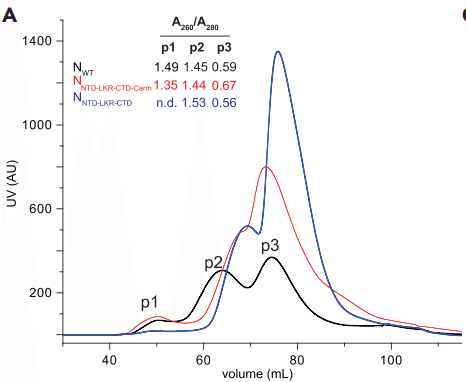
Truncation of the Narm results in an increase of the RNA-free peak (p3), suggesting that N truncations can alter the structure of N and correspondingly impact RNA binding and oligomerization.
Narm の切断により RNA フリーピーク (p3) が増加し、N 切断により N の構造が変化し、それに応じて RNA 結合とオリゴマー化に影響が及ぶ可能性があることが示唆されています。
When both Narm and Carm were removed, we observed an even greater shift to p3, suggesting that both arms contribute to RNA-binding interactions.
Narm と Carm の両方を除去すると、p3 へのシフトがさらに大きくなり、両方のアームが RNA 結合相互作用に寄与していることが示唆されています。
To gain a better understanding of the two RNA-bound populations p1 and p2, we visualized these samples using negative-stain electron microscopy in near-physiological salt concentrations (150 mM).
RNA 結合集団 p1 と p2 の 2 つの理解を深めるため、生理学的塩濃度 (150 mM) に近い濃度でネガティブ染色電子顕微鏡を使用してこれらのサンプルを視覚化しました。
We observed that NWT p1 contains N-RNA with a loose-coil appearance (Figure 4B, top left), similar to that observed for other RNA-bound Ns.
NWT p1 には、他の RNA 結合 N で観察されるものと同様に、ゆるいコイル状の外観を持つ N-RNA が含まれていることがわかりました (図 4B、左上)。

In contrast, we mostly observe spheres in NWT p2 that correspond to liquid droplets separated from the surrounding buffer (Figure 4B, top right), consistent with many recent studies that describe liquid-liquid phase separation of N in the presence of RNA.
対照的に、NWT p2 では、周囲のバッファーから分離された液滴に対応する球体が主に観察されます (図 4B、右上)。これは、RNA 存在下での N の液液相分離を説明する最近の多くの研究と一致しています。
To determine if N regions that impact RNA binding also impact liquid droplet formation, we next examined the role of Narm and Carm.
RNA 結合に影響を与える N 領域が液滴形成にも影響を与えるかどうかを判断するために、次に Narm と Carm の役割を調べました。
N-NTD-LKR-CTD-Carm behaves similarly to NWT, having loose coils in p1 (Figure 4B, middle left) and forming spherical liquid droplets in p2 (Figure 4B, middle right).
N-NTD-LKR-CTD-Carm は NWT と同様に動作し、p1 に緩いコイルがあり (図 4B、中央左)、p2 に球状の液滴を形成します (図 4B、中央右)。
However, examination of p2 from NNTD-LKR-CTD (Figure 4B, bottom right) revealed a much smaller population of liquid droplets (red arrow) and mostly crystal-like needle aggregates, suggesting that the Carm is important for droplet formation.
しかし、NNTD-LKR-CTD の p2 を調べたところ (図 4B、右下)、液滴 (赤い矢印) の数がはるかに少なく、ほとんどが結晶のような針状凝集体であることが明らかになりました。これは、Carm が液滴形成に重要であることを示唆しています。
A transition from spherical liquids to needle-like solids is consistent with the liquid-to-solid transitions observed for other proteins that undergo phase separation.
球状の液体から針状の固体への遷移は、相分離を起こす他のタンパク質で観察される液体から固体への遷移と一致しています。
Phosphorylation of LKR modulates RNA binding and higher-order assembly
LKRのリン酸化はRNA結合と高次アセンブリを調節
Recently, N protein phosphorylation has been qualitatively shown to modulate both RNA binding and phase separation, and we successfully used the improved solubility of phosphomimics to map the RNA binding interface within the NTD-LKR region.
最近、N タンパク質のリン酸化は RNA 結合と相分離の両方を調節することが定性的に示されており、私たちはリン酸化模倣体の溶解性の向上を利用して、NTD-LKR 領域内の RNA 結合界面をマッピングすることに成功しました。
To further test how phosphorylation quantitatively impacts RNA binding and solution properties of SARS-CoV-2 N-RNA, we generated additional N phosphomimics in the context of NWT and NNTD-LKR. Size-exclusion chromatography showed that, compared with NWT, NS188D/S206D (priming serine residues for S176 by GSK-3) produced a reduced RNA-free peak (p3) and an increased RNA-bound peak (p1) (Figure 4C).
リン酸化が SARS-CoV-2 N-RNA の RNA 結合と溶解特性に定量的にどのような影響を与えるかをさらにテストするために、NWT と NNTD-LKR のコンテキストで追加の N リン酸化模倣体を生成しました。サイズ排除クロマトグラフィーにより、NWT と比較して、NS188D/S206D (GSK-3 によって S176 のセリン残基をプライミング) は RNA フリーピーク (p3) が減少し、RNA 結合ピーク (p1) が増加することが示されました (図 4C)。
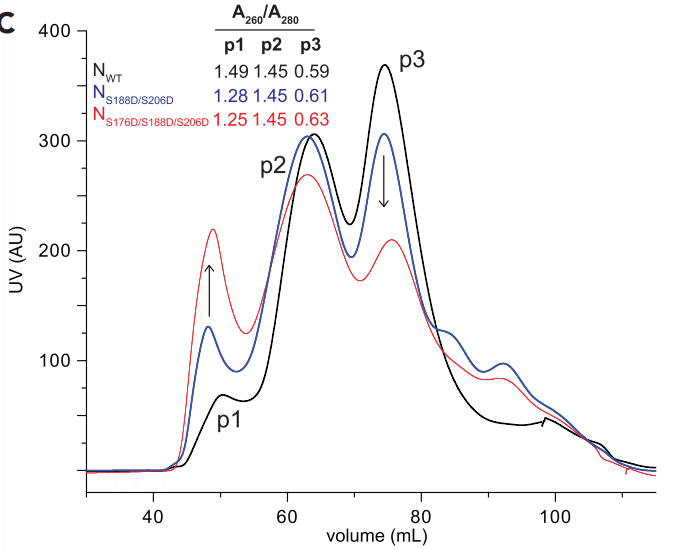
Introduction of S176D to generate NS176D/S188D/S206D resulted in an even greater shift in p1 and p3 distributions, showing how phosphorylation can affect N interactions with RNA.
S176D を導入して NS176D/S188D/S206D を生成すると、p1 と p3 の分布がさらに大きくシフトし、リン酸化が N と RNA の相互作用にどのように影響するかが示されました。
The height of p2 remains relatively the same for all preparations.
p2 の高さは、すべての調製物でほぼ同じままです。
Examination of these protein peaks using electron microscopy revealed that NS188D/S206D displays similar loose coils in p1 (Figure 4D, top left) and spherical droplets in p2 (Figure 4D, top right) for the RNA-bound species.
電子顕微鏡を使用してこれらのタンパク質ピークを調べたところ、RNA 結合種の場合、NS188D/S206D は p1 で同様の緩いコイル (図 4D、左上) を、p2 で球状の液滴 (図 4D、右上) を示すことがわかりました。

Similar observations were made for NS176D/S188D/S206D (Figure 4D).
NS176D/S188D/S206D についても同様の観察結果が得られました (図 4D)。
To describe this interaction further, we measured ssRNA binding to the N phosphomimics (Figures 4E and S5A).
この相互作用をさらに説明するために、N ホスホミミックへの ssRNA 結合を測定しました (図 4E および S5A)。
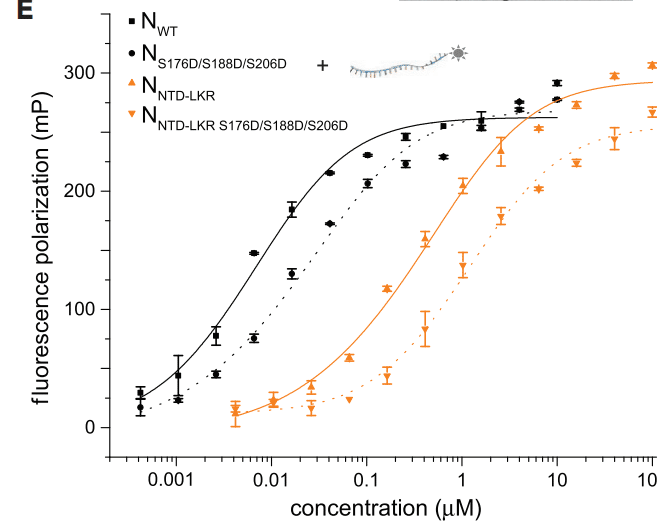
NS176D/S188D/S206D displays ∼5-fold lower binding affinity to ssRNA compared with NWT binding, a result in trend with previous work examining the impact of LKR phosphorylation on RNA binding through simulations.
NS176D/S188D/S206D は、NWT 結合と比較して ssRNA への結合親和性が約 5 倍低く、これはシミュレーションを通じて LKR リン酸化が RNA 結合に与える影響を調べた以前の研究の傾向と一致しています。
We observed a similar trend of lower binding to phosphomimics for the NNTD-LKR construct.
NNTD-LKR 構造についても、リン酸化模倣体への結合が低下するという同様の傾向が観察されました。
Furthermore, binding to slRNA is also affected by these mutations (Figure S5B).
さらに、slRNA への結合もこれらの変異の影響を受けます (図 S5B)。
Collectively, our data suggest that phosphorylation of the LKR region can impact N interactions with RNA, causing changes in properties, including solvent accessibility and RNA binding.
総合的に、私たちのデータは、LKR 領域のリン酸化が N と RNA の相互作用に影響を与え、溶媒のアクセシビリティや RNA の結合などの特性の変化を引き起こす可能性があることを示唆しています。
Interestingly, there are 14 serine residues in the SR motif of LKR in SARS-CoV-2 N, of which 13 are found in SARS-CoV N, and an increase in phosphorylation in this region may further enhance these changes for RNA interaction and N associated functions.
興味深いことに、SARS-CoV-2 N の LKR の SR モチーフには 14 個のセリン残基があり、そのうち 13 個は SARS-CoV N に存在し、この領域のリン酸化の増加は RNA 相互作用と N 関連機能に対するこれらの変化をさらに強化する可能性があります。
NCTD is a sensitive serological marker
Given that the different domains of N impact the various biochemical and physicochemical properties of N, we next assessed if particular domains contribute more significantly to N immunodominance that has been observed worldwide.
N のさまざまなドメインが N のさまざまな生化学的および物理化学的特性に影響を与えることを考慮して、次に、世界中で観察されている N 免疫優位性に特定のドメインがより大きく寄与しているかどうかを評価しました。
Plasma samples collected from two cohorts of patients with real-time polymerase chain reaction (RT-PCR)-confirmed COVID-19, one in St. Louis, USA (n = 45) and one in Hong Kong (n = 23), at different time points of infection.
リアルタイムポリメラーゼ連鎖反応(RT-PCR)でCOVID-19と確定診断された2つのコホート患者から血漿サンプルを採取しました。 1つは米国セントルイス(n = 45)で、もう1つは香港(n = 23)で、感染の異なる時点で採取されました。
Using these samples, we performed enzyme-linked immunosorbent assays (ELISAs) to detect IgG present in plasma of patients with COVID-19 using different N domains that we have purified and characterized.
これらのサンプルを使用して、精製して特性評価したさまざまなNドメインを使用して、COVID-19患者の血漿中に存在するIgGを検出する酵素結合免疫吸着測定(ELISA)を実施しました。
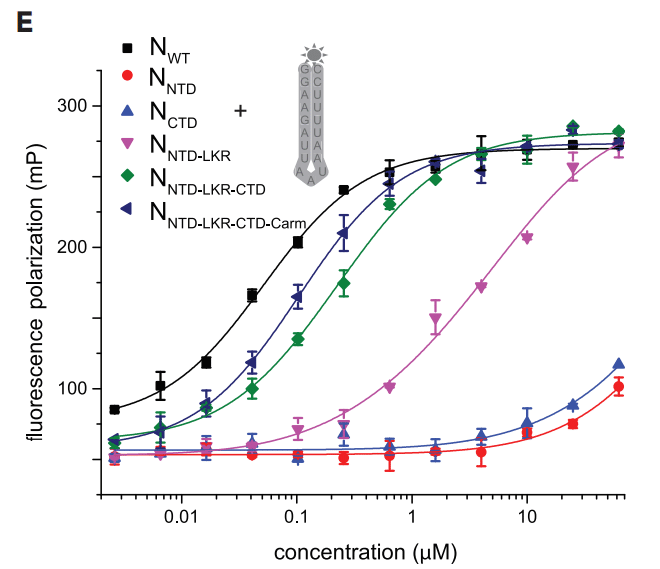
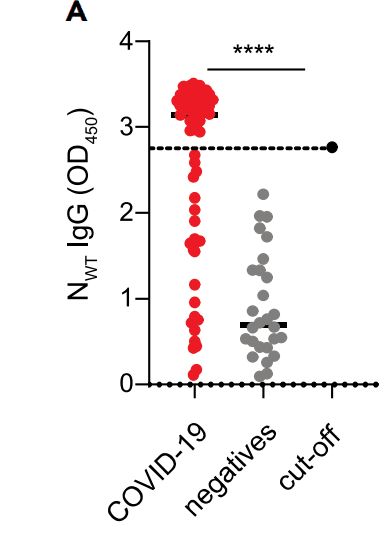
First, we confirmed that purified NWT is a sensitive serological marker to differentiate between COVID-19-positive and COVID-19-negative individuals (Figure 5A).
まず、精製されたNWTがCOVID-19陽性者とCOVID-19陰性者を区別する感度の高い血清学的マーカーであることを確認しました(図5A)。
As shown in Figure 5B, antibodies against all five N constructs were detected in the COVID-19 cohort (p < 0.0001 versus negative controls for all).
図5Bに示すように、COVID-19コホートでは5つのN構成体すべてに対する抗体が検出されました(すべて陰性対照と比較してp < 0.0001)。
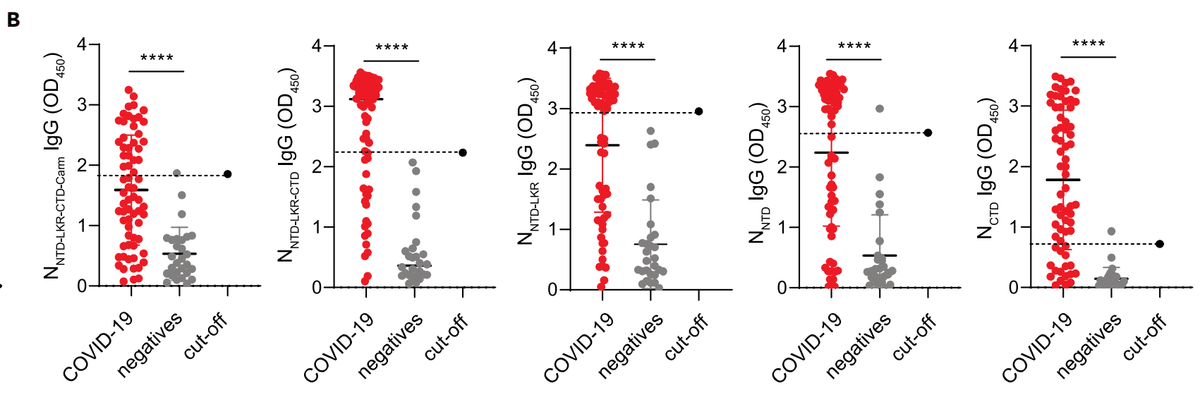
A cutoff based on the mean of the negatives plus three standard deviations allowed us to assess the performance of each N construct at detecting IgG antibodies in COVID-19-positive individuals (Figure 5C).
陰性者の平均と3つの標準偏差に基づくカットオフにより、COVID-19陽性者のIgG抗体検出における各N構成体の性能を評価することができました(図5C)。

We find that NNTD-LKR-CTD-Carm shows the lowest sensitivity (41.2%), whereas the truncated NNTD-LKR-CTD can detect more COVID-19-positive individuals (70.6%).
NNTD-LKR-CTD-Carm は最も低い感度 (41.2%) を示しましたが、短縮された NNTD-LKR-CTD はより多くの COVID-19 陽性者 (70.6%) を検出できることが分かりました。
Furthermore, NCTD shows the highest combination of sensitivity (75%) and specificity (96.4%) over the other N constructs tested.
さらに、NCTD は、テストされた他の N 構成体よりも高い感度 (75%) と特異度 (96.4%) の組み合わせを示しています。
This is demonstrated by the lowest cutoff score for the NCTD for negative control samples, despite a comparable level of amino acid sequence conservation of the NCTD (29–41%) to the NNTD (32–48%) and NLKR (28–42%) domains with common cold coronaviruses (Figure 1B).
これは、風邪コロナウイルスの NNTD (32~48%) および NLKR (28~42%) ドメインと同等のレベルのアミノ酸配列保存性 (29~41%) があるにもかかわらず (図 1B)、陰性対照サンプルの NCTD のカットオフ スコアが最も低いことで示されています。
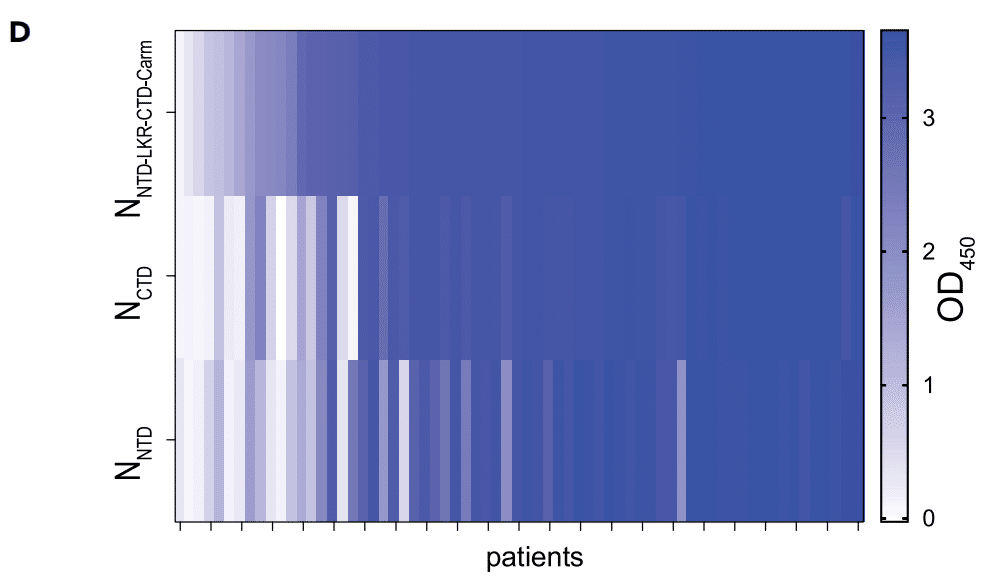
We next compared the immunogenicity of NNTD-LKR-CTD-Carm to NCTD and NNTD on an IgG heatmap during natural infection to an independent panel of 67 COVID-19 samples from Hong Kong.
次に、香港の67のCOVID-19サンプルの独立したパネルへの自然感染中のIgGヒートマップ上で、NNTD-LKR-CTD-Carmの免疫原性をNCTDおよびNNTDと比較しました。
The magnitude of the IgG response to the NNTD-LKR-CTD-Carm tends to follow the same trend as that of NCTD (Figure 5D).
NNTD-LKR-CTD-Carm に対する IgG 反応の大きさは、NCTD と同じ傾向をたどる傾向があります (図 5D)。
When we assessed the kinetics of NCTD and the NNTD-LKR-CTD-Carm responses, we find that the magnitude of the NCTD IgG detection tends to reach a similar level to that of NNTD-LKR-CTD-Carm at convalescent time points (after day 14) (Figure 5E).
NCTD と NNTD-LKR-CTD-Carm の反応の動態を評価したところ、回復期 (14 日目以降) の時点で NCTD IgG 検出の大きさが NNTD-LKR-CTD-Carm と同程度に達する傾向があることがわかりました (図 5E)。
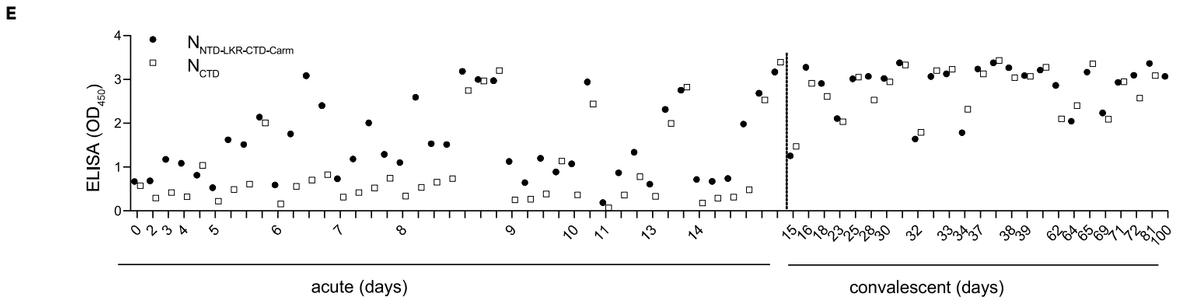
The ELISA ratio of NCTD/NNTD-LKR-CTD-Carm demonstrates this finding, pointing to a maturation of the humoral immune response toward the NCTD with time after infection (Figure 5F, p < 0.0001 for acute versus convalescent time points).
NCTD/NNTD-LKR-CTD-Carm の ELISA 比率はこの結果を実証しており、感染後の時間とともに NCTD に対する体液性免疫反応が成熟することを示しています (図 5F、急性期と回復期の時点で p < 0.0001)。
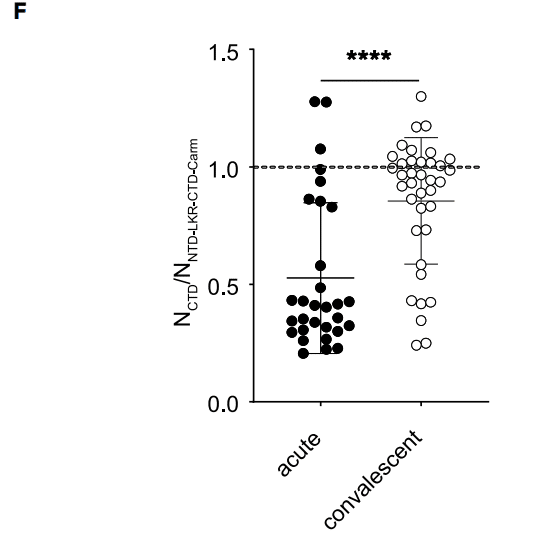
The early dominance of the NNTD-LKR-CTD-Carm IgG response may reflect the recruitment of a cross-reactive preexisting N-specific response.
NNTD-LKR-CTD-Carm IgG 応答の初期の優位性は、交差反応性の既存の N 特異的応答の採用を反映している可能性があります。
This response becomes more specific with time for the NCTD domain as a de novo antibody response is made.
この応答は、新規抗体応答が行われるにつれて、時間の経過とともに NCTD ドメインに対してより特異的になります。
Given that some RNA viral N proteins are known immune antagonists, including prior studies of SARS-CoV, we hypothesized that N from SARS-CoV-2 may also suppress the type-I interferon (IFN) signaling pathway.
SARS-CoV の以前の研究を含め、一部の RNA ウイルス N タンパク質が免疫拮抗薬として知られていることから、SARS-CoV-2 の N も I 型インターフェロン (IFN) シグナル伝達経路を抑制する可能性があるという仮説を立てました。
Using an IFNβ promoter reporter assay, we showed that N has a role in suppressing IFN signaling pathway when stimulated by Sendai virus (SeV) infection (Figure 5G).
IFNβプロモーターレポーターアッセイを使用して、Nはセンダイウイルス(SeV)感染によって刺激されたときにIFNシグナル伝達経路を抑制する役割を果たすことを示しました(図5G)。
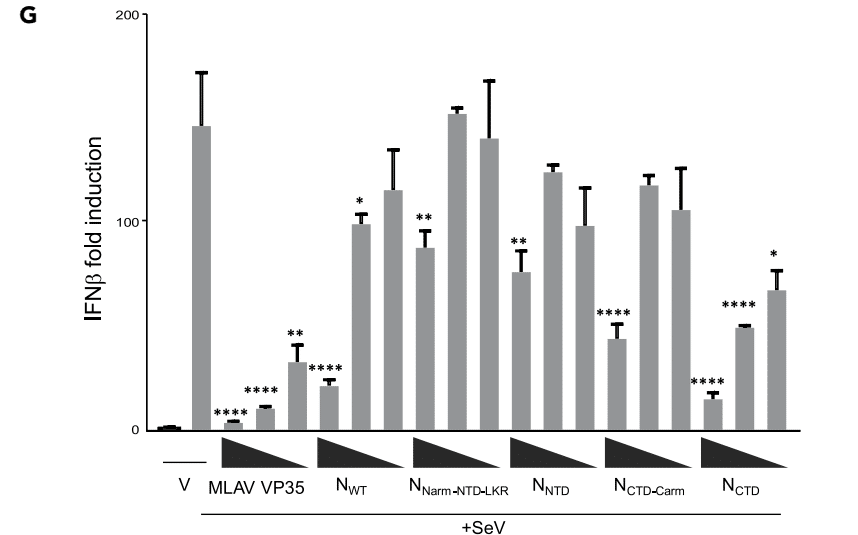
NWT can inhibit IFNβ promoter activity, although not as well as Měnglà virus (MLAV) VP35, a potent inhibitor of IFN signaling.
NWTはIFNβプロモーター活性を阻害できますが、IFNシグナル伝達の強力な阻害剤であるMěnglàウイルス(MLAV)VP35ほどではありません。
Both NNarm-NTD-LKR and NNTD show modest inhibition at the highest concentration tested.
NNarm-NTD-LKR と NNTD はどちらも、テストした最高濃度で中程度の阻害を示します。
However, NCTD-Carm shows similar levels of inhibition as NWT, and NCTD displays the highest inhibition even at lower concentrations.
ただし、NCTD-Carm は NWT と同程度の阻害を示し、NCTD はより低い濃度でも最高の阻害を示します。
In summary, N is a potent inhibitor of IFN signaling and the NCTD appears to be the region critical for mediating this function.
要約すると、N は IFN シグナル伝達の強力な阻害剤であり、NCTD はこの機能の媒介に重要な領域であると思われます。
Discussion
SARS-CoV-2 N protein is a core viral protein produced by the subgenomic RNA, positioned proximal to the 3′ end of genome, displays high transcription levels, and is in high abundance in virions.
SARS-CoV-2 Nタンパク質は、サブゲノムRNAによって生成されるコアウイルスタンパク質で、ゲノムの3'末端に近い位置にあり、高い転写レベルを示し、ビリオンに多く含まれています。
N is prone to forming higher-order oligomers that is impacted by the presence of different domains.
Nは、異なるドメインの存在によって影響を受ける高次オリゴマーを形成しやすい傾向があります。
We found that the isolated NNTD and NCTD form well-behaved monomers and dimers, respectively.
分離されたNNTDとNCTDは、それぞれ適切に動作するモノマーとダイマーを形成することがわかりました。
Linkage of the two domains through the LKR and addition of the Narm and Carm contribute to N oligomerization, demonstrated in our DLS studies.
LKR を介した 2 つのドメインの連結と Narm および Carm の追加は、DLS 研究で実証された N オリゴマー化に寄与します。
It also contributes to enhancement of RNA binding to a continuous platform that was quantitatively measured using a sensitive fluorescence polarization assay.
また、感度の高い蛍光偏光アッセイを使用して定量的に測定された連続プラットフォームへの RNA 結合の強化にも寄与します。
Using HDX-MS for the NNTD-LKR phosphomimic, we confirmed a positively charged groove as an RNA-binding region in the NNTD but also revealed that unstructured regions flanking the SR motif in the LKR are important for RNA interactions, which was not included in previous studies and crystal structures.
NNTD-LKR リン酸化模倣体の HDX-MS を用いて、NNTD の RNA 結合領域として正に帯電した溝を確認しましたが、LKR の SR モチーフに隣接する非構造化領域が RNA 相互作用に重要であることも明らかにしました。これは、以前の研究や結晶構造には含まれていませんでした。
The high extent of HDX observed for the LKR domain in the absence of RNA is consistent with minimal hydrogen bonding (i.e., minimal defined structure).
RNA が存在しない状態で LKR ドメインに観察された HDX の程度が高いことは、最小限の水素結合 (つまり、最小限の定義された構造) と一致しています。
Thus, with a lack of structural information from other techniques, RNA-induced decreased HDX in the LKR domain could indicate RNA binding-induced secondary or tertiary structure, direct interactions with the RNA, or oligomerization.
したがって、他の技術からの構造情報が不足しているため、RNA によって誘発された LKR ドメインの HDX の減少は、RNA 結合によって誘発された二次または三次構造、RNA との直接相互作用、またはオリゴマー化を示している可能性があります。
Overall, these results demonstrate the functional importance of the LKR domain for oligomerization and RNA binding.
全体として、これらの結果は、オリゴマー化と RNA 結合における LKR ドメインの機能的重要性を示しています。
Thus, the coupling of oligomerization and RNA binding likely provides multilayered regulation that is important for immune evasion, viral replication, and N assembly.
したがって、オリゴマー化と RNA 結合の結合は、免疫回避、ウイルス複製、および N アセンブリにとって重要な多層制御を提供すると考えられます。
N is also regulated by post-translational modifications and phosphorylation appears to play an important role that modulates RNA binding and changes the physicochemical properties of N.
N は翻訳後修飾によっても調節されており、リン酸化は RNA 結合を調節し、N の物理化学的特性を変える重要な役割を果たしているようです。
Our data revealed that mutation of S176, S188, and S206 in the SR motif to generate N phosphomimics resulted in decreased binding to RNA and a shift in protein-RNA populations with different solution properties.
私たちのデータから、SR モチーフの S176、S188、および S206 を変異させて N リン酸化模倣体を生成すると、RNA への結合が減少し、異なる溶液特性を持つタンパク質-RNA 集団に変化が生じることが明らかになりました。
In addition to characterizing the domain-specific biochemical and biophysical properties of N, we also gained insight into the antigenicity of individual domains of N and their potential utility in serological studies.
N のドメイン固有の生化学的および生物物理学的特性を特徴付けるだけでなく、N の個々のドメインの抗原性と血清学的研究におけるその潜在的な有用性についての洞察も得られました。
Our data reveal that NCTD acts more specifically in detecting infection of SARS-CoV-2, from patient plasma in comparison with NWT, consistent with predictions that the NCTD region encompasses major antigenic sites of N.
私たちのデータは、NCTD が患者の血漿からの SARS-CoV-2 感染の検出において NWT よりも特異的に作用することを明らかにし、NCTD 領域が N の主要な抗原部位を包含するという予測と一致しています。
Interestingly, 2 of 10 individuals assessed in this study of longitudinal donors unexposed to SARS-CoV-2 by Edridge et al., produced broadly reactive antibodies toward SARS-CoV-2 NWT.
興味深いことに、Edridge らによる SARS-CoV-2 に曝露していない縦断的ドナーを対象としたこの研究で評価された 10 人のうち 2 人が、SARS-CoV-2 NWT に対して広域反応性抗体を産生しました。
The possibility of broadly reactive antibodies in unexposed individuals highlights the need for domain-specific serology, such as our use of the NCTD for increased sensitivity to discriminate COVID-19 cases, while reducing the false-positive rate from cross-reactive antibodies generated by infections of the common cold coronaviruses.
曝露していない個人に広域反応性抗体が存在する可能性は、COVID-19 症例を区別する感度を高めるために NCTD を使用するなど、ドメイン特異的な血清学の必要性を浮き彫りにし、同時に風邪コロナウイルスの感染によって生成される交差反応性抗体による偽陽性率を低減します。
Current serodiagnostic assays to identify COVID-19-positive individuals are based on the detection of antibodies against N due to its abundant expression and corresponding high immune response.
COVID-19陽性者を特定するための現在の血清診断検査は、Nの豊富な発現とそれに伴う高い免疫反応によるNに対する抗体の検出に基づいています。
However, these N-directed serological assays are highly variable, and their sensitivity depend on the sampling time-points, ranging from 0% to 93.75%, suggesting that serological markers for SARS-CoV-2 infection can be further improved to include the NCTD.
しかし、これらのN指向性血清学的アッセイは非常に変動性が高く、その感度はサンプリング時点に依存し、0%から93.75%の範囲です。これは、SARS-CoV-2感染の血清学的マーカーがNCTDを含むようにさらに改善できることを示唆しています。
In conclusion, we describe our efforts to characterize how different domains contribute to the biochemical and physicochemical properties of SARS-CoV-2 N.
結論として、さまざまなドメインが SARS-CoV-2 N の生化学的および物理化学的特性にどのように寄与しているかを特徴付けるための取り組みについて説明します。
Our results advance the understanding of the different levels of regulation involved in modulating viral replication and highlight the utility of using the NCTD as a highly specific and sensitive diagnostic marker of COVID-19.
私たちの結果は、ウイルス複製の調節に関与するさまざまなレベルの制御についての理解を深め、NCTD を COVID-19 の非常に特異的で感度の高い診断マーカーとして使用することの有用性を強調しています。
以下省略。
この記事が気に入ったらサポートをしてみませんか?
