SARS-CoV-2 ヌクレオカプシドタンパク質: ウイルスのライフサイクル、構造、機能における役割、ワクチンや診断薬の開発における潜在的なターゲットとしての利用
The SARS-CoV-2 nucleocapsid protein: its role in the viral life cycle, structure and functions, and use as a potential target in the development of vaccines and diagnostics
SARS-CoV-2 ヌクレオカプシドタンパク質: ウイルスのライフサイクル、構造、機能における役割、ワクチンや診断薬の開発における潜在的なターゲットとしての利用
Abstract
Coronavirus disease 2019 (COVID-19) continues to take a heavy toll on personal health, healthcare systems, and economies around the globe.
新型コロナウイルス感染症(COVID-19)は、世界中の個人の健康、医療制度、経済に大きな打撃を与え続けています。
Scientists are expending tremendous effort to develop diagnostic technologies for detecting positive infections within the shortest possible time, and vaccines and drugs specifically for the prevention and treatment of COVID-19 disease.
科学者たちは、可能な限り短時間で陽性感染を検出するための診断技術、およびCOVID-19疾患の予防と治療に特化したワクチンと薬の開発に多大な努力を費やしています。
At the same time, emerging novel variants have raised serious concerns about vaccine efficacy.
同時に、新たな変異株の出現により、ワクチンの有効性について深刻な懸念が生じています。
The SARS-CoV-2 nucleocapsid (N) protein plays an important role in the coronavirus life cycle, and participates in various vital activities after virus invasion.
SARS-CoV-2 ヌクレオカプシド (N) タンパク質は、コロナウイルスのライフサイクルで重要な役割を果たし、ウイルス侵入後のさまざまな重要な活動に参加しています。
It has attracted a large amount of attention for vaccine and drug development.
ワクチンや医薬品の開発において大きな注目を集めています。
Here, we summarize the latest research of the N protein, including its role in the SARS-CoV-2 life cycle, structure and function, and post-translational modifications in addition to its involvement in liquid–liquid phase separation (LLPS) and use as a basis for the development of vaccines and diagnostic techniques.
ここでは、SARS-CoV-2 ライフサイクルでの役割、構造と機能、翻訳後修飾、液液相分離 (LLPS) への関与、ワクチンや診断技術の開発の基礎としての使用など、N タンパク質の最新の研究をまとめます。
Introduction
The SARS-CoV-2 global pandemic has had a significant impact on public health and economies around the world.
SARS-CoV-2の世界的パンデミックは、世界中の公衆衛生と経済に大きな影響を与えています。
Since the end of 2020, a variety of mutations of the SARS-CoV-2 strain have appeared, which can potentially seriously threaten the health of human beings and impose significant long-term impacts on human production and living activities.
2020年末以来、SARS-CoV-2株のさまざまな変異が出現しており、これらは潜在的に人間の健康を深刻に脅かし、人間の生産活動や生活活動に重大な長期的影響を及ぼす可能性があります。
Thus, vaccines and antiviral drugs against SARS-CoV-2 are urgently needed.
したがって、SARS-CoV-2に対するワクチンと抗ウイルス薬が緊急に必要とされている。
The current strategy for addressing SARS-CoV-2 are aimed at attacking the main link in the life cycle of the virus.
SARS-CoV-2に対処するための現在の戦略は、ウイルスのライフサイクルにおける主要なリンクを攻撃することを目的としている。
It is therefore becomes imperative to understand the life cycle of SARS-CoV-2 at the molecular level and to analyze the structure and function of its constituent proteins.
したがって、SARS-CoV-2のライフサイクルを分子レベルで理解し、その構成タンパク質の構造と機能を分析することが不可欠になります。
Among the four structural proteins and sixteen nonstructural proteins encoded by the SARS-CoV-2 genome, the majority of attention has been focused on the coronavirus spike (S) proteins due to their importance in the viral life cycle.
SARS-CoV-2ゲノムによってコードされる4つの構造タンパク質と16の非構造タンパク質のうち、ウイルスのライフサイクルにおける重要性から、コロナウイルススパイク(S)タンパク質に最も多くの注目が集まっています。
However, many other SARS-CoV-2 proteins play an equally important role in the virus life cycle, but we know relatively little about their structures or biophysical properties.
しかし、他の多くのSARS-CoV-2タンパク質もウイルスのライフサイクルで同様に重要な役割を果たしていますが、その構造や生物物理学的特性についてはほとんどわかっていません。
Among them, the N protein is a particularly attractive antiviral target.
その中でも、Nタンパク質は特に魅力的な抗ウイルス標的です。
In fact, the N protein is not only the basis of viral RNA genome packaging into ribonucleotide complex (RNP) and assembly into virus particles but also is the most abundant protein in virions and a high immunogenicity antigen.
実際、Nタンパク質は、ウイルスRNAゲノムをリボヌクレオチド複合体(RNP)にパッケージングしてウイルス粒子に組み立てる基礎であるだけでなく、ビリオンの中で最も豊富なタンパク質であり、免疫原性の高い抗原でもあります。
Moreover, it is also the determinant of virulence and pathogenesis.
さらに、毒性と病原性の決定因子でもあります。
The N protein can not only be used as a potential target for therapy or vaccines but also as an important diagnostic marker for COVID-19.
Nタンパク質は、治療やワクチンの潜在的なターゲットとしてだけでなく、COVID-19の重要な診断マーカーとしても使用できます。
Therefore, this paper focuses on the SARS-CoV-2 life cycle, the structure and function of N protein domains, the post-translational modification of the N protein, the mechanism and function of N protein-related LLPS, and the N protein-based development of vaccines and diagnostics.
したがって、本論文では、SARS-CoV-2 のライフサイクル、N タンパク質ドメインの構造と機能、N タンパク質の翻訳後修飾、N タンパク質関連 LLPS のメカニズムと機能、および N タンパク質に基づくワクチンと診断の開発に焦点を当てています。
This information may help in designing effective drugs and vaccines to end the SARS-CoV-2 pandemic.
この情報は、SARS-CoV-2 パンデミックを終息させるための効果的な薬剤とワクチンの設計に役立つ可能性があります。
SARS-CoV-2
SARS-CoV-2 is a pleomorphic particle with a diameter of approximately 60–100 nm.
SARS-CoV-2は、直径約60~100nmの多形性粒子です。
The length of its RNA genome is approximately 30 kb, and the specific structural composition is shown in Fig. 1a and b: it contains two large overlapping open reading frames, ORF1a and ORF1b, which are further processed to produce 16 nonstructural proteins (Nsp1 to 16).
そのRNAゲノムの長さは約30kbで、具体的な構造構成は図1aとbに示されています:ORF1aとORF1bという2つの大きな重複オープンリーディングフレームが含まれており、さらに処理されて16の非構造タンパク質(Nsp1~16)が生成されます。
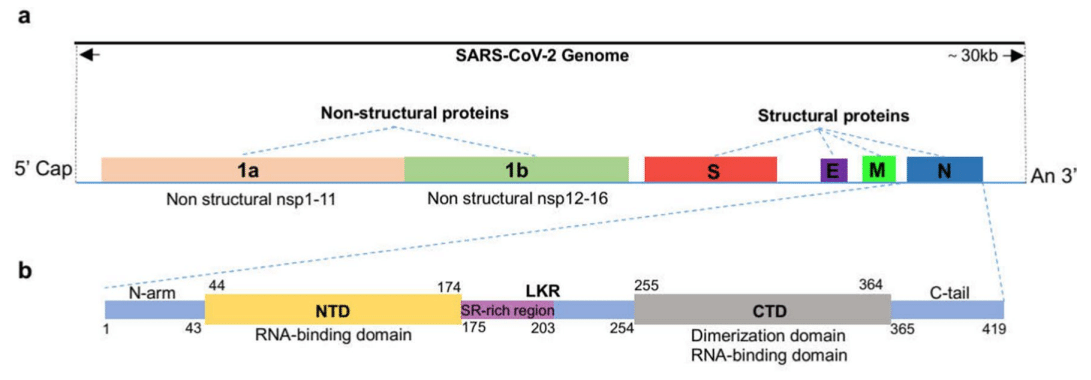
Additionally, it encodes 4 structural proteins, namely the spike (S), membrane (M), envelope (E), and nucleocapsid (N) proteins and 9 auxiliary proteins (ORF3a, ORF3b, ORF6, ORF7a, ORF7b, ORF8, ORF9b, ORF9c, and ORF10).
さらに、スパイク (S)、膜 (M)、エンベロープ (E)、ヌクレオカプシド (N) タンパク質の 4 つの構造タンパク質と、9 つの補助タンパク質 (ORF3a、ORF3b、ORF6、ORF7a、ORF7b、ORF8、ORF9b、ORF9c、ORF10) をコードします。
The structures of some of these proteins have been resolved, and their functions have been studied in depth.
これらのタンパク質のいくつかは構造が解明されており、その機能も詳細に研究されている。
The role of the N protein in SARS-CoV-2 life cycle
The life cycle of the SARS-CoV-2 is shown in Fig. 2.
SARS-CoV-2のライフサイクルを図2に示します。
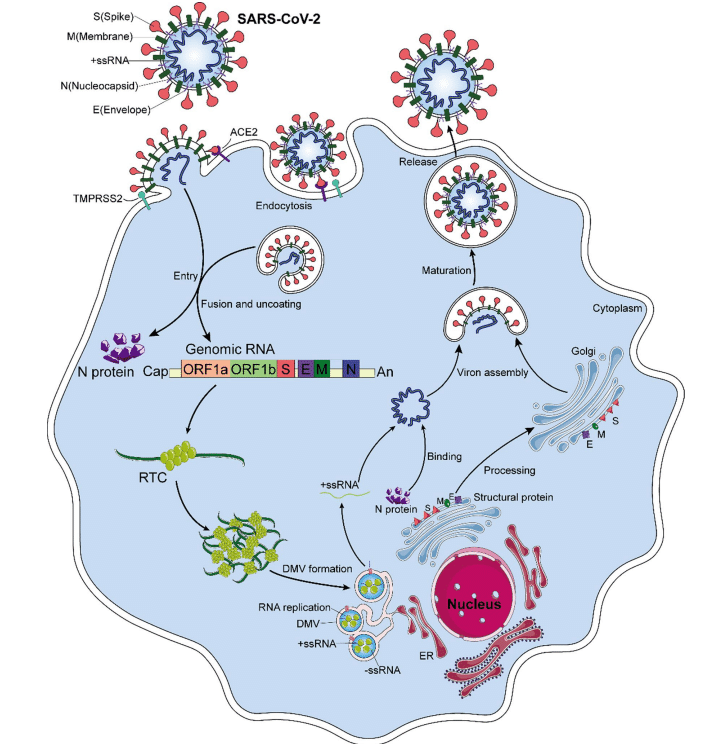
The initial stage of coronavirus infection involves the specific binding of the SARS-CoV-2 spike (S) protein to host cell entry receptors.
コロナウイルス感染の初期段階では、SARS-CoV-2 スパイク (S) タンパク質が宿主細胞侵入受容体に特異的に結合する。
The spike protein, which is a homotrimeric class I fusion glycoprotein, consists of S1 and S2 subunits with different functions.
スパイクタンパク質はホモ三量体クラス I 融合糖タンパク質であり、異なる機能を持つ S1 サブユニットと S2 サブユニットで構成されています。
The surface-exposed S1 subunit receptor binding domain (RBD) specifically binds to the host cell receptor angiotensin-converting enzyme 2 (ACE2).
表面に露出した S1 サブユニット受容体結合ドメイン (RBD) は、宿主細胞受容体アンジオテンシン変換酵素 2 (ACE2) に特異的に結合します。
The transmembrane S2 domain comprises heptapeptide repeat regions and fusion peptides.
膜貫通S2ドメインは、ヘプタペプチド繰り返し領域と融合ペプチドから構成されています。
When hydrolyzed by host cell-derived serine protease TMPRSS2, they mediate fusion of the virus and host cell membrane through extensive conformational rearrangement.
宿主細胞由来のセリンプロテアーゼTMPRSS2によって加水分解されると、広範な構造再配置を介してウイルスと宿主細胞膜の融合を媒介します。
Host cysteine protease cathepsin B (CatB) and CATL facilitate this process.
宿主システインプロテアーゼカテプシンB(CatB)とCATLがこの過程を促進する。
In addition, a multi-alkali cleavage site (PRRAR) in the prototype protein converting enzyme Furin can be found at the boundary of S1–S2, and Furin cleavage leads to increased infection.
さらに、プロトタイプタンパク質変換酵素フーリンのマルチアルカリ切断部位(PRRAR)はS1-S2の境界に存在し、フーリン切断は感染の増加につながる。
In addition, SARS-CoV-2 can enter the cells through endocytosis, and fusion is then induced by S cleavage of endosomal/lysosomal proteins at low Ph.
さらに、SARS-CoV-2はエンドサイトーシスを介して細胞に侵入し、その後、低Phでエンドソーム/リソソームタンパク質のS切断によって融合が誘導されます。
When SARS-CoV-2 enters the host cell, the N protein is dissociated from the positive strand (+) RNA genome of the virus, and the viral gene replication and expression program begins, which is highly regulated in space and time.
SARS-CoV-2が宿主細胞に侵入すると、Nタンパク質がウイルスのプラス鎖(+)RNAゲノムから解離し、空間的および時間的に高度に制御されたウイルス遺伝子複製および発現プログラムが開始されます。
In host cells, there is an inherent antiviral immune defense mechanism, called RNA interference (RNAi), which can lead to degradation of the virus genome to inhibit virus replication.
宿主細胞には、RNA干渉(RNAi)と呼ばれる固有の抗ウイルス免疫防御機構があり、ウイルスゲノムの分解を招き、ウイルスの複製を阻害します。
The N protein acts as a viral inhibitor of RNAi in host cells.
Nタンパク質は、宿主細胞におけるRNAiのウイルス阻害剤として作用する。
In the initial step, the dsRNA in infected cells can be intercepted by the N protein, thus preventing the recognition and cleavage of viral dsRNA.
最初の段階では、感染細胞内のdsRNAはNタンパク質によって遮断され、ウイルスdsRNAの認識と切断が防止されます。
On the one hand, the positive strand (+) RNA genome of the virus translates ORF1a and ORF1b into polyprotein pp1a and pp1ab, respectively.
一方、ウイルスのプラス鎖(+)RNAゲノムは、ORF1aとORF1bをそれぞれポリタンパク質pp1aとpp1abに翻訳します。
After the cleavage of two cysteine proteases, nsp3 (papain-like protease, PLpro) and nsp5 (chymotrypsin-like protease, also known as 3C protease 3CLpro, or major proteolytic enzyme, Mpro), nonstructural proteins are produced.
2 つのシステイン プロテアーゼ、nsp3 (パパイン様プロテアーゼ、PLpro) と nsp5 (キモトリプシン様プロテアーゼ、3C プロテアーゼ 3CLpro、または主要タンパク質分解酵素、Mpro とも呼ばれる) が切断されると、非構造タンパク質が生成されます。
NSP1 shuts down host translation and promotes host mRNA degradation.
NSP1 は宿主の翻訳を停止し、宿主の mRNA 分解を促進します。
Nsp2-11 regulates the intracellular environment to facilitate viral replication.
Nsp2-11 は細胞内環境を調節してウイルスの複製を促進します。
Nsp12-16 contains core enzymes needed for RNA synthesis, including RNA-dependent RNA polymerase (RdRp; NSP 12), while NSP2-16 and N protein establish a viral replication-transcriptional complex (RTC) and reshape the cell membrane to form replicating organelles, which are essential for keeping RNA replicated and transcribed in an orderly conformation.
Nsp12-16には、RNA依存性RNAポリメラーゼ(RdRp; NSP 12)などのRNA合成に必要なコア酵素が含まれていますが、NSP2-16とNタンパク質はウイルス複製転写複合体(RTC)を確立し、細胞膜を再形成して複製細胞小器官を形成します。これは、RNAを秩序ある構造で複製および転写するために不可欠です。
These organelles are connected to the endoplasmic reticulum (ER), providing the best environment for viral RNA replication.
これらの細胞小器官は小胞体(ER)に接続されており、ウイルスRNAの複製に最適な環境を提供します。
On the other hand, viral RNAs are replicated in double-membrane vesicles (DMVs).
一方、ウイルスRNAは二重膜小胞(DMV)で複製されます。
Replication begins with the synthesis of negative-strand RNA copies, which are used as templates to synthesize new plus-strand RNA genomes that may enter additional rounds of translation or be incorporated into new virions.
複製はマイナス鎖RNAコピーの合成から始まり、これがテンプレートとして新しいプラス鎖RNAゲノムを合成し、それが追加の翻訳ラウンドに入るか、新しいウイルス粒子に組み込まれることがあります。
The discontinuous transcription of positive-strand genomic RNA produces subgenomic minus-strand RNA, which is used as a template to synthesize subgenomic positive-strand RNA that encodes structural proteins and helper proteins.
プラス鎖ゲノムRNAの不連続転写によりサブゲノムマイナス鎖RNAが生成され、これがテンプレートとしてサブゲノムプラス鎖RNAを合成し、構造タンパク質とヘルパータンパク質をコードする。
The newborn virus RNA exits DMVs via transmembrane pores to reach the location for translation or virion assembly.
新生ウイルスRNAは膜貫通孔を介してDMVから出て、翻訳またはビリオンの組み立て場所に到達します。
Subsequently, the positive-strand RNA of the genome is encapsulated with the N protein and undergoes assembly with structural proteins S, M, and E in the ER–Golgi intermediate compartment (ERGIC), and new virions are formed by budding into the lumen at ERGIC.
その後、ゲノムのプラス鎖RNAはNタンパク質でカプセル化され、ER-ゴルジ中間体(ERGIC)で構造タンパク質S、M、Eと組み立てられ、ERGICの内腔に出芽することで新しいウイルス粒子が形成される。
Finally, the offspring virions may be released from the host cells via exocytosis.
最後に、子孫ウイルス粒子はエキソサイトーシスによって宿主細胞から放出される可能性がある。
However, recent evidence suggests that SARS-CoV-2 is more likely to exit infected cells through lysosomal transport.
しかし、最近の証拠は、SARS-CoV-2がリソソーム輸送を介して感染細胞から出る可能性が高いことを示唆している。
In general, N proteins are responsible for regulating host cell cycle progression, host–pathogen interactions, and apoptosis.
一般的に、Nタンパク質は宿主細胞周期の進行、宿主と病原体の相互作用、およびアポトーシスの調節を担っています。
In addition, the N protein has strong immunogenicity, can induce protective immune responses, and is highly expressed during infection.
さらに、Nタンパク質は免疫原性が強く、防御免疫反応を誘導することができ、感染時に高度に発現します。
The exploitation of host cellular mechanisms plays a regulatory role in the virus life cycle and is the key to the integration of viral RNA into virus offspring particles.
宿主細胞メカニズムの利用は、ウイルスのライフサイクルにおいて調節的な役割を果たし、ウイルスRNAをウイルスの子孫粒子に統合するための鍵となります。
Thus, the N protein plays an important role in the virus infecting host cells.
したがって、Nタンパク質は、ウイルスが宿主細胞に感染する際に重要な役割を果たします。
Structure and function of the N protein
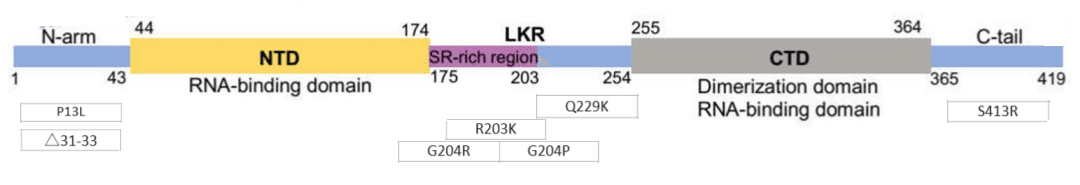
The N protein is a structurally heterogeneous, 419-amino acid-long, multi-domain RNA-binding protein (Fig. 1b and c).
Nタンパク質は構造的に不均一で、419アミノ酸長のマルチドメインRNA結合タンパク質です(図1bおよびc)。
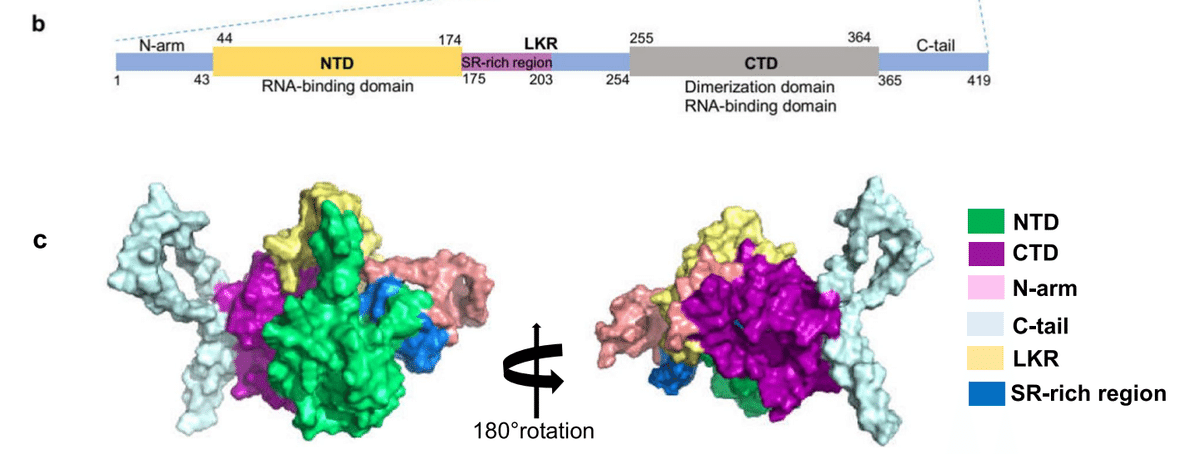
Like other coronaviruses, the N protein also has two conserved, independently folded domains, namely the N-terminal domain (NTD) and the C-terminal domain (CTD) (Fig. 1b).
他のコロナウイルスと同様に、Nタンパク質にも、N末端ドメイン(NTD)とC末端ドメイン(CTD)という2つの保存された独立して折り畳まれたドメインがあります(図1b)。
These two domains are connected by an inherently disordered region (IDR) called the central linking region (LKR).
これら 2 つのドメインは、中央連結領域 (LKR) と呼ばれる本質的に無秩序な領域 (IDR) によって接続されています。
The LKR includes a Ser/Arg (SR)-rich region, which contains putative phosphorylation sites.
LKR には、推定リン酸化部位を含む Ser/Arg (SR) に富んだ領域が含まれています。
In addition, there are two IDRs on both sides of the NTD and CTD, which are called N-arm and C-tail.
さらに、NTDとCTDの両側にはNアームとCテールと呼ばれる2つのIDRがあります。
NTD is responsible for RNA binding, CTD is responsible for RNA binding and dimerization, and IDR is responsible for regulating the RNA-binding activity and oligomerization of NTD and CTD.
NTDはRNA結合を担い、CTDはRNA結合と二量体化を担い、IDRはNTDとCTDのRNA結合活性とオリゴマー化を調節します。
Next, this paper will present the latest research progress on characterizing the structure and function of N protein domains.
次に、本論文では、Nタンパク質ドメインの構造と機能の特徴づけに関する最新の研究の進歩を紹介します。
Structure and function of NTD
A number of scientific teams have successfully resolved the structure of SARS-CoV-2 NTD.
数多くの科学チームがSARS-CoV-2 NTDの構造を解明することに成功しました。
Its structure is very similar to the N proteins of other coronaviruses.
その構造は他のコロナウイルスのNタンパク質と非常によく似ています。
The SARS-CoV-2 NTD takes the shape of a right-handed fist (Fig. 3 a).
SARS-CoV-2 NTD は右の拳の形をしています (図 3 a)。
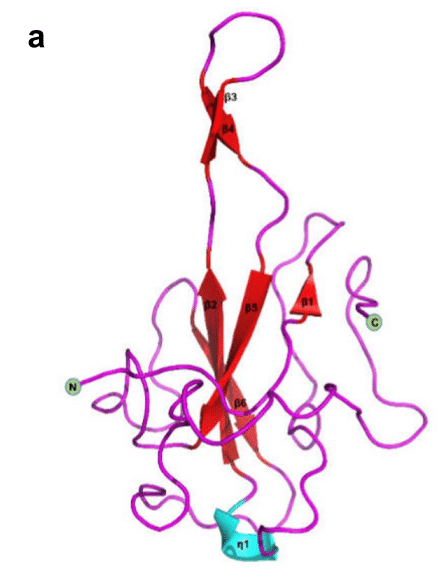
It consists of a four-strand antiparallel β-fold core subdomain, which is located between the annular or short 310 helix and the prominent β-hairpin region formed by β2 and β3 chains outside the nucleus(Fig. 3c).
これは 4 本の鎖からなる平行 β フォールド コア サブドメインで構成されており、環状または短い 310 ヘリックスと、核の外側の β2 鎖と β3 鎖によって形成される顕著な β ヘアピン領域の間に位置しています (図 3c)。
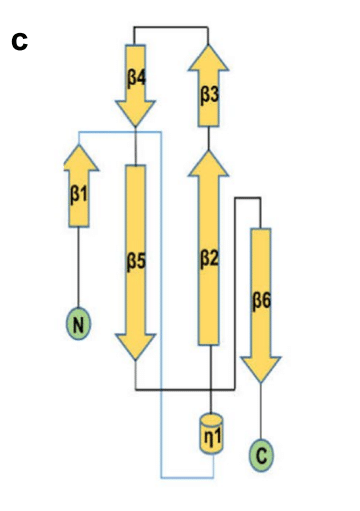
There is a large protruding β-hairpin between β2 and β3 that acts as a bridge connecting them and protrudes from the core (PDBID:6YI3 and 7CDZ); the β-hairpin has a high degree of flexibility.
β2とβ3の間には、両者をつなぐ橋渡しとして機能し、コアから突き出ている大きなβヘアピンがあります(PDBID:6YI3および7CDZ)。βヘアピンは柔軟性に優れています。
The N protein has the ability to recognize and bind RNA.
Nタンパク質はRNAを認識して結合する能力がある。
In NTD, the prominent β-hairpin is mainly composed of basic amino acid residues.
NTDでは、目立つβヘアピンは主に塩基性アミノ酸残基で構成されています。
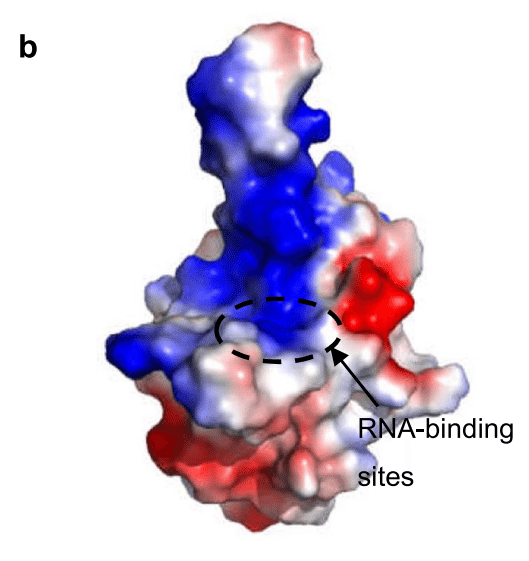
Further analysis of the surface electrostatic potential shows that there is a positively charged pocket at the connection between the basic hairpin and the core structure that is considered to be an RNA-binding site, and this site is conserved between different coronaviruses that infect humans (Fig. 3b).
表面静電ポテンシャルをさらに分析すると、塩基性ヘアピンとコア構造の接続部にRNA結合部位と考えられる正に帯電したポケットがあり、この部位はヒトに感染するさまざまなコロナウイルス間で保存されていることがわかります(図3b)。
By constructing an atomic model of the protein–RNA complex, Dinesh et al. demonstrated that both dsRNA and ssRNA bind to the positively charged canyon between the alkaline β-hairpin and the core of NTD in a similar way, and the arginine residues R92, R107, and R149 that directly bind RNA are located in the canyon.
Dineshらは、タンパク質-RNA複合体の原子モデルを構築することで、dsRNAとssRNAの両方がアルカリβヘアピンとNTDのコアの間の正に帯電した峡谷に同じように結合し、RNAに直接結合するアルギニン残基R92、R107、およびR149が峡谷に位置することを実証しました。
Although the overall structure of SARS-CoV-2 NTD is very similar to that of other coronavirus NTD structures, there are significant structural differences in several regions.
SARS-CoV-2 NTDの全体的な構造は他のコロナウイルスNTD構造と非常に似ていますが、いくつかの領域で構造上大きな違いがあります。
First, compared with all other existing CoV NTD structures that cause human infections, the N-terminal loop of SARS-CoV-2 extends outward, while the circumferential core subdomain of HCoV-OC43 and HCoV-NL63 NTD rotates.
まず、ヒト感染を引き起こす他のすべての既存のCoV NTD構造と比較すると、SARS-CoV-2のN末端ループは外側に伸びていますが、HCoV-OC43およびHCoV-NL63 NTDの円周コアサブドメインは回転しています。
Second, the β-hairpin region protruding from SARS-CoV-2 NTD is flexible, but the flexible ring cannot even be seen in HCoV-OC43 or HCoV-NL63 NTD structures.
第二に、SARS-CoV-2 NTDから突出しているβヘアピン領域は柔軟ですが、HCoV-OC43やHCoV-NL63 NTD構造では柔軟なリングは見られません。
Additionally, the residues that bind to AMP are also different.
さらに、AMPに結合する残基も異なります。
The N-terminal region of the SARS-CoV-2, SARS-CoV, and MERS-CoV NTDs is similar to that of HCoV-OC43 and contains conserved residues.
SARS-CoV-2、SARS-CoV、およびMERS-CoV NTDのN末端領域はHCoV-OC43のものと類似しており、保存された残基が含まれています。
The phenolic hydroxyl of Y124 interacts with the adenine ring of AMP through a hydrogen bond, while the skeleton of G68 forms a hydrogen bond with the monophosphate group of AMP.
Y124のフェノール性ヒドロキシルはAMPのアデニン環と水素結合を介して相互作用し、G68の骨格はAMPの一リン酸基と水素結合を形成する。
For HCoV-NL63, residues H77 and P24 lack the ability to form hydrogen bonds, which may further block the binding of AMP.
HCoV-NL63の場合、残基H77とP24は水素結合を形成する能力を欠いており、AMPの結合をさらに阻害する可能性があります。
Additionally, in different coronavirus NTD structures, the electrostatic surface potential also shows different charge distribution patterns, especially corresponding to the N-terminal ring, the top of the protruding region, and the bottom of the core subdomain.
さらに、コロナウイルスNTDの構造によって、静電表面電位も異なる電荷分布パターンを示し、特にN末端リング、突出領域の上部、コアサブドメインの下部に対応しています。
In addition, the N-terminal tail residues (Asn48, Asn49, Thr50, and Ala51) of SARS-CoV-2 NTD are more flexible and extend outward to a greater extent than their equivalent residues in HCoV-OC43 NTD, and this may facilitate opening of the binding pocket to adequately fit the high-order structure of the viral RNA genome.
さらに、SARS-CoV-2 NTDのN末端残基(Asn48、Asn49、Thr50、Ala51)は、HCoV-OC43 NTDの同等の残基よりも柔軟性が高く、外側に大きく伸びているため、結合ポケットが開き、ウイルスRNAゲノムの高次構造に適切に適合する可能性がある。
Structure and function of N-CTD
Similarly, the SARS-CoV-2 CTD crystal structure has been characterized by several research teams.
同様に、SARS-CoV-2 CTD結晶構造は、いくつかの研究チームによって特徴付けられています。
Usually, two CTD monomers combine to form dimers (Fig. 3d) (PDB:6YUN,7CE0 and 6ZCO), which are diamond-shaped and tile-shaped, and each monomer consists of five α-helices, two 310-helices, and two β-strands (Fig. 3f).
通常、2 つの CTD モノマーが結合してダイアモンド型とタイル型のダイマーを形成します (図 3d) (PDB:6YUN、7CE0、6ZCO)。ダイアモンド型とタイル型で、各モノマーは 5 つの α ヘリックス、2 つの 310 ヘリックス、および 2 つの β ストランドで構成されます (図 3f)。
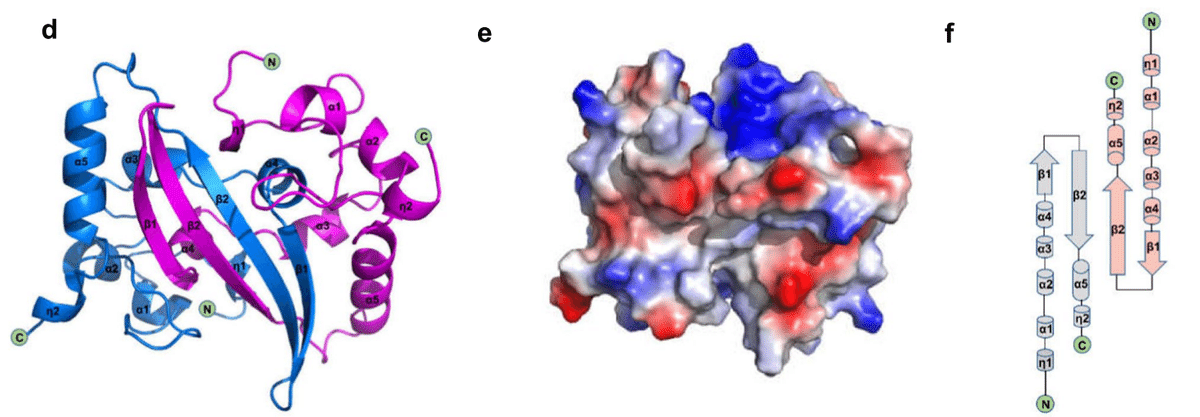
The β-hairpin from one prototype is inserted into the cavity of another prototype, resulting in the formation of a four-chain, antiparallel β-strand at the dimer interface.
あるプロトタイプのβヘアピンが別のプロトタイプの空洞に挿入され、その結果、二量体界面に4本の鎖からなる反平行βストランドが形成される。
The β-strand forms one side of the dimer, while on the other side of the dimer, the surface is formed by an α-helix and a ring.
βストランドは二量体の片側を形成し、二量体のもう一方の側の表面はαヘリックスとリングによって形成される。
The extensive hydrogen bonding interaction between the two hairpins and the hydrophobic interaction between the β-sheet and α-helix result in the dimer structure being highly stable.
2つのヘアピン間の広範な水素結合相互作用と、βシートとαヘリックス間の疎水性相互作用により、二量体構造は非常に安定している。
The CTD structure of SARS-CoV-2 is highly similar to that of SARS-CoV, MERS-CoV, and HCoV-NL63.
SARS-CoV-2のCTD構造は、SARS-CoV、MERS-CoV、HCoV-NL63のCTD構造と非常に類似しています。
All of them show a conservative positively charged groove on the helicoid of the N-CTD dimer, and it is speculated that they contain RNA-binding sites.
いずれもN-CTD二量体のヘリコイド上に保存的な正電荷の溝を示しており、RNA結合部位が含まれていると推測されています。
In SARS-CoV CTD, the hypothetical RNA-binding site is located at 248–280 aa and is conserved.
SARS-CoV CTDでは、仮説上のRNA結合部位は248~280アミノ酸に位置し、保存されている。
The residues identified as RNA-binding sites in SARS-CoV2 CTD correspond to Arg319, Thr334, and Ala336.
SARS-CoV2 CTD で RNA 結合部位として特定された残基は、Arg319、Thr334、および Ala336 に対応します。
The analysis of the electrostatic surface potential of these positions shows that the positively charged residues belonging to these amino acid regions gather at the relative edge of the basic groove, extending laterally relative to the dimer interface line (Fig. 3e).
これらの位置の静電表面電位の分析により、これらのアミノ酸領域に属する正に帯電した残基は、基本溝の相対的な端に集まり、二量体界面線に対して横方向に伸びていることが示されています(図3e)。
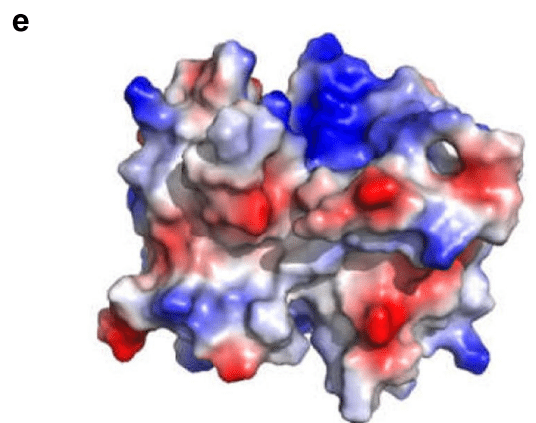
The positive charge in this region of SARS-CoV-2 CTD is mainly due to the distribution of several positively charged residues, including K256, K257, K261, and R262.
SARS-CoV-2 CTDのこの領域における正電荷は、主にK256、K257、K261、R262を含むいくつかの正電荷残基の分布によるものです。
However, the electrostatic potential surfaces of the β-strand surfaces of these proteins show different characteristics.
しかし、これらのタンパク質のβ鎖表面の静電ポテンシャル表面は異なる特性を示しています。
The MERS-CoV structure shows a positively charged central region, while the SARS-CoV-2 and SARS-CoV structures both show a negatively charged region.
MERS-CoVの構造は正に帯電した中央領域を示し、SARS-CoV-2とSARS-CoVの構造は両方とも負に帯電した領域を示します。
For HCoV-NL63, the central region of its β-folding surface shows a highly negatively charged region.
HCoV-NL63の場合、β折り畳み表面の中央領域は、非常に負に帯電した領域を示します。
These differences may affect the binding pattern of RNA recognition.
これらの違いは、RNA認識の結合パターンに影響を与える可能性があります。
According to the proposed model of the coronavirus RNP complex, CTD primordia are packaged into a spiral core, around which the ssRNA of the genome is distorted, so each CTD contains a single-stranded RNA channel consisting of seven bases in its positively charged groove.
コロナウイルスRNP複合体の提案モデルによれば、CTD原基はらせん状のコアにパッケージ化されており、その周りにゲノムのssRNAが歪んでいるため、各CTDには正に帯電した溝に7塩基からなる一本鎖RNAチャネルが含まれています。
Microscale thermophoresis demonstrated that binding occurs to a 7-nucleotide fragment corresponding to the CTD of the single-stranded SARS-CoV-2 RNA genome, showing micromolar affinity, and this fragment is surrounded by the basic grooves of the CTD.
マイクロスケール熱泳動により、一本鎖SARS-CoV-2 RNAゲノムのCTDに対応する7ヌクレオチド断片への結合がマイクロモル親和性を示し、この断片はCTDの基本溝に囲まれていることが実証された。
In addition, it has been demonstrated that CTD can self-bind to form oligomers (dimers, trimers, tetramers, or even octamers), and the instantaneous interaction between CTD dimers leads to the formation of higher-order oligomers.
さらに、CTDは自己結合してオリゴマー(二量体、三量体、四量体、さらには八量体)を形成し、CTD二量体間の瞬間的な相互作用により高次のオリゴマーが形成されることが実証されています。
The degree of aggregation depends on the protein concentration.
凝集の程度はタンパク質濃度に依存します。
Similarly, the high-resolution crystal structure of CTD shows that it exists as a dimer in solution due to chain exchange resulting from close contact.
同様に、CTDの高解像度結晶構造は、密接な接触による鎖交換により、溶液中で二量体として存在することを示しています。
The CTD detection and analysis of SARS-CoV-2 by static light scattering and chemical crosslinking showed that the SARS-CoV-2 CTD dimer is stable in solution, and the self-binding of this domain plays an important role in the overall N stability of SARS-CoV-2.
静的光散乱と化学架橋によるSARS-CoV-2のCTD検出と分析により、SARS-CoV-2 CTD二量体は溶液中で安定しており、このドメインの自己結合がSARS-CoV-2の全体的なN安定性に重要な役割を果たしていることが示された。
Most importantly, it was found that the C-terminal domain could also self-assemble and further mediate formation of the N protein tetramer.
最も重要なことは、C末端ドメインも自己組織化し、Nタンパク質テトラマーの形成をさらに媒介できることがわかったことです。
In addition, the SARS-CoV-2 CTD is crucial for liquid–liquid phase separation (LLPS) and NF-κB regulation of the N protein.
さらに、SARS-CoV-2 CTDは、液液相分離(LLPS)とNタンパク質のNF-κB調節に極めて重要である。
The formation mechanism contributing to LLPS is described in detail below.
LLPSに寄与する形成メカニズムについては、以下で詳しく説明する。
Structure and function of LKR and arms of the N protein
α-Helices and β-sheets are traditionally recognized as important elements of a protein’s secondary structure, and the intrinsically disordered regions (IDR) is becoming increasingly regarded as an important part of protein function.
αヘリックスとβシートは、伝統的にタンパク質の二次構造の重要な要素として認識されています。 そして、本質的に無秩序な領域(IDR)は、タンパク質機能の重要な部分としてますます認識されるようになっています。
The IDR lacks an inherent structure and is flexible and variable in form, which is why it can interact with biological macromolecules such as RNA, DNA, and proteins.
IDR には固有の構造がなく、柔軟で多様な形状であるため、RNA、DNA、タンパク質などの生物学的高分子と相互作用することができます。
Previous studies have shown that there are three IDRs in the N proteins of SARS CoV-1, two of which are either at the N-terminal or the carboxyl terminal (N-arm and C-tail), whereas the third is in the central region (LKR) (Fig. 1 b).
これまでの研究では、SARS CoV-1のNタンパク質には3つのIDRがあり、そのうち2つはN末端またはカルボキシル末端(NアームとCテール)にあり、3つ目は中央領域(LKR)にあることが示されています(図1b)。
Similarly, there are also three IDRs in the N protein of SARS-CoV-2.
同様に、SARS-CoV-2のNタンパク質にも3つのIDRがあります。
The disordered C-tail of the N protein is thought to play a key role in the interaction with virus M protein and packaging signal.
Nタンパク質の無秩序なCテールは、ウイルスMタンパク質およびパッケージングシグナルとの相互作用において重要な役割を果たすと考えられています。
The structural prediction of the C-tail of SARS-CoV-2 N protein shows that this region can form an instantaneous helix.
SARS-CoV-2 Nタンパク質のCテールの構造予測は、この領域が瞬間ヘリックスを形成できることを示している。
Cubuk et al. reported formation of an instantaneous helix in the leucine-rich region using molecular dynamics simulation and suggested it provides an interface for oligomerization.
Cubuk らは、分子動力学シミュレーションを使用してロイシンに富む領域に瞬間的なヘリックスが形成されることを報告し、それがオリゴマー化のインターフェースを提供することを示唆しました。
Using structure prediction tools, Zhao et al. confirmed the existence of a helix across residues 215–235 and found evidence of its role in protein oligomerization and co-assembly with N-terminal nucleic acid(NA).
Zhao らは、構造予測ツールを使用して、残基 215~235 にわたるヘリックスの存在を確認し、タンパク質オリゴマー化および N 末端核酸 (NA) との共集合におけるその役割の証拠を発見しました。
Regarding the conserved leucine-rich sequence 218–231, its potential role comes from its position in the junction region at 210–246, which has been found to be essential for RNA-mediated LLPS.
保存されたロイシンに富む配列 218~231 については、その潜在的な役割は 210~246 のジャンクション領域の位置から来ており、これは RNA を介した LLPS に必須であることがわかっています。
In addition, hydrogen–deuterium exchange mass spectrometry analysis has shown that the conserved junction region rich in serine/arginine also has RNA-binding capacity.
さらに、水素-重水素交換質量分析により、セリン/アルギニンに富む保存された接合領域にもRNA結合能力があることが示されています。
At the N-terminal of the disordered linker, the region rich in SR is due to charged residues and groups serving as phosphorylation sites.
無秩序なリンカーのN末端では、SRに富む領域は、リン酸化部位として機能する荷電残基と基によるものである。
Their phosphorylation status is thought to regulate the function of the N protein through interactions of viral NSP3 protein with host proteins such as glycogen synthase 3, CDK-1, and 14–3-3 proteins.
これらのリン酸化状態は、ウイルスのNSP3タンパク質とグリコーゲン合成酵素3、CDK-1、14-3-3タンパク質などの宿主タンパク質との相互作用を通じて、Nタンパク質の機能を制御すると考えられています。
The R203K/G204R mutation has been experimentally shown to enhance the ability of the N protein to undergo agglutination, while R203M, which is commonly found in delta variants, can enhance virus replication.
R203K/G204R変異は、Nタンパク質の凝集能を高めることが実験的に示されており、デルタ変異体によく見られるR203Mはウイルスの複製を高めることができる。
The most recently discovered mutation, G215C, is located in the linker between SR-rich and leucine-rich regions.
最も最近発見された変異であるG215Cは、SRに富む領域とロイシンに富む領域の間のリンカーに位置している。
G215C enhanced dimer–dimer interactions under reducing conditions and the possibility of forming disulfide bonds between different protoplasts.
G215Cは還元条件下での二量体間相互作用と、異なるプロトプラスト間のジスルフィド結合形成の可能性を高めた。
In general, these regions are involved in interactions with viral RNA and proteins.
一般的に、これらの領域はウイルスRNAおよびタンパク質との相互作用に関与しています。
Biological function of SARS-CoV-2 N protein
The N protein is the core component of the SARS-CoV-2 virus.
Nタンパク質はSARS-CoV-2ウイルスのコア成分です。
It is mainly responsible for identifying and wrapping the virus RNA into a helical symmetrical structure and plays an important multi-functional role in the life cycle of the coronavirus.
ウイルスRNAを識別し、らせん状の対称構造に包み込むことを主に担っており、コロナウイルスのライフサイクルにおいて重要な多機能的役割を果たしている。
It binds to virus genomic RNA to form ribonucleoprotein complex (RNP).
ウイルスゲノムRNAに結合してリボ核タンパク質複合体(RNP)を形成する。
In addition to assembly, N proteins have other functions, including roles in viral mRNA transcription and replication, cytoskeletal tissue, and immune regulation.
Nタンパク質は、組み立てに加えて、ウイルスmRNAの転写と複製、細胞骨格組織、免疫調節などの役割を含む他の機能も有する。
In particular, N protein has been found to counteract host RNAi-mediated antiviral responses through its RNA binding activity, acting as a viral inhibitor through RNA silencing.
特に、Nタンパク質はRNA結合活性を介して宿主RNAi媒介抗ウイルス反応を打ち消し、RNAサイレンシングを介してウイルス阻害剤として作用することが分かっている。
In addition, N protein can induce humoral and cellular immune response after infection, making it a key target for the development of diagnostics and vaccines.
さらに、N タンパク質は感染後に体液性および細胞性免疫反応を誘導できるため、診断薬やワクチンの開発の重要なターゲットとなっています。
N protein usually exists as a stable dimer.
Nタンパク質は通常、安定した二量体として存在します。
Both NTD and CTD domains and connectors contribute to the binding of hybrid NA.
NTD ドメインと CTD ドメインおよびコネクタの両方がハイブリッド NA の結合に寄与します。
The binding of NA induces a more ordered conformation, allowing dimer–dimer interactions, which in turn coordinate the N protein with scaffolding on NA, resulting in the co-assembly of polymers.
NA の結合により、より秩序だった構造が形成され、二量体間の相互作用が可能になり、その結果、N タンパク質が NA 上の足場と調整され、ポリマーの共集合が起こります。
Recent studies have shown that N proteins can be separated by liquid–liquid phase separation (LLPS).
最近の研究では、Nタンパク質は液液相分離(LLPS)によって分離できることが示されています。
NA binding also promotes LLPS, and the high concentration of N protein and NA co-condensates allows the formation of ribonucleoprotein particles.
NA結合もLLPSを促進し、Nタンパク質とNA共凝縮物の高濃度によりリボ核タンパク質粒子の形成が可能になる。
The N protein also interacts with the SARS-CoV-2 membrane (M) protein, which seems to play a role in promoting N protein aggregates, fixing ribonucleoprotein particles on the virus membrane, and recognizing viral RNA.
Nタンパク質はSARS-CoV-2膜(M)タンパク質とも相互作用し、Nタンパク質の凝集を促進し、リボ核タンパク質粒子をウイルス膜に固定し、ウイルスRNAを認識する役割を果たしていると思われる。
A recent study found that SARS-CoV-2 N protein binds to mannan-binding lectin (MBL)-associated serine protease 2 (masp2) and leads to complement overactivation and the aggravation of inflammatory lung injury.
最近の研究では、SARS-CoV-2 Nタンパク質がマンナン結合レクチン(MBL)関連セリンプロテアーゼ2(masp2)に結合し、補体過剰活性化と炎症性肺損傷の悪化につながることが判明しました。
In addition, Oh and Shin identified the role of the N protein in regulating antiviral immunity.
さらに、Oh と Shin は、抗ウイルス免疫の調節における N タンパク質の役割を特定しました。
N protein overexpression leads to retinoic acid-induced gene-I (RIG-I)-like receptor-mediated interferon production and a reduction in interferon-induced gene expression.
Nタンパク質の過剰発現は、レチノイン酸誘導遺伝子I(RIG-I)様受容体を介したインターフェロン産生と、インターフェロン誘導遺伝子発現の減少につながる。
N protein inhibits the interaction between three-part motif protein 25 (TRIM25) and RIG-I.
Nタンパク質は、3部モチーフタンパク質25(TRIM25)とRIG-Iの相互作用を阻害する。
In addition, N protein inhibits polyinosinic:polycytidylic-mediated interferon signal transduction at the level of Tank binding protein 1 (Tank-binding kinase1, TBK1), which interferes with the binding of TK1 to interferon regulatory factor 3 (IRF3), thus preventing nuclear translocation of IRF3.
さらに、Nタンパク質は、タンク結合タンパク質1(タンク結合キナーゼ1、TBK1)レベルでポリイノシン:ポリシチジル酸を介したインターフェロンシグナル伝達を阻害し、TK1のインターフェロン調節因子3(IRF3)への結合を阻害して、IRF3の核移行を防止します。
Another study showed that 11 s proteasome activator PA28γ can regulate intracellular abundance of the N protein.
別の研究では、11 s プロテアソーム活性化因子 PA28γ が N タンパク質の細胞内存在量を調節できることが示されています。
Immunoprecipitation has been used to show that proteasome activator PA28γ is a nucleocapsid binding protein, whereby PA28γ binding plays an important role in regulating 20 s proteasome activity, which in turn regulates the level of SARS-CoV-2 key nucleocapsid protein.
免疫沈降法は、プロテアソーム活性化因子PA28γがヌクレオカプシド結合タンパク質であり、PA28γ結合が20sプロテアソーム活性の調節に重要な役割を果たしていることを実証するために使用されました。 これは、SARS-CoV-2の重要なヌクレオカプシドタンパク質のレベルを調節します。
N protein post-translational modifications
The N protein is post-translationally modified, and studying these modifications is very important for developing potential medical applications based on N protein.
Nタンパク質は翻訳後修飾されており、これらの修飾を研究することは、Nタンパク質に基づく潜在的な医療用途を開発する上で非常に重要です。
Post-translational modifications of the N protein are shown in Fig. 4: First, phosphorylation plays an important role in regulating RNA binding and changing the physical and chemical properties of the N protein.
Nタンパク質の翻訳後修飾を図4に示す:まず、リン酸化はRNA結合の調節とNタンパク質の物理的および化学的特性の変化に重要な役割を果たしている。
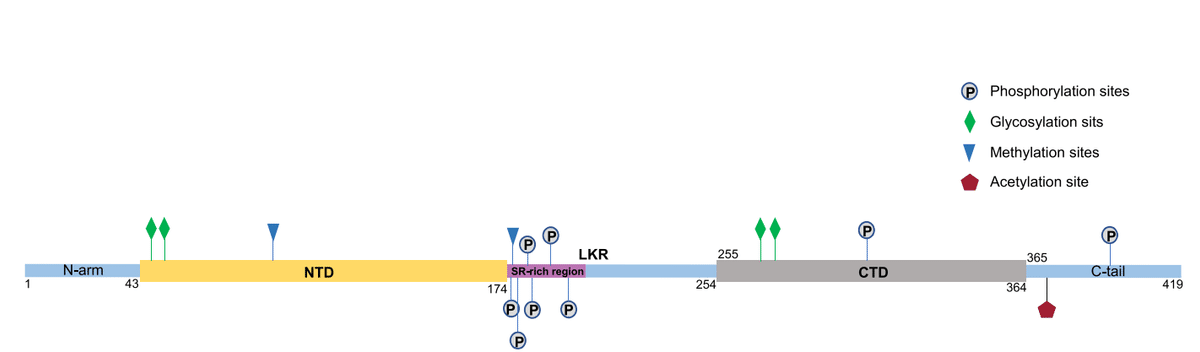
In the early stage of infection, the SR region is rapidly phosphorylated by cytoplasmic kinases at multiple sites.
感染の初期段階では、SR領域は複数の部位で細胞質キナーゼによって急速にリン酸化される。
Phosphorylation leads to binding to RNA helicase DDX1, which promotes the structural RNA changes needed for long subgenomic RNA transcription in RTC.
リン酸化はRNAヘリカーゼDDX1への結合につながり、RTCにおける長いサブゲノムRNA転写に必要な構造的RNA変化を促進する。
Multivalent RNA–protein and protein–protein interactions based on unmodified N proteins lead to the formation partially ordered gel-like aggregates and discrete particles.
修飾されていないNタンパク質に基づく多価RNA-タンパク質およびタンパク質-タンパク質相互作用は、部分的に秩序だったゲル状の凝集体と個別の粒子の形成につながる。
Phosphorylation of the C-terminal region disrupts these interactions, and the phosphorylated proteins form droplets that resemble liquids for viral genome processing.
C末端領域のリン酸化はこれらの相互作用を阻害し、リン酸化タンパク質はウイルスゲノム処理のための液体に似た液滴を形成する。
In the late stage of infection, nucleocapsid formation and virus assembly do not seem to be dependent on N protein phosphorylation, which is significantly reduced in the nucleocapsids of MHV and SARS-CoV viruses.
感染後期には、ヌクレオカプシド形成とウイルスの組み立てはNタンパク質のリン酸化に依存していないようで、MHVおよびSARS-CoVウイルスのヌクレオカプシドではリン酸化が大幅に減少している。
N proteins form structured oligomers suitable for nucleocapsid assembly.
N タンパク質は、ヌクレオカプシドの組み立てに適した構造化されたオリゴマーを形成します。
Studies by Wu et al. showed that mutations in S176, S188, and S206 in SR motifs lead to reduced RNA binding and a transition in protein–RNA populations with different solution properties.
Wu らによる研究では、SR モチーフの S176、S188、および S206 の変異により、RNA 結合が減少し、異なる溶液特性を持つタンパク質-RNA 集団の遷移が起こることが示されました。
In addition, the highly phosphorylated state of the SR domain can regulate the interactions of the N protein with viral NSP3 proteins and host proteins such as glycogen synthase 3, CDK-1, and 14–3-3 proteins.
さらに、SR ドメインの高度にリン酸化された状態は、N タンパク質とウイルスの NSP3 タンパク質、およびグリコーゲン合成酵素 3、CDK-1、14–3-3 タンパク質などの宿主タンパク質との相互作用を調節することができます。
The phosphorylated N protein binds to the 14–3-3 protein in the host cytoplasm and regulates nucleoplasmic N protein shuttling.
リン酸化Nタンパク質は宿主細胞質内の14-3-3タンパク質に結合し、核質Nタンパク質のシャトル輸送を調節する。
The phosphorylation of residues in the serine/arginine of LKR regulates discontinuous transcription, especially for shorter subgenomic mRNA that is closer to the 3' end in the early stage of replication.
LKR のセリン/アルギニン残基のリン酸化は、特に複製の初期段階で 3' 末端に近い短いサブゲノム mRNA の不連続転写を制御します。
Second, the N protein exhibits methylation modification.
第二に、N タンパク質はメチル化修飾を受ける。
Cai et al. demonstrated that the R95 and R177 residues in the RGG/RG motif of PRMT1-methylated N protein regulate the binding of N protein to its 5'-UTR genomic RNA.
Cai らは、PRMT1 メチル化 N タンパク質の RGG/RG モチーフの R95 および R177 残基が、N タンパク質とその 5'-UTR ゲノム RNA との結合を制御することを実証した。
The methylation of R95 regulates the N protein by inhibiting the formation of stress granules (SGs).
R95 のメチル化は、ストレス顆粒 (SG) の形成を阻害することで N タンパク質を制御する。
Arginine methylation affects nearby phosphorylation sites, which are usually antagonistic.
アルギニンのメチル化は、通常は拮抗する近くのリン酸化部位に影響を与える。
The methylation of N proteins R95 and R177 is required for RNA binding.
Nタンパク質R95とR177のメチル化はRNA結合に必要である。
Since S176, S180, S183, and S184 are phosphorylated by SRPK1 and GSK3 cell cycle-dependent kinase 1, there is likely to be an interaction between phosphorylation and methylation, especially in the vicinity of R177, in regulating binding to the 5'UTR of SARS-CoV-2 RNA.
S176、S180、S183、およびS184はSRPK1およびGSK3細胞周期依存性キナーゼ1によってリン酸化されるため、SARS-CoV-2 RNAの5'UTRへの結合を制御する上で、特にR177付近でリン酸化とメチル化の間に相互作用がある可能性が高い。
Finally, the N protein can undergo glycosylation and acetylation modification.
最後に、N タンパク質はグリコシル化およびアセチル化修飾を受ける可能性がある。
Positions 48 and 270 of the N protein are N-glycosylation sites.
N タンパク質の位置 48 と 270 は N グリコシル化部位である。
The Lys375 site is acetylated by host acetyltransferase, and acetylation-mimicking mutations occur frequently at this site, all of which adversely impact liquid–liquid separation of the P protein and RNA.
Lys375部位は宿主アセチルトランスフェラーゼによってアセチル化され、この部位ではアセチル化を模倣した変異が頻繁に発生します。 これらはすべて、Pタンパク質とRNAの液体-液体分離に悪影響を及ぼします。
In addition, the post-translational modification of the N protein is also related to its production pathway.
さらに、Nタンパク質の翻訳後修飾もその生成経路に関連している。
The results of MS/MS and 18O labeling experiments showed that N47 and N269 of the N protein are modified by N-glycosylation when expressed in HEK293 cells.
MS/MSおよび18O標識実験の結果、HEK293細胞で発現するとNタンパク質のN47とN269がNグリコシル化によって修飾されることが示された。
Phosphorylation of the T393 site was also observed.
T393部位のリン酸化も観察された。
On the other hand, the natural N protein produced in the cell exhibits O-phosphorylation, at Ser176, but not glycosylation.
一方、細胞内で生成される天然のNタンパク質はSer176でOリン酸化を示すが、グリコシル化は示さない。
Formation mechanism and function of liquid–liquid phase separation (LLPS)
Many RNA-binding proteins, especially those with a high proportion of inherently disordered regions, participate in liquid–liquid phase separation (LLPS).
多くのRNA結合タンパク質、特に本質的に無秩序な領域の割合が高いタンパク質は、液体-液体相分離(LLPS)に関与しています。
The protein LLPS is a physical and chemical phenomenon that is considered to be the key mechanism for organizing macromolecules, such as proteins and nucleic acids, into membrane-free organelles.
タンパク質 LLPS は物理的および化学的現象であり、タンパク質や核酸などの高分子を膜のない細胞小器官に組織化する重要なメカニズムであると考えられています。
These membraneless cell compartments are dynamically assembled by LLPS and endow cells with the important ability to initiate biological functions or responses to a range of pressures.
これらの膜のない細胞区画は、LLPS によって動的に組み立てられ、細胞に生物学的機能を開始したり、さまざまな圧力に反応したりする重要な能力を与えます。
After RNA virus infection, LLPS mediates the formation of stress granules and P-bodies.
RNAウイルス感染後、LLPSはストレス顆粒とPボディの形成を媒介します。
These substances play an important role in antiviral immunity by inhibiting the translation of viral mRNA and promoting RNA degradation.
これらの物質は、ウイルスmRNAの翻訳を阻害し、RNA分解を促進することで、抗ウイルス免疫において重要な役割を果たします。
LLPS is also considered to be the key to virus assembly.
LLPS はウイルスの組み立ての鍵とも考えられている。
A key step in coronavirus replication is the association of the N protein with virus genomic RNA, which then condenses into a higher-order RNA–protein complex, thus initiating the assembly of virions.
コロナウイルスの複製における重要なステップは、Nタンパク質とウイルスゲノムRNAの結合であり、これが高次のRNA-タンパク質複合体に凝縮され、ウイルス粒子の組み立てが開始されます。
To date, phase separation has been invoked or suggested in many virus environments.
現在までに、多くのウイルス環境で相分離が示唆または示唆されている。
LLPS is involved in the interaction between the SARS-CoV-2 N protein–virus RNA complex and other viral proteins, such as nsp12.
LLPS は、SARS-CoV-2 N タンパク質-ウイルス RNA 複合体と nsp12 などの他のウイルスタンパク質との相互作用に関与しています。
Confocal fluorescence microscopy has shown that the N protein can easily self-bind into many micron-sized spherical aggregates, and its aggregates then fuse and condense into larger aggregates when reaching confluence, which serves as verification of the liquid characteristics of the N protein aggregates.
共焦点蛍光顕微鏡法では、Nタンパク質が容易に自己結合して多数のミクロンサイズの球状凝集体を形成し、その凝集体が合流点に達すると融合してより大きな凝集体に凝縮することが示されており、これはNタンパク質凝集体の液体特性の検証となる。
Fluorescence recovery after photobleaching was used to study the dynamics of internal molecules of N protein agglutinate.
光退色後の蛍光回復を利用して、Nタンパク質凝集体の内部分子の動態を研究した。
It was shown that N proteins can diffuse partly freely in the condensed phase, which is consistent with their liquid behavior.
Nタンパク質は凝縮相で部分的に自由に拡散できることが示されており、これはその液体挙動と一致している。
In addition, the phase condensation of the N protein is very sensitive to increases in ionic strength, which indicates that the electrostatic interaction is very important for its condensation.
さらに、Nタンパク質の相凝縮はイオン強度の増加に非常に敏感であり、これは静電相互作用がその凝縮に非常に重要であることを示している。
The N protein can also experience LLPS in cells and exhibit liquid-like behavior.
Nタンパク質は細胞内でLLPSを経験し、液体のような挙動を示すこともできる。
Matsuo summarized the formation mechanism of LLPS: RNA binding triggers LLPS of the N protein.
松尾は、LLPSの形成メカニズムを次のようにまとめた:RNA結合がNタンパク質のLLPSを誘発する。
The N protein easily binds RNA, they are effectively co-separated at physiological salt concentration.
Nタンパク質はRNAに容易に結合し、生理的塩濃度で効果的に共分離します。
These droplets are formed by electrostatic interactions between positively charged N proteins and negatively charged RNA.
これらの液滴は、正に帯電したNタンパク質と負に帯電したRNAとの間の静電相互作用によって形成される。
The shape of the droplet depends on the ratio of the N protein to RNA concentration.
液滴の形状は、Nタンパク質とRNAの濃度の比率に依存します。
The size of the droplet is affected by the used RNA length.
液滴のサイズは、使用されるRNAの長さによって影響を受けます。
The phase separation behavior also depends on the pH value and salt concentration and ATP can biphasically regulate LLPS by specifically binding to Arg/Lys residues in IDD, which is induced at low ATP concentrations but dissipates at high concentrations.
相分離挙動はpH値と塩濃度にも依存し、ATPはIDDのArg/Lys残基に特異的に結合することでLLPSを二相性に制御することができます。 これは低ATP濃度で誘導されますが、高濃度では消散します。
In addition, each domain of SARS CoV-2 N contributes to its phase separation.
さらに、SARS CoV-2 Nの各ドメインは相分離に寄与している。
Nsp12 cannot undergo spontaneous separation, even if it is mixed with virus RNA, and it can be easily transformed into amorphous agglutinate with poor dynamic performance.
Nsp12は、ウイルスRNAと混合されても自発的に分離することはできず、動的性能の低い非晶質凝集体に容易に変形される。
However, nsp12 can be easily recruited into N protein–RNA aggregates without changing its shape or arrangement.
しかし、nsp12は、その形状や配置を変えることなく、Nタンパク質-RNA凝集体に容易にリクルートされる。
Similar results were obtained using RdRp complexes and ubiquitin-like domain 1 (Ubl1), which means that the N protein-driven LLPS may play an important role in the life cycle of SARS-CoV-2.
RdRp複合体とユビキチン様ドメイン1(Ubl1)を用いた場合も同様の結果が得られており、これはNタンパク質駆動型LLPSがSARS-CoV-2のライフサイクルにおいて重要な役割を果たす可能性があることを意味している。
LLPS also mediates the interaction between N protein/RNA and host.
LLPS は、N タンパク質/RNA と宿主との相互作用も媒介します。
By binding to viral RNA, the N protein undergoes liquid–liquid phase separation and forms functional membraneless organelles to recruit TAK1 and IKK complexes, thus promoting the activation of NF-κB.
ウイルス RNA に結合することで、N タンパク質は液液相分離を起こし、機能的な膜のない細胞小器官を形成して TAK1 および IKK 複合体をリクルートし、NF-κB の活性化を促進します。
Consistently, 1.6-hexanediol, an inhibitor of LLPS, attenuates SARS-CoV-2-induced NF-κB activation.
一貫して、LLPS 阻害剤である 1.6-ヘキサンジオールは、SARS-CoV-2 誘導性 NF-κB 活性化を弱めます。
LLPS of N protein/RNA contributes to virus-induced inflammation.
N タンパク質/RNA の LLPS は、ウイルス誘導炎症に寄与します。
In cells, N protein forms aggregates and recruits stress granule protein G3BP1, highlighting the potential role of the N protein in isolating G3BP1 and inhibiting stress granules.
細胞内では、Nタンパク質は凝集体を形成し、ストレス顆粒タンパク質G3BP1をリクルートし、G3BP1を分離しストレス顆粒を阻害するNタンパク質の潜在的な役割を浮き彫りにする。
The phase separation of the N protein helps to inhibit G3BP1-dependent host immune response and package genomic RNA during virion assembly.
Nタンパク質の相分離は、G3BP1依存性宿主免疫応答を阻害し、ウイルス粒子の組み立て中にゲノムRNAをパッケージ化するのに役立ちます。
Vaccine research and development
The N protein, spike protein, and membrane protein have been used in vaccine development due to their high immunogenicity.
Nタンパク質、スパイクタンパク質、膜タンパク質は、免疫原性が高いため、ワクチン開発に使用されています。
Nucleocapsid-specific antibodies can improve protection against SARS-CoV-2.
ヌクレオカプシド特異的抗体は、SARS-CoV-2に対する防御力を向上させることができる。
Gao et al. used high-affinity glycan ligand-decorated glyconanoparticles to develop a universal SARS-CoV-2 vaccine (TCCSia-Ace-Dex-N-Rd vaccine), the TCCSia-Ace-Dex-N-Rd vaccine carries SARS-CoV-2 nucleocapsid protein (N) and can trigger strong N-specific CTL responses against target cells infected with SARS-CoV-2 and its variants of concern.
Gao らは、高親和性グリカンリガンド修飾糖ナノ粒子を使用して、普遍的な SARS-CoV-2 ワクチン (TCCSia-Ace-Dex-N-Rd ワクチン) を開発しました。TCCSia-Ace-Dex-N-Rd ワクチンは SARS-CoV-2 ヌクレオカプシドタンパク質 (N) を運び、SARS-CoV-2 およびその懸念される変異体に感染した標的細胞に対して強力な N 特異的 CTL 応答を誘発することができます。
Thura et al. demonstrate novel vaccine candidates against SARS-CoV-2 by using the whole conserved N-protein or its fragment/peptides.
Thuraらは、保存されたNタンパク質全体またはその断片/ペプチドを使用して、SARS-CoV-2に対する新しいワクチン候補を実証しています。
The high titers of specific anti-N antibodies maintained for a reasonably long duration, suggesting that N-protein is a excellent immunogen to stimulate host immune system and enhance B-cells activation.
特異的抗N抗体の高い力価がかなり長い期間維持されたことから、Nタンパク質は宿主の免疫系を刺激し、B細胞の活性化を高める優れた免疫原であることが示唆されています。
Purified inactivated viruses manufactured by Sinovac and Sinopharm, such as CoronaVac and BBIBP-CorV, are options because they integrate not only S protein, but also other viral proteins, including matrix (M), envelope (E) and nucleocapsid (N).
CoronaVacやBBIBP-CorVなど、SinovacとSinopharmが製造する精製不活化ウイルスは、Sタンパク質だけでなく、マトリックス(M)、エンベロープ(E)、ヌクレオカプシド(N)などの他のウイルスタンパク質も組み込んでいるため、選択肢となります。
Studies have shown that in populations immunized with inactivated virus and the seropositivity rate is low, enhanced vaccinations significantly improves immunogenicity.
研究によると、不活化ウイルスで免疫化され、血清陽性率が低い集団では、強化ワクチン接種によって免疫原性が大幅に改善されることが示されています。
Appelberg et al. designed a universal SARS-CoV-2 DNA vaccine containing receptor-binding domain loops from the huCoV-19/WH01, the Alpha, and the Beta variants, combined with the membrane and nucleoproteins.
Appelbergらは、huCoV-19/WH01、アルファ、ベータ変異体の受容体結合ドメインループと膜タンパク質および核タンパク質を組み合わせた、ユニバーサルSARS-CoV-2 DNAワクチンを設計した。
The vaccine induced spike antibodies cross-reactive that neutralized huCoV-19/WH01, Beta, Delta, and Omicron virus in vitro, and primed nucleoprotein-specific T cells.
ワクチンは、huCoV-19/WH01、ベータ、デルタ、オミクロンウイルスをin vitroで中和し、核タンパク質特異的T細胞をプライミングする交差反応性のスパイク抗体を誘導した。
Priming of cross-reactive nucleoprotein-specific T cells alone was 60% protective.
交差反応性の核タンパク質特異的T細胞のみのプライミングは、60%の防御率であった。
Similarly, other teams also designed and developed multiple recombinant vaccines based on nucleoproteins.
同様に、他のチームも核タンパク質をベースにした複数の組み換えワクチンを設計・開発しました。
The results showed that recombinant vaccines could significantly increase the levels of serum neutralizing antibody and total immunoglobulin, and induce strong specific lymphocyte proliferative response and T cell response.
結果は、組み換えワクチンが血清中和抗体と総免疫グロブリンのレベルを大幅に増加させ、強力な特異的リンパ球増殖反応とT細胞反応を誘発できることを示した。
The antigen specificity level of interferon-γ in splenocytes of immunized mice increased.
免疫マウスの脾臓細胞におけるインターフェロン-γの抗原特異性レベルは上昇した。
The recently published reviews provided a good summary of the role of SARS-CoV-2 nucleocapsid protein in antiviral immunity and vaccine development.
最近発表されたレビューでは、抗ウイルス免疫とワクチン開発における SARS-CoV-2 ヌクレオカプシドタンパク質の役割について、優れた要約が提供されています。
In addition, the N protein gene has attracted much attention as a potential drug target because it is more conservative, is more stable, and has fewer mutations than other viral proteins, such as spike proteins.
さらに、Nタンパク質遺伝子は、スパイクタンパク質などの他のウイルスタンパク質よりも保守的で、より安定しており、変異が少ないため、潜在的な薬剤ターゲットとして大きな注目を集めています。
The drug research and development of the nucleocapsid protein is mainly based on its structure, function, and life cycle.
ヌクレオカプシドタンパク質の医薬品研究開発は、主にその構造、機能、ライフサイクルに基づいています。
Since the RNA-binding activity of the N protein is very important for viral RNP formation and genome replication, the development of drugs that block RNA binding of the NTD or CTD has been proven to be a good antiviral strategy.
Nタンパク質のRNA結合活性はウイルスのRNP形成とゲノム複製に非常に重要であるため、NTDまたはCTDのRNA結合を阻害する薬剤の開発は優れた抗ウイルス戦略であることが証明されています。
In recent studies, molecular docking and molecular dynamics simulation were used to identify potential antiviral drugs and to study the stability of NTD drug complexes.
最近の研究では、分子ドッキングと分子動力学シミュレーションを使用して、潜在的な抗ウイルス薬を特定し、NTD薬物複合体の安定性を研究しました。
New strategies for the use of old drugs can be used in the treatment of new outbreaks of disease in a very short time and at a low cost.
古い薬を利用する新しい戦略は、非常に短期間かつ低コストで、新たな病気の発生を治療するために使用できます。
As a result, 34 drugs that have been approved or are under development were studied.
その結果、承認済みまたは開発中の34の薬が研究されました。
The results showed that rapamycin had the best binding affinity for NTD, and other compounds, such as saracatinib, camostat, trimetini and nafamostat, also showed high binding affinity and high stability.
結果によると、ラパマイシンはNTDに対して最も優れた結合親和性を示し、サラカチニブ、カモスタット、トリメチニ、ナファモスタットなどの他の化合物も高い結合親和性と高い安定性を示した。
Another study focused on compounds from medicinal plants.
別の研究では、薬用植物由来の化合物に焦点を当てました。
Five compounds were successfully isolated from 100 plant compounds: aloe-emodin, anthrarine, alizarine, dantron, and emodin.
100種類の植物化合物から、アロエエモジン、アントラリン、アリザリン、ダントロン、エモジンの5つの化合物を単離することに成功しました。
Previous studies have shown that the compound PJ34, which targets the ribonucleotide binding site in NTD, can effectively inhibit the RNA-binding activity of the HCoV-OC43 N protein and inhibit viral replication.
これまでの研究では、NTDのリボヌクレオチド結合部位を標的とする化合物PJ34が、HCoV-OC43 Nタンパク質のRNA結合活性を効果的に阻害し、ウイルスの複製を阻害できることが示されている。
By comparing the binding sites of PJ34 in the SARS-CoV-2 NTD structure with those in the HCoV-OC43 NTD structure, it is found that the key residues involved in the interaction are conserved.
SARS-CoV-2 NTD構造のPJ34の結合部位をHCoV-OC43 NTD構造のPJ34の結合部位と比較すると、相互作用に関与する主要な残基が保存されていることがわかります。
Dhankhar et al. identified three small molecules with a conformation similar to guanosine monophosphate (GMP) at the active site of the NTD, and they exhibited high binding affinity and stable binding through the formation of hydrogen bonds with Arg107, Tyr111, and Arg149 of the N-terminal domain.
Dhankhar らは、NTD の活性部位にグアノシン一リン酸 (GMP) に類似した構造を持つ 3 つの小分子を同定し、それらは N 末端ドメインの Arg107、Tyr111、および Arg149 との水素結合の形成を介して高い結合親和性と安定した結合を示した。
Second, we should consider screening inhibitors that block normal N protein oligomerization so as to prevent RNP formation or to induce abnormal aggregation.
第二に、RNP形成を阻止したり異常な凝集を誘発したりするために、正常なNタンパク質オリゴマー化を阻害する阻害剤のスクリーニングを検討すべきである。
Recently, a new inhibitor, 5-benzyloxy Grammer (P3), was discovered by virtual screening.
最近、仮想スクリーニングによって新しい阻害剤である5-ベンジルオキシグラマー(P3)が発見された。
This compound can mediate the NTD nonnative dimerization of MERS-CoV and induce N protein aggregation.
この化合物は、MERS-CoVのNTD非ネイティブ二量体化を媒介し、Nタンパク質の凝集を誘導することができる。
It has been proven to have strong antiviral activity against MERS-CoV.
MERS-CoVに対して強力な抗ウイルス活性があることが証明されている。
By comparing the binding cavity of P3 with the corresponding parts of the SARS CoV-2 N-NTD structure, it was found that almost all the residues involved in the interaction are conserved.
P3の結合空洞をSARS CoV-2 N-NTD構造の対応する部分と比較すると、相互作用に関与する残基のほぼすべてが保存されていることが判明した。
Third, many conserved coronavirus proteins, especially N proteins, need to be phosphorylated to be fully functional.
第三に、多くの保存されたコロナウイルスタンパク質、特にNタンパク質は、完全に機能するためにはリン酸化される必要がある。
Accordingly, Yaron et al. identified alectinib through screening, a kinase inhibitor approved by the FDA that can inhibit N protein phosphorylation of SRPK1/2 and restrict SARS-CoV-2 replication.
したがって、Yaronらはスクリーニングを通じて、FDA承認のキナーゼ阻害剤であるアレクチニブを特定した。これはSRPK1/2のNタンパク質リン酸化を阻害し、SARS-CoV-2の複製を制限できる。
It is reported that the phosphorylated N protein dimer directly binds to the dimer 14–3-3 protein in a phosphorylation-dependent manner.
リン酸化Nタンパク質二量体は、リン酸化依存的に二量体14-3-3タンパク質に直接結合することが報告されている。
The relatively tight 14–3-3/N protein binding can regulate nucleocytoplasmic shuttling and other functions of the N protein by blocking the SR enrichment region and hijacking the cell pathway by 14–3-3 protein isolation.
比較的緊密な14–3-3/Nタンパク質結合は、SR濃縮領域をブロックし、14–3-3タンパク質の単離によって細胞経路を乗っ取ることによって、Nタンパク質の核質シャトルやその他の機能を調節することができる。
Therefore, this component may be a valuable target for therapeutic intervention.
したがって、この成分は治療介入の貴重な標的となる可能性がある。
GSK-3 inhibitors block N protein phosphorylation and reduce the accumulation of viral RNA in cells.
GSK-3阻害剤はNタンパク質のリン酸化を阻害し、細胞内のウイルスRNAの蓄積を減らす。
Targeting GSK-3 may provide a new strategy for addressing COVID-19.
GSK-3を標的とすることで、COVID-19に対処するための新しい戦略が得られる可能性がある。
The final antiviral therapy strategy involves indirect targeting of N protein regulation by inhibiting host cell kinases.
最終的な抗ウイルス療法戦略は、宿主細胞キナーゼを阻害することによってNタンパク質調節を間接的に標的とすることです。
The site-specific phosphorylation of the SR domain by the host cell kinase seems to indicate that the N protein hijacks the host cell kinase for spatiotemporal regulation during the virus life cycle.
宿主細胞キナーゼによる SR ドメインの部位特異的リン酸化は、N タンパク質がウイルスのライフサイクル中に時空間制御のために宿主細胞キナーゼを乗っ取ることを示唆しているようです。
SRPK1 seems to play an important role in the viral replication of many different viruses, as the inhibition or activation of SRPK1 may be beneficial to viruses.
SRPK1 は、SRPK1 の阻害または活性化がウイルスに有益である可能性があるため、さまざまなウイルスのウイルス複製において重要な役割を果たしているようです。
The inhibition of SRPK1 and overactivation of SRPK1 may be effective antiviral strategies alone.
SRPK1の阻害とSRPK1の過剰活性化は、単独でも効果的な抗ウイルス戦略となる可能性がある。
In addition, in cell culture models, the inhibition of SRPK1/2 has been shown to inhibit virus replication.
さらに、細胞培養モデルでは、SRPK1/2の阻害がウイルスの複製を阻害することが示されています。
Similarly, viral replication SRPK1/2 inhibitors that have been shown to reduce SAR-SCoV-2 continue to be developed as potential cancer drugs, e.g., feasible strategies to interfere with SARS-CoV-2 replication and transmission include use of the FDA-approved kinase inhibitor alectinib, which strongly reacts with SRPK1 and inhibits SRPK1; the reuse of SRPK1/2 or GSK-3 inhibitors; or the development of new inhibitors.
同様に、SAR-SCoV-2 を減らすことが示されているウイルス複製 SRPK1/2 阻害剤は、潜在的な抗がん剤として開発が続けられています。たとえば、SARS-CoV-2 の複製と伝播を阻害するための実行可能な戦略には、SRPK1 と強く反応して SRPK1 を阻害する FDA 承認のキナーゼ阻害剤アレクチニブの使用、SRPK1/2 または GSK-3 阻害剤の再利用、または新しい阻害剤の開発が含まれます。
Furthermore, the formation and regulation of biomolecule condensates may be an important activity of the N protein that is essential for SARS-CoV-2.
さらに、生体分子凝縮物の形成と調節は、SARS-CoV-2に不可欠なNタンパク質の重要な活動である可能性がある。
Cascarina and Ross suggested that the N protein may use its ability to form or bind biomolecule condensates to disrupt stress particles, enhance viral replication or viral protein translation, and package the viral RNA genome into new virions.
カスカリナとロスは、Nタンパク質が生体分子凝縮体を形成または結合する能力を利用して、ストレス粒子を破壊し、ウイルスの複製またはウイルスタンパク質の翻訳を促進し、ウイルスRNAゲノムを新しいウイルス粒子にパッケージ化する可能性があると示唆した。
Considering the important role of the N protein in many stages of the virus life cycle, the regulation of the N protein through treatment with host cell kinase or nonmembrane organelle may be a feasible strategy to combat existing SARSCoV-2 infection.
ウイルスライフサイクルの多くの段階でNタンパク質が重要な役割を果たしていることを考慮すると、宿主細胞キナーゼまたは非膜細胞小器官による処理を通じてNタンパク質を制御することは、既存のSARSCoV-2感染と戦うための実行可能な戦略である可能性がある。
Diagnostic technology development
In addition to being a potential therapeutic or vaccine target, the N protein can also be used as an important diagnostic marker of COVID-19.
Nタンパク質は、潜在的な治療薬やワクチンの標的であることに加えて、COVID-19の重要な診断マーカーとしても使用できます。
The presence of N protein peptides in gargles and nasopharyngeal swabs can be used for the immediate high-throughput detection of SARS-CoV-2.
うがいや鼻咽頭スワブに含まれるNタンパク質ペプチドは、SARS-CoV-2の即時ハイスループット検出に利用できる。
Fabiani et al. used electrochemical immunosensors to detect SARS-CoV-2 S and N proteins in saliva at concentrations as low as 19 and 8 ng/mL, respectively.
Fabianiらは電気化学免疫センサーを使用して、唾液中のSARS-CoV-2 Sタンパク質とNタンパク質をそれぞれ19 ng/mLと8 ng/mLという低濃度で検出しました。
Cai et al. developed an ultra-sensitive, rapid, and double digital enzyme-linked immunosorbent assay (dELISA) based on a single-molecule array, which is used to simulate the detection of the spike protein (S-RBD) and N protein.
Caiらは、単一分子アレイに基づく超高感度、高速、二重デジタル酵素結合免疫吸着測定法(dELISA)を開発しました。これは、スパイクタンパク質(S-RBD)とNタンパク質の検出をシミュレートするために使用されます。
It shows supersensitivity and a high signal-to-noise ratio, which are helpful to improve the accuracy of COVID-19 diagnosis.
これは超高感度と高い信号対雑音比を示し、COVID-19診断の精度を向上させるのに役立ちます。
The detection of SARS-CoV-2 peptides through tandem mass spectrometry can be used as an alternative to polymerase chain reaction (PCR) and immunodiagnosis.
タンデム質量分析法によるSARS-CoV-2ペプチドの検出は、ポリメラーゼ連鎖反応(PCR)や免疫診断の代替として使用できます。
Clinical studies have shown that the peptide 41RPQGLPNNTASWFTALTQHGK61 in the N protein can be detected in the saliva of patients with COVID-19.
臨床研究では、Nタンパク質のペプチド41RPQGLPNNTASWFTALTQHGK61がCOVID-19患者の唾液で検出できることが示されています。
Another study showed that strong signals for the N protein peptides 375ADETQALPQR385 and 170GYAQGSR177 can be rapidly detected in nasopharyngeal samples of patients with COVID-19.
別の研究では、COVID-19患者の鼻咽頭サンプルでNタンパク質ペプチド375ADETQALPQR385と170GYAQGSR177の強いシグナルが迅速に検出できることが示されています。
This detection method based on the N protein serum level is helpful to accurately distinguish PCR-positive patients with COVID-19 from healthy and uninfected individuals.
Nタンパク質血清レベルに基づくこの検出方法は、PCR陽性のCOVID-19患者を健康な未感染者と正確に区別するのに役立ちます。
Li et al. detected the N protein in the serum of COVID-19 patients with a sensitivity of 92% and a specificity of 97%.
Liらは、COVID-19患者の血清中のNタンパク質を感度92%、特異度97%で検出した。
In another study, S and N proteins were detected in the plasma of patients with COVID-19 at concentrations ranging from 8 to 20,000 pg/mL and 0.8 to 1700 pg/mL, respectively.
別の研究では、COVID-19患者の血漿中のSタンパク質とNタンパク質が、それぞれ8~20,000 pg/mLと0.8~1700 pg/mLの濃度で検出された。
Tan et al. developed a microfluidic chemiluminescence ELISA platform that can detect S and N proteins in tenfold diluted serum within 40 min.
Tanらは、10倍希釈血清中のSタンパク質とNタンパク質を40分以内に検出できるマイクロ流体化学発光ELISAプラットフォームを開発した。
Haljasmägi et al. developed sensitive and specific LIPS methods for binding different SARS-CoV-2 antigens.
Haljasmägiらは、さまざまなSARS-CoV-2抗原に結合するための高感度で特異的なLIPS法を開発した。
The LIPS detection of S and N antigen fragments may provide useful information for the immune response of COVID-19 patients with different clinical courses.
S抗原とN抗原フラグメントのLIPS検出は、さまざまな臨床経過を持つCOVID-19患者の免疫応答に有用な情報を提供する可能性がある。
Torrente-Rodriguez et al. reported a multiplex electrochemical immunoassay for the detection of the N protein and S protein IgG and IgM in 100 × diluted serum samples.
Torrente-Rodriguezらは、100倍希釈血清サンプル中のNタンパク質とSタンパク質のIgGとIgMを検出するための多重電気化学免疫測定法を報告した。
These studies have shown that the quantitative measurement of SARS-CoV-2 antigens, such as N and S proteins in serum/plasma, can be used for the accurate early detection of COVID-19.
これらの研究は、血清/血漿中のNタンパク質やSタンパク質などのSARS-CoV-2抗原の定量測定が、COVID-19の正確な早期検出に使用できることを示している。
Conclusion
The outbreak of COVID-19 at the end of 2019 has had a wide range of medical, social, political, and financial implications.
2019年末のCOVID-19の流行は、医療、社会、政治、経済の面で幅広い影響を及ぼしました。
The high morbidity and mortality rate of COVID-19 has far exceeded that of seasonal influenza and other diseases.
COVID-19の罹患率と死亡率の高さは、季節性インフルエンザやその他の疾患をはるかに上回っています。
In order to curb the rapid spread of SARS-CoV-2 around the world, people have made great efforts to discover effective methods for diagnosis and treatment.
世界中でSARS-CoV-2が急速に蔓延するのを抑えるために、人々は効果的な診断と治療方法の発見に多大な努力を払ってきました。
Therefore, a detailed understanding of molecular events in the life cycle of SARS-CoV-2 and their underlying mechanisms, including virus replication and assembly, is urgently needed.
したがって、SARS-CoV-2のライフサイクルにおける分子イベントと、ウイルスの複製や組み立てなどのその基礎となるメカニズムの詳細な理解が緊急に必要です。
Here, we summarize the progress in the research on SARS-CoV-2 N protein.
ここでは、SARS-CoV-2 Nタンパク質の研究の進捗状況をまとめます。
It is important to deepen and develop our understanding of the structure and function of SARS-CoV-2 N proteins, their role in the life cycle of the virus, and their potential in vaccine and drug development.
SARS-CoV-2 Nタンパク質の構造と機能、ウイルスのライフサイクルにおける役割、ワクチンや医薬品の開発における可能性についての理解を深め、発展させることが重要です。
In addition, to date, the molecular mechanisms involved in the SARS-CoV-2 life cycle after invading cells are not fully understood.
また、現在までに、細胞に侵入した後のSARS-CoV-2のライフサイクルに関与する分子メカニズムは完全には解明されていません。
In particular, SARS-CoV-2 has been constantly mutating and evolving new adaptation mechanisms, such as the "escape mechanism".
特に、SARS-CoV-2は絶えず変異し、「脱出メカニズム」などの新しい適応メカニズムを進化させています。
More in-depth research is needed to discern the details.
詳細を見極めるには、より詳細な研究が必要です。
New coronavirus vaccines and diagnostic techniques robust to virus mutation have been developed.
ウイルスの変異に強い新しいコロナウイルスワクチンと診断技術が開発されました。
Finally, specific drugs and treatments for COVID-19 have not yet been popularized; accurate diagnosis and a series of prevention and control measures are still the most effective means to prevent disease spread.
最後に、COVID-19 の特定の薬や治療法はまだ普及しておらず、正確な診断と一連の予防・制御措置が依然として病気の蔓延を防ぐ最も効果的な手段です。
Therefore, everyone must be vigilant, on the one hand, by strengthening their physical fitness levels to improve their own immunity and, on the other hand, by gathering scientific knowledge of self-protection to prevent infection in addition to establishing a routine for COVID-19 disease treatment in the case of future infections.
したがって、誰もが警戒する必要があります。 一方では、体力を強化して免疫力を高め、他方では、感染を防ぐための自己防衛に関する科学的知識を収集し、将来の感染に備えて COVID-19 疾患の治療ルーチンを確立する必要があります。
以下省略。
この記事が気に入ったらサポートをしてみませんか?
