BA.1、BA.2、および BA.5 のウイルス適応度と固有の病原性
The viral fitness and intrinsic pathogenicity of dominant SARS-CoV-2 Omicron sublineages BA.1, BA.2, and BA.5
優勢な SARS-CoV-2 オミクロン亜系統 BA.1、BA.2、および BA.5 のウイルス適応度と固有の病原性
Summary
Background
Among the Omicron sublineages that have emerged, BA.1, BA.2, BA.5, and their related sublineages have resulted in the largest number of infections. While recent studies demonstrated that all Omicron sublineages robustly escape neutralizing antibody response, it remains unclear on whether these Omicron sublineages share any pattern of evolutionary trajectory on their replication efficiency and intrinsic pathogenicity along the respiratory tract.
出現したオミクロン亜系統のうち、BA.1、BA.2、BA.5、およびそれらの関連亜系統は、最も多くの感染を引き起こしています。最近の研究では、すべてのオミクロン亜系統が中和抗体反応を確実に回避していることが実証されていますが、これらのオミクロン亜系統が、呼吸器系における複製効率と固有の病原性に関して、進化の軌跡のパターンを共有しているかどうかは不明です。
Methods
We compared the virological features, replication capacity of dominant Omicron sublineages BA.1, BA.2 and BA.5 in the human nasal epithelium, and characterized their pathogenicity in K18-hACE2, A129, young C57BL/6, and aged C57BL/6 mice.
ヒトの鼻上皮における優勢なオミクロン亜系統 BA.1、BA.2、および BA.5 のウイルス学的特徴、複製能力を比較し、K18-hACE2、A129、若い C57BL/6、および高齢の C57BL/6 マウスにおける病原性を特徴付けました。
Findings
We found that BA.5 replicated most robustly, followed by BA.2 and BA.1, in the differentiated human nasal epithelium. Consistently, BA.5 infection resulted in higher viral gene copies, infectious viral titres and more abundant viral antigen expression in the nasal turbinates of the infected K18-hACE2 transgenic mice. In contrast, the Omicron sublineages are continuously attenuated in lungs of infected K18-hACE2 and C57BL/6 mice, leading to decreased pathogenicity. Nevertheless, lung manifestations remain severe in Omicron sublineages-infected A129 and aged C57BL/6 mice.
分化したヒトの鼻上皮では、BA.5 が最も活発に複製され、続いて BA.2、BA.1 であることが分かりました。一貫して、BA.5 感染は、感染した K18-hACE2 トランスジェニック マウスの鼻甲介におけるウイルス遺伝子コピー数の増加、感染力価の上昇、およびウイルス抗原の発現の増加をもたらしました。対照的に、Omicron 亜系統は感染した K18-hACE2 マウスおよび C57BL/6 マウスの肺で継続的に弱毒化され、病原性が低下しました。しかし、Omicron 亜系統に感染した A129 マウスおよび高齢の C57BL/6 マウスでは、肺の症状は依然として重篤です。
Interpretation
Our results suggested that the Omicron sublineages might be gaining intrinsic replication fitness in the upper respiratory tract, therefore highlighting the importance of global surveillance of the emergence of hyper-transmissive Omicron sublineages. On the contrary, replication and intrinsic pathogenicity of Omicron is suggested to be further attenuated in the lower respiratory tract. Effective vaccination and other precautions should be in place to prevent severe infections in the immunocompromised populations at risk.
我々の研究結果は、オミクロン亜系統が上気道で本来の複製適応性を獲得している可能性を示唆しており、そのため、高感染性のオミクロン亜系統の出現を世界規模で監視することの重要性を強調している。逆に、オミクロンの複製と本来の病原性は下気道でさらに弱まることが示唆されている。リスクのある免疫不全集団における重篤な感染を防ぐには、効果的なワクチン接種やその他の予防措置を講じる必要がある。
Funding
A full list of funding bodies that contributed to this study can be found in the Acknowledgements section.
Introduction
Reported after Alpha (PANGO lineage B.1.1.7), Beta (B.1.351), Gamma (P.1), and Delta (B.1.617.2), Omicron (B.1.1.529) was designated as the fifth SARS-CoV-2 variant of concern (VOC) by the World Health Organization on November 26th, 2021 and has quickly replaced Delta as the predominant circulating SARS-CoV-2 variant.
オミクロン(B.1.1.529)は、アルファ(PANGO系統B.1.1.7)、ベータ(B.1.351)、ガンマ(P.1)、デルタ(B.1.617.2)に続いて報告され、2021年11月26日に世界保健機関によって5番目の懸念されるSARS-CoV-2変異体(VOC)に指定され、急速にデルタに取って代わり、主要な循環SARS-CoV-2変異体となりました。
Omicron BA.1 (B.1.1.529.1) contains a large number of mutations when compared with ancestral SARS-CoV-2, including 30 substitutions, three short deletions, and one insertion in the spike protein.
オミクロンBA.1(B.1.1.529.1)には、祖先のSARS-CoV-2と比較して、30の置換、3つの短い欠失、およびスパイクタンパク質への1つの挿入を含む多数の変異が含まれています。
Due to these changes, BA.1 demonstrated substantially altered virological features including reduced spike cleavage, attenuated pathogenicity, and robust immune evasion to neutralization antibodies.
これらの変化により、BA.1は、スパイク切断の減少、病原性の弱毒化、中和抗体に対する強力な免疫回避など、大幅に変化したウイルス学的特徴を示しました。
Continuous surveillance of Omicron evolution revealed that BA.1 was replaced by BA.2 and related sublineages, which became the predominant circulating SARS-CoV-2 strains until mid-2022, and were subsequently replaced by BA.5 and related sublineages.
オミクロンの進化を継続的に監視した結果、BA.1 は BA.2 および関連亜系統に置き換えられ、2022 年半ばまで SARS-CoV-2 の優勢な循環株となり、その後 BA.5 および関連亜系統に置き換えられたことが明らかになりました。
Recent clinical investigations revealed a decreased pathogenicity of BA.1 and BA.2 when compared to previous VOCs, which are in agreement with previous in vitro and in vivo studies reported from us and others.
最近の臨床調査では、以前の VOC と比較して BA.1 および BA.2 の病原性が低下していることが明らかになりました。これは、私たちおよび他の研究者が報告した以前の in vitro および in vivo 研究と一致しています。
However, clinical severity of BA.5 and its related sublineages remain incompletely understood.
ただし、BA.5 およびその関連亜系統の臨床的重症度は完全には解明されていません。
Moreover, Omicron and its sublineages have remained as the most prevalent SARS-CoV-2 variant until today.
さらに、オミクロンおよびその亜系統は、今日まで最も一般的な SARS-CoV-2 変異体のままです。
Whether these sequentially emerged Omicron sublineages share a pattern of evolutionary trajectory on their intrinsic infectability and pathogenicity remains unclear.
これらの順次出現したオミクロン亜系統が、その固有の感染性と病原性に関する進化の軌跡のパターンを共有しているかどうかは不明です。
In this study, we systemically compared the virological features, replication capacity in the upper and lower respiratory tract, and intrinsic pathogenicity of BA.1, BA.2, BA.4, and BA.5 sublineages.
本研究では、BA.1、BA.2、BA.4、BA.5 亜系統のウイルス学的特徴、上気道および下気道での複製能力、および固有の病原性を体系的に比較しました。
We demonstrated that the virus replication of Omicron sublineages is becoming increasingly robust in primary human nasal epithelial cells and in the nasal turbinates of K18-hACE2, young C57BL/6, aged C57BL/6, and A129 mouse models.
Omicron 亜系統のウイルス複製は、ヒトの初代鼻腔上皮細胞および K18-hACE2、若い C57BL/6、老齢 C57BL/6、および A129 マウス モデルの鼻甲介でますます強力になっていることを実証しました。
Meanwhile, the virus replication of Omicron sublineages has a trend of further attenuation in the lungs of K18-hACE2 and young C57BL/6 mice, leading to attenuated pathogenicity.
一方、Omicron 亜系統のウイルス複製は、K18-hACE2 および若い C57BL/6 マウスの肺でさらに弱毒化する傾向があり、病原性が弱毒化しています。
Notably, Omicron sublineages can still cause severe diseases in the lungs of A129 and aged C57BL/6 mice, suggesting preventative measures should be in place for the highly susceptible populations including the elderlies and immunocompromised patients.
特に、オミクロン亜系統は、A129 マウスや高齢の C57BL/6 マウスの肺に依然として重篤な疾患を引き起こす可能性があるため、高齢者や免疫不全患者など、感染しやすい集団に対しては予防措置を講じる必要があることが示唆されています。
Methods
Experimental model and subject details
Viruses and biosafety
Wild type SARS-CoV-2 HKU-001a (GenBank: MT230904), B.1.351/Beta (GISAID: EPI_ISL_2423556), B.1.617.2/Delta (GenBank: OM212471), BA.1 (GenBank: OM212472), BA.2 (GISAID: EPI_ISL_9845731), BA.2.12.1 (GISAID: EPI_ISL_13777659), BA.4.1 (GISAID: EPI_ISL_13777657) and BA.5.2 (GISAID: EPI_ISL_13777658) were isolated from laboratory-confirmed COVID-19 patients in Hong Kong.
ウイルスとバイオセーフティ野生型 SARS-CoV-2 HKU-001a (GenBank: MT230904)、B.1.351/Beta (GISAID: EPI_ISL_2423556)、B.1.617.2/Delta (GenBank: OM212471)、BA.1 (GenBank: OM212472)、BA.2 (GISAID: EPI_ISL_9845731)、BA.2.12.1 (GISAID: EPI_ISL_13777659)、BA.4.1 (GISAID: EPI_ISL_13777657)、および BA.5.2 (GISAID: EPI_ISL_13777658) は、香港で検査により確認された COVID-19 患者から分離されました。
All variants of SARS-CoV-2 were cultured and titrated by plaque assays using VeroE6-TMPRSS2 cells.
SARS-CoV-2 のすべての変異体は、VeroE6-TMPRSS2 細胞を用いたプラークアッセイによって培養および滴定されました。
Sequences of all variants used in this study were confirmed with nanopore sequencing.
この研究で使用されたすべての変異体の配列は、ナノポアシーケンシングによって確認されました。
In vivo and in vitro experiments with infectious SARS-CoV-2 were performed according to the approved standard operating procedures of the Biosafety Level 3 facility at Department of Microbiology, HKU.
感染性SARS-CoV-2のin vivoおよびin vitro実験は、香港大学微生物学部のバイオセーフティレベル3施設の承認された標準操作手順に従って実施されました。
Cell cultures
293T, and VeroE6 were maintained in Dulbecco’s modified Eagle’s medium (DMEM) (11965092, Gibco, Amarillo, Texas, USA) containing 10% fetal bovine serum, 100 units penicillin, and 100ug/ml streptomycin.
293T および VeroE6 は、10% ウシ胎児血清、100 単位ペニシリン、100 ug/ml ストレプトマイシンを含むダルベッコ改変イーグル培地 (DMEM) (11965092、Gibco、テキサス州アマリロ、米国) で維持されました。
Calu3 was maintained in DMEM/F12 (11320033, Gibco) containing 10% fetal bovine serum, 100 units penicillin, and 100ug/ml streptomycin.
Calu3 は、10% ウシ胎児血清、100 単位ペニシリン、100 ug/ml ストレプトマイシンを含む DMEM/F12 (11320033、Gibco) で維持されました。
VeroE6-TMPRSS2 was cultured in DMEM supplemented with 10% fetal bovine serum, 100 units penicillin,100ug/ml streptomycin and 2% G418.
VeroE6-TMPRSS2 は、10% ウシ胎児血清、100 単位ペニシリン、100 ug/ml ストレプトマイシン、2% G418 を添加した DMEM で培養されました。
All cells were cultured at 37 °C in an incubator with 5% CO2.
すべての細胞は、5% CO2 のインキュベーターで 37 °C で培養されました。
All cell lines used are routinely tested for mycoplasma and are maintained mycoplasma-free.
使用したすべての細胞株は、マイコプラズマについて定期的に検査されており、マイコプラズマフリーで維持されています。
Virus infection in human nasal epithelial cell air-liquid interface (ALI) culture
ヒト鼻上皮細胞気液界面 (ALI) 培養におけるウイルス感染
The human nasal epithelial cells in air-liquid interface (ALI) culture were purchased from Epithelix (EP02MP, Epithelix, Switzerland) and maintained with MucilAir culture medium (EP04MM, Epithelix) until virus challenge.
気液界面(ALI)培養中のヒト鼻上皮細胞は、Epithelix(EP02MP、Epithelix、スイス)から購入し、ウイルス感染までMucilAir培地(EP04MM、Epithelix)で維持しました。
On the day of virus challenge, cells were washed with MucilAir culture medium to remove the apical mucin, followed by virus inoculation at the apical side at 2 multiplicity of infection (MOI).
ウイルス感染当日、細胞を MucilAir 培地で洗浄して頂端粘液を除去し、続いて頂端側に 2 倍の感染多重度 (MOI) でウイルスを接種しました。
Cells were incubated for 2 h at 37 °C to allow virus entry.
細胞を 37 °C で 2 時間インキュベートし、ウイルスが侵入できるようにしました。
Residual inoculum was removed and washed away after incubation.
残った接種物は培養後に除去し、洗い流しました。
Supernatants and cell lysates were harvested from the apical side for viral genome copy quantification with RNA extraction followed by one-step RT-qPCR and infectious virus titration with TCID50 assays at the designated time points.
上清と細胞溶解物を頂端側から採取し、RNA 抽出によるウイルスゲノムコピーの定量、続いてワンステップ RT-qPCR、および指定された時点での TCID50 アッセイによる感染性ウイルスの滴定を行いました。
For immunofluorescence staining, cells were fixed with neutral-buffered formalin followed by permeabilization with 0.1% Triton-X-100 (11332481001, Sigma–Aldrich, USA).
免疫蛍光染色では、細胞を中性緩衝ホルマリンで固定し、その後 0.1% Triton-X-100 (11332481001、Sigma–Aldrich、米国) で透過処理しました。
The SARS-CoV-2 nucleocapsid protein and ciliated cells were detected with in-house rabbit anti-SARS-CoV-2 nucleocapsid serumand mouse anti-beta-tubulin (T7941, Sigma–Aldrich, USA), respectively.
SARS-CoV-2 ヌクレオカプシドタンパク質と繊毛細胞は、それぞれ自社製のウサギ抗 SARS-CoV-2 ヌクレオカプシド血清とマウス抗ベータチューブリン (T7941、Sigma–Aldrich、米国) で検出されました。
Primary antibodies were visualized with Alexa Fluor 594-conjugated goat anti-mouse secondary antibody (A-11005, Thermo Fisher Scientific, USA) and Alexa Fluor 488-conjugated goat anti-rabbit secondary antibody (A-11034, Thermo Fisher Scientific), followed by mounting with ProLong™ Diamond Antifade Mountant with DAPI (P36962, Thermo Fisher Scientific).
一次抗体は、Alexa Fluor 594 結合ヤギ抗マウス二次抗体 (A-11005、Thermo Fisher Scientific、米国) と Alexa Fluor 488 結合ヤギ抗ウサギ二次抗体 (A-11034、Thermo Fisher Scientific) で可視化し、その後、ProLong™ Diamond Antifade Mountant with DAPI (P36962、Thermo Fisher Scientific) でマウントしました。
Images were acquired with a Carl Zeiss LSM880 confocal microscopy (Zeiss, USA).
画像は、Carl Zeiss LSM880 共焦点顕微鏡 (Zeiss、米国) で取得しました。
To acquire the monolayer image of hNECs, Z-stack was applied to capture each layer in the z-axis of hNECs.
hNEC の単層画像を取得するために、Z スタックを適用して、hNEC の Z 軸の各層をキャプチャしました。
Orthogonal projection was used to stack the Z-stack into one image.
直交投影を使用して、Z スタックを 1 つの画像にスタックしました。
Images were processed and analysed by ZEISS Zen (blue edition) software using the maximum intensity projection setting.
画像は、最大強度投影設定を使用して、ZEISS Zen (ブルー エディション) ソフトウェアで処理および分析されました。
In vivo virus challenge in mice
マウスでの生体内ウイルス チャレンジ
Heterozygous K18-hACE2 C57BL/6J mice (2B6.Cg-Tg(K18-ACE2)2Prlmn/J) were obtained from The Jackson Laboratory.
ヘテロ接合性 K18-hACE2 C57BL/6J マウス (2B6.Cg-Tg(K18-ACE2)2Prlmn/J) は、ジャクソン研究所から入手しました。
The C57BL/6J and A129 mice were obtained from the Centre for Comparative Medicine Research, the University of Hong Kong.
C57BL/6J マウスと A129 マウスは、香港大学比較医学研究センターから入手しました。
The mice were kept in cages with individual ventilation under 65% humidity and an ambient temperature of 21–23 °C and a 12–12 h day–night cycle for housing and husbandry.
マウスは、湿度 65%、室温 21~23 °C、飼育と飼育管理のための昼夜サイクル 12~12 時間のケージで個別に換気して飼育されました。
Group sizes were chosen based on statistical power analysis and our prior experience in examining viral titres in SARS-CoV-2-infected K18-hACE2 transgenic and C57BL/6 mice.
グループのサイズは、統計的検出力分析と、SARS-CoV-2 に感染した K18-hACE2 トランスジェニックマウスおよび C57BL/6 マウスのウイルス力価を調べたこれまでの経験に基づいて選択されました。
Gender- and age-matched mice were randomized into different experimental groups.
性別と年齢が一致したマウスは、異なる実験グループにランダムに割り当てられました。
For virus challenge in transgenic mice, female and male K18-hACE2 transgenic mice (aged 6–10 weeks) were anaesthetized with ketamine and xylazine, followed by intranasal inoculation with 20 μl per mouse of Omicron subvariants BA.1, BA.2, BA.2.12.1, BA.4.1 or BA.5.2 at 5000 PFU per mouse as we previously described.
トランスジェニックマウスへのウイルス感染では、メスおよびオスの K18-hACE2 トランスジェニックマウス (6~10 週齢) をケタミンおよびキシラジンで麻酔し、その後、以前に説明したように、マウス 1 匹あたり 20 μl の Omicron 亜種 BA.1、BA.2、BA.2.12.1、BA.4.1、または BA.5.2 を 1 匹あたり 5000 PFU で鼻腔内接種しました。
For infection in the wildtype and A129 mice, female C57BL6/J (aged 6–8 weeks and aged 10–11 months) and female A129 mice (aged 6–8 weeks) were intranasally challenged with B.1.351, BA.2, BA.4.1 or BA.5.2 at 1 × 105 PFU.
野生型マウスと A129 マウスの感染については、メスの C57BL6/J (6~8 週齢および 10~11 か月齢) とメスの A129 マウス (6~8 週齢) に、1 × 105 PFU の B.1.351、BA.2、BA.4.1、または BA.5.2 を鼻腔内投与しました。
Mice were euthanized at 2 dpi. to collect nasal turbinate and lung tissues for virological assessment, histological examination and pathology scoring.
マウスは 2 dpi で安楽死させ、ウイルス学的評価、組織学的検査、病理学的スコアリングのために鼻甲介と肺組織を採取しました。
Histology and immunohistochemistry staining
組織学および免疫組織化学染色
Animal tissues were collected and fixed with 10% neutral-buffered formalin.
動物組織を採取し、10%中性緩衝ホルマリンで固定しました。
Nasal turbinates were decalcified with 10% formic acid for 10 days before being processed with a semi-enclosed benchtop tissue processor (TP1020, Leica, Germany) and sectioned at 5 μm with the Leica RM2125 RTS benchtop microtome.
鼻甲介は10%ギ酸で10日間脱灰した後、半密閉式ベンチトップ組織プロセッサー(TP1020、Leica、ドイツ)で処理し、Leica RM2125 RTSベンチトップミクロトームで5μmに切片化しました。
Immunohistochemistry (IHC) staining was performed with in-house rabbit polyclonal anti-SARS-CoV-2 nucleocapsid protein antibodies (1:4000), followed by incubation with biotinylated goat anti-rabbit IgG (H + L) secondary antibody (1:500) (BA-1000, Vector laboratories, USA).
免疫組織化学 (IHC) 染色は、社内のウサギポリクローナル抗 SARS-CoV-2 ヌクレオカプシドタンパク質抗体 (1:4000) で実施し、続いてビオチン化ヤギ抗ウサギ IgG (H + L) 二次抗体 (1:500) (BA-1000、Vector Laboratories、米国) でインキュベートしました。
Color was developed with the VECTASTAIN® ABC-AP Kit (AK-5000) and VectorRed substrate kit (SK-5100, Vector Laboratories) according to the manufacturer’s instructions.
色は、メーカーの指示に従って、VECTASTAIN® ABC-APキット(AK-5000)およびVectorRed基質キット(SK-5100、Vector Laboratories)で発色しました。
The nuclei were counterstained with Gill’s haematoxylin before the tissue sections were mounted using the VectaMount permanent mounting medium (H-5000, Vector Laboratories).
核はギルズヘマトキシリンで対比染色した後、組織切片をVectaMount永久封入剤(H-5000、Vector Laboratories)で封入した。
The nucleocapsid protein-covered area was quantified with the IHC Image analysis Toolbox in ImageJ (v.1.8.0_345).
核カプシドタンパク質で覆われた領域は、ImageJ(v.1.8.0_345)のIHC画像分析ツールボックスを使用して定量化した。
For H&E staining, tissue sections were stained with Gill’s haematoxylin and eosin-Y.
H&E 染色では、組織切片をギルのヘマトキシリンおよびエオシン Y で染色しました。
Images were acquired using the Olympus BX53 light microscope (Olympus Life Science, Japan).
画像は、オリンパス BX53 光学顕微鏡 (オリンパス ライフ サイエンス、日本) を使用して取得しました。
Three to four mice were sampled in each group (as specified in the figure legends) and four to six sections from each animal were used for histology analysis.
各グループで3~4匹のマウスを採取し(図の説明で指定)、各動物から4~6つの切片を組織学的分析に使用しました。
To obtain the semiquantitative histology scoring, tissue sections were graded in a blinded manner according to the pathological changes in the nasal turbinate and lung tissues by an experienced pathologist as previously reported by us and others.
半定量的な組織学的スコアリングを得るために、私たちと他の人が以前に報告したように、経験豊富な病理学者が鼻甲介と肺組織の病理学的変化に応じて盲検法で組織切片を等級付けしました。
In vivo competition assay
6-to-8-week-old female K18-hACE2 transgenic mice were intranasally challenged with mixture of BA.2 and BA.5.2 or BA.4.1 and BA.5.2 at 1:1 ratio based on their infectious viral tires at 5000 PFU of each virus.
6~8週齢のメスのK18-hACE2トランスジェニックマウスに、BA.2とBA.5.2またはBA.4.1とBA.5.2の混合物を、それぞれのウイルスの感染力に基づいて1:1の割合で、各ウイルス5000 PFUで鼻腔内投与した。
Mice were euthanized at 2 dpi. to collect nasal turbinate for RNA extraction with the RNeasy Mini kit (74106, Qiagen, MD, USA) and first strand cDNA synthesis using Transcriptor First Strand cDNA Synthesis Kit (04897030001, Roche, USA).
マウスは2日後に安楽死させ、RNeasy Miniキット(74106、Qiagen、MD、米国)によるRNA抽出と、Transcriptor First Strand cDNA合成キット(04897030001、Roche、米国)による第一鎖cDNA合成のために鼻甲介を採取した。
The viral genome was amplified using Q5® High-Fidelity DNA Polymerase (M0491L, New England Biolabs, USA) with ARTIC network nCoV-2019 primers v4 (Integrated DNA Technologies, USA).
ウイルスゲノムは、ARTICネットワークnCoV-2019プライマーv4(Integrated DNA Technologies、米国)とQ5® High-Fidelity DNAポリメラーゼ(M0491L、New England Biolabs、米国)を使用して増幅した。
The PCR amplicons were purified with SPRIselect beads (B23319, Beckman Coulter, USA) and quantified with Qubit Fluorometer (Thermo Fisher Scientific).
PCR アンプリコンは SPRIselect ビーズ (B23319、Beckman Coulter、米国) で精製され、Qubit Fluorometer (Thermo Fisher Scientific) で定量化されました。
The Illumina libraries were prepared using the KAPA HyperPrep Kit (KK8505, Roche Applied Science, Penzberg, Germany) using 100 ng as input following the manufacture protocol with double-sided size selection to select adapter ligated libraries with the size range 300–750 bp.
Illumina ライブラリは、KAPA HyperPrep キット (KK8505、Roche Applied Science、ペンツベルク、ドイツ) を使用して、100 ng を入力として、製造元のプロトコルに従って、両面サイズ選択でサイズ範囲 300~750 bp のアダプター連結ライブラリを選択し、準備されました。
PCR was performed for library enrichment followed by purification using AMPure XP beads (A63882, Beckman Coulter).
ライブラリの濃縮のためにPCRを行い、その後AMPure XPビーズ(A63882、Beckman Coulter)を使用して精製しました。
The enriched libraries were validated using gel electrophoresis, Qubit and qPCR for quality control analysis.
濃縮されたライブラリは、品質管理分析のためにゲル電気泳動、Qubit、qPCRを使用して検証されました。
The libraries were denatured and diluted to optimal concentration prior sequencing on the Illumina MiSeq System using the MiSeq Reagent Kit v3 (600-cycle) (Illumina, San Diego, USA) for paired-end sequencing (PE301).
ライブラリは変性され、最適な濃度に希釈された後、ペアエンドシーケンシング(PE301)用のMiSeq試薬キットv3(600サイクル)(Illumina、米国サンディエゴ)を使用してIllumina MiSeqシステムでシーケンシングされました。
The output fastq files were subjected to adapter removal using FASTP.
出力された fastq ファイルは、FASTP を使用してアダプター除去を受けました。
The pair end reads were then combined into one read using Paired-End reAd merger (PEAR).
その後、ペアエンド リードは、Paired-End reAd merger (PEAR) を使用して 1 つのリードに統合されました。
Then, the assembled reads were filtered by Quality by FASTP by retaining reads of average Q score of 30, removing reads with at least 10% unqualified bases and length shorter than 300.
次に、組み立てられたリードは、平均 Q スコアが 30 のリードを保持し、少なくとも 10% の不適格な塩基と 300 未満の長さを持つリードを削除することで、FASTP による品質でフィルタリングされました。
The PCR primers were trimmed from the front and the end of each read. Pairwise alignments were performed between the BA.2 (GISAID: EPI_ISL_9845731) and BA.5.2 (GISAID: EPI_ISL_13777658) and between BA.4.1 (GISAID: EPI_ISL_13777657) and BA.5.2 genome sequence using SnapGene (v.3.2.1).
PCR プライマーは、各リードの先頭と末尾から切り取られました。BA.2 (GISAID: EPI_ISL_9845731) と BA.5.2 (GISAID: EPI_ISL_13777658) の間、および BA.4.1 (GISAID: EPI_ISL_13777657) と BA.5.2 のゲノム配列の間で、SnapGene (v.3.2.1) を使用してペアワイズ アライメントが実行されました。
Single nucleotide polymorphism (SNP) sites between the pair of genomes were extracted using SNP-SITES (v.2.5.1).
ゲノムのペア間の一塩基多型 (SNP) サイトは、SNP-SITES (v.2.5.1) を使用して抽出されました。
Clean reads were mapped to the BA.2 and BA.4.1 genome for the competition assay between BA.2 and BA.5.2 and that between BA.4.1 and BA.5.2, respectively using BWA MEM (v.0.7.17) with default setting.
BA.2 と BA.5.2 間の競合アッセイ、および BA.4.1 と BA.5.2 間の競合アッセイでは、それぞれ BWA MEM (v.0.7.17) をデフォルト設定で使用して、クリーン リードが BA.2 および BA.4.1 ゲノムにマッピングされました。
The coverage statistics of both comparisons were retrieved from the BAM files with PYSAMSTATS (v.1.1.2) (https://github.com/alimanfoo/pysamstats).
両方の比較のカバレッジ統計は、PYSAMSTATS (v.1.1.2) を使用して BAM ファイルから取得されました。
The abundance of BA.2, BA.4.1 and BA.5.2 was calculated based on the ratio of reads containing the variant-specific SNPs.
BA.2、BA.4.1、および BA.5.2 の存在量は、バリアント固有の SNP を含むリードの比率に基づいて計算されました。
Infectious virus titration by TCID50 assays and plaque assays
TCID50アッセイとプラークアッセイによる感染性ウイルスの滴定
To quantify infectious viral titre with TCID50 assays, nasal turbinates and lung tissues harvested from infected mice were homogenized in DMEM with Tissue Lyzer II (Qiagen) and clarified supernatants were 10-fold serially diluted and inoculated to monolayered VeroE6-TMPRSS2 cells.
TCID50 アッセイで感染性ウイルス力価を定量化するために、感染マウスから採取した鼻甲介と肺組織を Tissue Lyzer II (Qiagen) を含む DMEM でホモジェナイズし、清澄化した上清を 10 倍連続希釈して単層 VeroE6-TMPRSS2 細胞に接種しました。
Supernatant samples from cells infected with SARS-CoV-2 WT, Delta, BA.1, BA.2, BA.2.12.1, BA.4.1 or BA.5.2 were harvested and 10-fold serially diluted before inoculated onto VeroE6-TMPRSS2 cells.
SARS-CoV-2 WT、Delta、BA.1、BA.2、BA.2.12.1、BA.4.1、または BA.5.2 に感染した細胞から上清サンプルを採取し、10 倍連続希釈してから VeroE6-TMPRSS2 細胞に接種しました。
Cytopathic effect (CPE) was observed at five days post infection for the quantification of the median tissue culture infectious dose.
感染後5日目に細胞変性効果(CPE)を観察し、組織培養感染量の中央値を定量化した。
To compare the plaque size developed by SARS-CoV-2 WT, Omicron BA.1, BA.2, BA.2.12.1, BA.4.1 or BA.5.2, monolayer of VeroE6-TMPRSS2 cells in 12-well plate were challenged with different virus strains at 40 PFU/well.
SARS-CoV-2 WT、Omicron BA.1、BA.2、BA.2.12.1、BA.4.1、またはBA.5.2によって生じたプラークの大きさを比較するため、12ウェルプレートのVeroE6-TMPRSS2細胞の単層に40 PFU/ウェルで異なるウイルス株を接種した。
After 2 h infection, the cells were covered with 1% low-melting agarose in DMEM with 1% FBS for following incubation.
2 時間の感染後、細胞は 1% FBS を含む DMEM 中の 1% 低融点アガロースで覆われ、その後の培養が行われました。
The infected plates were fixed with 4% paraformaldehyde at 1, 2, 3, 4, or 5 dpi, followed by staining with 0.5% crystal violet in 25% ethanol/distilled water for plaque visualization.
感染したプレートは1、2、3、4、または5 dpiで4%パラホルムアルデヒドで固定され、その後プラークを可視化するために25%エタノール/蒸留水中の0.5%クリスタルバイオレットで染色されました。
The diameter of plaques was measured by Adobe Photoshop CC software (v. 2018).
プラークの直径はAdobe Photoshop CCソフトウェア(v. 2018)で測定されました。
Cell viability assays
細胞生存率アッセイ
VeroE6-TMPRSS2 cells were infected with SARS-CoV-2 WT, Delta, BA.1, BA.2, BA.2.12.1, BA.4.1 or BA.5.2 at 0.1 MOI.
VeroE6-TMPRSS2 細胞に、0.1 MOI で SARS-CoV-2 WT、Delta、BA.1、BA.2、BA.2.12.1、BA.4.1、または BA.5.2 を感染させました。
Cell viability was quantified by CellTiter-Glo luminescent cell viability assay kit (G7573, Promega, WI, USA), following manufacturer’s manual with the EnSight Multimode Microplate Reader (Perkin Elmer, USA) at the designated time points.
細胞生存率は、メーカーのマニュアルに従い、EnSight マルチモード マイクロプレート リーダー (Perkin Elmer、米国) で指定された時点で CellTiter-Glo 発光細胞生存率アッセイ キット (G7573、Promega、ウィスコンシン州、米国) によって定量化されました。
Package of SARS-CoV-2-spike pseudoviruses and pseudovirus entry assays
SARS-CoV-2 スパイク擬似ウイルスのパッケージングと擬似ウイルス エントリー アッセイ
SARS-CoV-2-spike pseudoviruses were packaged as previously described.
SARS-CoV-2 スパイク擬似ウイルスは、前述のとおりパッケージングされました。
Briefly, 293T cells were transfected with different spikes with Lipofectamine 3000 (L3000-015, Thermo Fisher Scientific).
簡単に説明すると、293T 細胞に Lipofectamine 3000 (L3000-015、Thermo Fisher Scientific) を用いてさまざまなスパイクをトランスフェクトしました。
At 24 h post transfection, the cells were transduced with VSV-deltaG-firefly pseudotyped with VSV-G.
トランスフェクション後 24 時間で、細胞に VSV-G で擬似型化した VSV-deltaG-firefly を導入しました。
At 2 h post transduction, the cells were washed three times with PBS and cultured in DMEM containing 1%FBS and anti-VSV-G (8G5F11) antibody (EB0010, kerafast, Boston, MA, USA).
形質導入後 2 時間で、細胞を PBS で 3 回洗浄し、1% FBS および抗 VSV-G (8G5F11) 抗体 (EB0010、kerafast、米国マサチューセッツ州ボストン) を含む DMEM で培養しました。
The pseudoviruses were then harvested at 16 h post transduction and titrated with TCID50 assays.
擬似ウイルスは形質導入後 16 時間で採取し、TCID50 アッセイで滴定しました。
For pseudovirus entry assays, target cells were inoculated with pseudoviruses for 2 h and cultured in media containing 1% FBS for 24 h, before washed and lysed for detection of luciferase signal with a luciferase assay system (E1501, Promega, Madison, WI, USA) according to manufacturer’s instructions.
擬似ウイルス侵入アッセイでは、標的細胞に擬似ウイルスを 2 時間接種し、1% FBS を含む培地で 24 時間培養した後、洗浄して溶解し、ルシフェラーゼ アッセイ システム (E1501、Promega、米国ウィスコンシン州マディソン) で製造元の指示に従ってルシフェラーゼ シグナルを検出しました。
All SARS-CoV-2 spike plasmids were obtained from GenScript (Nanjing, China).
すべての SARS-CoV-2 スパイク プラスミドは GenScript (中国南京) から入手しました。
RNA extraction and real-time reverse-transcription polymerase chain reaction
RNA抽出とリアルタイム逆転写ポリメラーゼ連鎖反応
Viral RNA was extracted from infected cells using QIAsymphony RNA Kit (931636, Qiagen).
ウイルスRNAは、QIAsymphony RNAキット(931636、Qiagen)を使用して感染細胞から抽出されました。
Viral RNA of supernatants collected from infected cells was extracted using QIAamp Viral RNA Mini Kit (52906, Qiagen).
感染細胞から収集した上清のウイルスRNAは、QIAamp Viral RNA Mini Kit(52906、Qiagen)を使用して抽出されました。
Viral RNA from mice lung and nasal turbinate samples were extracted with the RNeasy Mini kit (74106, Qiagen).
マウスの肺と鼻甲介のサンプルからのウイルスRNAは、RNeasy Mini kit(74106、Qiagen)を使用して抽出されました。
After RNA extraction, qRT-PCR was performed using QuantiNova Probe RT-PCR Kit (208354, Qiagen) or QuantiNova SYBR Green RT-PCR Kit (208154, Qiagen) with the LightCycler 480 Real-Time PCR System (Roche).
RNA 抽出後、QuantiNova Probe RT-PCR キット (208354、Qiagen) または QuantiNova SYBR Green RT-PCR キット (208154、Qiagen) を LightCycler 480 リアルタイム PCR システム (Roche) とともに使用して qRT-PCR を実行しました。
The primer and probe sequences are available upon request.
プライマーとプローブの配列は、リクエストに応じて入手できます。
Protease inhibitor treatment assay
プロテアーゼ阻害剤処理アッセイ
The serine protease inhibitor, camostat (HY-13512), and the cysteine protease inhibitor, E64D (HY-100229), were purchased from MedChemExpress (Monmouth Junction, NJ, USA).
セリンプロテアーゼ阻害剤カモスタット (HY-13512) およびシステインプロテアーゼ阻害剤 E64D (HY-100229) は MedChemExpress (米国ニュージャージー州モンマスジャンクション) から購入しました。
VeroE6-TMPRSS2 cells were treated with DMSO, camostat, or E64D at concentration of 50 μM for 2 h before pseudovirus transduction.
VeroE6-TMPRSS2 細胞は、擬似ウイルスの導入前に 2 時間、濃度 50 μM の DMSO、カモスタット、または E64D で処理しました。
At 24 hpi, the cell lysates were lysed for detection of luciferase signal.
24 hpi で、細胞溶解物を溶解してルシフェラーゼシグナルを検出しました。
The DMSO-treated cells transduced with pseudovirus carrying the same spike served as the control.
同じスパイクを持つ擬似ウイルスを導入した DMSO 処理細胞を対照として用いた。
Luciferase readings from the inhibitor-treated cells were normalized with the mock-treated controls.
阻害剤処理細胞からのルシフェラーゼの測定値は、模擬処理対照で正規化された。
For protease inhibitor treatment on Calu3 and VeroE6 cells, Calu3 and VeroE6 cells were infected with the indicated Omicron sublineages at 0.5 MOI or 0.1 MOI after treatment with DMSO, camostat, or E64D at 25 μM for 2 h.
Calu3 細胞および VeroE6 細胞に対するプロテアーゼ阻害剤処理では、DMSO、カモスタット、または E64D を 25 μM で 2 時間処理した後、Calu3 細胞および VeroE6 細胞を、0.5 MOI または 0.1 MOI で指定された Omicron サブ系統に感染させました。
At 24 hpi, virus infected cells were lysed for detection of viral sgE gene copies.
24 hpi で、ウイルス感染細胞を溶解し、ウイルス sgE 遺伝子コピーを検出しました。
The human nasal epithelial cells were infected with the indicated Omicron sublineages at 2 MOI after treatment with DMSO, camostat, or E64D at 10 μM for 2 h.
ヒト鼻上皮細胞は、DMSO、カモスタット、またはE64D 10 μMで2時間処理した後、2 MOIで指定されたオミクロン亜系統に感染しました。
At 72 hpi, supernatants were lysed for detection of viral RdRp gene copies.
72 hpiで、ウイルスRdRp遺伝子コピーを検出するために上清を溶解しました。
Western blot analysis of spike cleavage
スパイク切断のウェスタンブロット分析
293T cells were transfected with spike plasmids of SARS-CoV-2 WT (614G), BA.1, BA.2, BA.2.12.1, BA.4/5 and S1/S2Del.
293T細胞に、SARS-CoV-2 WT(614G)、BA.1、BA.2、BA.2.12.1、BA.4/5、およびS1/S2Delのスパイクプラスミドを導入しました。
For infection experiment, VeroE6 cells were challenged with SARS-CoV-2 WT, BA.1, BA.2 or BA.5.2 at 2 MOI.
感染実験では、VeroE6 細胞に SARS-CoV-2 WT、BA.1、BA.2、または BA.5.2 を 2 MOI で導入しました。
Cell lysates were harvested in RIPA buffer (89901, Thermo Fisher Scientific) at 24 h post transfection or infection for Western blot analysis.
ウェスタンブロット分析のために、導入または感染の 24 時間後に RIPA バッファー (89901、Thermo Fisher Scientific) で細胞溶解物を採取しました。
The membranes were blocked with 5% milk for 2h at room temperature and incubated with a rabbit anti-SARS-CoV-2 spike S2 antibody (40590-T62, Sino Biological, China) at 4 °C for overnight incubation, followed by detection with horseradish peroxidase (HRP) conjugated secondary antibodies (31460, Thermo Fisher Scientific) for 1h at room temperature.
メンブレンは室温で2時間5%ミルクでブロックされ、ウサギ抗SARS-CoV-2スパイクS2抗体(40590-T62、Sino Biological、中国)とともに4℃で一晩インキュベートされ、その後、西洋ワサビペルオキシダーゼ(HRP)結合二次抗体(31460、Thermo Fisher Scientific)で室温で1時間検出されました。
The signal was developed using SuperSignal West Pico PLUS Chemiluminescent Substrate (34580, Thermo Scientific) and detected using Alliance Imager apparatus (Uvitec, Cambridge, UK).
シグナルはSuperSignal West Pico PLUS化学発光基質(34580、Thermo Scientific)を使用して開発され、Alliance Imager装置(Uvitec、ケンブリッジ、英国)を使用して検出されました。
β-actin was detected with a β-actin antibody (clone AC-74, A5316, Sigma–Aldrich) (1:5000).
β-アクチンはβ-アクチン抗体(クローンAC-74、A5316、Sigma–Aldrich)(1:5000)で検出されました。
Cell-cell fusion assay
細胞間融合アッセイ
293T cells were co-transfected with different SARS-CoV-2 spike plasmids with GFP1-10 plasmid (cat#68715, Addgene, USA) as effector cells.
293T 細胞は、エフェクター細胞として GFP1-10 プラスミド (cat#68715、Addgene、米国) を含むさまざまな SARS-CoV-2 スパイクプラスミドで共トランスフェクトされました。
Another population of 293T cells was co-transfected with ACE2, TMPRSS2, and GFP11 (cat#68716, Addgene) as target cells.
別の 293T 細胞集団は、ターゲット細胞として ACE2、TMPRSS2、および GFP11 (cat#68716、Addgene) で共トランスフェクトされました。
After 24 h post-transfection, the effector and target cells were digested by EDTA–Trypsin (25200-072, Gibco) and mixed at a 1:1 ratio.
トランスフェクション後 24 時間後、エフェクター細胞とターゲット細胞は EDTA-トリプシン (25200-072、Gibco) で消化され、1:1 の比率で混合されました。
The mixed cells were co-cultured at a 37 °C incubator for another 24 h.
混合細胞は、37 °C のインキュベーターでさらに 24 時間共培養されました。
The mixed cells were fixed in 10% formalin and then permeabilized with 0.1% Triton-X100 (Sigma, USA) at room temperature.
混合細胞を 10% ホルマリンで固定し、室温で 0.1% Triton-X100 (Sigma、米国) で透過処理しました。
The antifade mounting medium with 4′,6-Diamidino-2-Phenylindole, Dihydrochloride (DAPI, H-1200, Vector Laboratories) was used for mounting and DAPI staining.
4′,6-ジアミジノ-2-フェニルインドール二塩酸塩 (DAPI、H-1200、Vector Laboratories) を含む退色防止封入剤を使用して封入および DAPI 染色を行いました。
Images were taken with the Olympus BX73 fluorescence microscope (Olympus Life Science, Tokyo, Japan).
画像は Olympus BX73 蛍光顕微鏡 (Olympus Life Science、東京、日本) で撮影しました。
The fusion area of images was quantified by ImageJ (v.1.8.0_345).
画像の融合領域は ImageJ (v.1.8.0_345) で定量化しました。
Ordinal logistic regression (OLR) of lung pathology score over time
経時的な肺病理スコアの順序ロジスティック回帰 (OLR)
Ordinal logistic regression (OLR) was used to model the lung pathology of K18-hACE2 mice infected by BA.1, BA.2, BA.2.12.1, BA.4.1, or BA.5.2 over time.
BA.1、BA.2、BA.2.12.1、BA.4.1、または BA.5.2 に感染した K18-hACE2 マウスの肺病理を経時的にモデル化するために、順序ロジスティック回帰 (OLR) が使用されました。
The peak count date of Omicron sublineages was retrieved from the SARS-CoV-2 PANGO lineage online tool (https://cov-lineages.org/).
Omicron サブ系統のピークカウント日は、SARS-CoV-2 PANGO 系統オンラインツールから取得されました。
The correlation between the peak count date of each Omicron sublineage since COVID-19 outbreak and the lung pathology score was fitted to the OLR model with the R (v. 4.1.2) package MASS.
COVID-19 の発生以降の各 Omicron サブ系統のピークカウント日と肺病理スコアの相関関係は、R (v. 4.1.2) パッケージ MASS を使用して OLR モデルに適合されました。
Ethics
The use of animals was approved by the Committee on the Use of Live Animals in Teaching and Research of The University of Hong Kong under CULATR 22-139, 5440-20 and 5193-19. Experiment procedures have complied with the approved operation protocol.
Statistical analysis
Statistical comparison between two experimental groups were performed with unpaired two-tailed Student’s t-test. Comparison between three or more experimental groups was performed with one-way or two-way ANOVA. Differences were considered statistically significant when p < 0.05. Data analysis was performed with GraphPad Prism 8.0.
Role of funders
All donors and funding sources had no role in the study design, data collection, analysis, interpretation, or writing of the manuscript.
Results
Comparative virological features of Omicron BA.1, BA.2, BA.4, and BA.5 sublineages
オミクロン BA.1、BA.2、BA.4、および BA.5株のウイルス学的特徴の比較
Over the past year, Omicron has continued to prevail as the dominant SARS-CoV-2 VOC worldwide.
過去 1 年間、オミクロンは世界中で主要な SARS-CoV-2 VOC として優勢を保ってきました。
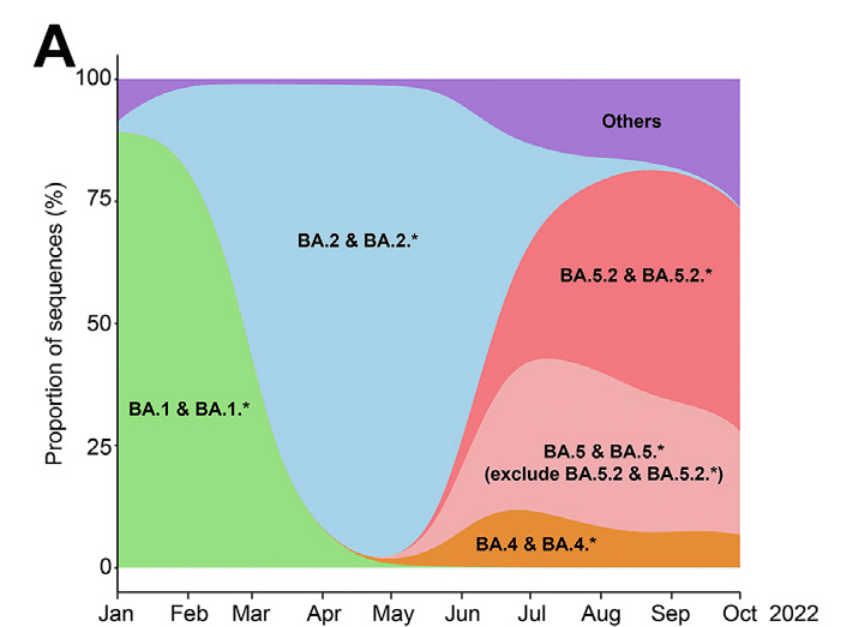
Among the emerged Omicron sublineages, BA.1, BA.2, BA.5, and their related sublineages resulted in the largest waves of infections (Fig. 1A).
出現したオミクロン亜系統のうち、BA.1、BA.2、BA.5、およびそれらの関連亜系統は、最大の感染波をもたらしました (図 1A)。
To perform in parallel comparison on the virological features of Omicron sublineages, we propagated the clinical isolates of BA.1, BA.2, BA.2.12.1, BA.4.1, and BA.5.2 in VeroE6-TMPRSS2 cells.
オミクロン亜系統のウイルス学的特徴を並行して比較するために、BA.1、BA.2、BA.2.12.1、BA.4.1、および BA.5.2 の臨床分離株を VeroE6-TMPRSS2 細胞で増殖させました。
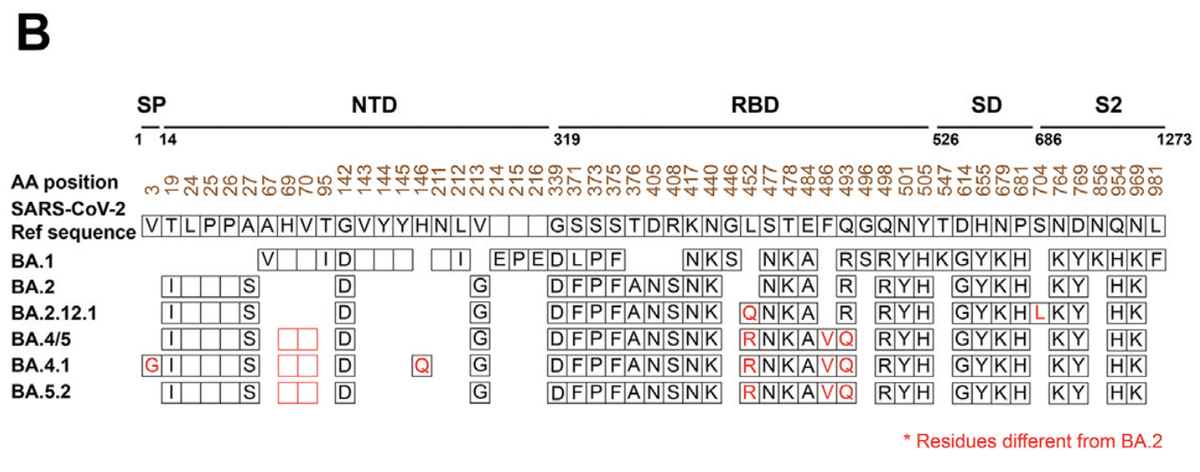
BA.2.12.1 spike differs from BA.2 spike at L452Q and S704L.
BA.2.12.1 スパイクは、L452Q および S704L で BA.2 スパイクと異なります。
BA.4 and BA.5 share the same spike sequence, which differs from BA.2 spike at 69-70del, L452R, F486V, and R493Q.
BA.4 と BA.5 は同じスパイク配列を共有していますが、これは BA.2 スパイクとは 69-70del、L452R、F486V、および R493Q で異なります。
Our BA.4.1 isolate contains a V3G substitution in the signal peptide region and a H146Q substitution when compared to BA.4 spike.
当社の BA.4.1 分離株には、シグナル ペプチド領域に V3G 置換があり、BA.4 スパイクと比較すると H146Q 置換があります。
BA.5.2 and BA.5.2 related sublineages (BA.5.2.∗) are the most dominant BA.5 related sublineages (Fig. 1A).
BA.5.2 および BA.5.2 関連サブ系統 (BA.5.2.∗) は、最も優勢な BA.5 関連サブ系統です (図 1A)。
BA5.2 shares the same spike sequence with BA.4 and BA.5 (Fig. 1B).
BA5.2 は、BA.4 および BA.5 と同じスパイク配列を共有しています (図 1B)。
To compare the replication kinetics of different sublineages, we first infected Calu3 and VeroE6 cells with SARS-CoV-2 wildtype (WT), Delta, BA.1, BA.2, BA.2.12.1, BA.4.1, or BA.5.2 and harvested samples at different time points post infection.
異なる亜系統の複製速度を比較するために、まずCalu3およびVeroE6細胞にSARS-CoV-2野生型(WT)、Delta、BA.1、BA.2、BA.2.12.1、BA.4.1、またはBA.5.2を感染させ、感染後のさまざまな時点でサンプルを採取しました。
We assessed virus replication in cell lysates by quantification of subgenomic envelope (E) gene expression, which is a replication intermediate of SARS-CoV-2.
SARS-CoV-2の複製中間体であるサブゲノムエンベロープ(E)遺伝子発現を定量化することで、細胞溶解物中のウイルス複製を評価しました。
In parallel, we quantified the level of infectious virus particles in the supernatants with 50% tissue culture infectious dose (TCID50) assays.
並行して、50%組織培養感染量(TCID50)アッセイで上清中の感染性ウイルス粒子のレベルを定量化しました。
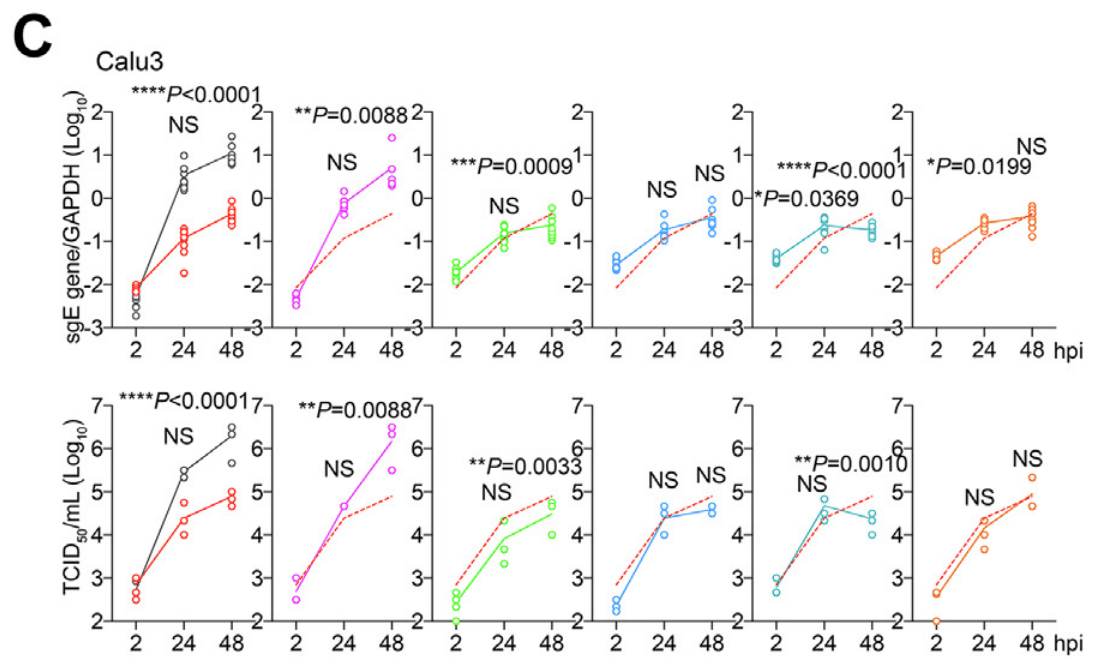
Our results demonstrated that all five Omicron sublineages replicated less efficiently in Calu3 cells when compared to that of WT and Delta.
結果は、5 つの Omicron サブ系統すべてが Calu3 細胞で WT および Delta と比較して複製効率が低いことを実証しました。
Among the Omicron sublineages, BA.5.2 replicated to the highest level in Calu3 cells, which was statistically different in infectious titre than that of BA.1 and BA.2.12.1 at 48 h post infection (hpi) (Fig. 1C).
Omicron サブ系統のうち、BA.5.2 は Calu3 細胞で最も高いレベルで複製され、感染後 48 時間 (hpi) の感染力価は BA.1 および BA.2.12.1 と統計的に異なっていました (図 1C)。
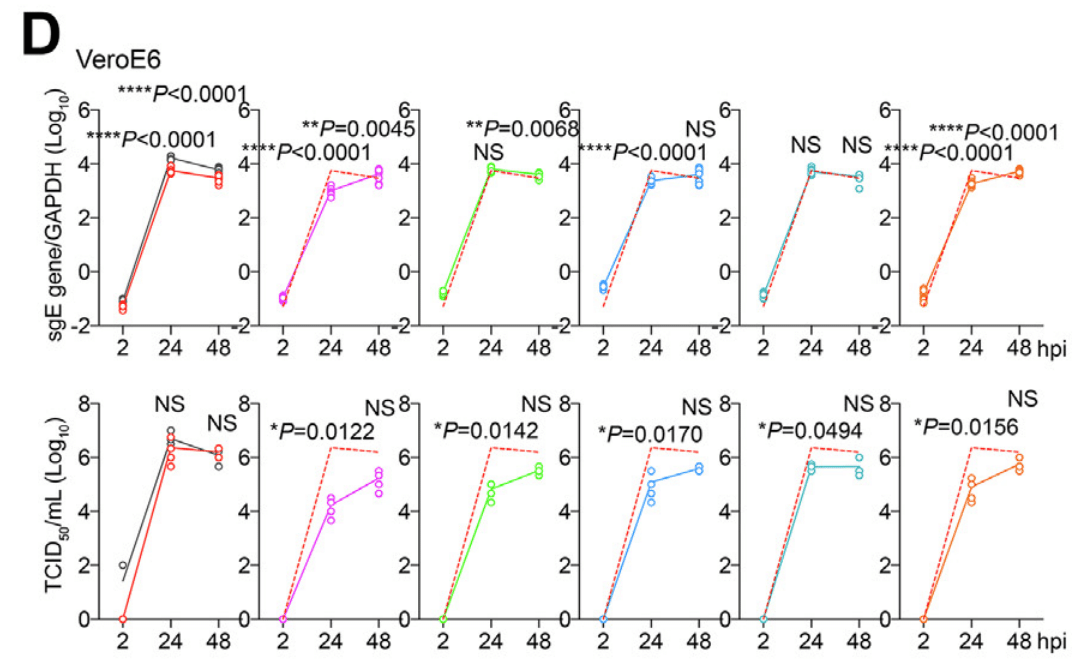
In VeroE6 cells, BA.5.2 infection generated higher infectious titre than that of Delta, BA.1, BA.2, BA.2.12.1, BA.4.1, and were comparable to that of WT (Fig. 1D).
VeroE6 細胞では、BA.5.2 感染は Delta、BA.1、BA.2、BA.2.12.1、BA.4.1 よりも高い感染力価を生成し、WT と同等でした (図 1D)。
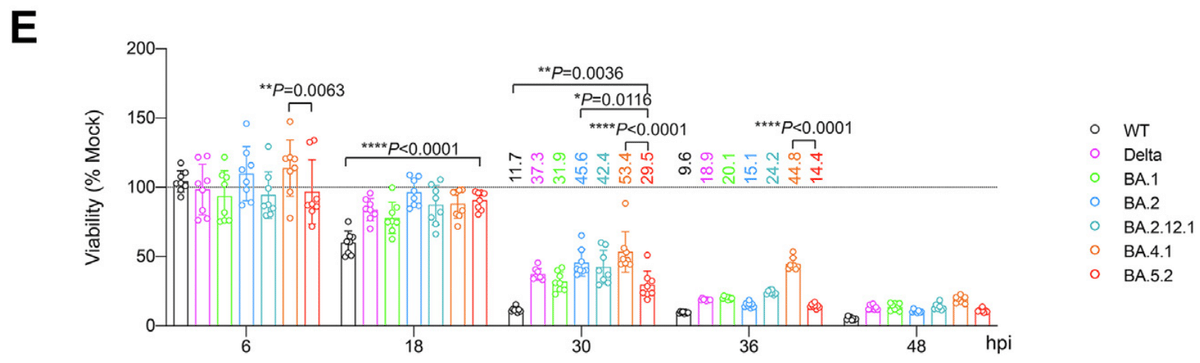
In keeping with its better replication, BA.5.2 infection resulted in lower cell viability in VeroE6-TMPRSS2 cells when compared to Delta, BA.1, BA.2, BA.2.12.1, and BA.4.1 at 30 and 36 hpi, while WT remained as the most cytopathic SARS-CoV-2 strain evaluated (Fig. 1E).
BA.5.2 感染は、その優れた複製と一致して、感染後 30 および 36 時間で、Delta、BA.1、BA.2、BA.2.12.1、および BA.4.1 と比較して VeroE6-TMPRSS2 細胞の細胞生存率が低下しましたが、WT は評価した SARS-CoV-2 株の中で最も細胞変性が進んでいました (図 1E)。
Next, we assessed the virological features of Omicron spikes.
次に、オミクロンスパイクのウイルス学的特徴を評価しました。
We found that cleavage of BA.1 and BA.2 spike at S1/S2 in the infected cells were less efficient than WT (D614G) (Fig. 2A), in keeping with previous studies from us and others.
感染細胞における S1/S2 での BA.1 および BA.2 スパイクの切断は、WT (D614G) よりも効率が低いことがわかりました (図 2A)。これは、私たちおよび他の研究者による以前の研究と一致しています。
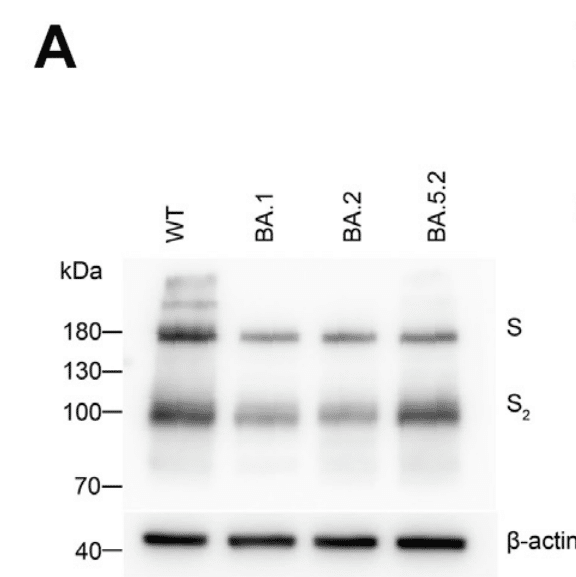
Interestingly, cleavage of BA.5 spike was more efficient than that of BA.1 and BA.2 (Fig. 2A).
興味深いことに、BA.5 スパイクの切断は BA.1 および BA.2 よりも効率的でした (図 2A)。
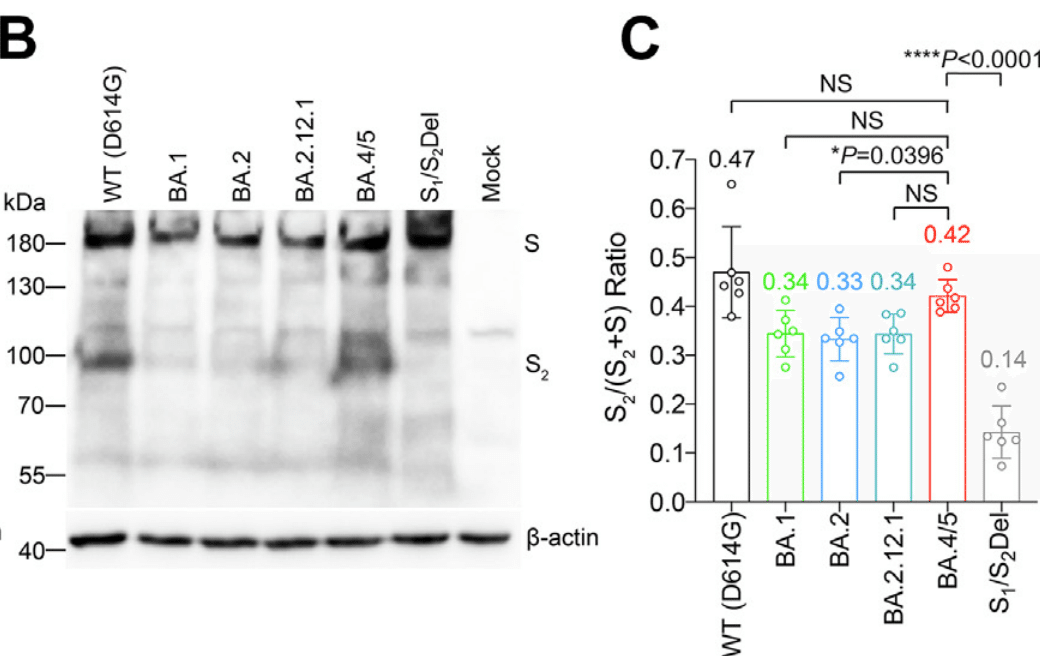
To further dissect spike cleavage irrespective of non-spike viral proteins, we transfected 293T cells with spike expression plasmid of WT (D614G), BA.1, BA.2, BA.2.12.1, BA.4/5, and S1/S2Del (a WT spike mutant with deletions flanking the S1/S2 site) as a negative control, and harvested cell lysates at 24 h post transfection to detect spike cleavage.
スパイク以外のウイルスタンパク質に関係なくスパイク切断をさらに分析するために、293T 細胞に WT (D614G)、BA.1、BA.2、BA.2.12.1、BA.4/5、および S1/S2Del (S1/S2 部位の両側に欠失がある WT スパイク変異体) のスパイク発現プラスミドをネガティブ コントロールとして導入し、導入後 24 時間で細胞溶解物を採取してスパイク切断を検出しました。
Consistent with the observations in infected cells, we showed that BA.1, BA.2, and S1/S2Del spikes were less readily cleaved at S1/S2 than WT (D614G).
感染細胞での観察と一致して、BA.1、BA.2、および S1/S2Del スパイクは WT (D614G) よりも S1/S2 で切断されにくいことがわかりました。
While the cleavage of BA.2.12.1 spike was highly comparable with that of BA.2 spike, the cleavage of BA.4/5 spike was significantly more efficient than that of BA.2 (P = 0.0396), and was comparable with that of WT (D614G) (P = 0.4321) (Fig. 2B and C).
BA.2.12.1 スパイクの切断は BA.2 スパイクの切断とほぼ同等でしたが、BA.4/5 スパイクの切断は BA.2 よりも大幅に効率的であり (P = 0.0396)、WT (D614G) の切断と同等でした (P = 0.4321) (図 2B および C)。
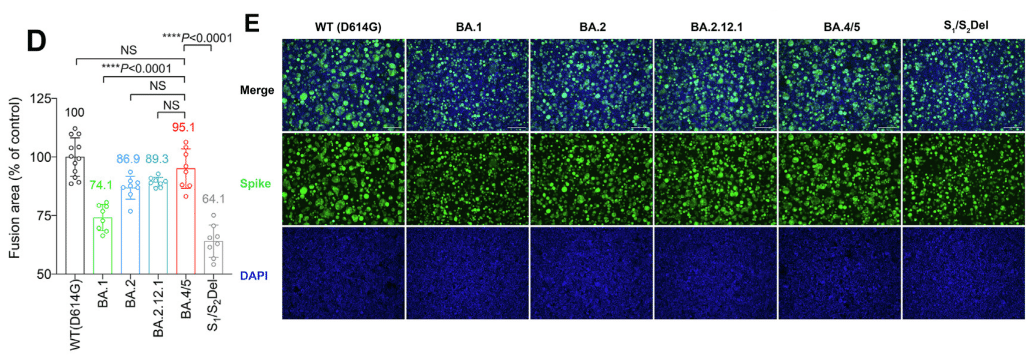
In parallel, we analyzed spike-mediated cell-cell fusion in 293T cells using the split GFP fusion assays.
並行して、スプリット GFP 融合アッセイを使用して、293T 細胞におけるスパイク媒介細胞間融合を分析しました。
In line with the spike cleavage results, BA.4/5 spike most efficiently mediated cell-cell fusion among the evaluated Omicron sublineages, and induced a comparable level of cell-cell fusion compared with that of WT (D614G) (P = 0.3379) (Fig. 2D and E).
スパイク切断の結果と一致して、BA.4/5 スパイクは、評価した Omicron サブ系統の中で最も効率的に細胞間融合を媒介し、WT (D614G) と比較して同等レベルの細胞間融合を誘導しました (P = 0.3379) (図 2D および E)。
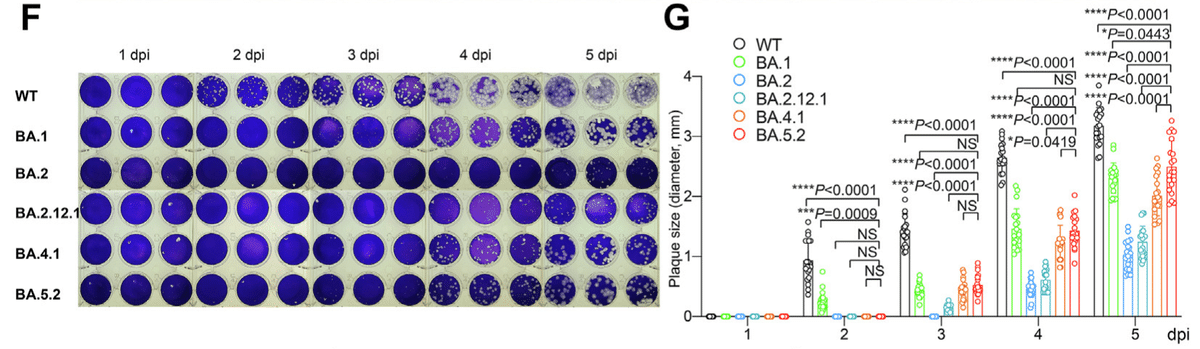
We next infected VeroE6-TMPRSS2 cells with WT, BA.1, BA.2, BA.2.12.1, BA.4.1, and BA.5.2, and quantified plaque formation at different time points post infection.
次に、VeroE6-TMPRSS2 細胞に WT、BA.1、BA.2、BA.2.12.1、BA.4.1、および BA.5.2 を感染させ、感染後のさまざまな時点でのプラーク形成を定量化しました。
We found that BA.5.2 infection resulted in the largest plaques among the evaluated Omicron sublineages on 5 days post infection, followed by BA.1.
BA.5.2 感染は、感染後 5 日目に評価した Omicron サブ系統の中で最大のプラークをもたらし、次に BA.1 が大きなプラークをもたらしたことがわかりました。
However, plaques formed upon WT infection remained significantly larger than that of BA.5.2 at 2 to 5 dpi (Fig. 2F and G).
しかし、WT 感染時に形成されたプラークは、感染後 2 ~ 5 日で BA.5.2 のプラークよりも大幅に大きいままでした (図 2F および G)。
The more efficient spike cleavage at S1/S2 suggests that BA.4/5 may be more efficient in transmembrane serine protease 2 (TMPRSS2)-mediated entry.
S1/S2 でのスパイク切断がより効率的であることから、BA.4/5 は膜貫通型セリンプロテアーゼ 2 (TMPRSS2) を介した侵入においてより効率的である可能性が示唆されます。
However, in pseudovirus entry assays, we found that the efficiency of TMPRSS2 usage by BA.4/5 pseudoviruses was similar to that of BA.2 pseudoviruses, and lower than that of WT (D614G) (P < 0.0001) and BA.1 pseudoviruses (P = 0.0002) (Fig. 2H).
ただし、擬似ウイルス侵入アッセイでは、BA.4/5 擬似ウイルスによる TMPRSS2 の使用効率は BA.2 擬似ウイルスと同程度で、WT (D614G) (P < 0.0001) および BA.1 擬似ウイルス (P = 0.0002) よりも低いことがわかりました (図 2H)。
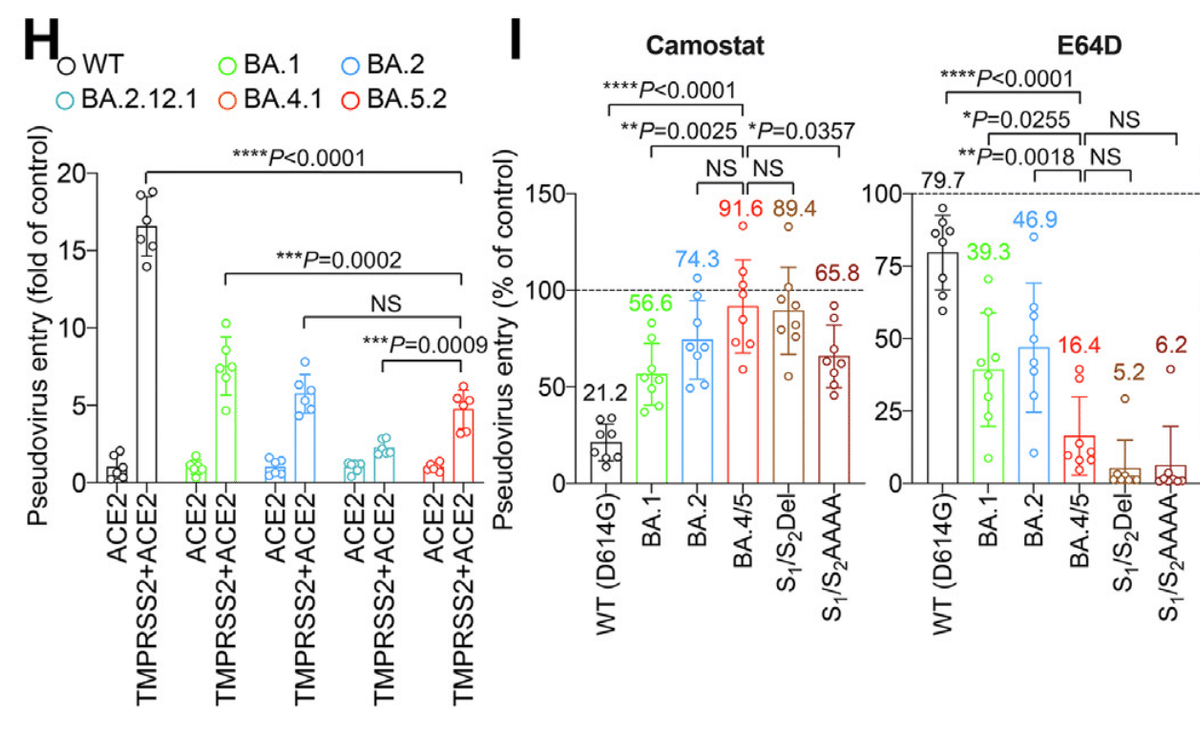
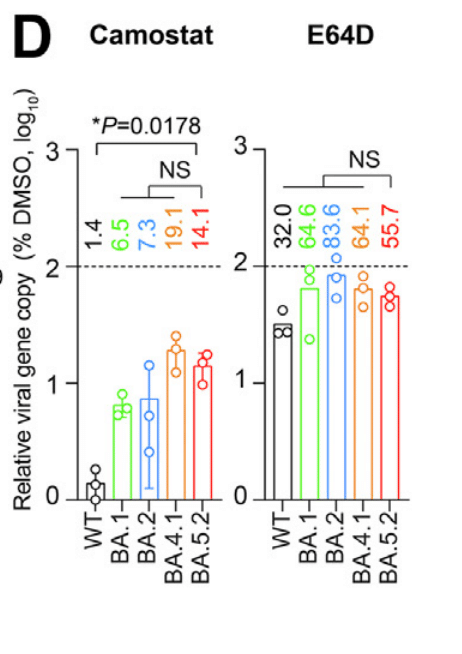
This observation agreed with results obtained in pseudovirus entry assays in the presence of camostat (pan-serine protease inhibitor), which demonstrated inefficient inhibition of BA.4/5 pseudovirus entry with camostat in VeroE6-TMPRSS2 cells (Fig. 2I).
この観察結果は、カモスタット(汎セリンプロテアーゼ阻害剤)存在下での擬似ウイルス侵入試験で得られた結果と一致した。 この試験では、カモスタットによる BA.4/5 擬似ウイルスの侵入阻害は VeroE6-TMPRSS2 細胞では不十分であることが示された(図 2I)。
In contrast, BA.4/5 pseudovirus entry was more efficiently inhibited by E64D (pan-cysteine protease inhibitor) when compared to WT (D614G), BA.1, and BA.2 pseudovirus, and was inhibited to a similar degree compared to S1/S2Del and S1/S2AAAA (a WT spike mutant with the S1/S2 PRRA motif changed to AAAA) (Fig. 2I).
対照的に、BA.4/5 擬似ウイルスの侵入は、WT(D614G)、BA.1、および BA.2 擬似ウイルスと比較して、E64D(汎システインプロテアーゼ阻害剤)によってより効率的に阻害され、S1/S2Del および S1/S2AAAA(S1/S2 PRRA モチーフが AAAA に変更された WT スパイク変異体)と比較しても同程度に阻害された(図 2I)。
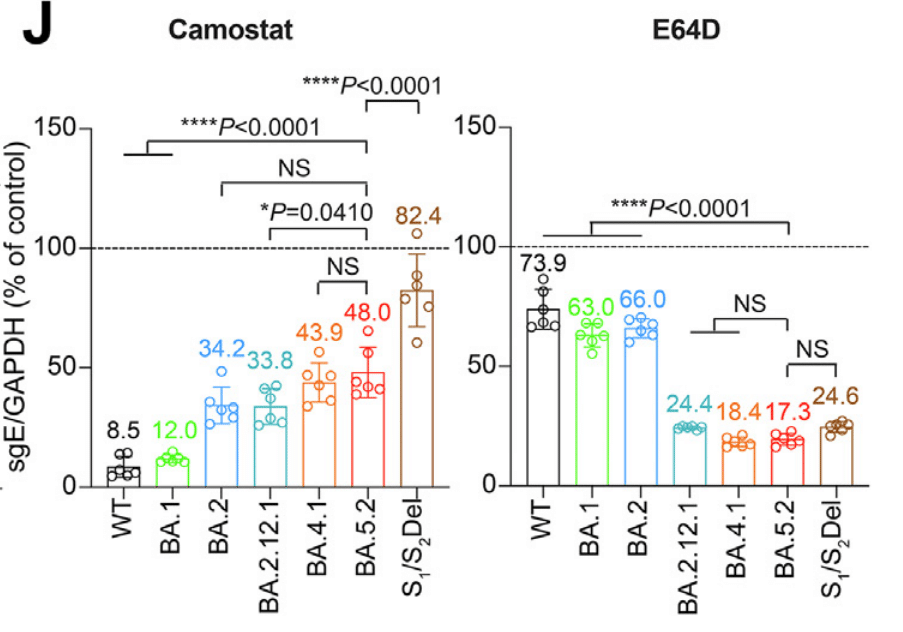
To verify the data obtained from using pseudoviruses, Calu3 and VeroE6 cells were pretreated with camostat or E64D, followed by infection with WT (D614G), BA.1, BA.2, BA2.12.1, BA.4.1, BA.5.2 and S1/S2Del.
擬似ウイルスを使用して得られたデータを検証するために、Calu3 細胞と VeroE6 細胞をカモスタットまたは E64D で前処理し、その後 WT (D614G)、BA.1、BA.2、BA2.12.1、BA.4.1、BA.5.2、および S1/S2Del で感染させました。
In keeping with the pseudovirus findings, BA.5.2 showed reduced sensitivity to camostat in Calu3 cells and increased sensitivity to E64D in VeroE6 cells when compared with WT and earlier Omicron sublineages (Fig. 2J).
擬似ウイルスの所見と一致して、BA.5.2 は WT および以前の Omicron 亜系統と比較して、Calu3 細胞ではカモスタットに対する感受性が低下し、VeroE6 細胞では E64D に対する感受性が上昇しました (図 2J)。
Replication fitness of Omicron BA.1, BA.2, BA.4, and BA.5 sublineages in primary human nasal epithelial cells
初代ヒト鼻粘膜上皮細胞におけるオミクロン BA.1、BA.2、BA.4、および BA.5 サブ系統の複製適応度
Omicron sublineages robustly evade neutralization antibody responses, which in part explained their highly efficient transmission in the immunized or previously exposed populations.
オミクロン亜系統は中和抗体反応を強力に回避しており、これが免疫付与された集団や以前に感染した集団における非常に効率的な伝播を部分的に説明している。
Meanwhile, the replication capacity of Omicron sublineages at the human nasal cavity, which may similarly play a role in their transmissibility, has not been thoroughly investigated.
一方、同様に伝染性に関与している可能性があるヒトの鼻腔におけるオミクロン亜系統の複製能力は、十分に調査されていません。
To this end, we sought to assess the replication fitness of SARS-CoV-2 WT, BA.1, BA.2, BA.4.1, or BA.5.2 in primary human nasal epithelial cells (hNECs).
この目的のために、私たちは、初代ヒト鼻上皮細胞(hNEC)におけるSARS-CoV-2 WT、BA.1、BA.2、BA.4.1、またはBA.5.2の複製適合性を評価しようとしました。
We first showed that the respiratory tract cell type markers for basal cells (P63, CK5), ciliated cells (FOXJ1, SNTN), and club cells (CC10) are significantly upregulated in the hNECs than that of lung epithelial Calu3 cells (Fig. 3A).
私たちはまず、基底細胞(P63、CK5)、繊毛細胞(FOXJ1、SNTN)、およびクラブ細胞(CC10)の呼吸器系細胞型マーカーが、肺上皮Calu3細胞よりもhNECで大幅にアップレギュレーションされていることを示しました(図3A)。
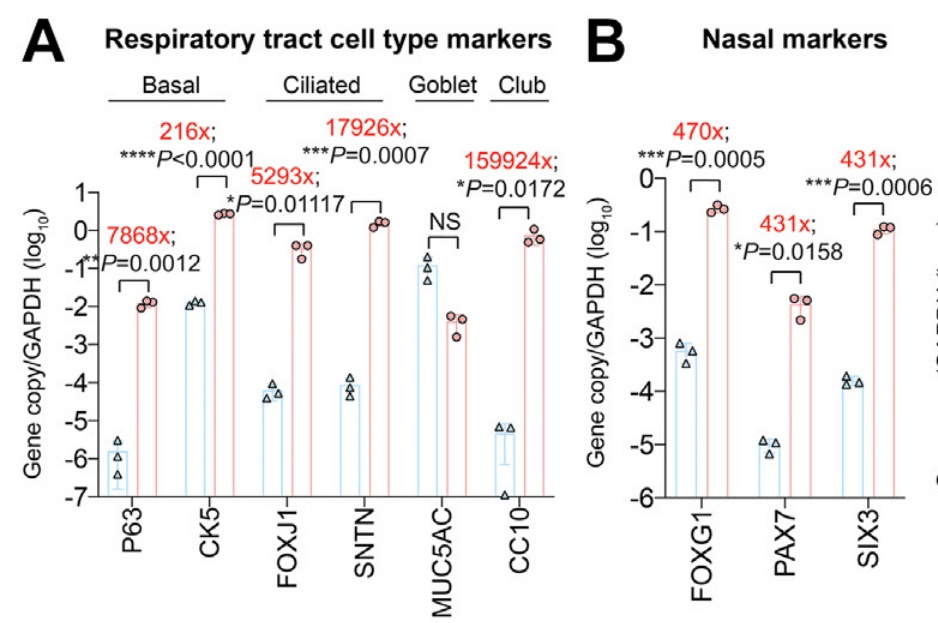
In addition, we evaluated the expression of nasal markers (FOXG1, PAX7, SIX3) in the hNECs, and found that they are more highly expressed than that in Calu3 cells (Fig. 3B), suggesting that the compartment-specific gene expression profile of the hNECs is consistent with the human nasal epithelium as previously reported in single-cell sequencing analysis.
さらに、hNEC における鼻マーカー (FOXG1、PAX7、SIX3) の発現を評価したところ、Calu3 細胞よりも高い発現が見られました (図 3B)。これは、hNEC のコンパートメント特異的遺伝子発現プロファイルが、以前に単一細胞シーケンス解析で報告されたように、ヒト鼻上皮と一致していることを示唆しています。
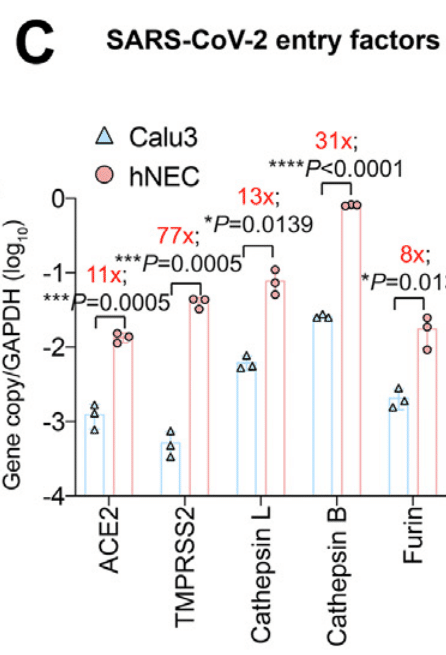
In parallel, we found that hNECs expressed SARS-CoV-2 entry factors (ACE2, TMPRSS2, cathepsin L, cathepsin B, and furin) at significantly higher levels than Calu3 cells (Fig. 3C).
同時に、hNEC は、Calu3 細胞よりも有意に高いレベルで SARS-CoV-2 侵入因子 (ACE2、TMPRSS2、カテプシン L、カテプシン B、およびフーリン) を発現していることもわかりました (図 3C)。
SARS-CoV-2 entry in hNECs predominantly occurred through the TMPRSS2-mediated plasma membrane pathway since SARS-CoV-2 entry was more readily inhibited by camostat than E64D (Fig. 3D).
SARS-CoV-2 の hNEC への侵入は主に TMPRSS2 を介した細胞膜経路を介して発生しました。これは、SARS-CoV-2 の侵入が E64D よりもカモスタットによって容易に阻害されたためです (図 3D)。
Upon virus infection, we harvested supernatants at 2, 24, 48, and 72 hpi and quantified the level of infectious virus production with TCID50 assays.
ウイルス感染後、2、24、48、72 hpi で上清を採取し、TCID50 アッセイで感染性ウイルスの産生レベルを定量化しました。
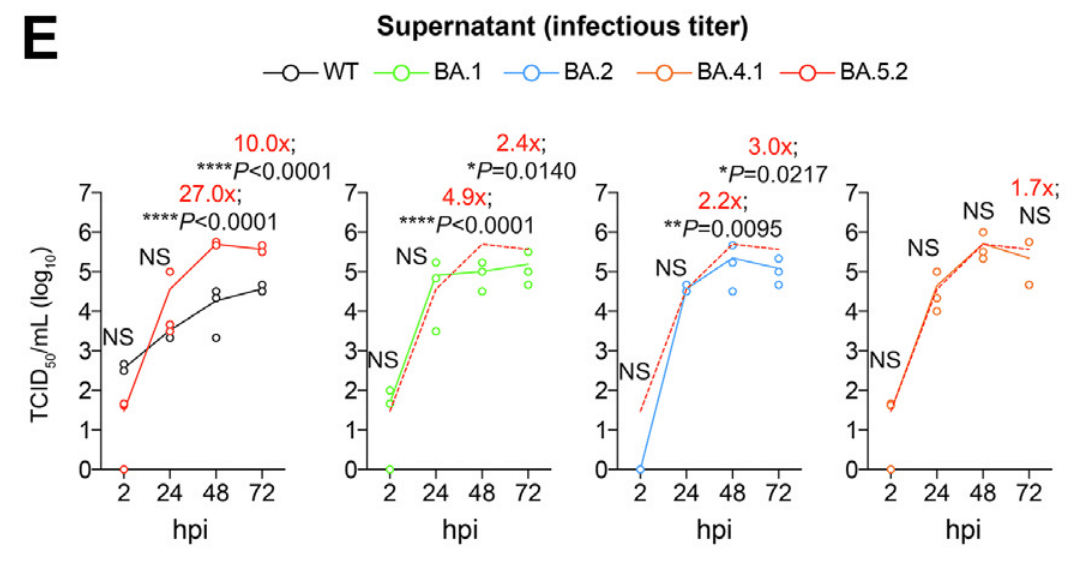
We found that while all Omicron sublineages replicated more efficiently than that of WT, BA.5.2 demonstrated the highest level of replication among all Omicron sublineages.
すべての Omicron 亜系統は WT よりも効率的に複製しましたが、BA.5.2 はすべての Omicron 亜系統の中で最も高いレベルの複製を示しました。
At 48 hpi, BA.5.2 produced 27.0-folds (P < 0.0001), 4.9-folds (P < 0.0001), and 2.2-folds (P = 0.0095) higher infectious titre than that of WT, BA.1, and BA.2, respectively, and was comparable with that of BA.4.1.
48 hpi では、BA.5.2 はそれぞれ WT、BA.1、BA.2 よりも 27.0 倍 (P < 0.0001)、4.9 倍 (P < 0.0001)、2.2 倍 (P = 0.0095) 高い感染力価を示し、BA.4.1 と同等でした。
At 72 hpi, BA.5.2 produced 10.0-folds (P < 0.0001), 2.4-folds (P = 0.0140), 3.0-folds (P = 0.0217), and 1.7-fold (P = 0.8277) higher infectious titre than that of WT, BA.1, BA.2, and BA.4.1, respectively (Fig. 3E).
72 hpiでは、BA.5.2はWT、BA.1、BA.2、BA.4.1と比較して、それぞれ10.0倍(P < 0.0001)、2.4倍(P = 0.0140)、3.0倍(P = 0.0217)、1.7倍(P = 0.8277)高い感染力価を示しました(図3E)。
Intriguingly, there is a trend of increasing fitness of SARS-CoV-2 replication in the hNECs, which was also evidenced by quantification of the RdRp gene in the supernatant samples and subgenomic E gene in the cell lysate samples at 72 hpi (Fig. 3F and G).
興味深いことに、hNEC における SARS-CoV-2 複製の適応度が増加する傾向があり、これは 72 hpi での上清サンプルの RdRp 遺伝子と細胞溶解物サンプルのサブゲノム E 遺伝子の定量によっても証明されました (図 3F および G)。
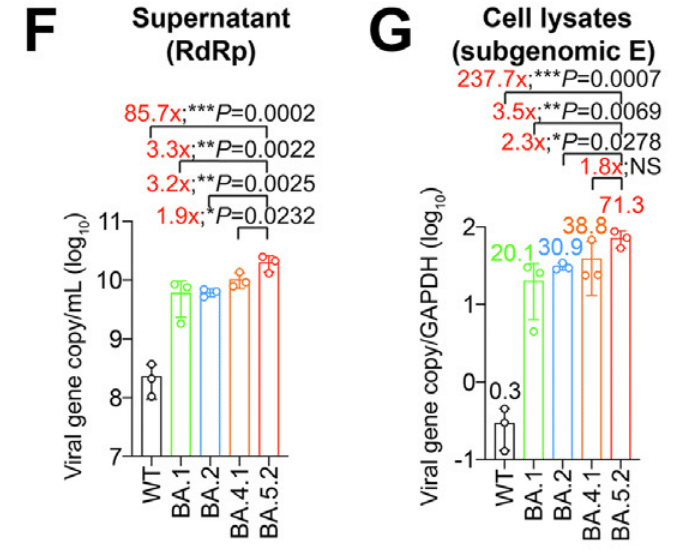
In the cell lysates, the subgenomic E gene level upon BA.5.2 infection was 237.7-folds (P = 0.0007), 3.5-folds (P = 0.0069), 2.3-folds (P = 0.0278), and 1.8-fold (P = 0.0787) higher than that of WT, BA.1, BA.2, and BA.4.1, respectively (Fig. 3G).
細胞溶解物では、BA.5.2 感染時のサブゲノム E 遺伝子レベルは、WT、BA.1、BA.2、および BA.4.1 と比較して、それぞれ 237.7 倍 (P = 0.0007)、3.5 倍 (P = 0.0069)、2.3 倍 (P = 0.0278)、および 1.8 倍 (P = 0.0787) 高くなりました (図 3G)。
We further evaluated the antigen expression of WT-, BA.1-, BA.2-, BA.4.1-, and BA.5.2-infected hNECs by immunofluorescence staining at 24 hpi.
さらに、感染後24時間で免疫蛍光染色により、WT、BA.1、BA.2、BA.4.1、およびBA.5.2に感染したhNECの抗原発現を評価しました。
Our data indicated that while all Omicron sublineages could target ACCTUB + ciliated cells in the hNECs, the highest number of nucleocapsid (N)-expressing cells were detected in BA.5.2-infected samples, followed by BA.4.1, BA.2, and BA.1 in a descending order (Fig. 3H).
データによると、すべてのOmicronサブ系統がhNECのACCTUB +繊毛細胞を標的としましたが、ヌクレオカプシド(N)発現細胞が最も多く検出されたのはBA.5.2感染サンプルで、次いでBA.4.1、BA.2、およびBA.1の順でした(図3H)。
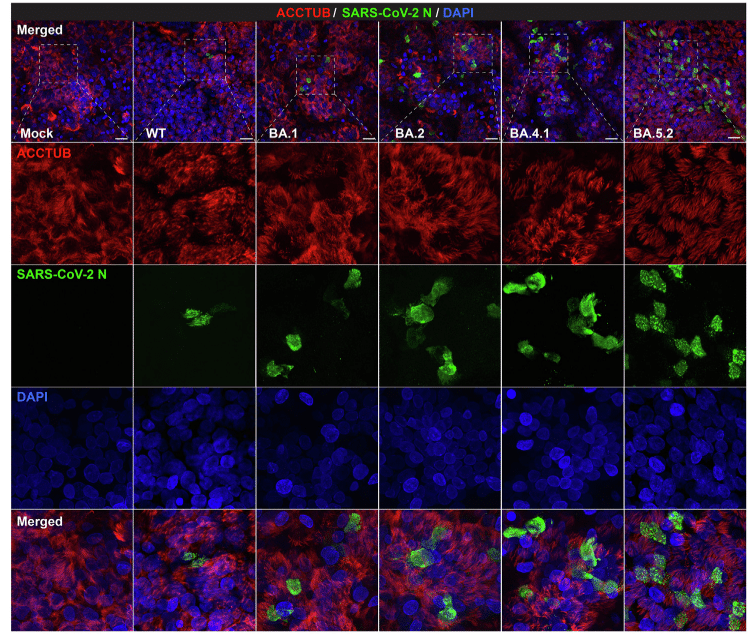
In contrast, N-expressing ciliated cells were scarcely detected in the WT-infected hNECs (Fig. 3H).
対照的に、N発現繊毛細胞はWT感染hNECではほとんど検出されませんでした(図3H)。
Together, these results revealed that all Omicron sublineages replicated significantly more robustly than WT in hNECs.
これらの結果を合わせると、すべてのOmicron亜系統がhNECでWTよりも大幅に強力に複製されたことが明らかになりました。
In addition, the sequentially emerged Omicron sublineages are gaining further replication fitness in the hNECs.
さらに、連続的に出現したオミクロンのサブ系統は、hNEC 内でさらなる複製適応性を獲得しています。
Replication and pathogenicity of Omicron BA.1, BA.2, BA.4, and BA.5 sublineages in K18-hACE2 transgenic mice
K18-hACE2 トランスジェニックマウスにおける Omicron BA.1、BA.2、BA.4、および BA.5 サブ系統の複製と病原性
To investigate the in vivo replication fitness of the dominant Omicron sublineages at different stages of the Omicron wave, we intranasally challenged 6-to-10-week-old K18-hACE2 transgenic mice with 5000 PFU of BA.1, BA.2, BA.2.12.1, BA.4.1 or BA.5.2, and euthanized the infected mice at 2 dpi to collect nasal turbinates and lung tissues for virological assessment (Fig. 4A–D).
オミクロン波のさまざまな段階での優勢なオミクロン亜系統の生体内複製適応度を調べるために、6~10週齢のK18-hACE2トランスジェニックマウスに5000 PFUのBA.1、BA.2、BA.2.12.1、BA.4.1、またはBA.5.2を鼻腔内投与し、感染マウスを2日後に安楽死させて鼻甲介と肺組織を採取し、ウイルス学的評価を行いました(図4A~D)。
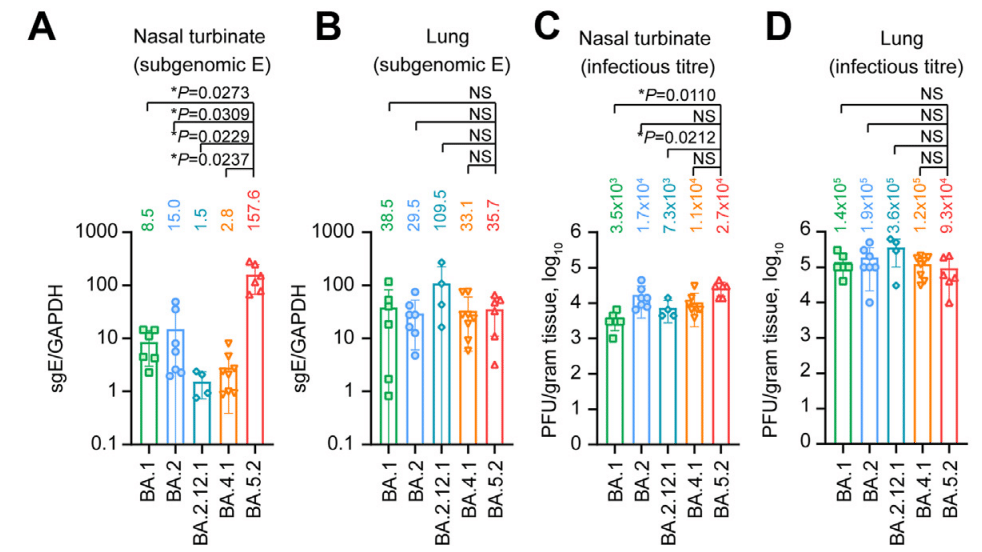
In the nasal turbinate samples, BA.5.2 infection resulted in the highest viral sgE gene copies, which was 18.5- (P = 0.0273) and 10.5-folds (P = 0.0309) higher than that of BA.1 and BA.2, respectively (Fig. 4A).
鼻甲介サンプルでは、BA.5.2感染によりウイルスsgE遺伝子コピー数が最も多くなり、BA.1およびBA.2よりもそれぞれ18.5倍(P = 0.0273)、10.5倍(P = 0.0309)高くなりました(図4A)。
Consistently, the infectious viral titre of BA.5.2-infected mouse nasal turbinates were 7.7- (P = 0.0110) and 1.6-fold (P = 0.4480) higher than that of the BA.1- and BA.2-infected mouse nasal turbinates, respectively (Fig. 4C).
一貫して、BA.5.2 に感染したマウスの鼻甲介の感染性ウイルス力価は、BA.1 および BA.2 に感染したマウスの鼻甲介のそれよりもそれぞれ 7.7 倍 (P = 0.0110) および 1.6 倍 (P = 0.4480) 高かった (図 4C)。
We next assessed the replication of BA.5.2 in the lungs of the infected K18-hACE2 mice, and detected comparable RNA levels for BA.1, BA.2, and BA.5.2 (Fig. 4B).
次に、感染した K18-hACE2 マウスの肺における BA.5.2 の複製を評価し、BA.1、BA.2、および BA.5.2 の RNA レベルが同等であることが検出されました (図 4B)。
The mean infectious titre of BA.5.2 (0.9 × 10⁵ PFU/g) in lungs of infected animals was slightly lower than that of BA.1 (1.4 × 10⁵ PFU/g) and BA.2 (1.9 × 10⁵ PFU/g), but did not reach statistical significance (Fig. 4D).
感染した動物の肺における BA.5.2 の平均感染力価 (0.9 × 10⁵ PFU/g) は、BA.1 (1.4 × 10⁵ PFU/g) および BA.2 (1.9 × 10⁵ PFU/g) よりもわずかに低かったものの、統計的有意性には達しませんでした (図 4D)。
To further characterize the viral antigen expression in vivo, we performed immunohistochemistry staining to detect the SARS-CoV-2 nucleocapsid (N) protein in nasal turbinates and lungs of the infected mice (Fig. 4E–H).
生体内でのウイルス抗原発現をさらに特徴付けるために、感染マウスの鼻甲介と肺で SARS-CoV-2 ヌクレオカプシド (N) タンパク質を検出する免疫組織化学染色を実施しました (図 4E~H)。
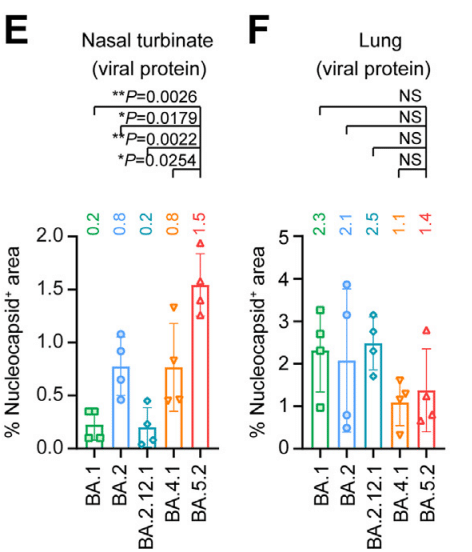
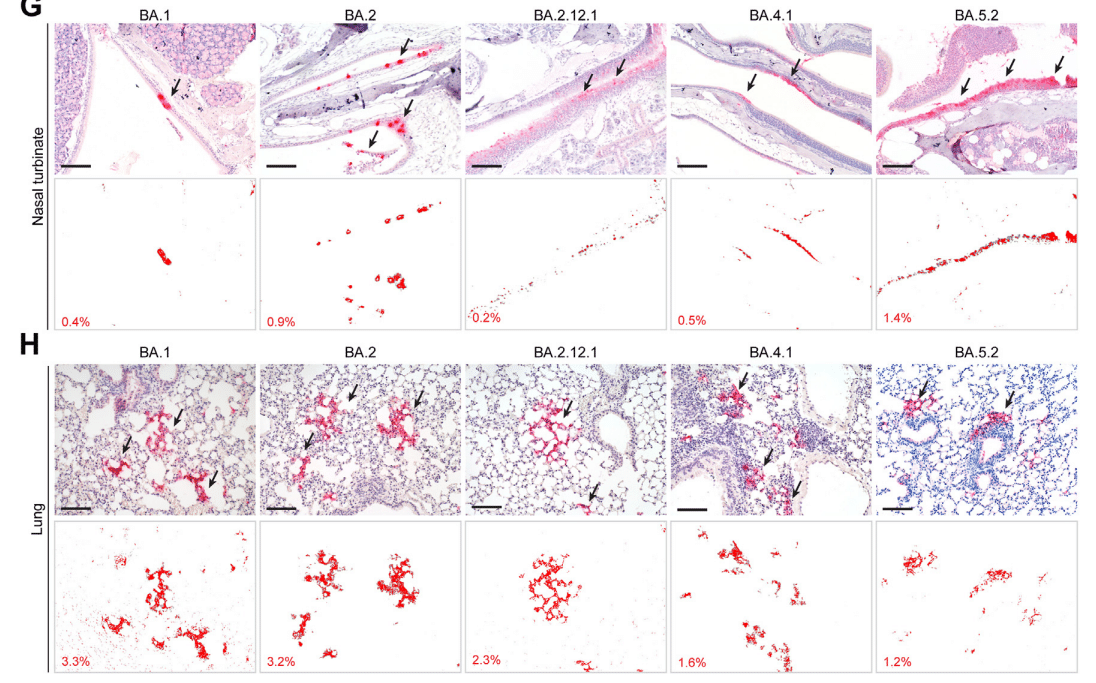
In keeping with the virus burden findings in the nasal turbinate, N protein was most abundantly detected in the nasal epithelium of BA.5.2-infected mice when compared to their counterparts infected with BA.1, BA.2, BA.2.12.1, or BA.4.1 (Fig. 4G).
鼻甲介におけるウイルス負荷の所見と一致して、BA.1、BA.2、BA.2.12.1、または BA.4.1 に感染したマウスと比較した場合、BA.5.2 に感染したマウスの鼻上皮で N タンパク質が最も多く検出されました (図 4G)。
With quantification, the N protein-covered area in nasal turbinates of BA.5.2-infected mice was 7.5- (P = 0.0026) and 1.9-folds (P = 0.0179) higher than that of BA.1- and BA.2-infected mice, respectively (Fig. 4E).
定量化により、BA.5.2 感染マウスの鼻甲介における N タンパク質で覆われた面積は、BA.1 感染マウスおよび BA.2 感染マウスの面積よりそれぞれ 7.5 倍 (P = 0.0026) および 1.9 倍 (P = 0.0179) 高くなりました (図 4E)。
Consistent with the infectious titre results, N protein in the lungs of BA.5.2-infected mice (1.4%) was less readily detected when compared to those of BA.1- (2.3%) and BA.2-infected mice (2.1%) (Fig. 4F and H).
感染力価の結果と一致して、BA.5.2 感染マウスの肺における N タンパク質 (1.4%) は、BA.1 感染マウス (2.3%) および BA.2 感染マウス (2.1%) と比較して検出されにくかった (図 4F および H)。
To investigate the pathogenicity caused by the Omicron sublineages, we performed comparative histopathological analysis with haematoxylin and eosin (H&E) staining on the nasal turbinate and lung tissues harvested at 2 dpi.
オミクロン亜系統によって引き起こされる病原性を調べるため、感染後2日目に採取した鼻甲介と肺組織をヘマトキシリン・エオシン(H&E)染色で比較組織病理学的分析を行った。
We first examined the nasal turbinate tissues of BA.5.2-infected mice and found that the pathological changes were featured by extensive epithelium damage.
まず、BA.5.2に感染したマウスの鼻甲介組織を検査したところ、病理学的変化は広範囲にわたる上皮損傷を特徴としていることが判明した。
Integrity of the epithelium lining was destroyed due to severe epithelial cell loss, while necrotic epithelial cell deposition and proteinaceous exudates were frequently identified in the nasal cavity (Fig. 5A).
上皮細胞の著しい損失により上皮内層の完全性が破壊され、鼻腔内には壊死性上皮細胞の沈着とタンパク質性滲出液が頻繁に確認された(図5A)。
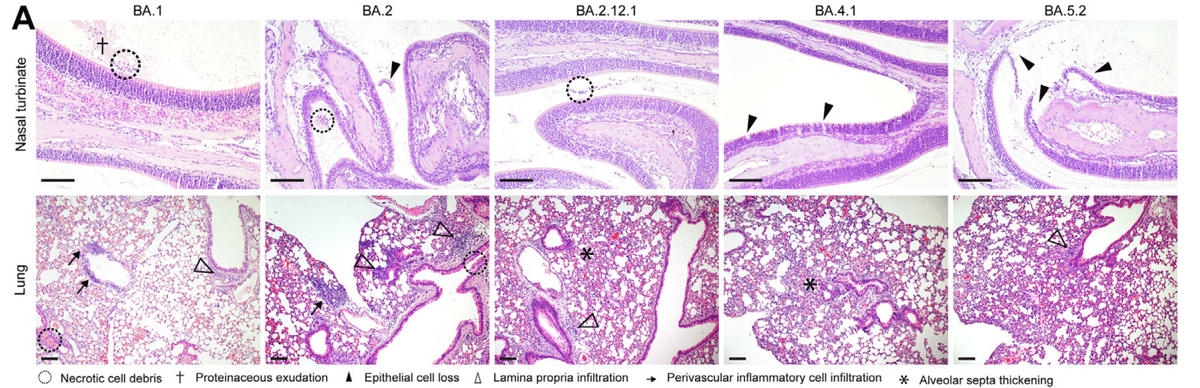
Similarly, significant epithelium desquamation and cell necrosis were also observed in the nasal turbinate of BA.2-infected mice (Fig. 5A).
同様に、BA.2 感染マウスの鼻甲介でも、顕著な上皮剥離と細胞壊死が観察されました (図 5A)。
In contrast, scattered nasal epithelial cell loss was occasionally spotted in the nasal epithelium of BA.4.1-infected mice, while the nasal epithelium lining in BA.1- and BA.2.12.1-infected animals remained largely intact, with only a small amount of inflammatory cell infiltrates accompanied with proteinaceous exudates found in the nasal cavity (Fig. 5A).
対照的に、BA.4.1 感染マウスの鼻上皮では、散発的な鼻上皮細胞の喪失が時折見られましたが、BA.1 および BA.2.12.1 感染動物の鼻上皮内層はほぼ無傷のままで、鼻腔内にはタンパク質性滲出液を伴う炎症細胞浸潤が少量見られるだけでした (図 5A)。
We adopted a semi-quantitative histopathological scoring system to describe the pathological findings as previously reported.
私たちは、以前に報告されたように、病理学的所見を説明するために半定量的な組織病理学的スコアリング システムを採用しました。
In the nasal turbinates, BA.5.2 infection contributed to the highest pathology scores, followed by BA.2 (Fig. 5B–D).
鼻甲介では、BA.5.2 感染が最も高い病理スコアに寄与し、次いで BA.2 でした (図 5B~D)。
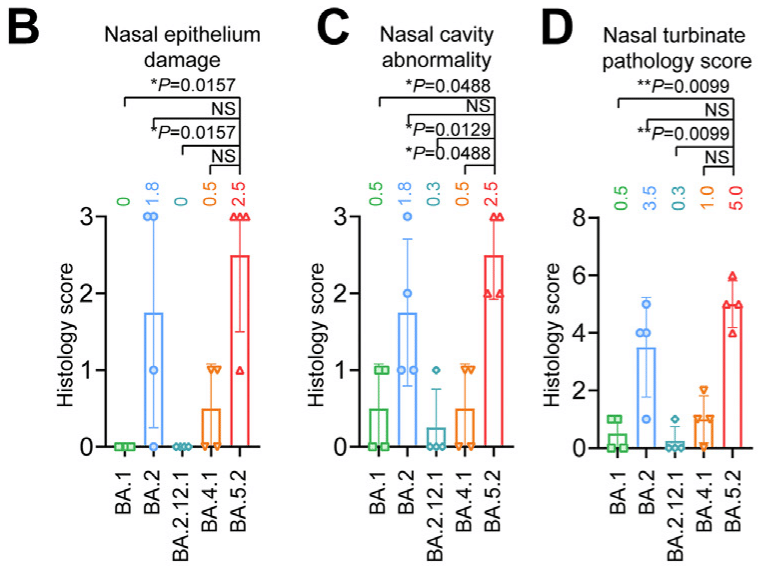
The mean pathology score in the nasal turbinate of BA.5.2-infected mice was 5.0, which was higher than that of BA.2-infected mice (3.5) and BA.1-infected mice (0.5) (Fig. 5D).
BA.5.2 感染マウスの鼻甲介の平均病理スコアは 5.0 で、BA.2 感染マウス (3.5) や BA.1 感染マウス (0.5) よりも高かったです (図 5D)。
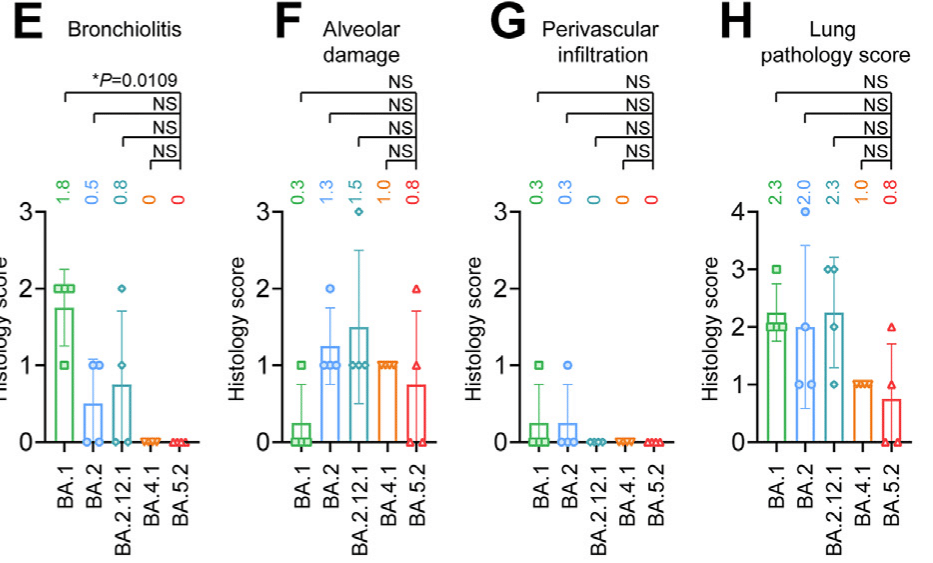
Next, we examined the histopathological changes in the lung tissues. In general, inflammation in the lung was mild among all Omicron sublineages examined.
次に、肺組織の組織病理学的変化を調べました。一般的に、調べたすべてのオミクロン亜系統において、肺の炎症は軽度でした。
Localized inflammatory infiltration in the lamina propria, alveolar septa, and perivascular area was universally identified in all experimental groups (Fig. 5A).
粘膜固有層、肺胞中隔、血管周囲領域における局所的な炎症性浸潤は、すべての実験グループで普遍的に確認されました (図 5A)。
Semi-quantitative scoring identified a total lung pathology score of 0.8 in BA.5.2-infected mice, which was lower than that of BA.2-infected mice (2.0) and BA.1-infected mice (2.3) (Fig. 5E–H).
半定量的スコアリングにより、BA.5.2 感染マウスの肺病理スコアは合計 0.8 と判定されましたが、これは BA.2 感染マウス (2.0) や BA.1 感染マウス (2.3) よりも低い値でした (図 5E~H)。
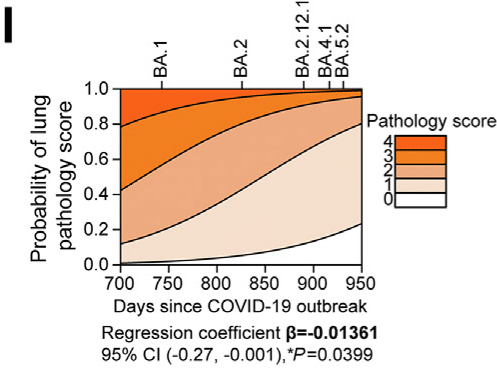
We performed an ordinal logistic regression (OLR) based on the total lung pathology score and the peak count date of BA.1, BA.2, BA.2.12.1, BA.4.1, and BA.5.2, and found that the sequentially emerged Omicron sublineages are significantly declining in intrinsic lung pathology over time (β = −0.01361, P = 0.0399) (Fig. 5I).
肺病理スコアの合計と BA.1、BA.2、BA.2.12.1、BA.4.1、および BA.5.2 のピークカウント日に基づいて順序ロジスティック回帰 (OLR) を実行したところ、順次出現した Omicron サブ系統は、時間の経過とともに内因性肺病理が著しく低下していることがわかりました (β = −0.01361、P = 0.0399) (図 5I)。
In comparison, lungs of the mock-infected littermates were absence of any identified pathological changes (Supplementary Fig. S1).
それに比べて、模擬感染した同腹仔の肺には、確認された病理学的変化は見られませんでした (補足図 S1)。
Collectively, our histopathological findings corroborated with the virological assessment results and provided further evidence that the increasing replication fitness of Omicron sublineages in the nasal turbinates results in more severe pathological damage in the nasal epithelium, with BA.5.2 infection causes the most severe damage among all evaluated Omicron sublineages.
全体として、私たちの組織病理学的所見は、ウイルス学的評価結果と裏付けられ、鼻甲介におけるオミクロン亜系統の複製適応度の増加が鼻上皮のより重篤な病理学的損傷をもたらし、評価されたすべてのオミクロン亜系統の中で BA.5.2 感染が最も重篤な損傷を引き起こすというさらなる証拠を提供しました。
In mouse lungs, Omicron sublineages are continuously attenuated and BA.5.2 resulted in milder pathology when compared to the previous dominant Omicron sublineages, BA.2 and BA.1.
マウスの肺では、オミクロン亜系統は継続的に弱毒化しており、BA.5.2 は、以前の優勢なオミクロン亜系統である BA.2 および BA.1 と比較して、より軽度の病理をもたらしました。
Replication fitness of BA.2, BA.4 and BA.5 in K18-hACE2 transgenic mice
K18-hACE2トランスジェニックマウスにおけるBA.2、BA.4、BA.5の複製適応度
As evidenced by the more robust replication fitness of BA.5.2 over BA.2 and BA.4.1 in the nasal turbinates, we postulated that BA.5.2 might be able to outcompete BA.2 and BA.4.1 in the nasal turbinates of infected mice.
鼻甲介における BA.5.2 の複製適応度が BA.2 および BA.4.1 より強固であることから、感染マウスの鼻甲介において BA.5.2 が BA.2 および BA.4.1 に打ち勝つ可能性があると仮定しました。
To evaluate this hypothesis, we carried out competition assays between BA.5.2 and BA.2 by inoculating a mixture of the two viruses (each at 5000 PFU) to the K18-hACE2 mice.
この仮説を評価するために、2 つのウイルスの混合物 (それぞれ 5000 PFU) を K18-hACE2 マウスに接種して、BA.5.2 と BA.2 の競合試験を実施しました。
At 2dpi, we harvested nasal turbinate tissues for detection of the viral genome abundance with next generation sequencing (NGS) analysis.
感染後2日目に、次世代シーケンシング(NGS)解析でウイルスゲノムの存在量を検出するために鼻甲介組織を採取しました。
The NGS results indicated that BA.5.2 rapidly outcompeted BA.2 in the nasal turbinate of all four infected mice at as early as 2dpi (Fig. 5J).
NGSの結果は、感染したマウス4匹すべての鼻甲介において、感染後2日目という早い段階でBA.5.2がBA.2を急速に打ち負かしたことを示しました(図5J)。
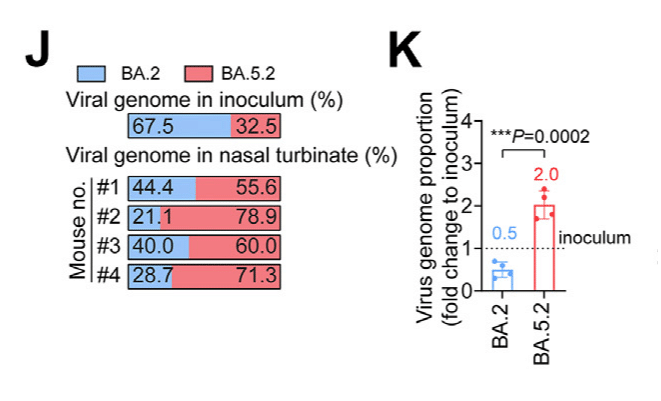
The mean virus genome proportion of BA.5.2 in the nasal turbinate samples increased to 2-folds when compared to that of the inoculum (32.5% in the inoculum vs 66.4% in harvested samples), while that of BA.2 was decreased to 0.5-fold (67.5% in the inoculum vs 33.6% in harvested samples) (P = 0.0002) (Fig. 5K).
鼻甲介サンプル中の BA.5.2 の平均ウイルスゲノム比率は、接種物と比較して 2 倍に増加しました (接種物では 32.5%、採取サンプルでは 66.4%)。一方、BA.2 は 0.5 倍に減少しました (接種物では 67.5%、採取サンプルでは 33.6%) (P = 0.0002) (図 5K)。
We next compared the viral fitness between BA.5.2 and BA.4.1, and showed that BA.5.2 similarly outcompeted BA.4.1 in the nasal turbinates of all three co-infected mice at 2 dpi (Fig. 5L).
次に、BA.5.2 と BA.4.1 のウイルス適応度を比較し、2 dpi で BA.5.2 が BA.4.1 を同様に上回ったことを示しました (図 5L)。
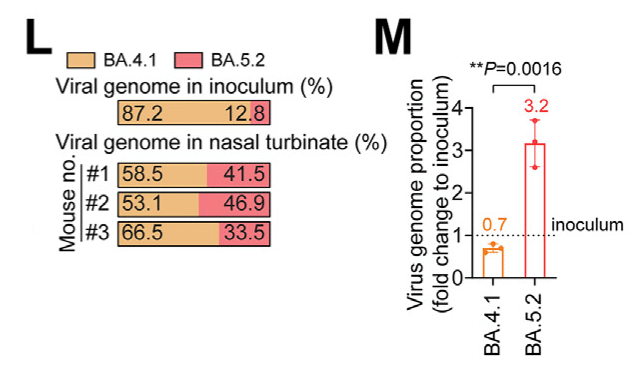
The mean virus genome abundance of BA.5.2 increased to 3.2-folds when compared to that of the inoculum while that of BA.4.1 diminished to 0.7-fold (P = 0.0016) (Fig. 5M).
BA.5.2 の平均ウイルスゲノム存在量は接種物と比較して 3.2 倍に増加しましたが、BA.4.1 は 0.7 倍に減少しました (P = 0.0016) (図 5M)。
Thus, these results indicated that BA.5.2 demonstrated better replication fitness than BA.2 and BA.4.1 in the nasal turbinates of infected mice.
したがって、これらの結果は、感染したマウスの鼻甲介において、BA.5.2 が BA.2 および BA.4.1 よりも優れた複製適合性を示したことを示しています。
Taken together, the nasal turbinate findings from K18-hACE2 mice agreed with results obtained from the hNECs, indicating that the Omicron sublineages are gaining replication fitness in the nasal epithelium of humans and mice, with BA.5.2 demonstrated the most robust replication capacity.
総合すると、K18-hACE2 マウスの鼻甲介の所見は hNEC から得られた結果と一致し、オミクロン亜系統がヒトおよびマウスの鼻上皮で複製適合性を獲得しつつあり、BA.5.2 が最も強力な複製能力を示したことを示しています。
Meanwhile, the trend is different in the lungs with BA.5.2 demonstrated lower antigen expression when compared with the previous dominant Omicron sublineages BA.1 and BA.2.
一方、肺では傾向が異なり、BA.5.2 は、以前の優勢なオミクロン亜系統 BA.1 および BA.2 と比較して、抗原発現が低いことが示されました。
Replication fitness and pathogenicity of Omicron sublineages in young C57BL/6, A129, and aged C57BL/6 mice
若いC57BL/6、A129、老齢C57BL/6マウスにおけるオミクロン亜系統の複製適応度と病原性
Since Omicron sublineages carry the N501Y mutation in spikes that renders infectivity to wildtype (WT) mice (Supplementary Fig. S2), we further infected young C57BL/6 mice (aged 6–8 weeks), aged C57BL/6 mice (aged 10–11 months), and the type I interferon receptor knockout A129 mice (aged 6–8 weeks) to investigate the differential replication and pathogenicity characteristics of Omicron sublineages between the immunocompromised and immunocompetent mouse models.
オミクロン亜系統はスパイクにN501Y変異を持ち、野生型(WT)マウスに感染性を与えるため(補足図S2)、我々はさらに若いC57BL/6マウス(6~8週齢)、高齢C57BL/6マウス(10~11か月齢)、およびI型インターフェロン受容体ノックアウトA129マウス(6~8週齢)に感染させ、免疫不全マウスモデルと免疫正常マウスモデル間のオミクロン亜系統の異なる複製および病原性特性を調査した。
We intranasally challenged the three animal models with 1 × 10⁵ PFU of BA.2, BA.4.1 and BA.5.2 and measured viral burdens in the nasal turbinate and lung tissues at 2 dpi.
3つの動物モデルに1×10⁵ PFUのBA.2、BA.4.1、およびBA.5.2を鼻腔内投与し、2 dpiで鼻甲介および肺組織のウイルス量を測定しました。
Additionally, we used the N501Y-carrying B.1.351 (Beta) as a control, which was previously shown to be capable of infecting WT murines.
さらに、N501Y を保有する B.1.351 (ベータ) を対照として使用しました。これは、以前に WT マウスに感染できることが示されています。
As expected, B.1.351 replicated at high levels in both nasal turbinates and lung tissues in all three mouse models (Fig. 6A and B).
予想通り、B.1.351 は 3 つのマウス モデルすべてにおいて鼻甲介と肺組織の両方で高レベルで複製されました (図 6A および B)。
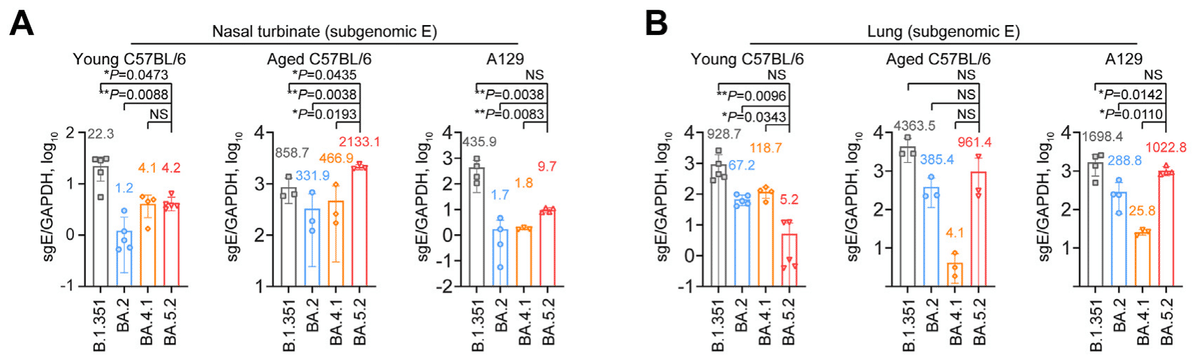
In the nasal turbinates of young C57BL/6 mice, while the replication of all evaluated Omicron sublineages was attenuated compared with B.1.351, the replication of BA.5.2 was significantly more efficient than that of BA.2 (P = 0.0088) (Fig. 6A), in keeping with our findings in K18-hACE2 mice.
若い C57BL/6 マウスの鼻甲介では、評価されたすべての Omicron サブ系統の複製は B.1.351 と比較して弱まりましたが、BA.5.2 の複製は BA.2 よりも大幅に効率的でした (P = 0.0088) (図 6A)。これは、K18-hACE2 マウスでの調査結果と一致しています。
In the nasal turbinates of aged C57BL/6 mice, while the replication of Omicron BA.2 was attenuated compared with B.1.351, the replication of BA.5.2 was significantly more robust than that of BA.2 (P = 0.0038) and B.1.351 (P = 0.0435) (Fig. 6A).
老齢 C57BL/6 マウスの鼻甲介では、Omicron BA.2 の複製は B.1.351 と比較して弱まりましたが、BA.5.2 の複製は BA.2 (P = 0.0038) および B.1.351 (P = 0.0435) よりも大幅に強力でした (図 6A)。
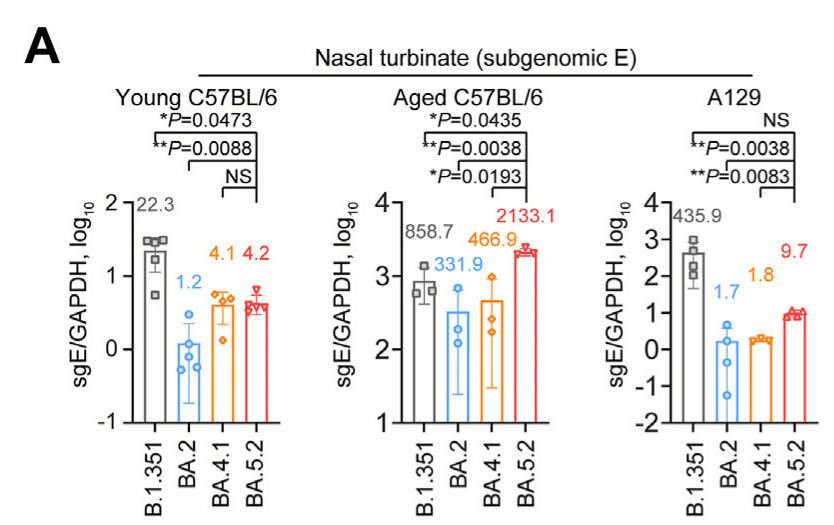
In the nasal turbinates of A129 mice, the replication of BA.5.2 was significantly more efficient than that of BA.2 but was lower than that of B.1.351, similar to the pattern observed in young C57BL/6 mice (Fig. 6A).
A129 マウスの鼻甲介では、BA.5.2 の複製は BA.2 よりも大幅に効率的でしたが、B.1.351 よりも低く、若い C57BL/6 マウスで観察されたパターンと同様でした (図 6A)。
Overall, the viral sgE copy in the nasal turbinates of BA.5.2-infected mice was 3.5- (P = 0.0088), 6.4- (P = 0.0038), and 5.7-folds (P = 0.0038) higher than that of BA.2 in young C57BL/6, aged C57BL/6, and A129 mice, respectively (Fig. 6A).
全体的に、BA.5.2 に感染したマウスの鼻甲介におけるウイルス sgE コピーは、若い C57BL/6、老齢の C57BL/6、および A129 マウスにおける BA.2 よりそれぞれ 3.5 倍 (P = 0.0088)、6.4 倍 (P = 0.0038)、および 5.7 倍 (P = 0.0038) 高かった (図 6A)。
In the lung tissues of young C57BL/6 mice, while BA.2 replication was reduced when compared to B.1.351, BA.5.2 replication was further attenuated by 12.9-folds when compared to BA.2 (P = 0.0096) (Fig. 6B).
若い C57BL/6 マウスの肺組織では、BA.2 の複製は B.1.351 と比較して減少したが、BA.5.2 の複製は BA.2 と比較してさらに 12.9 倍弱まった (P = 0.0096) (図 6B)。
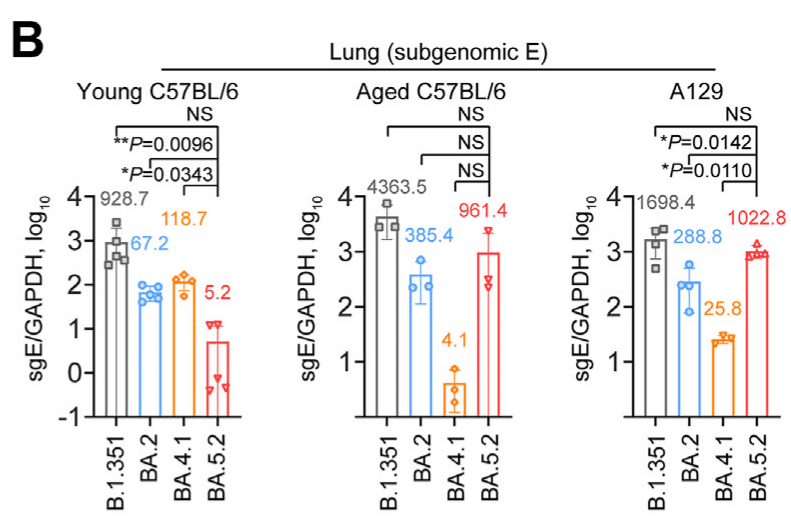
In contrast to the observation from young C57BL/6 mice, in the lung tissues of aged C57BL/6 mice and A129 mice, the replication of BA.5.2 was not attenuated when compared to BA.2 (Fig. 6B).
若い C57BL/6 マウスの観察とは対照的に、高齢の C57BL/6 マウスおよび A129 マウスの肺組織では、BA.2 と比較して BA.5.2 の複製は弱まっていませんでした (図 6B)。
We next determined the production of progeny viruses in lungs with plaque assays.
次に、プラーク アッセイで肺における子孫ウイルスの生成を測定しました。
Our data indicated that the infectious viral titre of BA.5.2 was 22-folds (P = 0.0100) lower in comparison to BA.2 in the lungs of young C57BL/6 mice (Fig. 6C).
データによると、若い C57BL/6 マウスの肺では、BA.5.2 の感染性ウイルス力価は BA.2 の 22 倍 (P = 0.0100) 低いことがわかりました (図 6C)。
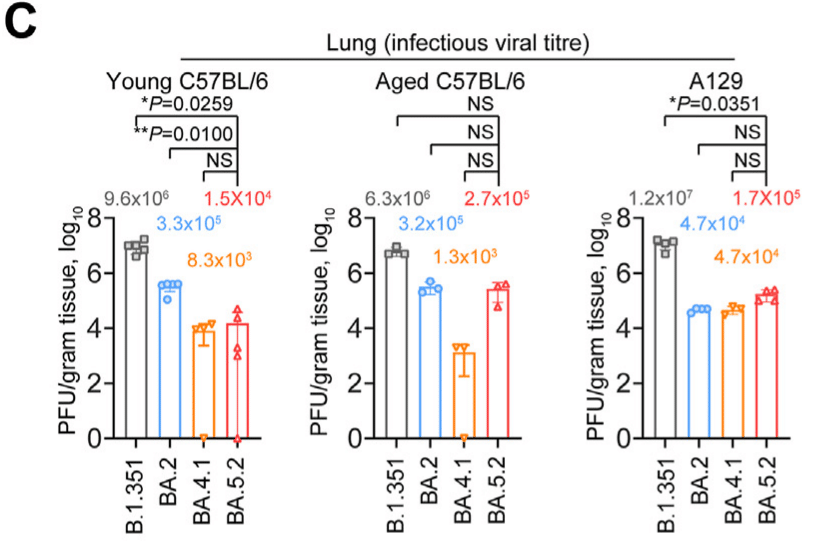
In lungs of aged C57BL/6 mice and A129 mice, the infectious viral titre of BA.5.2 was largely comparable with that of BA.2 (Fig. 6C).
老齢C57BL/6マウスおよびA129マウスの肺では、BA.5.2の感染性ウイルス力価はBA.2とほぼ同等であった(図6C)。
To evaluate the differences in viral antigen expression among the Omicron sublineages, we detected the abundance of viral N protein in lung tissues with immunohistochemistry staining.
オミクロン亜系統間のウイルス抗原発現の違いを評価するために、免疫組織化学染色により肺組織中のウイルスNタンパク質の豊富さを検出した。
In corroboration with the virological assessment results, viral antigen expression in lungs of BA.5.2-infected young C57BL/6 mice were 19.3- (P = 0.0163) and 11.3-folds (P = 0.0266) lower than those of B.1.351 and BA.2, respectively.
ウイルス学的評価結果の裏付けとして、BA.5.2 に感染した若い C57BL/6 マウスの肺におけるウイルス抗原発現は、それぞれ B.1.351 および BA.2 のそれより 19.3 倍 (P = 0.0163) および 11.3 倍 (P = 0.0266) 低かった。
In contrast, viral N protein was expressed at comparable levels between BA.5.2 and BA.2 in the lungs of aged C57BL/6 mice and A129 mice (Fig. 6D and E).
対照的に、ウイルス N タンパク質は、高齢の C57BL/6 マウスおよび A129 マウスの肺において、BA.5.2 と BA.2 の間で同等のレベルで発現した (図 6D および E)。
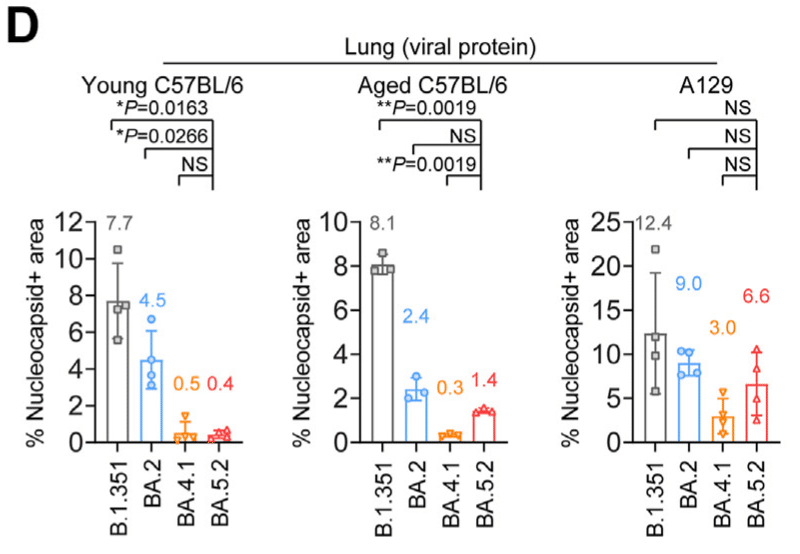
In parallel, we assessed histopathological changes and corresponding pathology scores with formalin-fixed right lungs collected from the three infection models.
並行して、3 つの感染モデルから採取したホルマリン固定右肺の組織病理学的変化と対応する病理スコアを評価しました。
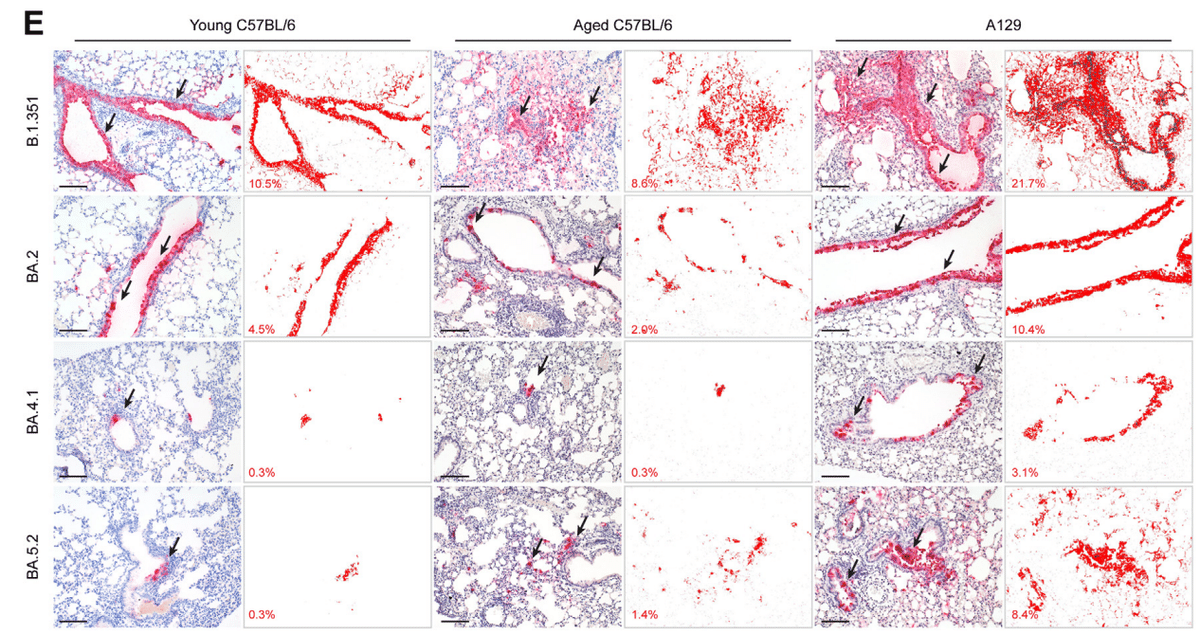
We found that compared to mock infection which contributes to no pathological changes in mouse lungs (Supplementary Fig. S1), B.1.351 infection uniformly led to obvious pathological damages in the lungs of young C57BL/6, aged C57BL/6, and A129 mice at 2 dpi, including moderate to severe bronchiolitis (Fig. 7A and B), multifocal alveolar haemorrhage (Fig. 7A and C), extensive alveolar destruction (Fig. 7A and C), and interstitial congestion with severe inflammatory infiltrations (Fig. 7A and D).
マウスの肺に病理学的変化をもたらさない模擬感染 (補足図 S1) と比較して、B.1.351 感染は、感染後 2 日目には、若い C57BL/6、高齢の C57BL/6、および A129 マウスの肺に一様に明らかな病理学的損傷を引き起こし、中等度から重度の細気管支炎 (図 7A および B)、多巣性肺胞出血 (図 7A および C)、広範囲の肺胞破壊 (図 7A および C)、および重度の炎症性浸潤を伴う間質うっ血 (図 7A および D) が見られました。
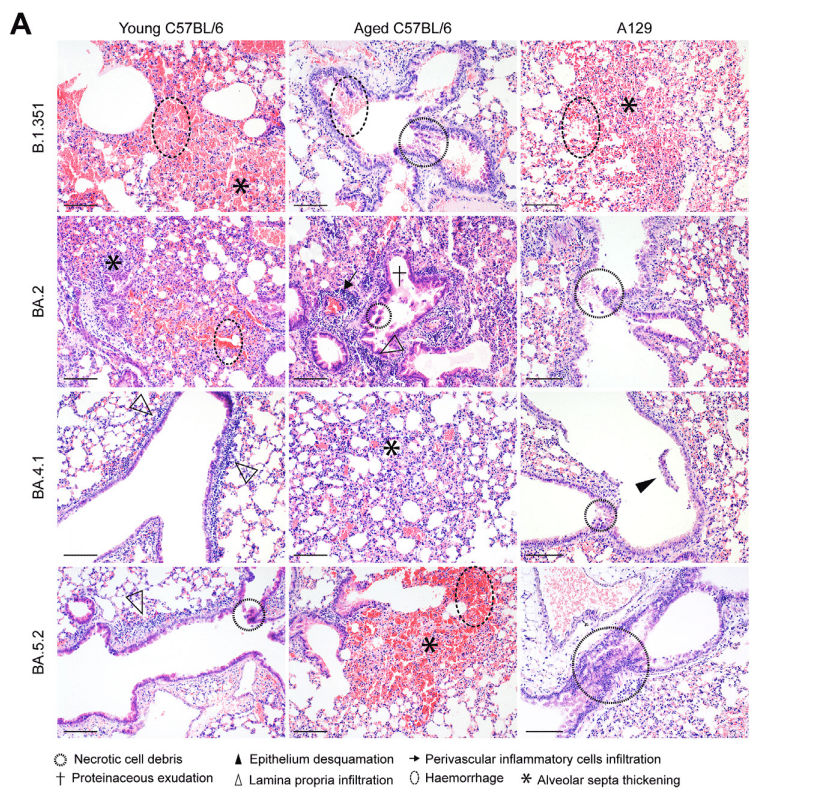
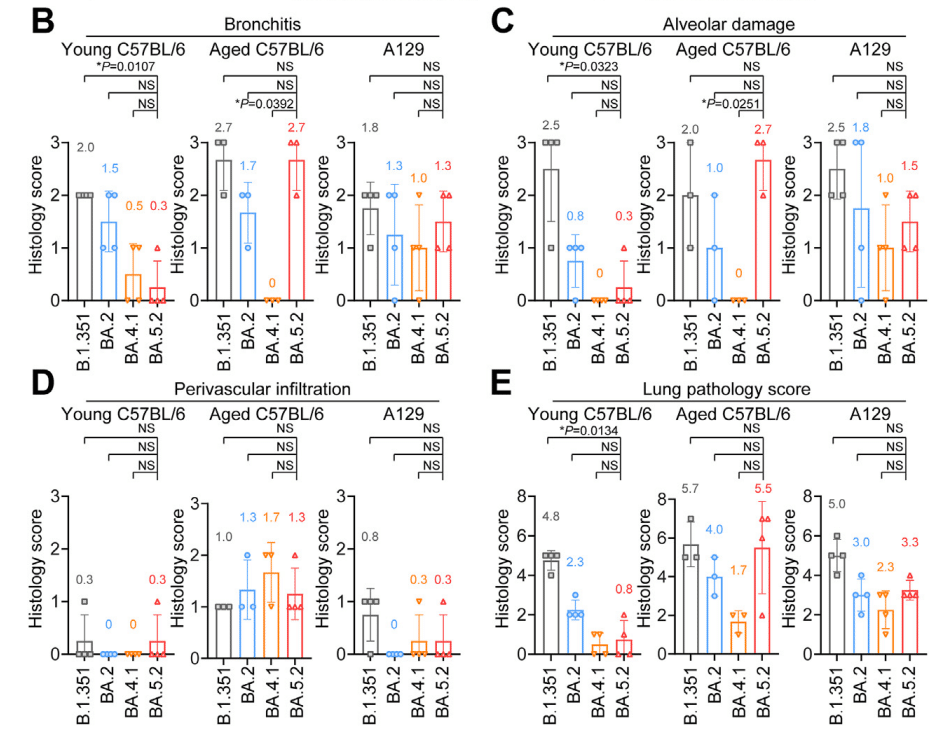
In keeping with the viral antigen expression results in lungs of young C57BL/6 mice, the mean total lung pathology score of BA.5.2 (0.8) was lower than those of B.1.351 (4.8) and BA.2 (2.3).
若い C57BL/6 マウスの肺におけるウイルス抗原発現結果と一致して、BA.5.2 の平均肺病理スコア (0.8) は、B.1.351 (4.8) および BA.2 (2.3) よりも低かった。
However, in aged C57BL/6 and A129 mice, the total lung pathology score of BA.5.2 was similar to those of B.1.351 and BA.2 (Fig. 7E).
ただし、高齢の C57BL/6 マウスおよび A129 マウスでは、BA.5.2 の肺病理スコアは B.1.351 および BA.2 と同程度であった (図 7E)。
Together, our results demonstrate that the dominant Omicron sublineages are gaining replication fitness in both primary human nasal epithelial cells and nasal epithelium of infected animals.
これらの結果を総合すると、優勢な Omicron サブ系統が、一次ヒト鼻上皮細胞および感染動物の鼻上皮の両方で複製適応度を獲得していることがわかる。
In contrast, the replication of all evaluated Omicron sublineages are attenuated in the lungs of K18-hACE2 and young C57BL/6 mice, leading to reduced lung pathology.
対照的に、評価されたすべての Omicron サブ系統の複製は、K18-hACE2 マウスおよび若い C57BL/6 マウスの肺で弱まり、肺病理が軽減されている。
Notably, the replication and pathogenicity of BA.5.2 remain comparable to BA.2 in the lungs of aged C57BL/6 and A129 mice, suggesting Omicron infection can still cause severe infection in the immunocompromised animals.
注目すべきことに、BA.5.2 の複製と病原性は、高齢の C57BL/6 マウスと A129 マウスの肺では BA.2 と同等のままであり、オミクロン感染は免疫不全動物に依然として重篤な感染を引き起こす可能性があることを示唆しています。
Discussion
Previous literature demonstrated that BA.1 had a replication advantage over Delta and could outcompete Delta in co-infection experiments in human nasal epithelial cells.
これまでの文献では、BA.1 は Delta よりも複製に優れており、ヒトの鼻上皮細胞での共感染実験で Delta に打ち勝つことができることが実証されています。
However, the intrinsic replication fitness of Omicron sublineages in the nasal epithelium has not been thoroughly investigated.
しかし、鼻上皮における Omicron サブ系統の固有の複製適応性は、十分に調査されていません。
Here, we revealed that while all Omicron sublineages replicated substantially more robustly than that of WT in human nasal epithelial cells of the upper respiratory tract, the dominant Omicron sublineages BA.1, BA.2 and BA.5 are gaining further replication fitness in human nasal epithelial cells in the order of BA.1, BA.2, BA.4.1, and BA.5.2.
ここでは、すべての Omicron サブ系統が上気道のヒト鼻上皮細胞で WT よりも大幅に堅牢に複製されている一方で、優勢な Omicron サブ系統 BA.1、BA.2、および BA.5 は、BA.1、BA.2、BA.4.1、および BA.5.2 の順にヒト鼻上皮細胞で複製適応性をさらに高めていることを明らかにしました。
In the nasal turbinates of K18-hACE2 mice, BA.5.2 replicated better than BA.2, which was in turn better than BA.1.
K18-hACE2 マウスの鼻甲介では、BA.5.2 は BA.2 よりもよく複製され、BA.2 は BA.1 よりもよく複製されました。
In keeping with these results, BA.5.2 outcompeted BA.2 and BA.4.1 in in-vivo competition assays in the nasal turbinates of K18-hACE2 mice.
これらの結果と一致して、BA.5.2 は、K18-hACE2 マウスの鼻甲介における in vivo 競合アッセイで BA.2 および BA.4.1 に勝ちました。
Similarly, BA.5.2 consistently replicated to higher levels than BA.2 in the nasal turbinates of young C57BL/6, aged C57BL/6, and A129 mice.
同様に、BA.5.2 は、若い C57BL/6、老齢の C57BL/6、および A129 マウスの鼻甲介で、一貫して BA.2 よりも高いレベルで複製されました。
Concordantly, a recent study also demonstrated an increased replication capacity of BA.5 in the upper airway in ferrets.
一致して、最近の研究では、フェレットの上気道における BA.5 の複製能力の増加も実証されました。
Collectively, these findings indicate that the dominant Omicron sublineages, including BA.1, BA.2, and BA.5, have become increasingly efficient in their replication at the nasal epithelium of the upper respiratory tract.
総合的に、これらの知見は、BA.1、BA.2、BA.5を含む主要なオミクロン亜系統が、上気道の鼻上皮での複製においてますます効率的になっていることを示しています。
Increased viral replication fitness in the upper respiratory tract was associated with enhanced virus transmissibility in vivo.
上気道でのウイルス複製適応度の向上は、in vivoでのウイルス伝染性の向上と関連していました。
Therefore, the intrinsically increased efficiency of Omicron replication in the nasal epithelium may contribute to their more efficient spread among the human populations observed clinically.
したがって、鼻上皮でのオミクロン複製の本質的に高い効率は、臨床的に観察されるヒト集団間でのより効率的な拡散に寄与している可能性があります。
In early 2022, we and others reported that BA.1 was attenuated in pathogenicity in vivo when compared to SARS-CoV-2 WT and previous VOCs.
2022年初頭、私たちと他の研究者は、SARS-CoV-2 WTおよび以前のVOCと比較して、BA.1は生体内で病原性が弱まっていると報告しました。
This observation was later confirmed by additional in vivo studies as well as recent clinical studies conducted in COVID-19 patients.
この観察結果は、その後、追加の生体内研究や、COVID-19患者を対象に実施された最近の臨床研究によって確認されました。
Subsequently, we reported that BA.2 replicated slightly less efficiently than BA.1 in the lungs of K18-hACE2 mice, resulting in slightly more attenuated lung pathology when compared to BA.1.
その後、BA.2はK18-hACE2マウスの肺でBA.1よりもわずかに効率よく複製されず、BA.1と比較して肺病変がわずかに軽減されたことを報告しました。
This finding was similarly confirmed in recent clinical studies.
この発見は、最近の臨床研究でも同様に確認されました。
In a large cohort study in England that included over one million patients, BA.2 cases had lower risks of death and hospital admission than BA.1 after adjustment for confounders.
100万人以上の患者を含むイギリスの大規模コホート研究では、交絡因子の調整後、BA.2症例はBA.1症例よりも死亡および入院のリスクが低かった。
In another study from South Africa, the risk of severe disease among hospitalised individuals upon BA.2 infection was lower than that of BA.1 cases (adjusted odds ratio = 0.78; P = 0.029).
南アフリカの別の研究では、BA.2 感染による入院患者の重症化リスクは BA.1 症例よりも低かった (調整オッズ比 = 0.78、P = 0.029)。
During the preparation of our manuscript, a study indicated BA.5 can infect lung cells more robustly than earlier Omicron subvariants both in vitro and in vivo.
本論文の準備中に、ある研究で、BA.5 は以前の Omicron 亜種よりも in vitro および in vivo の両方で肺細胞に強力に感染できることが示された。
Such discrepancies might be attributed to the amino acid substitutions in the viral genome as well as the differences in animal models used.
このような相違は、ウイルスゲノムのアミノ酸置換と、使用された動物モデルの違いに起因する可能性がある。
The pathogenicity of BA.5 has been evaluated by additional groups but with inconsistent conclusions.
BA.5 の病原性は他のグループによって評価されているが、結論は一貫していない。
In this study, we showed that the infectious titre and viral protein expression of BA.5.2 was 2.1- and 1.5-fold lower than that of BA.2 in the lungs of K18-hACE2 mice.
この研究では、BA.5.2 の感染力価とウイルスタンパク質発現が、K18-hACE2 マウスの肺で BA.2 の 2.1 倍と 1.5 倍低いことを示しました。
Meanwhile, the total lung pathology score of BA.5.2 was 2.5-folds lower than that of BA.2, suggesting that BA.5.2 was slightly further attenuated when compared to BA.2.
一方、BA.5.2 の肺病理スコア全体は BA.2 の 2.5 倍低く、BA.5.2 は BA.2 と比較してわずかに弱毒化されていることを示しています。
Since the receptor expression in the K18-hACE2 transgenic mice may not be optimally physiological, we additionally evaluated the replication and pathogenicity of BA.5.2 and BA.2 in wildtype C57BL/6 mice and A129 mice.
K18-hACE2 トランスジェニック マウスにおける受容体の発現は生理学的に最適ではない可能性があるため、野生型 C57BL/6 マウスと A129 マウスにおける BA.5.2 と BA.2 の複製と病原性をさらに評価しました。
Importantly, we found that the subgenomic E gene, infectious titre, and viral protein expression in lungs of BA.5.2-infected young C57BL/6 mice were 12.9- (P = 0.0096), 22- (P = 0.0100), and 11.3-folds (P = 0.0266) lower than that of BA.2-infected young C57BL/6 mice, respectively.
重要なことに、BA.5.2 に感染した若い C57BL/6 マウスの肺におけるサブゲノム E 遺伝子、感染力価、およびウイルスタンパク質発現は、BA.2 に感染した若い C57BL/6 マウスと比較して、それぞれ 12.9 倍 (P = 0.0096)、22 倍 (P = 0.0100)、および 11.3 倍 (P = 0.0266) 低いことがわかりました。
In keeping with the virus replication results, the total lung pathological score of BA.5.2-infected young C57BL/6 mice was 2.9-folds lower when compared to that of BA.2-infected young C57BL/6 mice.
ウイルス複製の結果と一致して、BA.5.2 に感染した若い C57BL/6 マウスの肺病理スコアは、BA.2 に感染した若い C57BL/6 マウスのスコアと比較して 2.9 倍低かった。
Together, our results from K18-hACE2 and wildtype C57BL/6 mice indicate that the dominant Omicron sublineages, including BA.1, BA.2, and BA.5, continue to attenuate in replicating in the lungs, leading to attenuated pathogenicity.
K18-hACE2 マウスと野生型 C57BL/6 マウスの結果を合わせると、BA.1、BA.2、BA.5 を含む優勢な Omicron 亜系統が肺での複製を弱め続け、病原性が弱まることが示唆される。
Nevertheless, in the lung tissue of immunodeficient aged C57BL/6 mice and the IFN alpha/beta receptor knockout A129 mice, BA.5.2 replicated to comparable level as BA.2 and could still result in severe lung diseases, suggesting that despite the attenuation of Omicron sublineages in the general populations with intact immunity, the ongoing transmission of Omicron sublineages remains a threat to the elderlies and immunocompromised patients.
それにもかかわらず、免疫不全の老齢C57BL/6マウスおよびIFNα/β受容体ノックアウトA129マウスの肺組織では、BA.5.2はBA.2と同程度に複製され、依然として重篤な肺疾患を引き起こす可能性があり、免疫が損なわれていない一般集団ではオミクロン亜系統が弱毒化しているにもかかわらず、オミクロン亜系統の継続的な伝播は高齢者および免疫不全患者にとって依然として脅威であることを示唆している。
Therefore, vaccination is an important countermeasure to reduce hospitalization and to prevent the spread of SARS-CoV-2 in the immunocompromised population.
したがって、ワクチン接種は入院を減らし、免疫不全集団におけるSARS-CoV-2の拡散を防ぐための重要な対策である。
However, since major differences regarding to virus tropism, transmissibility, clinical symptoms remain unneglectable between infected animal models and humans, continuous clinical surveillance to reflect the replication fitness and pathogenicity of Omicron sublineages in humans should be conducted in future studies.
しかし、感染した動物モデルとヒトの間では、ウイルスの向性、伝染性、臨床症状に関する大きな違いが依然として無視できないため、今後の研究では、ヒトにおけるオミクロン亜系統の複製適応度と病原性を反映する継続的な臨床監視を実施する必要があります。
We and others previously demonstrated that BA.1 and BA.2 spikes are characterized with reduced spike cleavage at the S1/S2 site, reduced efficiency in spike-mediated cell-cell fusion, reduced plaque formation, and decreased TMPRSS2 usage while increase in capacity in cell entry through the endocytic pathway.
私たちと他の研究者は以前、BA.1 および BA.2 スパイクは、S1/S2 部位でのスパイク切断の減少、スパイクを介した細胞間融合の効率の低下、プラーク形成の減少、および TMPRSS2 の使用量の減少を特徴とし、エンドサイトーシス経路を介した細胞侵入の容量が増加することを実証しました。
In this study, we demonstrated that BA.5.2 spike had increased spike cleavage at the S1/S2 site and gained efficiency in spike-mediated cell-cell fusion leading to larger plaque size when compared with BA.1 and BA.2 spikes.
この研究では、BA.5.2スパイクはS1/S2部位でのスパイク切断が増加し、スパイクを介した細胞間融合の効率が向上し、BA.1およびBA.2スパイクと比較してプラークサイズが大きくなることを実証しました。
Interestingly, the BA.4/5 spike pseudovirus was enhanced in utilizing the endosomal entry pathway when compared to the BA.2 spike pseudovirus, which cannot be explained by its increased spike cleavage and gained efficiency in spike-mediated cell-cell fusion.
興味深いことに、BA.4/5スパイク擬似ウイルスは、BA.2スパイク擬似ウイルスと比較してエンドソーム侵入経路の利用が強化されていましたが、これはスパイク切断の増加とスパイクを介した細胞間融合の効率の向上だけでは説明できません。
We further verified this observation by infection with authentic BA.1, BA.2 and BA.5 viruses and the data suggested BA.5 consistently showed reduced TMPRSS2-mediated virus entry while increased in dependency on the endocytic entry pathway when compared with wildtype SARS-CoV-2 and BA.1.
さらに、本物の BA.1、BA.2、および BA.5 ウイルスに感染させてこの観察結果を検証したところ、野生型 SARS-CoV-2 および BA.1 と比較した場合、BA.5 では一貫して TMPRSS2 を介したウイルス侵入が減少し、エンドサイトーシス侵入経路への依存度が高まっていることがデータから示唆されました。
Although discrepancies remained between our current study and findings from a recent study which suggested an enhanced TMPRSS2 usage for BA.5, it should be aware that the predominant cell entry pathway utilized by SARS-CoV-2 is cell type-dependent.
BA.5 で TMPRSS2 の使用が強化されていることを示唆した最近の研究結果と現在の研究結果の間には依然として食い違いがありますが、SARS-CoV-2 が利用する主な細胞侵入経路は細胞の種類に依存していることを認識する必要があります。
To this end, physiological-relevant cells or cell lines such as primary human airway cells should be used when investigating virus entry pathways.
このため、ウイルス侵入経路を調査する際には、初代ヒト気道細胞などの生理学的に関連する細胞または細胞株を使用する必要があります。
While previous studies provided compelling evidence that suggested the spike gene is likely a major determinant of the observed virological and pathogenicity phenotypes of Omicron, additional reports have also suggested major determinants of Omicron pathogenicity to reside outside of spike.
これまでの研究では、スパイク遺伝子がオミクロンの観察されたウイルス学的および病原性の表現型の主要な決定因子である可能性が高いことを示唆する説得力のある証拠が示されていましたが、追加の報告では、オミクロンの病原性の主要な決定因子はスパイク外に存在することも示唆されています。
For example, nsp6 was shown to contribute to the attenuated pathogenicity of the Omicron BA.1, although detailed mechanism remained to be elucidated.
例えば、nsp6 は Omicron BA.1 の病原性の弱毒化に寄与することが示されていますが、詳細なメカニズムはまだ解明されていません。
The clinical isolate BA.5.2 that we used in the current study (GISAID: EPI_ISL_13777658) differs from BA.2 (GISAID: EPI_ISL_9845731) at multiple non-spike regions, including NSP3 (G489S), NSP4 (F438L), M (D3N) and ORF3 (F140L).
現在の研究で使用した臨床分離株 BA.5.2 (GISAID: EPI_ISL_13777658) は、NSP3 (G489S)、NSP4 (F438L)、M (D3N)、ORF3 (F140L) など、複数の非スパイク領域で BA.2 (GISAID: EPI_ISL_9845731) と異なります。
Substitutions at these sites might also contribute to the discrepant pathogenicity of BA.5.2 in immunocompetent mice (K18-hACE2 and young C57BL/6) and immunodeficient mice (A129 and aged C57BL/6), as well as the differences between spike-only recombinant Omicron and authentic Omicron strains reported in previous studies.
これらの部位での置換は、免疫能マウス (K18-hACE2 および若い C57BL/6) と免疫不全マウス (A129 および高齢の C57BL/6) における BA.5.2 の矛盾した病原性、および以前の研究で報告されたスパイクのみの組み換え Omicron 株と本物の Omicron 株間の違いにも寄与している可能性があります。
Overall, our study investigated the virological features, replication, and pathogenicity of the dominant Omicron sublineages for the past year (BA.1, BA.2, and BA.5).
全体として、私たちの研究では、過去 1 年間の主要なオミクロン亜系統 (BA.1、BA.2、および BA.5) のウイルス学的特徴、複製、および病原性を調査しました。
Our data suggested that the Omicron sublineages might be gaining replication fitness in the upper respiratory tract while that in the lung are continuously attenuated.
私たちのデータは、オミクロン亜系統が上気道で複製適応度を高めている一方で、肺での複製適応度は継続的に弱まっている可能性があることを示唆しています。
These findings provide scientific foundations to the public health authorities for setting up control and management measures against the emerging Omicron sublineages.
これらの調査結果は、公衆衛生当局が出現しつつあるオミクロン亜系統に対する制御および管理対策を講じるための科学的根拠を提供します。
以下省略。
この記事が気に入ったらサポートをしてみませんか?
