ACE2受容体に結合したスパイク受容体結合ドメインの構造
Structure of the SARS-CoV-2 spike receptor-binding domain bound to the ACE2 receptor
ACE2受容体に結合したSARS-CoV-2スパイク受容体結合ドメインの構造
元→Structure of the SARS-CoV-2 spike receptor-binding domain bound to the ACE2 receptor | Nature
Abstract
A new and highly pathogenic coronavirus (severe acute respiratory syndrome coronavirus-2, SARS-CoV-2) caused an outbreak in Wuhan city, Hubei province, China, starting from December 2019 that quickly spread nationwide and to other countries around the world.
2019年12月から中国湖北省武漢市で、病原性の高い新型コロナウイルス(重症急性呼吸器症候群コロナウイルス2、SARS-CoV-2)が流行し、中国全土および世界各国に急速に広がりました。
Here, to better understand the initial step of infection at an atomic level, we determined the crystal structure of the receptor-binding domain (RBD) of the spike protein of SARS-CoV-2 bound to the cell receptor ACE2.
ここでは、感染の初期段階を原子レベルでよりよく理解するために、細胞受容体 ACE2 に結合した SARS-CoV-2 のスパイクタンパク質の受容体結合ドメイン (RBD) の結晶構造を決定しました。
The overall ACE2-binding mode of the SARS-CoV-2 RBD is nearly identical to that of the SARS-CoV RBD, which also uses ACE2 as the cell receptor.
SARS-CoV-2 RBD の全体的な ACE2 結合モードは、ACE2 を細胞受容体として使用する SARS-CoV RBD のモードとほぼ同じです。
Structural analysis identified residues in the SARS-CoV-2 RBD that are essential for ACE2 binding, the majority of which either are highly conserved or share similar side chain properties with those in the SARS-CoV RBD.
構造解析により、SARS-CoV-2 RBD 内の ACE2 結合に必須の残基が特定されました。その大部分は高度に保存されているか、SARS-CoV RBD 内のものと類似した側鎖特性を共有しています。
Such similarity in structure and sequence strongly indicate convergent evolution between the SARS-CoV-2 and SARS-CoV RBDs for improved binding to ACE2, although SARS-CoV-2 does not cluster within SARS and SARS-related coronaviruses.
構造と配列のこのような類似性は、SARS-CoV-2 と SARS-CoV RBD 間の収束進化によって ACE2 への結合が改善されたことを強く示唆していますが、SARS-CoV-2 は SARS および SARS 関連コロナウイルス内にはクラスター化していません。
The epitopes of two SARS-CoV antibodies that target the RBD are also analysed for binding to the SARS-CoV-2 RBD, providing insights into the future identification of cross-reactive antibodies.
RBD を標的とする 2 つの SARS-CoV 抗体のエピトープも SARS-CoV-2 RBD への結合について分析され、交差反応性抗体の将来の特定に関する知見が提供されます。
Main
The emergence of the highly pathogenic coronavirus SARS-CoV-2 in Wuhan and its rapid international spread has posed a serious global public-health emergency.
武漢で高病原性のコロナウイルス SARS-CoV-2 が出現し、急速に国際的に広がったことで、深刻な世界的な公衆衛生上の緊急事態が発生しました。
Similar to individuals who were infected by pathogenic SARS-CoV in 2003 and Middle East respiratory syndrome coronavirus (MERS-CoV) in 2012, patients infected by SARS-CoV-2 showed a range of symptoms including dry cough, fever, headache, dyspnoea and pneumonia with an estimated mortality rate ranging from 3 to 5%.
2003年に病原性SARS-CoVに感染した人や2012年に中東呼吸器症候群コロナウイルス(MERS-CoV)に感染した人と同様に、SARS-CoV-2に感染した患者は、乾いた咳、発熱、頭痛、呼吸困難、肺炎などのさまざまな症状を示し、推定死亡率は3~5%です。
Since the initial outbreak in December of 2019, SARS-CoV-2 has spread throughout China and to more than 80 other countries and areas worldwide.
2019年12月の最初の流行以来、SARS-CoV-2は中国全土および世界80カ国以上の国と地域に広がっています。
As of 5 March 2020, 80,565 cases in China have been confirmed with the infection and 3,015 infected patients have died (https://www.who.int/emergencies/diseases/novel-coronavirus-2019/situation-reports/).
2020年3月5日現在、中国では80,565人の感染が確認され、感染者3,015人が死亡しています。
As a result, the epicentre Wuhan and the neighbouring cities have been under lockdown to minimize the continued spread and the WHO (World Health Organization) has announced a Public Health Emergency of International Concern owing to the rapid and global dissemination of SARS-CoV-2.
その結果、震源地である武漢と近隣の都市は、感染拡大の継続を最小限に抑えるために封鎖され、WHO(世界保健機関)はSARS-CoV-2の急速かつ世界的な拡散により国際的に懸念される公衆衛生上の緊急事態を宣言しました。
Phylogenetic analyses of the coronavirus genomes have revealed that SARS-CoV-2 is a member of the Betacoronavirus genus, which includes SARS-CoV, MERS-CoV, bat SARS-related coronaviruses (SARSr-CoV), as well as others identified in humans and diverse animal species.
コロナウイルスゲノムの系統発生解析により、SARS-CoV-2はベータコロナウイルス属のメンバーであることが明らかになりました。ベータコロナウイルス属には、SARS-CoV、MERS-CoV、コウモリSARS関連コロナウイルス(SARSr-CoV)、およびヒトとさまざまな動物種で特定されているその他のコロナウイルスが含まれます。
Bat coronavirus RaTG13 appears to be the closest relative of the SARS-CoV-2, sharing more than 93.1% sequence identity in the spike (S) gene. SARS-CoV and other SARSr-CoVs, however, are distinct from SARS-CoV-2 and share less than 80% sequence identity.
コウモリコロナウイルス RaTG13 は、SARS-CoV-2 に最も近い親戚のようで、スパイク (S) 遺伝子の配列同一性が 93.1% 以上あります。ただし、SARS-CoV およびその他の SARSr-CoV は SARS-CoV-2 とは異なり、配列同一性は 80% 未満です。
Coronaviruses use the homotrimeric spike glycoprotein (comprising a S1 subunit and S2 subunit in each spike monomer) on the envelope to bind to their cellular receptors.
コロナウイルスは、エンベロープ上のホモ三量体スパイク糖タンパク質(各スパイクモノマーに S1 サブユニットと S2 サブユニットを含む)を使用して細胞受容体に結合します。
Such binding triggers a cascade of events that leads to the fusion between cell and viral membranes for cell entry.
このような結合により、細胞とウイルスの膜が融合して細胞に侵入する一連のイベントが引き起こされます。
Previous cryo-electron microscopy studies of the SARS-CoV spike protein and its interaction with the cell receptor ACE2 have shown that receptor binding induces the dissociation of the S1 with ACE2, prompting the S2 to transit from a metastable pre-fusion to a more-stable post-fusion state that is essential for membrane fusion.
SARS-CoV スパイクタンパク質と細胞受容体 ACE2 との相互作用に関するこれまでのクライオ電子顕微鏡研究では、受容体結合によって S1 と ACE2 の解離が誘導され、S2 が融合前の準安定状態から膜融合に不可欠なより安定した融合後状態に移行することが示されています。
Therefore, binding to the ACE2 receptor is a critical initial step for SARS-CoV to enter into target cells.
したがって、ACE2 受容体への結合は、SARS-CoV が標的細胞に侵入するための重要な初期ステップです。
Recent studies also highlighted the important role of ACE2 in mediating entry of SARS-CoV-2.
最近の研究では、SARS-CoV-2 の侵入を媒介する ACE2 の重要な役割も強調されています。
HeLa cells expressing ACE2 are susceptible to SARS-CoV-2 infection whereas those without ACE2 are not.
ACE2 を発現する HeLa 細胞は SARS-CoV-2 感染に感受性があるが、ACE2 を発現しない HeLa 細胞は感受性がない。
In vitro binding measurements also showed that the SARS-CoV-2 RBD binds to ACE2 with an affinity in the low nanomolar range, indicating that the RBD is a key functional component within the S1 subunit that is responsible for binding of SARS-CoV-2 by ACE2.
また、in vitro 結合測定では、SARS-CoV-2 RBD が低ナノモル範囲の親和性で ACE2 に結合することが示され、RBD が ACE2 による SARS-CoV-2 の結合を担う S1 サブユニット内の重要な機能成分であることが示唆されました。
The cryo-electron microscopy structure of the SARS-CoV-2 spike trimer has recently been reported in two independent studies.
SARS-CoV-2 スパイク三量体のクライオ電子顕微鏡構造が、最近 2 つの独立した研究で報告されました。
However, inspection of one available spike structure revealed the incomplete modelling of the RBD, particularly for the receptor-binding motif (RBM) that interacts directly with ACE2.
しかし、入手可能なスパイク構造の 1 つを調べたところ、特に ACE2 と直接相互作用する受容体結合モチーフ (RBM) について、RBD のモデリングが不完全であることが明らかになりました。
Computer modelling of the interaction between the SARS-CoV-2 RBD and ACE2 has identified some residues that are potentially involved in the interaction; however, the actual residues that mediate the interaction remained unclear.
SARS-CoV-2 RBDとACE2の相互作用のコンピューターモデリングにより、相互作用に関与している可能性のある残基がいくつか特定されました;が、相互作用を媒介する実際の残基は不明のままでした。
Furthermore, despite detectable cross-reactive SARS-CoV-2-neutralizing activity of serum or plasma from patients who recovered from SARS-CoV infections, no isolated SARS-CoV monoclonal antibodies are able to neutralize SARS-CoV-2.
さらに、SARS-CoV感染から回復した患者の血清または血漿にはSARS-CoV-2に対する交差反応性中和活性が検出されているにもかかわらず、単離されたSARS-CoVモノクローナル抗体はSARS-CoV-2を中和することができない。
These findings highlight some of the intrinsic sequence and structure differences between the SARS-CoV and SARS-CoV-2 RBDs.
これらの発見は、SARS-CoV と SARS-CoV-2 RBD の本質的な配列と構造の違いを浮き彫りにしています。
To elucidate the interaction between the SARS-CoV-2 RBD and ACE2 at a higher resolution, we determined the structure of the SARS-CoV-2 RBD–ACE2 complex using X-ray crystallography.
SARS-CoV-2 RBD と ACE2 の相互作用をより高い解像度で解明するために、X 線結晶構造解析を使用して SARS-CoV-2 RBD-ACE2 複合体の構造を決定しました。
This atomic-level structural information greatly improves our understanding of the interaction between SARS-CoV-2 and susceptible cells, provides a precise target for neutralizing antibodies, and assists the structure-based vaccine design that is urgently needed in the ongoing fight against SARS-CoV-2.
この原子レベルの構造情報は、SARS-CoV-2 と感受性細胞との相互作用に関する理解を大幅に向上させ、中和抗体の正確なターゲットを提供し、進行中の SARS-CoV-2 との戦いで緊急に必要とされている構造ベースのワクチン設計に役立ちます。
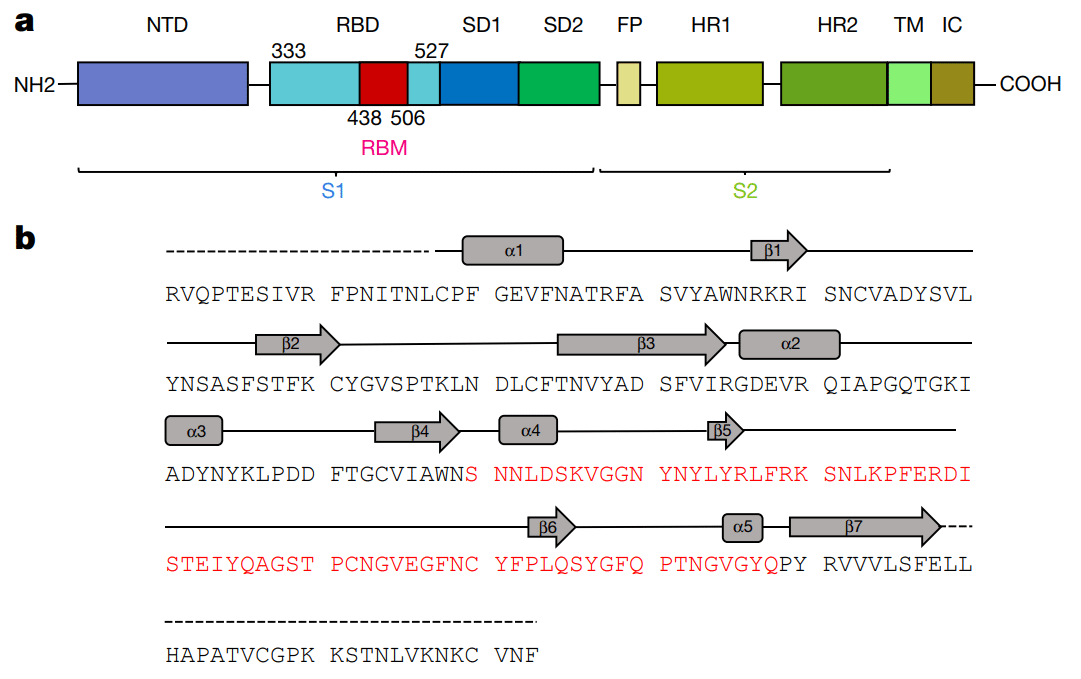
Specifically, we expressed the SARS-CoV-2 RBD (residues Arg319–Phe541) (Fig. 1a, b) and the N-terminal peptidase domain of ACE2 (residues Ser19–Asp615) in Hi5 insect cells and purified them by Ni-NTA affinity purification and gel filtration (Extended Data Fig. 1).
具体的には、Hi5昆虫細胞でSARS-CoV-2 RBD(残基Arg319~Phe541)(図1a、b)とACE2のN末端ペプチダーゼドメイン(残基Ser19~Asp615)を発現させ、Ni-NTAアフィニティー精製とゲル濾過によって精製しました(拡張データ図1)。
The structure of the complex was determined by molecular replacement using the SARS-CoV RBD and ACE2 structures as search models, and refined to a resolution of 2.45 Å with final Rwork and Rfree factors of 19.6% and 23.7%, respectively (Extended Data Fig. 2 and Extended Data Table 1).
複合体の構造は、SARS-CoV RBDとACE2構造を検索モデルとして使用して分子置換によって決定され、2.45Åの解像度に精製され、最終的なRworkとRfree係数はそれぞれ19.6%と23.7%でした(拡張データ図2と拡張データ表1)。
The final model contains residues Thr333–Gly526 of the SARS-CoV-2 RBD, residues Ser19–Asp615 of the ACE2 N-terminal peptidase domain, one zinc ion, four N-acetyl-β-glucosaminide (NAG) glycans linked to ACE2 Asn90, Asn322 and Asn546 and to RBD Asn343, as well as 80 water molecules.
最終モデルには、SARS-CoV-2 RBD の残基 Thr333~Gly526、ACE2 N 末端ペプチダーゼドメインの残基 Ser19~Asp615、亜鉛イオン 1 個、ACE2 Asn90、Asn322、Asn546 および RBD Asn343 に結合した 4 つの N-アセチル-β-グルコサミニド (NAG) グリカン、および 80 個の水分子が含まれています。
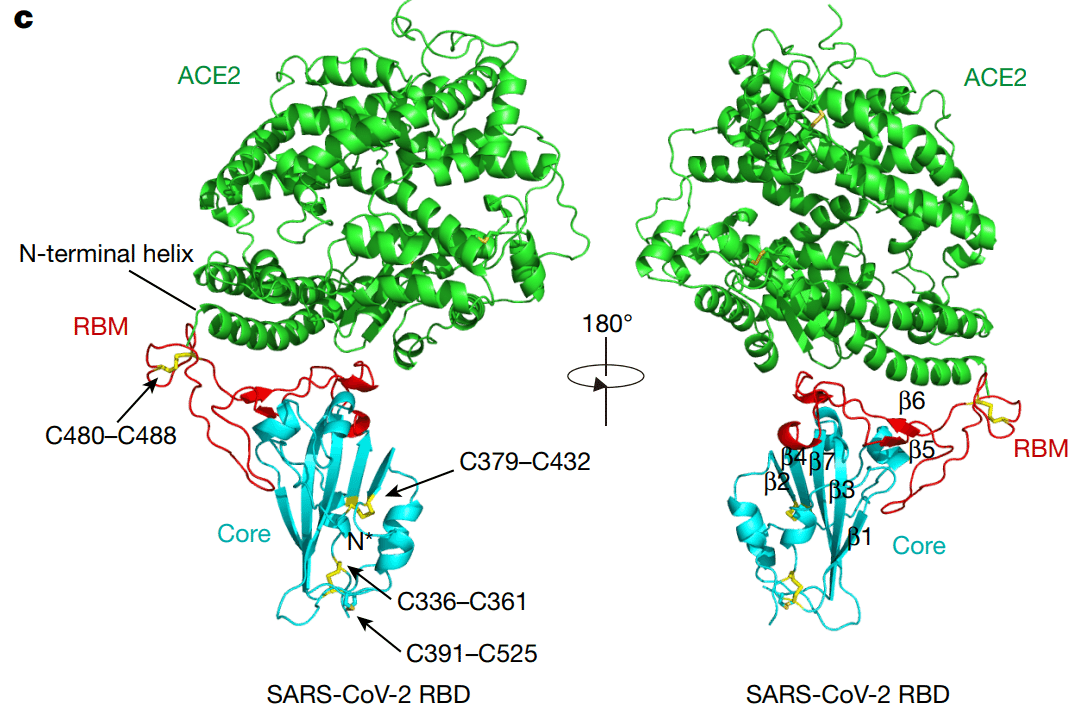
The SARS-CoV-2 RBD has a twisted five-stranded antiparallel β sheet (β1, β2, β3, β4 and β7) with short connecting helices and loops that form the core (Fig. 1b, c).
SARS-CoV-2 RBD には、短い接続ヘリックスとループがコアを形成する、ねじれた 5 本鎖の逆平行 β シート (β1、β2、β3、β4、β7) があります (図 1b、c)。
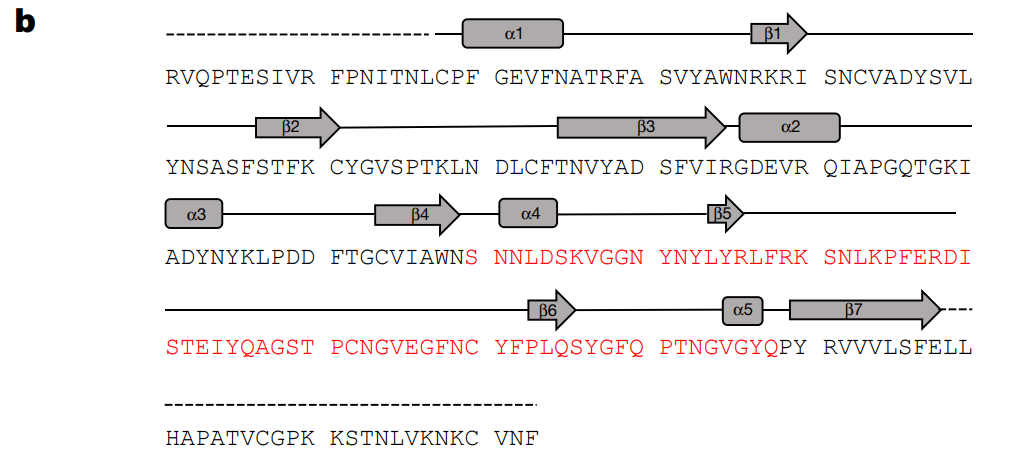
Between the β4 and β7 strands in the core, there is an extended insertion containing the short β5 and β6 strands, α4 and α5 helices and loops (Fig. 1b, c).
コアのβ4鎖とβ7鎖の間には、短いβ5鎖とβ6鎖、α4ヘリックスとα5ヘリックス、ループを含む延長挿入部があります(図1b、c)。
This extended insertion is the RBM, which contains most of the contacting residues of SARS-CoV-2 that bind to ACE2.
この延長挿入部はRBMで、ACE2に結合するSARS-CoV-2の接触残基のほとんどが含まれています。
A total of nine cysteine residues are found in the RBD, eight of which form four pairs of disulfide bonds that are resolved in the final model.
RBD には合計 9 つのシステイン残基があり、そのうち 8 つは最終モデルで解決される 4 対のジスルフィド結合を形成します。
Among these four pairs, three are in the core (Cys336–Cys361, Cys379–Cys432 and Cys391–Cys525), which help to stabilize the β sheet structure (Fig. 1c); the remaining pair (Cys480–Cys488) connects the loops in the distal end of the RBM (Fig. 1c).
これらの 4 対のうち 3 つはコアにあり (Cys336–Cys361、Cys379–Cys432、Cys391–Cys525)、β シート構造の安定化に役立ちます (図 1c)。残りの 1 対 (Cys480–Cys488) は RBM の遠位端のループを接続します (図 1c)。
The N-terminal peptidase domain of ACE2 has two lobes, forming the peptide substrate binding site between them.
ACE2 の N 末端ペプチダーゼドメインには 2 つのローブがあり、その間にペプチド基質結合部位を形成します。
The extended RBM in the SARS-CoV-2 RBD contacts the bottom side of the small lobe of ACE2, with a concave outer surface in the RBM that accommodates the N-terminal helix of the ACE2 (Fig. 1c).
SARS-CoV-2 RBD の拡張 RBM は ACE2 の小さなローブの底部に接し、RBM の凹状の外面には ACE2 の N 末端ヘリックスが収まります (図 1c)。
The overall structure of the SARS-CoV-2 RBD is similar to that of the SARS-CoV RBD (Extended Data Fig. 3a), with a root mean square deviation (r.m.s.d.) of 1.2 Å for 174 aligned Cα atoms.
SARS-CoV-2 RBD の全体構造は SARS-CoV RBD の構造と類似しており (拡張データ図 3a)、174 個の整列した Cα 原子の二乗平均平方根偏差 (r.m.s.d.) は 1.2 Å です。
Even in the RBM, which has more sequence variation, the overall structure is also highly similar (r.m.s.d. of 1.3 Å) to the SARS-CoV RBD, with only one obvious conformational change in the distal end (Extended Data Fig. 3a).
配列のバリエーションが多い RBM でも、全体構造は SARS-CoV RBD と非常に類似しており (r.m.s.d. は 1.3 Å)、遠位端に明らかな構造変化が 1 つだけ見られます (拡張データ図 3a)。
The overall binding mode of the SARS-CoV-2 RBD to ACE2 is also nearly identical to that observed in the previously determined structure of the SARS-CoV RBD–ACE2 complex (Extended Data Fig. 3b).
SARS-CoV-2 RBD の ACE2 への全体的な結合モードも、以前に決定された SARS-CoV RBD–ACE2 複合体の構造で観察されたものとほぼ同一です (拡張データ図 3b)。
The cradling of the N-terminal helix of ACE2 by the outer surface of the RBM results in a large buried surface of 1,687 Å2 (864 Å2 on the RBD and 823 Å2 on the ACE2) at the SARS-CoV-2 RBD–ACE2 interface.
ACE2 の N 末端ヘリックスが RBM の外表面で包まれることで、SARS-CoV-2 RBD-ACE2 界面に 1,687 Å2 (RBD で 864 Å2、ACE2 で 823 Å2) の大きな埋没表面が生じます。
A highly similar buried surface of 1,699 Å2 contributed by SARS-CoV RBD (869 Å2) and ACE2 (830 Å2) is also observed at the SARS-CoV RBD–ACE2 interface.
SARS-CoV RBD (869 Å2) と ACE2 (830 Å2) によってもたらされる 1,699 Å2 の非常に類似した埋没表面も、SARS-CoV RBD-ACE2 界面に観察されます。
With a distance cut-off of 4 Å, a total of 17 residues of the RBD are in contact with 20 residues of ACE2 (Fig. 2a and Extended Data Table 2).
距離カットオフが 4 Å の場合、RBD の合計 17 残基が ACE2 の 20 残基と接触しています (図 2a および拡張データ表 2)。
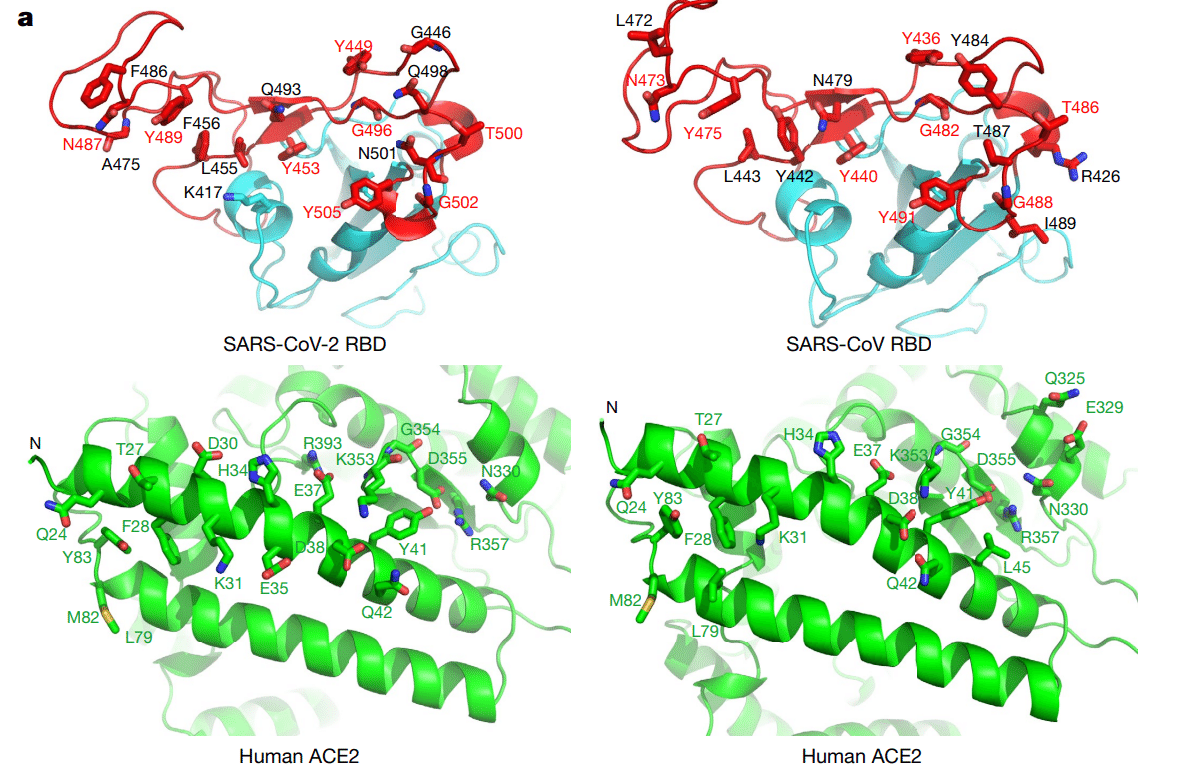
Analysis of the interface between the SARS-CoV RBD and ACE2 revealed a total of 16 residues of the SARS-CoV RBD in contact with 20 residues of ACE2 (Fig. 2a and Extended Data Table 2).
SARS-CoV RBD と ACE2 のインターフェイスを分析すると、SARS-CoV RBD の合計 16 残基が ACE2 の 20 残基と接触していることが明らかになりました (図 2a および拡張データ表 2)。
Among the 20 ACE2 residues that interact with the two different RBDs, 17 residues are shared between both interactions and most of the contacting residues are located at the N-terminal helix (Fig. 2a and Extended Data Table 2).
2 つの異なる RBD と相互作用する 20 個の ACE2 残基のうち、17 残基は両方の相互作用で共有されており、接触残基のほとんどは N 末端ヘリックスにあります (図 2a および拡張データ表 2)。
To compare the ACE2-interacting residues on the SARS-CoV-2 and SARS-CoV RBDs, we used structure-guided sequence alignment and mapped them to their respective sequences (Fig. 2b).
SARS-CoV-2 および SARS-CoV RBD 上の ACE2 相互作用残基を比較するために、構造ガイド配列アラインメントを使用し、それぞれの配列にマッピングしました (図 2b)。

Among 14 shared amino acid positions used by both RBMs for the interaction with ACE2, 8 have the identical residues between the two RBDs, including Tyr449/Tyr436, Tyr453/Tyr440, Asn487/Asn473, Tyr489/Tyr475, Gly496/Gly482, Thr500/Thr486, Gly502/Gly488 and Tyr505/Tyr491 of SARS-CoV-2/SARS-CoV, respectively (Fig. 2b).
ACE2 との相互作用のために両方の RBM で使用される 14 個の共有アミノ酸位置のうち、8 個は 2 つの RBD 間で同一の残基を持ち、SARS-CoV-2/SARS-CoV のそれぞれ Tyr449/Tyr436、Tyr453/Tyr440、Asn487/Asn473、Tyr489/Tyr475、Gly496/Gly482、Thr500/Thr486、Gly502/Gly488、および Tyr505/Tyr491 が含まれます (図 2b)。
Five positions have residues that have similar biochemical properties despite of having different side chains, including Leu455/Tyr442, Phe456/Leu443, Phe486/Leu472, Gln493/Asn479 and Asn501/Thr487 of SARS-CoV-2/SARS-CoV, respectively (Fig. 2b).
5 つの位置には、異なる側鎖を持つにもかかわらず、同様の生化学的特性を持つ残基があり、それぞれ SARS-CoV-2/SARS-CoV の Leu455/Tyr442、Phe456/Leu443、Phe486/Leu472、Gln493/Asn479、Asn501/Thr487 です (図 2b)。
The remaining position is at the Gln498/Tyr484 location (Fig. 2b), at which Gln498 of SARS-CoV-2 and Tyr484 of SARS-CoV both interact with Asp38, Tyr41, Gln42, Leu45 and Lys353 of ACE2.
残りの位置は Gln498/Tyr484 の位置 (図 2b) で、SARS-CoV-2 の Gln498 と SARS-CoV の Tyr484 はどちらも ACE2 の Asp38、Tyr41、Gln42、Leu45、Lys353 と相互作用します。
Among the six RBD positions with changed residues, SARS-CoV residues Tyr442, Leu472, Asn479 and Thr487 have previously been shown to be essential for binding ACE2.
残基が変化した6つのRBD位置のうち、SARS-CoV残基Tyr442、Leu472、Asn479、Thr487は、ACE2の結合に必須であることが以前に示されています。
At the Leu455/Tyr442 position, Leu455 of SARS-CoV-2 and Tyr442 of SARS-CoV have similar interactions with Asp30, Lys31 and His34 of ACE2 (Fig. 3a).
Leu455/Tyr442位置では、SARS-CoV-2のLeu455とSARS-CoVのTyr442は、ACE2のAsp30、Lys31、His34と同様の相互作用を示します(図3a)。
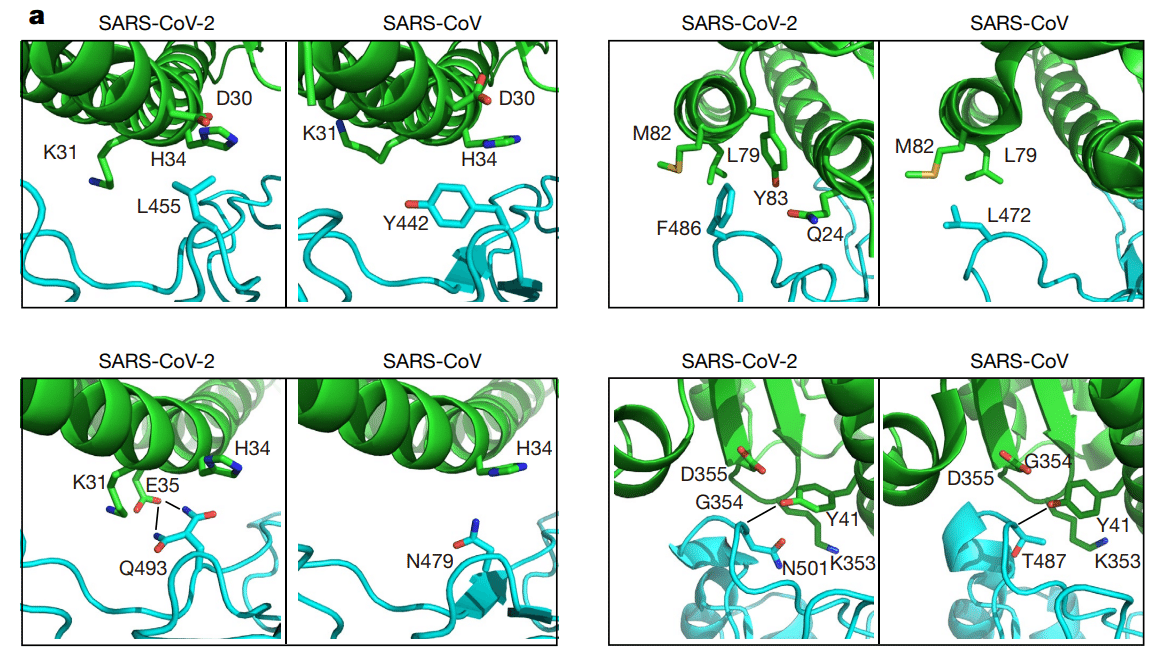
At the Phe486/Leu472 position, Phe486 of SARS-CoV-2 interacts with Gln24, Leu79, Met82 and Tyr83 of ACE2, whereas Leu472 of SARS-CoV has less interactions with Leu79 and Met82 of ACE2 (Fig. 3a).
Phe486/Leu472 の位置では、SARS-CoV-2 の Phe486 は ACE2 の Gln24、Leu79、Met82、Tyr83 と相互作用しますが、SARS-CoV の Leu472 は ACE2 の Leu79 および Met82 と相互作用する度合いは低くなります (図 3a)。
At the Gln493/Asn479 position, Gln493 of SARS-CoV-2 interacts with Lys31, His34 and Glu35 of ACE2 and forms a hydrogen bond with Glu35; Asn479 of SARS-CoV interacts with only His34 of ACE2 (Fig. 3a).
Gln493/Asn479 の位置では、SARS-CoV-2 の Gln493 は ACE2 の Lys31、His34、Glu35 と相互作用し、Glu35 と水素結合を形成します。SARS-CoV の Asn479 は ACE2 の His34 とのみ相互作用します (図 3a)。
Asn501 of SARS-CoV-2 and Thr487 of SARS-CoV both form a hydrogen bond with Tyr41 of ACE2 (Fig. 3a).
Asn501/Thr487 の位置では、両方の残基が ACE2 の Tyr41、Lys353、Gly354、Asp355 と類似の相互作用を示します (図 3a)。
Outside the RBM, there is a unique ACE2-interacting residue (Lys417) in SARS-CoV-2, which forms salt-bridge interactions with Asp30 of ACE2 (Fig. 3b).
RBM の外側には、SARS-CoV-2 に固有の ACE2 相互作用残基 (Lys417) があり、ACE2 の Asp30 と塩橋相互作用を形成します (図 3b)。
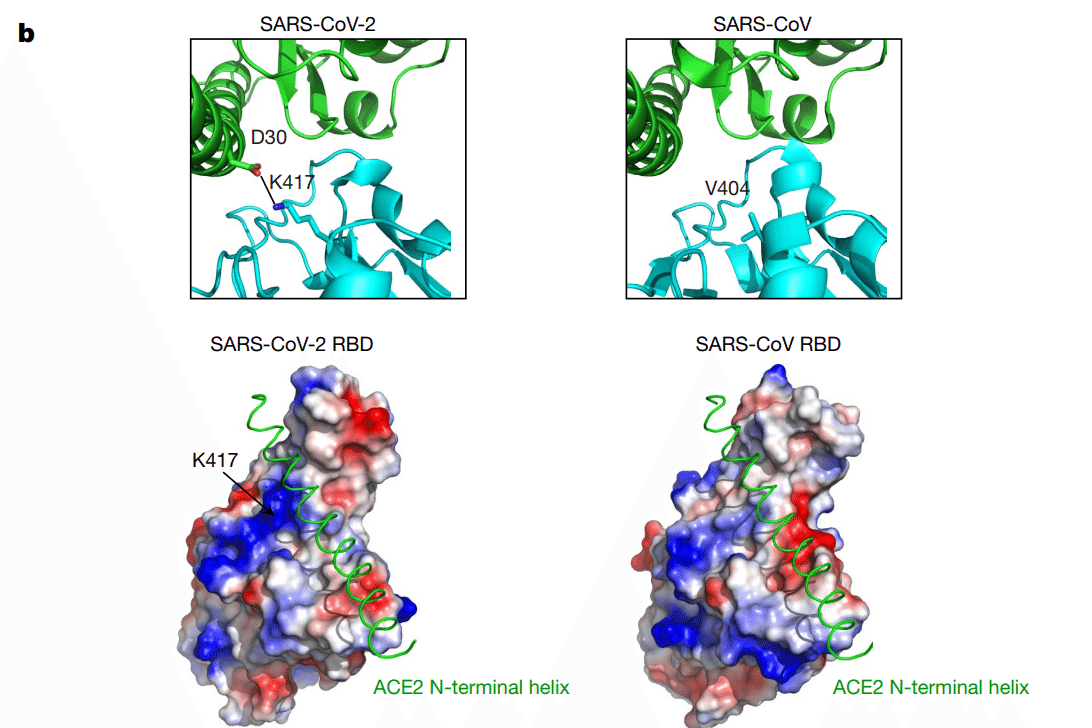
This position is replaced by a valine in the SARS-CoV RBD that fails to participate in ACE2 binding (Figs. 2b, 3b).
この位置は、SARS-CoV RBD では ACE2 結合に関与しないバリンに置き換えられています (図 2b、3b)。
Furthermore, a comparison of the surface electrostatic potential also identified a positive charged patch on the SARS-CoV-2 RBD contributed by Lys417 that is absent on the SARS-CoV RBD (Fig. 3b).
さらに、表面静電ポテンシャルの比較により、SARS-CoV-2 RBD には Lys417 によって生じた正電荷のパッチが特定されましたが、これは SARS-CoV RBD には存在しません (図 3b)。
These subtly different ACE2 interactions may contribute to the difference in binding affinity of the SARS-CoV-2 and SARS-CoV to the ACE2 receptor (4.7 nM compared with 31 nM, respectively) (Extended Data Fig. 4).
これらの微妙に異なる ACE2 相互作用は、SARS-CoV-2 と SARS-CoV の ACE2 受容体への結合親和性の違い (それぞれ 4.7 nM と 31 nM を比較) に寄与している可能性があります (拡張データ図 4)。
One notable and common feature that was found for both RBD–ACE2 interfaces is the networks of hydrophilic interactions.
両方の RBD–ACE2 インターフェースで発見された注目すべき共通点は、親水性相互作用のネットワークです。
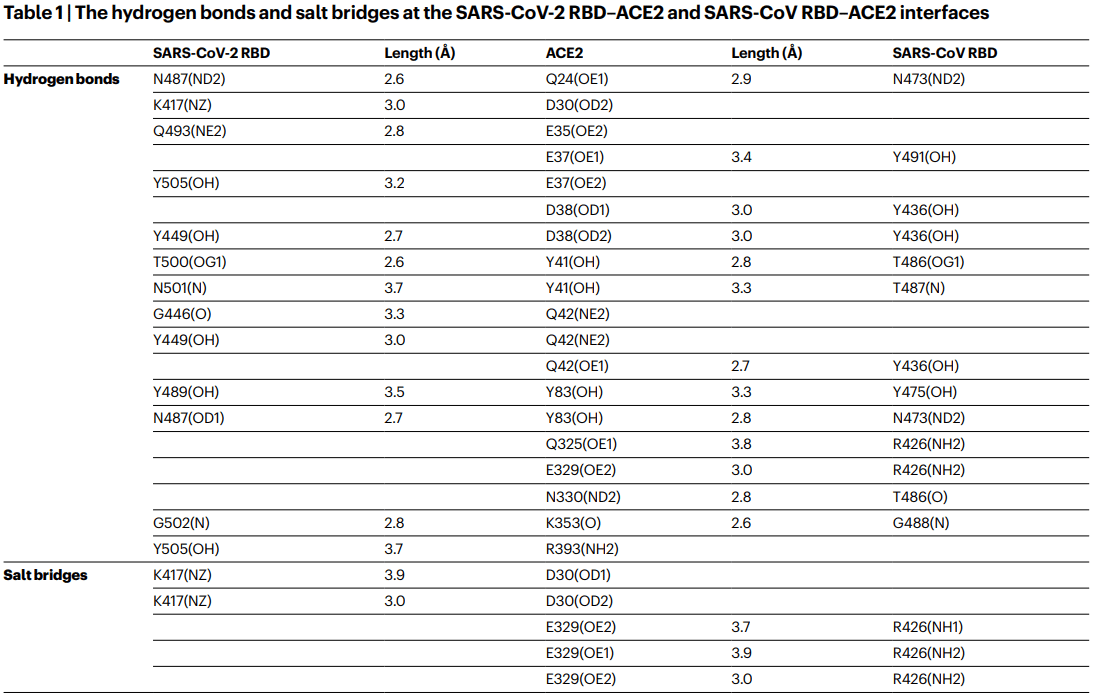
There are 13 hydrogen bonds and 2 salt bridges at the SARS-CoV-2 RBD–ACE2 interface, and 13 hydrogen bonds and 3 salt bridges at the SARS-CoV RBD–ACE2 interface (Table 1).
SARS-CoV-2 RBD-ACE2 インターフェースには 13 個の水素結合と 2 個の塩橋があり、SARS-CoV RBD-ACE2 インターフェースには 13 個の水素結合と 3 個の塩橋があります (表 1)。
The second shared feature is the involvement of multiple tyrosine residues that form hydrogen-bonding interactions with the polar hydroxyl group.
2 つ目の共通点は、極性ヒドロキシル基と水素結合相互作用を形成する複数のチロシン残基の関与です。
These include Tyr449, Tyr489 and Tyr505 from the SARS-CoV-2 RBD and Tyr436, Tyr475 and Tyr491 from the SARS-CoV RBD (Table 1).
これらには、SARS-CoV-2 RBD の Tyr449、Tyr489、Tyr505 と、SARS-CoV RBD の Tyr436、Tyr475、Tyr491 が含まれます (表 1)。
The third shared feature may reside in the Asn90-linked glycans of the ACE2 that bind to different RBDs.
3 つ目の共通点は、異なる RBD に結合する ACE2 の Asn90 結合グリカンにある可能性があります。
In the structure of the SARS-CoV RBD–ACE2 complex, a chain of Asn90-linked NAG–NAG–β-D-mannose is in contact with Thr402 of the SARS-CoV RBD (Extended Data Fig. 5a), and this glycan–RBD interaction has been proposed to have important roles in the binding of SARS-CoV RBD by ACE2.
SARS-CoV RBD-ACE2 複合体の構造では、Asn90 結合 NAG-NAG-β-D-マンノースの鎖が SARS-CoV RBD の Thr402 と接触しており (拡張データ図 5a)、このグリカン-RBD 相互作用は、ACE2 による SARS-CoV RBD の結合に重要な役割を果たすことが提案されています。
In the SARS-CoV-2 RBD–ACE2 structure, the density enabled only the modelling of the first NAG linked to ACE2 Asn90, and no interactions between this NAG and the SARS-CoV-2 RBD were observed (Extended Data Fig. 5b).
SARS-CoV-2 RBD-ACE2構造では、密度によってACE2 Asn90にリンクされた最初のNAGのみをモデル化でき、このNAGとSARS-CoV-2 RBD間の相互作用は観察されませんでした(拡張データ図5b)。
However, this does not exclude that glycans after the first NAG may interact with the SARS-CoV-2 RBD and may have important roles in the binding of SARS-CoV-2 RBD by ACE2.
ただし、これは、最初のNAGの後のグリカンがSARS-CoV-2 RBDと相互作用し、ACE2によるSARS-CoV-2 RBDの結合に重要な役割を果たす可能性を排除するものではありません。
Taken together, our results show that the SARS-CoV-2 RBD–ACE2 and SARS-CoV RBD–ACE2 interfaces share substantial similarity in the buried surface area, the number of interacting residues and hydrophilic interaction networks, although some of the ACE2 interactions observed both inside and outside the RBM were different (Fig. 3a, b).
まとめると、私たちの結果は、SARS-CoV-2 RBD-ACE2 と SARS-CoV RBD-ACE2 のインターフェースは、埋め込まれた表面積、相互作用する残基の数、親水性相互作用ネットワークにおいてかなりの類似性を共有していることを示していますが、RBM の内側と外側の両方で観察された ACE2 相互作用の一部は異なっていました (図 3a、b)。
Such similarities argue strongly for the convergent evolution of the SARS-CoV-2 and SARS-CoV RBD structures to improve binding affinity to the same ACE2 receptor, although SARS-CoV-2 does not cluster within SARS-CoV and SARSr-CoV in the Betacoronavirus genus.
このような類似性は、SARS-CoV-2 と SARS-CoV RBD 構造の収束進化によって同じ ACE2 受容体への結合親和性が向上したことを強く示唆しているが、SARS-CoV-2 はベータコロナウイルス属の SARS-CoV および SARSr-CoV 内にクラスター化していない。
Consistent with the high structural similarity, we found that the binding affinities between ACE2 and SARS-CoV-2 and SARS-CoV RBDs also fall into a similar range.
高い構造類似性と一致して、ACE2 と SARS-CoV-2 および SARS-CoV RBD 間の結合親和性も同様の範囲に収まることがわかりました。
Specifically, the equilibrium dissociation constant (KD) of ACE2 and SARS-CoV-2 RBD is 4.7 nM, and of ACE2 and SARS-CoV RBD is 31 nM (Extended Data Fig. 4).
具体的には、ACE2 と SARS-CoV-2 RBD の平衡解離定数 (KD) は 4.7 nM で、ACE2 と SARS-CoV RBD の平衡解離定数 (KD) は 31 nM です (拡張データ図 4)。
Similar results have also been reported by other groups.
同様の結果は他のグループからも報告されている。
However, this is slightly different from a recent report in which an approximately 20-fold increased binding between ACE2 and the SARS-CoV-2 spike trimer was found (KD of 14.7 nM) compared with that between ACE2 and SARS-CoV RBD–SD1 (KD of 325 nM).
ただし、これは、ACE2とSARS-CoV-2スパイクトリマー間の結合が、ACE2とSARS-CoV RBD-SD1間の結合(KD 325 nM)と比較して約20倍増加した(KD 14.7 nM)という最近の報告とは若干異なる。
This is perhaps due to the different proteins used in the assay or because of other unknown reasons.
これは、アッセイで使用されたタンパク質が異なるためか、その他の未知の理由によるものと思われる。
Nevertheless, the binding affinity alone is unlikely to explain the unusual transmissibility of SARS-CoV-2.
とはいえ、結合親和性だけでは、SARS-CoV-2の異常な伝染性を説明することはできないと思われる。
Other factors such as the unique ‘RRAR’ furin cleavage site at the S1–S2 boundary of the SARS-CoV-2 spike protein may have more-important roles in facilitating the rapid human-to-human transmission of SARS-CoV-2.
SARS-CoV-2スパイクタンパク質のS1-S2境界にある独自の「RRAR」フーリン切断部位などの他の要因は、SARS-CoV-2のヒトからヒトへの急速な伝染を促進する上でより重要な役割を果たしている可能性がある。
Neutralizing antibodies represent an important component of the immune system in the fight against viral infections.
中和抗体は、ウイルス感染と戦う免疫システムの重要な構成要素です。
It has been reported that SARS-CoV-2 could be cross-neutralized by horse anti-SARS-CoV serum and convalescent serum from a patient with a SARS-CoV infection, reinforcing the structural similarity between the RBDs of SARS-CoV-2 and SARS-CoV.
SARS-CoV-2 は、馬の抗 SARS-CoV 血清と SARS-CoV 感染患者の回復期血清によって交差中和される可能性があることが報告されており、SARS-CoV-2 と SARS-CoV の RBD の構造的類似性が強化されています。
Such similarity also increased the hope of the rapid application of previously characterized SARS-CoV monoclonal antibodies in the clinical setting.
このような類似性により、以前に特徴付けられたSARS-CoVモノクローナル抗体を臨床現場で迅速に適用できるという期待も高まりました。
However, no antibody that targeted SARS-CoV (m396, S230, 80R and CR3014) has so far demonstrated any notable cross-binding and neutralization activity against spike protein or RBD of SARS-CoV-2.
しかし、SARS-CoVを標的とした抗体(m396、S230、80R、CR3014)は、これまでのところ、SARS-CoV-2のスパイクタンパク質またはRBDに対する顕著な交差結合および中和活性を示していません。
One exception is SARS-CoV antibody CR3022 that binds to the SARS-CoV-2 RBD with a KD of 6.2 nM, although its neutralizing activity against SARS-CoV-2 has not yet been reported.
1つの例外は、SARS-CoV抗体CR3022で、これは6.2 nMのKDでSARS-CoV-2 RBDに結合しますが、SARS-CoV-2に対する中和活性はまだ報告されていません。
Currently, we are uncertain where exactly the epitope of CR3022 on the RBDs of SARS-CoV or SARS-CoV-2 is located.
現在、SARS-CoV または SARS-CoV-2 の RBD 上の CR3022 のエピトープが正確にどこに位置しているかは不明です。
Among the three antibodies that are incapable of binding to the SARS-CoV-2 RBD, two (m396 and 80R) have their epitopes resolved by the high-resolution crystal-structure determination of SARS-CoV RBD–Fab complexes.
SARS-CoV-2 RBD に結合できない 3 つの抗体のうち、2 つ (m396 と 80R) は、SARS-CoV RBD-Fab 複合体の高解像度結晶構造決定によってエピトープが解明されています。
By mapping these epitope residues onto the sequence of SARS-CoV RBD aligned with the sequence of SARS-CoV-2 RBD (Fig. 4), we found that antibody m396 has 7 residue changes in the SARS-CoV-2 RBD among 21 epitope positions (Fig. 4).
これらのエピトープ残基を、SARS-CoV-2 RBDの配列と整列させたSARS-CoV RBDの配列にマッピングすることで(図4)、抗体m396は、21のエピトープ位置のうちSARS-CoV-2 RBDに7つの残基変化があることを発見しました(図4)。

There are 16 residue changes in the SARS-CoV-2 RBD among 25 epitope positions of antibody 80R (Fig. 4).
抗体80Rの25のエピトープ位置のうちSARS-CoV-2 RBDに16の残基の変化があります(図4)。
This may provide a structural basis for the lack of cross-reactivity of m396 and 80R with SARS-CoV-2.
これは、m396と80RがSARS-CoV-2と交差反応性がない構造的根拠となる可能性があります。
The cross-neutralization of SARS-CoV-2 by horse anti-SARS-CoV serum and serum or plasma from patients recovered from SARS-CoV infections reveals a great potential in identifying antibodies with cross-reactivity between these two coronaviruses.
ウマ抗SARS-CoV血清とSARS-CoV感染から回復した患者の血清または血漿によるSARS-CoV-2の交差中和は、これら2つのコロナウイルス間で交差反応性を持つ抗体を特定する上で大きな可能性を示しています。
The conserved non-RBD regions in the spike protein, such as the S2 subunit, are the potential targets for cross-reactive antibodies.
S2サブユニットなどのスパイクタンパク質内の保存された非RBD領域は、交差反応性抗体の潜在的なターゲットです。
Although the RBD is less conserved, identical residues between SARS-CoV-2 and SARS-CoV RBD exist, even in the more variable RBM (Fig. 4).
RBDはあまり保存されていませんが、より可変性の高いRBMであっても、SARS-CoV-2とSARS-CoV RBDの間には同一の残基が存在します(図4)。
Considering that the RBD is the important region for receptor binding, antibodies that target the conserved epitopes in the RBD will also present a great potential for developing highly potent cross-reactive therapeutic agents against diverse coronavirus species, including SARS-CoV-2.
RBDは受容体結合に重要な領域であることを考慮すると、RBD内の保存されたエピトープを標的とする抗体は、SARS-CoV-2を含む多様なコロナウイルス種に対する非常に強力な交差反応性治療薬を開発する大きな可能性も提供します。
以下省略。
この記事が気に入ったらサポートをしてみませんか?
