ハムスター血清を使用した抗原地図作成により、ヒト XBB.1.5 ブースター血清に見られる JN.1 回避が特定
元→https://www.biorxiv.org/content/10.1101/2024.04.05.588359v1.full.pdf
Antigenic cartography using hamster sera identifies SARS-CoV-2 JN.1 evasion seen in human XBB.1.5 booster sera
ハムスター血清を使用した抗原地図作成により、ヒト XBB.1.5 ブースター血清に見られる SARS-CoV-2 JN.1 回避が特定される
Abstract
Antigenic assessments of SARS-CoV-2 variants inform decisions to update COVID-19 vaccines.
SARS-CoV-2 変異体の抗原評価は、COVID-19ワクチンの更新に関する決定に役立ちます。
Primary infection sera are often used for assessments, but such sera are rare due to population immunity from SARS-CoV-2 infections and COVID-19 vaccinations.
一次感染血清は評価によく使用されますが、SARS-CoV-2 感染や COVID-19ワクチン接種による集団免疫のため、そのような血清はまれです。
Here, we show that neutralization titers and breadth of matched human and hamster pre-Omicron variant primary infection sera correlate well and generate similar antigenic maps.
今回我々は、ヒトとハムスターのプレオミクロン株一次感染血清の中和力価と幅が一致し、よく相関し、同様の抗原マップを生成することを示します。
The hamster antigenic map shows modest antigenic drift among XBB sub-lineage variants, with JN.1 and BA.4/BA.5 variants within the XBB cluster, but with five to six-fold antigenic differences between these variants and XBB.1.5.
ハムスターの抗原マップは、XBB 亜系統株間の抗原のわずかな変動を示しています。 XBB クラスター内には JN.1 および BA.4/BA.5 変異体が存在しますが、これらの変異体と XBB.1.5 の間には 5 ~ 6 倍の抗原性の違いがあります。
Compared to sera following only ancestral or bivalent COVID-19 vaccinations, or with post-vaccination infections, XBB.1.5 booster sera had the broadest neutralization against XBB sub-lineage variants, although a five-fold titer difference was still observed between JN.1 and XBB.1.5 variants.
祖先型または二価COVID-19ワクチン接種のみ後の血清、またはワクチン接種後感染と比較して、 XBB.1.5 ブースター血清は XBB 亜系統変異体に対して最も広範な中和を示しましたが、JN.1 株と XBB.1.5 株の間では依然として 5 倍の力価差が観察されました。
These findings suggest that antibody coverage of antigenically divergent JN.1 could be improved with a matched vaccine antigen.
これらの発見は、抗原的に異なる JN.1 の抗体適用率が、適合するワクチン抗原によって改善される可能性があることを示唆しています。
Main text
Introduction
New SARS-CoV-2 variants continue to cause significant morbidity and mortality particularly among vulnerable groups despite increasing population immunity to SARS-CoV-2 from infections and vaccinations.
感染症やワクチン接種によってSARS-CoV-2に対する人口免疫が増加しているにもかかわらず、新たなSARS-CoV-2変異種は、特に脆弱な集団の間で重大な罹患率と死亡率を引き起こし続けています。
COVID-19 vaccines remain an important to for protecting against infection, severe illness, and Long COVID, but vaccine antigen updates may be needed for new variants.
COVID-19ワクチンは、感染症、重篤な病気、ロングCOVIDから身を守るために依然として重要ですが、新しい変異種についてはワクチン抗原の更新が必要になる可能性があります。
Neutralizing antibody titers help assess whether new variants evade vaccine-induced immunity and have been shown to correlate with protection against COVID-19.
中和抗体力価は、新しい変異体がワクチン誘発免疫を回避するかどうかを評価するのに役立ち、COVID-19に対する防御と相関することが示されています。
Antigenic cartography, initially introduced as a new analysis tool to aide interpretation of antigenic differences among influenza strains, uses antibody titers to visualize antigenic differences among strains.
抗原地図作成は、当初、インフルエンザ株間の抗原の違いの解釈を支援する新しい分析ツールとして導入されましたが、抗体力価を使用して株間の抗原の違いを視覚化します。
Convalescent sera from a first infection (primary infection sera) are often used to generate maps that discriminate viruses based on recognition by antibodies.
最初の感染からの回復期血清 (一次感染血清) は、抗体による認識に基づいてウイルスを識別するマップを作成するためによく使用されます。
However, human primary infection sera of new SARS-CoV-2 variants are now rare due to population immunity from prior SARS-CoV-2 infections, vaccinations, or both.
しかし、以前の SARS-CoV-2 感染、ワクチン接種、またはその両方による集団免疫のため、新しい SARS-CoV-2 変異体のヒト初感染血清は現在では稀です。
Syrian golden hamsters are among the preferred animal models for studying SARS-CoV-2 and have been used for generating primary infection sera for antigenic analyses.
シリアンゴールデンハムスターは、SARS-CoV-2を研究するための好ましい動物モデルの1つであり、抗原分析のための一次感染血清を生成するために使用されています。
Yet, data directly comparing the similarity between hamster and human antibody responses to the variants are limited, especially to the most recent Omicron variants.
しかし、変異株に対するハムスターとヒトの抗体反応の類似性を直接比較したデータは、特に最新の Omicron 亜種に対して、限られています。
Landscape analyses provide a method for visualizing how complex sera from multiple antigenic exposures from infections and vaccinations recognize different variants of a pathogen.
ランドスケープ分析は、感染症やワクチン接種による複数の抗原曝露による複雑な血清が病原体のさまざまな変異体をどのように認識するかを視覚化する方法を提供します。
Antibody titers against different viruses are modeled as a landscape over the primary sera antigenic map that defines the antigenic space.
さまざまなウイルスに対する抗体力価は、抗原空間を定義する一次血清抗原マップ上のランドスケープとしてモデル化されます。
Landscapes may allow interpolation of complex serum titers against variants in the map for which direct measurement have not been made.
ランドスケープにより、直接測定が行われていないマップ内の変異体に対する複雑な血清力価の内挿が可能になる場合があります。
Landscape analyses have previously been included in discussions regarding vaccine updates.
ランドスケープ分析はこれまで、ワクチンの更新に関する議論に含まれてきました。
However, prior antigenic exposures can shape subsequent responses to vaccines or infections, so complex and primary infection sera may recognize some variants differently.
ただし、以前の抗原曝露がワクチンや感染症に対するその後の反応を形作る可能性があるため、複雑な一次感染血清は一部の変異体を異なる方法で認識する可能性があります。
In this study, we generated a unique dataset of neutralization titers against a large panel of SARS-CoV-2 variants, including pre-Omicron and Omicron variants through JN.1, using hamster sera from primary, sequence-confirmed variant infections.
この研究では、配列が確認された一次感染由来のハムスター血清を使用して、プレオミクロンおよびJN.1によるオミクロン変異体を含むSARS-CoV-2変異体の大規模なパネルに対する中和力価の独自のデータセットを生成しました。
We applied antigenic cartography analyses to this serum set to characterize antigenic drift among recent SARS-CoV-2 variants.
私たちは、最近の SARS-CoV-2 変異体間の抗原変動を特徴付けるために、この血清セットに抗原地図作成分析を適用しました。
We further found good comparability between our antigenic map made with hamster sera and our prior antigenic map made with human sera, suggesting that hamster primary infection sera may be valuable for assessing antigenic differences among future variants.
さらに、ハムスター血清を使用して作成した抗原マップと、ヒト血清を使用して作成した以前の抗原マップとの間に良好な比較性があることも発見し、ハムスターの一次感染血清が将来の変異体間の抗原の違いを評価するのに貴重である可能性があることを示唆しました。
Our analyses of complex sera from persons with well-documented, COVID-19 vaccinations and infection exposures also suggests that antigen-matched vaccine boosters could improve protection against the most recent SARS-CoV-2 variants.
十分に文書化されたCOVID-19ワクチン接種と感染症への曝露を受けた人から採取した複雑な血清の我々の分析でも、抗原が一致したワクチンブースターが最新のSARS-CoV-2変異種に対する防御を向上させる可能性があることが示唆されている。
Results
Hamster primary infection sera exhibit poor cross-neutralization between pre-Omicron and Omicron variants.
ハムスターの一次感染血清は、プレオミクロンとオミクロン株の間で不十分な交差中和を示します。
To assess the suitability of hamster primary infection sera for antigenic analyses to aid selection of antigens for COVID-19 vaccines, we first determined neutralization patterns against a large panel of pre-Omicron and Omicron variants (Fig. 1).
COVID-19ワクチン用の抗原の選択を支援するための抗原分析に対するハムスターの一次感染血清の適合性を評価するために、まずプレオミクロンおよびオミクロン変異体の大規模なパネルに対する中和パターンを決定しました(図1)。
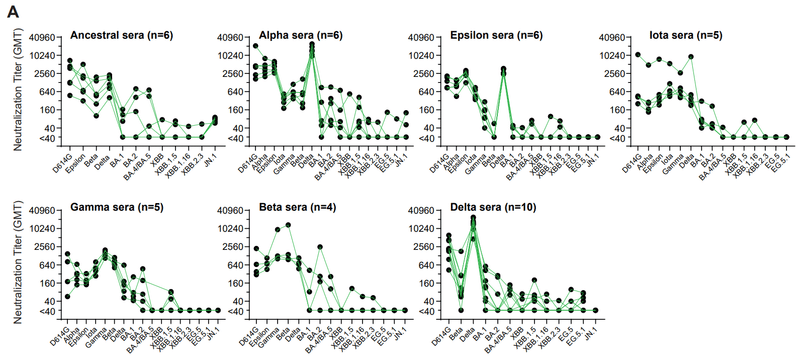
Variants tested for pseudovirus neutralization ranged from ancestral to JN.1 variants, but the number of variants tested for each primary infection serum sample was limited by available serum.
シュードウイルス中和についてテストされた変異体は祖先変異体から JN.1 変異体までの範囲でしたが、各一次感染血清サンプルについてテストされた変異体の数は利用可能な血清によって制限されました。
For pre-Omicron variant infections, neutralization titers were generally highest against the infecting variant, with variable cross-neutralization among pre-Omicron variants and much less cross-neutralization against Omicron variants (Fig. 1A).
プレオミクロン変異体感染の場合、中和力価は一般に感染変異体に対して最も高く、プレオミクロン変異体間では交差中和が異なり、オミクロン変異体に対する交差中和ははるかに低かった(図 1A)。
Infection by the Delta (B.1.617.2) variant generated the highest titers, possibly related to a more robust infection by this variant.
デルタ(B.1.617.2)変異体による感染は最も高い力価を生成しましたが、これはおそらくこの変異体によるより強力な感染に関連していると考えられます。
At the same time, the Delta variant appeared to be more sensitive to cross-neutralization than other pre-Omicron variants, possibly due to better exposure of neutralizing determinants.
同時に、デルタ変異体は他のオミクロン以前の変異体よりも交差中和に対してより敏感であるように見えましたが、これはおそらく中和決定基への曝露が良好だったためと考えられます。
Omicron sera also showed the highest titers against the infecting variant, with modest cross-neutralization against other Omicron variants and little or no cross-neutralization to pre-Omicron variants (Fig. 1B).
また、オミクロン血清は、感染変異体に対して最も高い力価を示し、他のオミクロン変異体に対しては中程度の交差中和が見られ、オミクロン以前の変異体に対しては交差中和がほとんどまたは全くありませんでした(図 1B)。
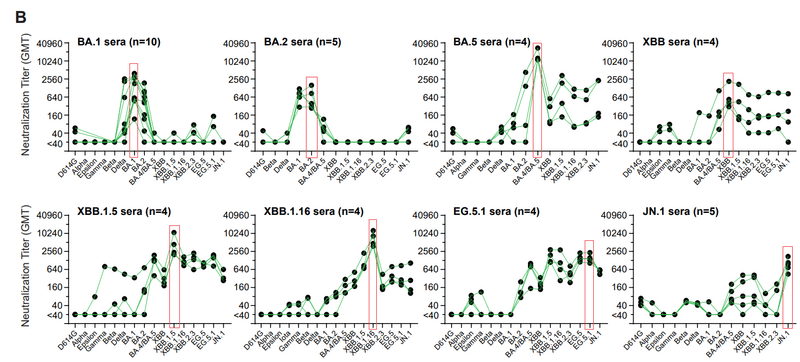
Notably, BA.1 and BA2 sera showed no cross-neutralization against JN.1 and XBB-lineage variants.
特に、BA.1 および BA2 血清は、JN.1 および XBB 系統変異体に対して交差中和を示さなかった。
In contrast, BA.5 sera showed cross-neutralization against the JN.1 and XBB-lineage variants, though titers were 24-75-fold lower than against the infecting BA.5 variant.
対照的に、BA.5 血清は JN.1 および XBB 系統変異体に対して交差中和を示しましたが、力価は感染している BA.5 変異体に対するものより 24 ~ 75 分の 1 でした。
XBB-lineage sera showed cross-neutralization against JN.1 and BA.4/BA.5.
XBB 系統血清は、JN.1 および BA.4/BA.5 に対して交差中和を示しました。
JN.1 sera also showed cross-neutralization against BA.4/BA.5 and XBB-lineage variants in a few animals.
JN.1 血清は、少数の動物において BA.4/BA.5 および XBB 系統変異体に対する交差中和も示しました。
XBB.1.5 sera showed high neutralization titers against the XBB-lineage variants.
XBB.1.5 血清は、XBB 系列変異体に対して高い中和力価を示しました。
The overall lower neutralization titers of BA.1, BA.2, XBB, EG.5.1 and JN.1 sera compared to BA.5, XBB.1.5 and XBB.1.16 sera may reflect variant differences in virus replication in hamsters or differences in immunogenicity and exposure of neutralizing epitopes.
BA.5、XBB.1.5、および XBB.1.16 血清と比較して BA.1 、BA.2、XBB、EG.5.1、および JN.1血清の中和力価が全体的に低いことは、ハムスターにおけるウイルス複製の変異の違い、または免疫原性および中和エピトープの露出の違いを反映している可能性があります。
Human and hamster neutralization titers correlate well.
ヒトとハムスターの中和力価はよく相関します。
We next compared neutralization titers from the hamster primary infection sera to the human primary infection sera that we previously reported for pre-Omicron variants and BA.1 variants.
次に、ハムスターの一次感染血清と、プレオミクロン変異体およびBA.1変異体について以前に報告したヒト一次感染血清の中和力価を比較しました。
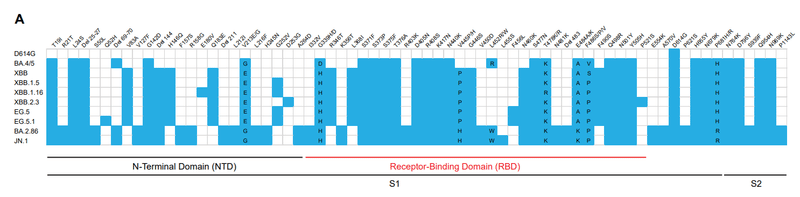
A comparison of spike amino acid sequences among primary human infection virus, hamster infection virus, and pseudovirus within given lineages revealed few differences (Fig. S1).
特定の系統内の初代ヒト感染ウイルス、ハムスター感染ウイルス、および偽ウイルス間のスパイクアミノ酸配列を比較すると、ほとんど違いがありませんでした(図S1)。
The number of differences within a lineage ranged from zero (B.1 in human infection versus pseudovirus) to six (BA.1 in human infection versus hamster infection) spike amino acid differences.
系統内の差異の数は、0 (ヒト感染対シュードウイルスにおける B.1) から 6 (ヒト感染対ハムスター感染における BA.1) のスパイクアミノ酸差異の範囲でした。
In the latter case, most spike substitutions are outside of the spike receptor binding domain.
後者の場合、ほとんどのスパイク置換はスパイク受容体結合ドメインの外側にあります。
Using the same pseudovirus neutralization assay, we found that human and hamster primary infection sera gave remarkably similar neutralization titers and specificity against the same SARS-CoV-2 variants (Figs. 2 and S2).
同じシュードウイルス中和アッセイを使用して、ヒトとハムスターの一次感染血清が、同じSARS-CoV-2変異体に対して著しく類似した中和力価と特異性を示すことがわかりました(図2およびS2)。
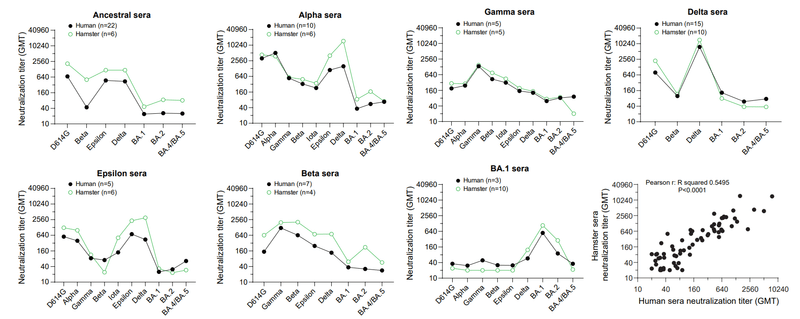
Overall, both human and hamster neutralization titers were typically highest against infecting variant, with modest cross-neutralization by pre-Omicron primary sera to pre-Omicron variants but comparatively little cross- neutralization to Omicron variants.
全体として、ヒトおよびハムスターの中和力価はいずれも感染変異体に対して通常最も高く、プレオミクロン一次血清によるプレオミクロン変異体に対する適度な交差中和が見られたが、オミクロン変異体に対する交差中和は比較的少なかった。
Likewise, the Omicron primary infection sera did not cross-neutralize pre-Omicron variants.
同様に、Omicron 一次感染血清は、Omicron 以前の変異体を交差中和しませんでした。
Antigenic maps created from human and hamster primary infection sera closely resemble each other.
ヒトとハムスターの一次感染血清から作成された抗原マップは、互いによく似ています。
We applied antigenic cartography to our extended hamster sera neutralization dataset, which allowed us to measure antigenic relationships to and among these newer Omicron variants that we could not measure using human primary infection sera alone.
私たちは抗原地図作成を拡張ハムスター血清中和データセットに適用しました。 これにより、ヒト初感染血清のみを使用した場合には測定できなかった、これらの新しい Omicron 変異体との間の抗原関係を測定できるようになりました。
As reported previously for primary human sera maps, Alpha, Delta, Epsilon, Beta, and Gamma variants formed a distinct antigenic cluster from the early Omicron variants BA.1, BA.2, BA.2.12.1, and BA.4/BA.5 (Fig. 3A, Table S1).
初代ヒト血清マップについて以前に報告されているように、アルファ、デルタ、イプシロン、ベータ、およびガンマ変異体は、初期のオミクロン変異体 BA.1、BA.2、BA.2.12.1、および BA.4/BA.5 とは異なる抗原クラスターを形成しました (図 3A、表 S1)。
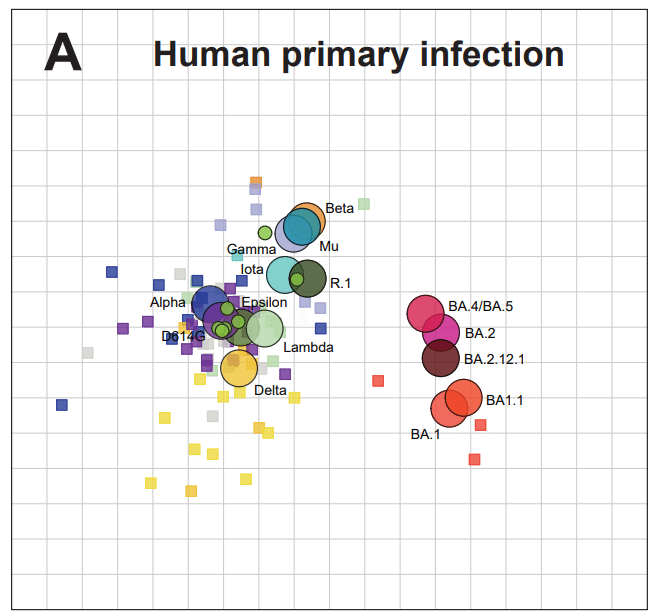
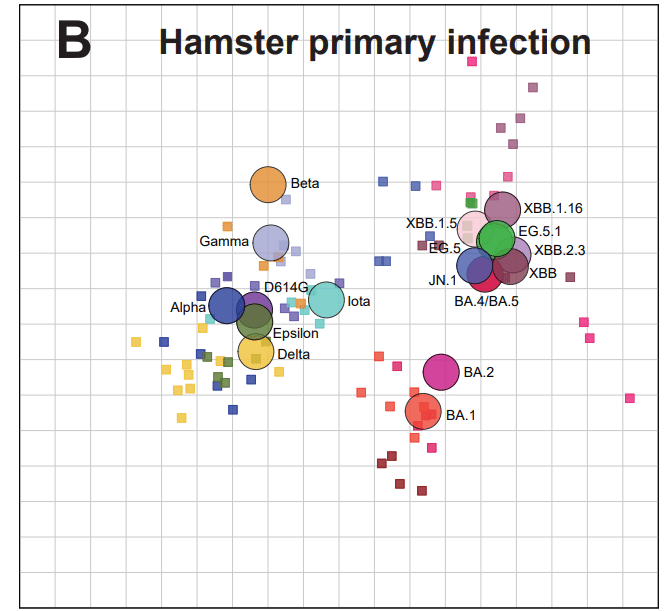
These variants have similar antigenic relationships to one another on a map made only with hamster sera (Fig. 3B, Procrustes arrows point from the position in one map to another in Fig. 3C), indicating the potential suitability of hamster primary infection as a substitute for primary human infection sera.
これらの変異体は、ハムスター血清のみで作成された地図上で互いに同様の抗原関係を示します (図 3B、プロクルステスの矢印は、図 3C の 1 つの地図上の位置から別の地図上の位置を指します)。 これは、ハムスターの一次感染血清がヒトの一次感染血清の代替として適している可能性があることを示しています。
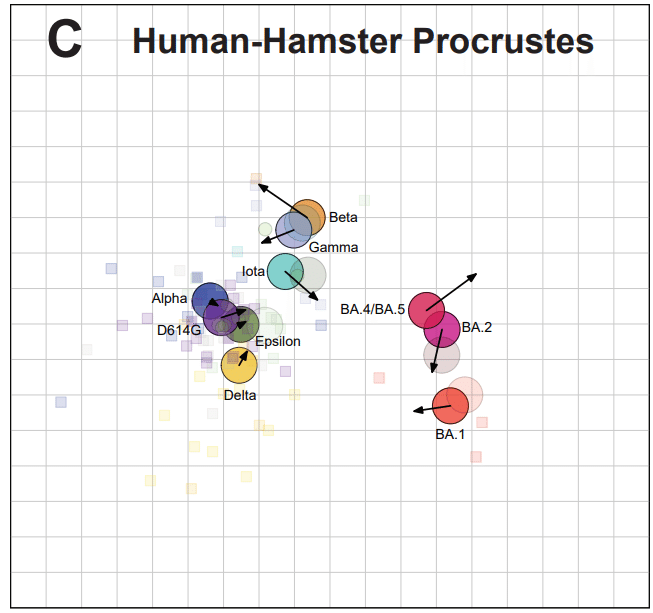
Distances from D614G to each of the variants is also similar between maps, with the greatest differences seen for BA.1 (1.3 antigenic units, AU) and BA.4/BA.5 (0.7 AU) (Table S1).
D614G から各変異体までの距離もマップ間で類似しており、BA.1 (1.3 抗原単位、AU) と BA.4/BA.5 (0.7 AU) で最も大きな違いが見られます (表 S1)。
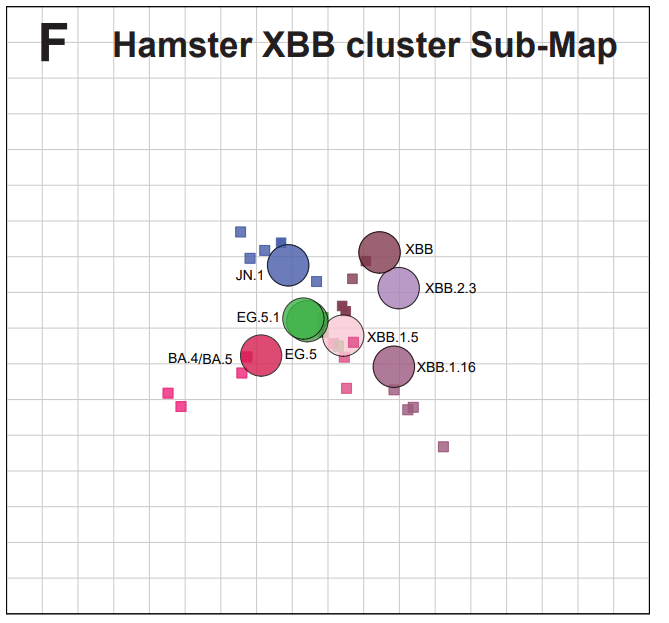
On the hamster map, the newly emerged XBB-lineage variants form an additional cluster (XBB cluster) furthest away from the ancestral variants and distinct from BA.1 and BA.2 (Fig. 3B).
ハムスターマップ上では、新たに出現した XBB 系統変異体は、祖先変異体から最も遠く離れ、BA.1 および BA.2 とは異なる追加のクラスター (XBB クラスター) を形成します (図 3B)。
With this addition of XBB-lineage variants in the hamster map, BA.4/BA.5 moves away from BA.1 and towards the more recent XBB-lineage variants (Fig. 3B).
ハムスターマップへの XBB 系統変異体のこの追加により、BA.4/BA.5 は BA.1 から離れ、より最近の XBB 系統変異体へと移行します (図 3B)。
Notably, JN.1 also clusters with BA.4/BA.5.
特に、JN.1 は BA.4/BA.5 ともクラスタ化します。
Given that the human and hamster sera measured similar antigenic relationships among the viruses, we created a merged human and hamster map including all data from both datasets (Fig. 3D).
ヒトとハムスターの血清がウイルス間で同様の抗原関係を測定したことを考慮して、両方のデータセットのすべてのデータを含むヒトとハムスターの統合マップを作成しました(図 3D)。
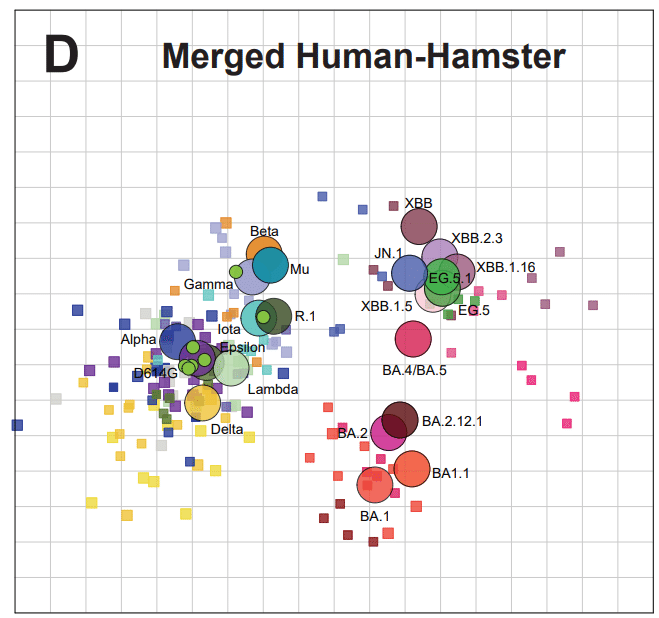
The merged antigenic map had similar error and fit to titer data as the individual human and hamster primary infection map (Fig. S3A) and had robustness in the positioning of viruses and sera when adding titer and antigen noise, as well as when resampling the data (Fig. S4).
統合された抗原マップには、個々のヒトおよびハムスターの一次感染マップと同様の誤差があり、力価データに適合しました(図 S3A)。 また、力価と抗原ノイズを追加するとき、およびデータをリサンプリングするときに、ウイルスと血清の位置決めに堅牢性がありました(図S4)。
The merged map shows similar antigenic relationships to the hamster map (Fig. 3E), with the greatest shifts in position in the XBB cluster.
統合されたマップは、ハムスター マップ (図 3E) と同様の抗原関係を示し、XBB クラスター内の位置が最も大きく変化しています。
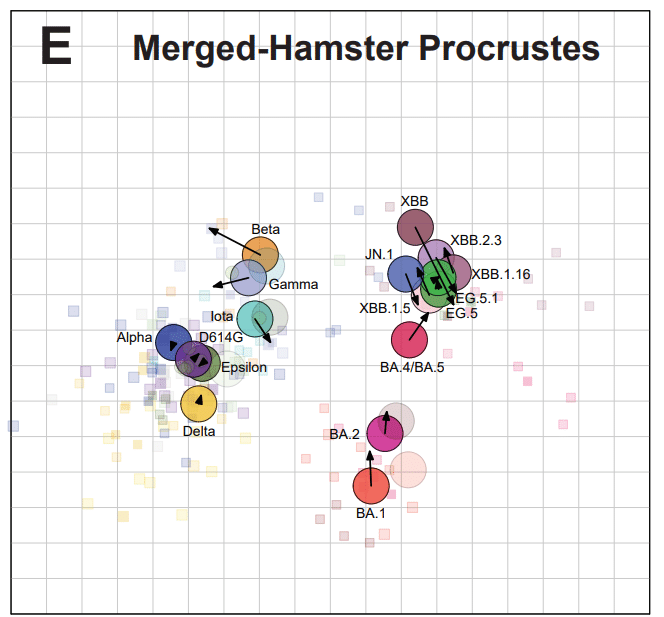
Compared to the hamster map, JN.1 and BA.4/BA.5 diverge in the merged human-hamster map, with JN.1 remaining with the XBB cluster and BA.4/BA.5 assuming a position between the XBB cluster and the early Omicron cluster (Fig. 3E).
ハムスター マップと比較すると、人間とハムスターの統合マップでは JN.1 と BA.4/BA.5 が分岐しています。 JN.1 は XBB クラスターに残り、BA.4/BA.5 は XBB クラスターと初期の Omicron クラスターの間の位置を想定します (図 3E)。
Based on dimensionality testing, we observed that the titers were better fit on maps made in three dimensions (Fig. S3C and S3D).
次元性テストに基づいて、力価が三次元で作成されたマップによりよく適合することが観察されました(図S3CおよびS3D)。
When we evaluated differences in antigenic positions between the 2D and 3D merged maps, we observed some of the most substantial shifts were within the XBB cluster, suggesting that the antigenic relationships in the 2D map were not as accurately representing the underlying data in this region of the map (Fig. S5A).
2D と 3D の統合マップ間の抗原位置の違いを評価したところ、最も大きな変化の一部が XBB クラスター内にあることが観察されました。 これは、2D マップの抗原関係がマップのこの領域の基礎となるデータを正確に表していないことを示唆しています (図 S5A)。
We thus evaluated whether antigenic relationships among the variants in the XBB cluster differed if evaluated only using hamster sera raised against these variants.
したがって、我々は、XBB クラスター内の変異間の抗原関係が、これらの変異に対して採取されたハムスター血清のみを使用して評価された場合に異なるかどうかを評価しました。
Both the serum and antigen positions shifted, in some cases substantially, between the full map and the XBB cluster map (Fig. 3F).
血清と抗原の位置は両方とも、完全なマップと XBB クラスター マップの間で、場合によっては大幅にシフトしました (図 3F)。
This shifting of antigenic positions within a cluster was not observed when the same analysis was performed on the earlier variant cluster (Fig. S5B).
クラスター内の抗原位置のこのシフトは、以前の変異クラスターに対して同じ分析を実行した場合には観察されませんでした(図S5B)。
Notably, distances were greater between JN.1, XBB.1.5, and BA.5 on the XBB cluster map (5 to 6-fold difference between each antigen and each other) compared to the full map (1 to 2.5-fold difference) (Fig. 3B and 3F).
特に、XBB クラスター マップ上の JN.1、XBB.1.5、および BA.5 間の距離は、完全なマップ (1 ~ 2.5 倍の差) と比較して (各抗原と相互の間で 5 ~ 6 倍の差) より大きかった (図3Bおよび3F)。
This suggests that the primary infection sera against XBB variants saw differences among these antigens that sera raised against early variants and Omicron were not able to detect, and the full map, which must accommodate all titers, did not fit the titers in the XBB cluster as well.これは、XBB 変異体に対する初感染血清には、初期変異体に対する血清では検出できなかったこれらの抗原間の差異が見られたことを示唆しており、Omicron は検出できませんでした。 そして、すべての力価を収容する必要がある完全なマップは、XBB クラスターの力価にも適合しませんでした。
Multiple antigenic exposures increase neutralization breadth against the variants.
抗原を複数回曝露すると、変異に対する中和の幅が広がります。
We next investigated neutralization breadth in sera from persons who had one of the following multiple antigenic exposures (Fig. 4A):
次に、以下の複数の抗原曝露のいずれかを経験した人の血清における中和幅を調査しました(図 4A)。
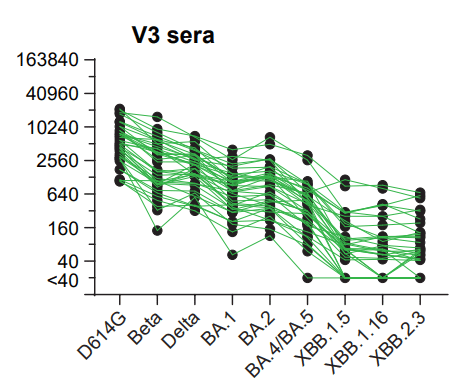
1) three doses of the ancestral mRNA COVID-19 vaccine (V3) ;
1) 祖先 mRNA COVID-19 ワクチン (V3) を 3 回投与。
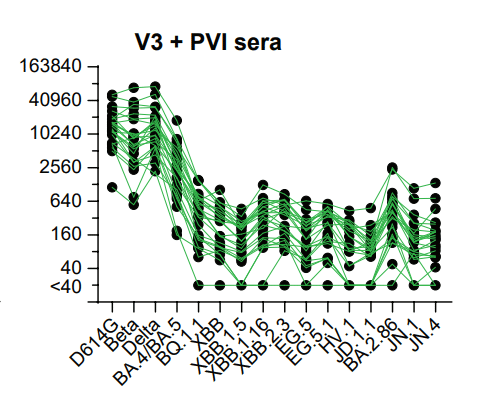
2) three doses of the ancestral mRNA COVID-19 vaccine and a post-vaccination infection (PVI) during BA.1 outbreak (V3+PVI);
2) BA.1 流行中の祖先 mRNA COVID-19 ワクチンの 3 回投与とワクチン接種後感染症 (PVI) (V3+PVI)。
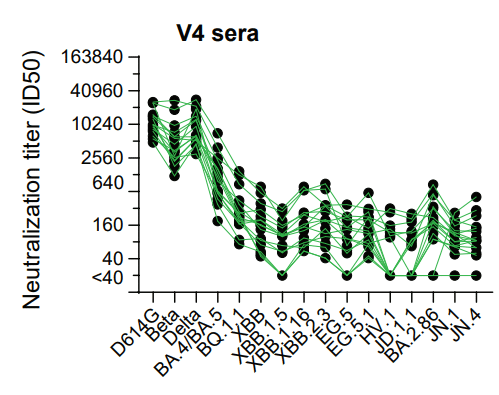
3) four doses of the ancestral mRNA COVID-19 vaccine (V4);
3) 祖先型mRNA COVID-19 ワクチン (V4) を 4 回投与。
4) three doses of the ancestral mRNA COVID-19 vaccine and one dose of the bivalent (ancestral+BA.5) mRNA COVID-19 vaccine (V3+Bi);
4)祖先mRNA COVID-19ワクチンを3回投与、二価(祖先+BA.5)mRNA COVID-19ワクチンを1回投与(V3+Bi)。
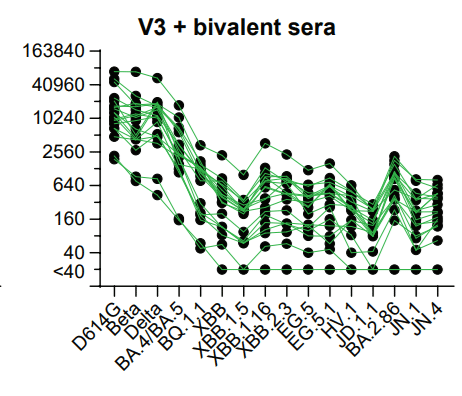
5) four doses of the ancestral mRNA COVID-19 vaccine and one bivalent mRNA COVID-19 vaccine (V4+Bi) ;
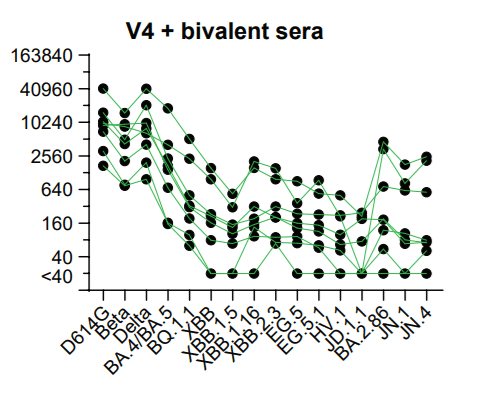
5) 祖先型 mRNA COVID-19 ワクチン 4 回と二価 mRNA COVID-19 ワクチン 1 回(V4+Bi)。
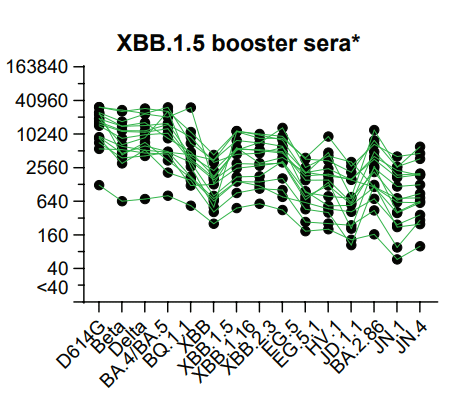
and 6) three or four doses of the ancestral mRNA COVID-19 vaccine, with or without a PVI before or after one bivalent mRNA COVID-19 vaccine followed by one XBB.1.5 mRNA vaccine (V3 or V4+bivalent+XBB.1.5 with or without PVI, referred to as XBB.1.5 booster group*).
6) 祖先 mRNA COVID-19 ワクチンを 3 回または 4 回接種。二価 mRNA COVID-19 ワクチン 1 回の前後に PVI の有無にかかわらず、XBB.1.5 mRNA ワクチン 1 回を接種 (V3 または V4+二価+XBB.1.5、PVI の有無にかかわらず、XBB.1.5 ブースター グループ* と呼ばれます)。
In the XBB.1.5 booster group*, six individuals had no reported PVI.
XBB.1.5 ブースター グループ* では、6 人は PVI の報告がありませんでした。
Based on time of infection, the PVIs in the XBB.1.5 group* included three presumed BA.1 PVIs, four presumed BA.5 PVIs after three doses of the ancestral COVID-19 vaccine, and three presumed XBB PVIs after the bivalent booster (Table S2).
感染時間に基づいてXBB.1.5 グループ* には、祖先型COVID-19ワクチンを3回接種した後に感染した人が推定 BA.1 株では3人、推定BA.5株では4人発生してました。 二価ブースター後では 3人の推定 XBB株感染が発生しました (表S2)。
Variant assignments of PVIs were based on the dominant circulating variant at the time of the documented infection.
PVI のバリアントの割り当ては、記録された感染時の主な循環バリアントに基づいていました。
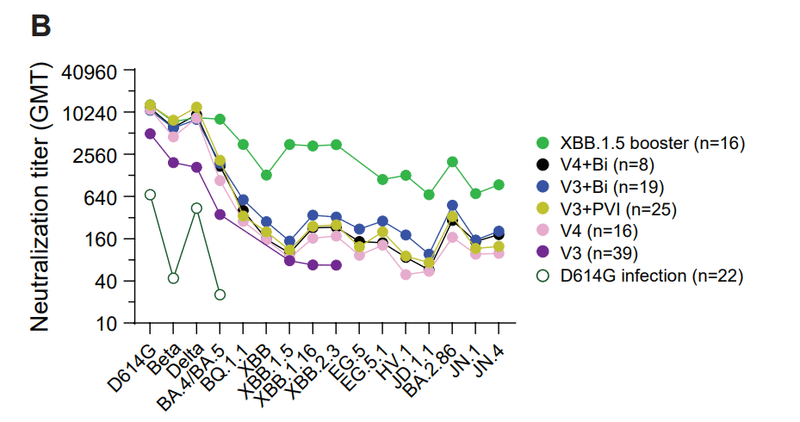
Overall, all groups that received vaccinations had higher neutralization titers and more breadth against the variants than the group that had only D614G infection (Fig. 4A-B).
全体として、ワクチン接種を受けたすべてのグループは、D614G 感染のみを持ったグループよりも高い中和力価と変異に対するより広い範囲を示しました (図 4A-B)。
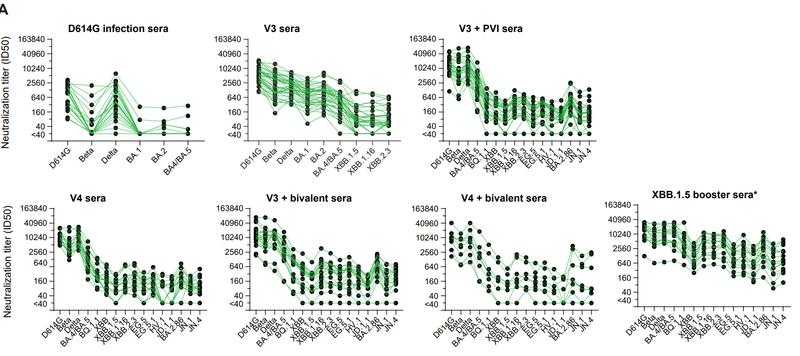
The different mutations in variant spikes compared to D614G are summarized in Fig. S6A.
D614Gと比較したバリアントスパイクの異なる変異を図S6Aにまとめます。
Groups that had heterologous antigenic exposures from XBB.1.5 or bivalent COVID-19 boosters or a BA.1 PVI after vaccinations with the ancestral COVID-19 vaccine had neutralization titers that trended higher than those from the group that had only the ancestral COVID-19 vaccine (V3) when measured against recently emerged XBB-lineage (XBB, XBB.1.5, XBB.1.16, XBB.2.3, EG5, EG.5.1, JD.1.1 and HV.1) and BA.2.86-lineage (BA.2.86, JN.1 and JN.4) variants (Fig. 4A-B).
祖先型COVID-19ワクチンのワクチン接種後に、XBB.1.5、二価COVID-19ブースター、またはBA.1 PVIによる異種抗原曝露を受けたグループは、中和力価が、最近出現したXBB系統(XBB、XBB.1.5、XBB.1.16、XBB.2.3、EG5、EG.5.1、JD.1.1、および HV.1) および BA.2.86系統 (BA.2.86、JN.1、および JN.4) のバリアントに対して測定した場合、祖先型COVID-19ワクチンのみを接種したグループ(V3)よりも高い傾向にあった(図4A-B)。
Neutralization titers against XBB-lineage and BA.2.86-lineage variants were 4-22-fold lower than titers against BA.4/BA.5 after the bivalent COVID-19 vaccine (Fig. 4B and S6B), indicating that XBB-lineage and BA.2.86-lineage variants exhibit substantial immune evasion from immunity elicited by prior vaccines, even after multiple immunizations.
XBB系統およびBA.2.86系統の変異体に対する中和力価は、二価COVID-19ワクチン接種後のBA.4/BA.5に対する力価よりも4~22倍低かった(図4BおよびS6B)。これは、XBB 系統および BA.2.86 系統の変異体が、複数回の免疫化後であっても、以前のワクチンによって誘発された免疫からの実質的な免疫回避を示すことを示しています。
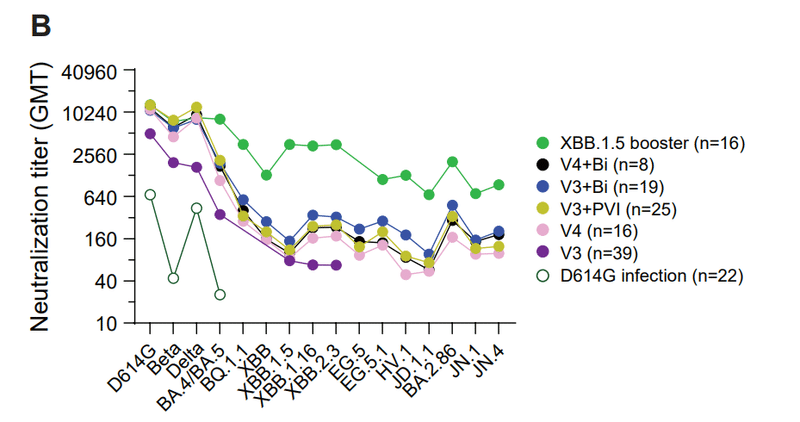
Notably, compared to the V4, V3+Bi, V3+PVI, and V4+Bi groups, the XBB.1.5 booster group* had higher neutralization titers against XBB-lineage and recently emerged variants, including JN.1.
特に、V4、V3+Bi、V3+PVI、および V4+Bi グループと比較して、XBB.1.5 ブースター グループ* は、XBB 系統および JN.1 を含む最近出現した変異体に対してより高い中和力価を示しました。
This finding is consistent with recent reports although the XBB.1.5 booster-induced neutralization titers against JN.1 are slightly different among the studies.
JN.1 に対する XBB.1.5 ブースター誘導性の中和力価は研究間でわずかに異なりますが、この発見は最近の報告と一致しています。
The XBB.1.5 booster also boosted titers against BA.4/BA.5 (Fig. 4B and S7B), consistent with other reports showing back-boosting to earlier variants.
XBB.1.5 ブースターも BA.4/BA.5 に対する力価を上昇させました (図 4B および S7B)。これは、以前の変異体に対するバックブーストを示す他の報告と一致しています。
However, neutralization titers against JN.1 were five-fold lower than titers against XBB.1.5 (Figs. 4B, S6B, and S7).
しかしながら、JN.1に対する中和力価は、XBB.1.5に対する力価よりも5倍低かった(図4B、S6B、およびS7)。
Sera from the XBB.1.5 booster group have flatter antibody landscapes and higher titers to newer variants than sera from groups with other combinations of vaccinations and pre-XBB variant infections.
XBB.1.5ブースターグループからの血清は、ワクチン接種とXBB以前の変異型感染の他の組み合わせを持つグループからの血清よりも、より平坦な抗体ランドスケープと新しい変異型に対するより高い力価を示します。
Antibody landscapes of the multi-exposure sera described above were constructed by fitting a cone-shaped landscape to neutralization titer data.
上述の多重曝露血清の抗体ランドスケープは、中和力価データに円錐形のランドスケープを当てはめることによって構築されました。
The hamster map was used as the base map in the antibody landscapes.
ハムスターマップは、抗体ランドスケープのベースマップとして使用されました。
All vaccination groups with or without a PVI had antibody titers, and thus detectable landscapes, across variants (Fig. 5A-E).
PVIの有無にかかわらず、すべてのワクチン接種グループは抗体力価を有しており、したがって変異体全体で検出可能な状況が見られました(図5A〜E)。
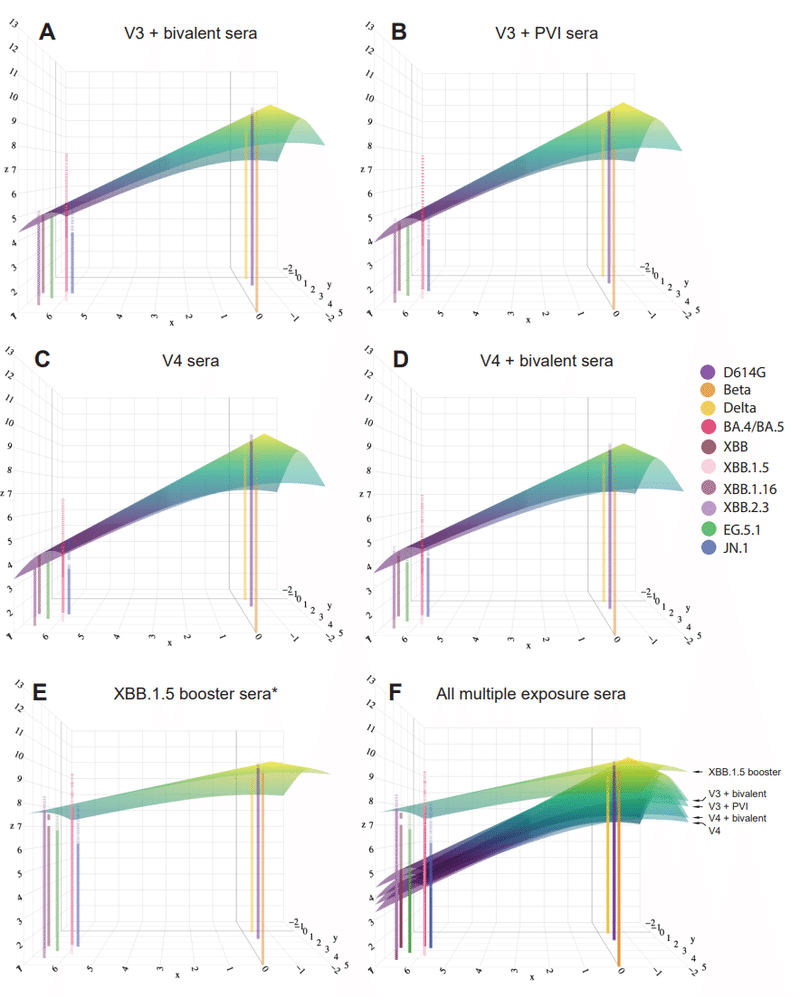
The landscapes were similar to one another, except for the XBB.1.5 booster sera* landscape.
XBB.1.5 ブースター セラ* ランドスケープを除いて、これらのランドスケープは互いに似ていました。
The V4 sera had the least breadth to antigenically distinct variants.
V4 血清は、抗原的に異なる変異の範囲が最も狭かった。
The V4+Bi sera had slightly increased breadth, followed by the V3+PVI, and V3+Bi sera.
V4+Bi 血清の幅はわずかに増加し、V3+PVI、V3+Bi 血清が続きました。
Landscapes were high for pre-Omicron and early Omicron variants (BA.4/BA.5) but dropped against XBB-lineage variants and JN.1.
Omicron 以前および初期の Omicron バリアント (BA.4/BA.5) ではランドスケープが高かったが、XBB 系統のバリアントと JN.1 に対しては低下しました。
In contrast, the XBB.1.5 booster sera* landscape was high across the map, including against all XBB-lineage variants.
対照的に、XBB.1.5 ブースター血清 * の状況は、すべての XBB 系統変異体を含め、マップ全体で高かった。
Similar patterns were observed for landscapes plotted above the XBB cluster hamster map (Fig. S8).
同様のパターンが、XBB クラスターハムスターマップの上にプロットされた風景でも観察されました(図 S8)。
Antibody titers against Beta, BA.4/BA.5, XBB.1.5 and JN.1 were less well-fit by the cone-shaped landscape as seen by their large residual titers.
ベータ、BA.4/BA.5、XBB.1.5、および JN.1 に対する抗体力価は、残留力価が大きいことからわかるように、円錐形のランドスケープにあまり適合しませんでした。
This was particularly true for BA.4/BA.5, which had measured titers at least two-fold (1 AU) greater than titers predicted from the cone landscape.
これは特に BA.4/BA.5 に当てはまり、錐体ランドスケープから予測された力価よりも少なくとも 2 倍 (1 AU) 大きい力価が測定されました。
In contrast, measured titers for JN.1 were more than 1 AU below the titers predicted from the cone landscape (Table S3).
対照的に、JN.1 について測定された力価は、錐体地形から予測された力価を 1 AU 以上下回っていました (表 S3)。
This suggests that a cone landscape may not accurately fit titers when plotted above an antigenic map that uses primary infection sera from newly emerged variants such as XBB.1.5.
これは、XBB.1.5 などの新たに出現した変異体からの一次感染血清を使用する抗原マップ上にプロットした場合、円錐形のランドスケープが力価と正確に一致しない可能性があることを示唆しています。
Notably, the large drop in titer for the vaccine sera between BA.4/BA.5 and the XBB variants, despite these viruses being close together on the primary antigenic map, suggests that any landscape or surface may not fit these data well and that primary infection sera may recognize epitopes on the variants differently from multi-exposure sera.
特に、 BA.4/BA.5 と XBB 変異体間のワクチン血清の力価の大幅な低下は、これらのウイルスが一次抗原マップ上で近接しているにもかかわらず、いかなる風景や表面もこれらのデータにうまく適合しない可能性があり、一次感染血清が多重曝露血清とは異なる方法で変異体上のエピトープを認識する可能性があることを示唆しています。
Repeat boosting decreases the antigenic distance from D614G to BA.5 more than to other variants, even for antigenic histories without BA.5 exposure.
追加免疫を繰り返すと、BA.5 に曝露されていない抗原病歴であっても、他の変異体よりも D614G から BA.5 までの抗原距離が減少します。
Multi-dimensional scaling analyses were also created for multi-antigen exposure sera groups that were described previously.
前述した多抗原曝露血清グループの多次元スケーリング分析も作成されました。
These visualizations cannot be interpreted as antigenic maps but can be useful for visualizing fold-drop in neutralization from infecting strains (e.g., D614G) to other strains.
これらの視覚化は抗原マップとして解釈できませんが、感染株 (D614G など) から他の株への中和におけるフォールドドロップを視覚化するのに役立ちます。
Maps made with multi-antigen exposure sera show that JN.1 and XBB.1.5, as well as other XBB-lineage variants, were more distant from D614G in all serum sets except following XBB1.5 booster vaccination, which decreased antigenic distances to these variants.
多重抗原曝露血清を用いて作成されたマップは、JN.1 および XBB.1.5、および他の XBB 系統変異体が、XBB1.5 追加ワクチン接種後を除くすべての血清セットにおいて D614G からより離れていることを示しています。 これにより、これらの変異体に対する抗原距離が減少しました。
BA.4/BA.5 was consistently situated closely to D614G (Fig. 6).
BA.4/BA.5 は一貫して D614G の近くに位置していました (図 6)。
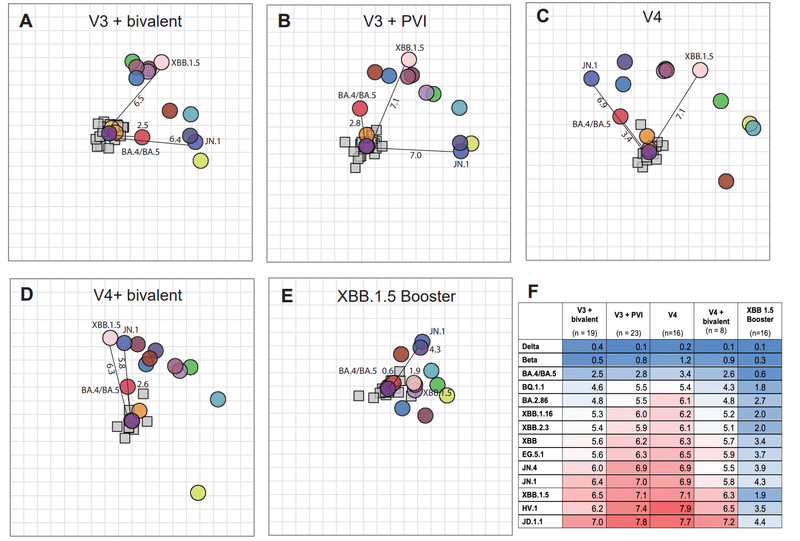
This contrasted with the hamster and merged human/hamster maps, where JN.1 and BA.5 clustered with the XBB variants, between 6.5-7.9 AU from D614G.
これは、JN.1 および BA.5 が D614G から 6.5 ~ 7.9 AU の範囲で XBB 変異体とクラスター化しているハムスターおよび統合ヒト/ハムスター マップとは対照的でした。
One hypothesis is that vaccination with the ancestral variant boosted immunity to a shared epitope on BA.4/BA.5 that is less present on the XBB variants.
1つの仮説は、祖先変異体によるワクチン接種により、XBB変異体にはあまり存在しないBA.4/BA.5上の共通エピトープに対する免疫が強化されたというものである。
This difference was not strongly recognized by primary infection sera, potentially because after only a single exposure, the shared epitope between the original variants and BA.4/BA.5 was only partially recognized.
この違いは、初感染血清では強く認識されませんでした。これは、1 回の曝露後では、元の変異体と BA.4/BA.5 の間の共有エピトープが部分的にしか認識されなかったためと考えられます。
Discussion
In this study, we extend prior studies by demonstrating that human and hamster primary infection serum titers correlate well.
この研究では、ヒトとハムスターの一次感染血清力価がよく相関していることを実証することで、先行研究を拡張します。
We also provide an extensive primary serum antigenic map that includes ancestral to JN.1 variants, which we discuss in relation to other published antigenic maps and human sera following multiple antigenic exposures.
また、祖先から JN.1 変異体までを含む広範な一次血清抗原マップも提供しており、他の公開されている抗原マップや複数の抗原曝露後のヒト血清と関連して議論します。
Finally, we show that those who received an XBB.1.5 booster had large increases in antibody titers against newly emerged variants.
最後に、XBB.1.5 ブースターを受けた人では、新たに出現した変異体に対する抗体力価が大幅に上昇したことを示します。
The combined antigenic and neutralization titer analyses provide insights into variant evolution and contribute to the development of a framework for evaluating antigenic differences among emerging SARS-CoV-2 variants to aide decisions about vaccine antigen updates.
抗原力価と中和力価を組み合わせた分析は、変異体の進化に関する洞察を提供し、ワクチン抗原の更新に関する決定を支援するために、新たなSARS-CoV-2変異体間の抗原の違いを評価するためのフレームワークの開発に貢献します。
Overall, our primary human and hamster maps show comparable orientations and similar topology to other maps created with human and hamster sera.
全体として、我々の主要なヒトおよびハムスターのマップは、ヒトおよびハムスターの血清を使用して作成された他のマップと同等の方向および同様のトポロジーを示します。
The consistency between human and hamster maps over a two-year span show that hamster sera are a suitable surrogate for creating antigenic maps to surveil SARS-CoV-2 antigenic variation and predict immune escape.
2年間にわたるヒトとハムスターのマップ間の一貫性は、ハムスター血清がSARS-CoV-2の抗原変異を監視し、免疫逃避を予測するための抗原マップを作成するための適切な代用物であることを示している。
Our merged human-hamster primary infection map positions BA.4/BA.5 and JN.1 in a cluster with newer XBB Omicron variants, suggesting shared epitopes.
我々の統合されたヒトハムスター一次感染マップでは、BA.4/BA.5 および JN.1 が新しい XBB Omicron 変異体を含むクラスターに位置しており、エピトープが共有されていることが示唆されています。
Previous studies have included limited number of samples or no sera raised against newer variants.
これまでの研究では、限られた数のサンプルが含まれているか、新しい変異体に対して採取された血清は含まれていませんでした。
By including additional single exposure hamster sera for BA.1, BA.4/BA.5, XBB variants and JN.1, our study helps to increase resolution in this region of antigenic space.
BA.1、BA.4/BA.5、XBB バリアントおよび JN.1 の追加の単回曝露ハムスター血清を含めることにより、我々の研究は抗原空間のこの領域の解像度を向上させるのに役立ちます。
Interestingly, we found that the full hamster and merged hamster-human antigenic maps did not accurately fit titers for sera and antigens in the XBB cluster.
興味深いことに、完全なハムスター抗原マップとハムスターとヒトを組み合わせた抗原マップは、XBB クラスター内の血清および抗原の力価に正確に適合しないことがわかりました。
When we made a map using only the XBB variants and sera raised against these viruses, we found that the antigens moved apart (Fig. 3F).
これらのウイルスに対して生成された XBB 変異体と血清のみを使用してマップを作成したところ、抗原が離れて移動することがわかりました (図 3F)。
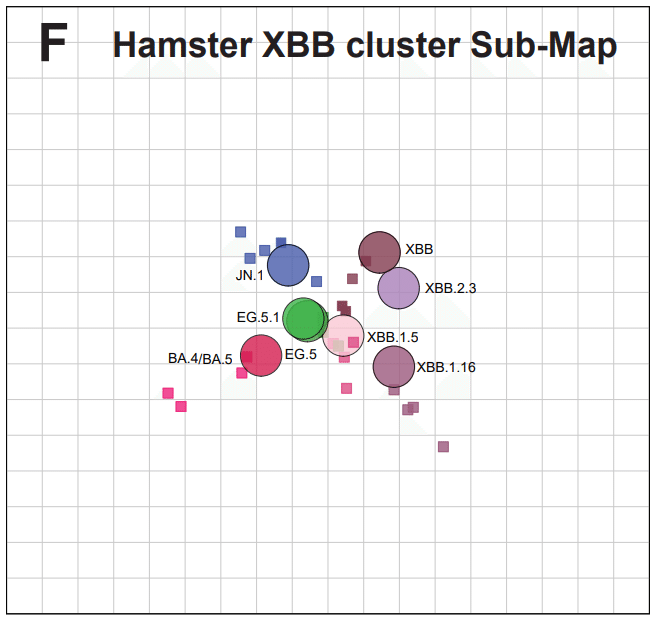
The antigenic distances observed in the XBB cluster map better matched the differences observed by XBB1.5 booster sera*.
XBB クラスター マップで観察された抗原距離は、XBB1.5 ブースター血清* で観察された差異とよりよく一致しました。
These findings suggest that there may be multiple ways to use antigenic maps to interpret complex serological data.
これらの発見は、抗原マップを使用して複雑な血清学的データを解釈する方法が複数ある可能性があることを示唆しています。
The map including all antigens and sera is useful for tracking antigenic evolution and identifying clusters.
すべての抗原と血清を含むマップは、抗原の進化を追跡し、クラスターを識別するのに役立ちます。
However, greater discrimination among variants within a cluster, which could be important for decisions about vaccine updates, may be achieved by making maps using only sera and antigens from that cluster.
ただし、ワクチンの更新に関する決定に重要となる可能性があるクラスター内の変異間のより大きな区別は、そのクラスターからの血清と抗原のみを使用してマップを作成することによって達成される可能性があります。
Building upon antigenic cartography, antibody landscapes have been useful for characterizing neutralization profiles of individuals who have had multiple antigenic exposures.
抗原地図作成に基づいて構築された抗体ランドスケープは、複数回抗原に曝露された個人の中和プロファイルを特徴付けるのに役立ちます。
The height of an accurate antibody landscape can be used to infer antibody titers against variants that were not used in making of the landscape.
正確な抗体ランドスケープの高さは、ランドスケープの作成に使用されなかった変異体に対する抗体力価を推測するために使用できます。
It has been common for antibody landscapes generated with multiple ancestral SARS-CoV-2 vaccinations and infection histories to assume a cone landscape, often with a peak at D614G (ancestral strain) with the landscape gradually tapering as antigenic distance from D614G increases.
複数の祖先のSARS-CoV-2ワクチン接種と感染歴で生成された抗体ランドスケープは円錐状のランドスケープを想定するのが一般的であり、多くの場合D614G(祖先株)にピークがあり、D614Gからの抗原距離が増加するにつれてランドスケープは徐々に先細りになっています。
Our study found that booster vaccines or PVIs containing Omicron moderately boosted neutralizing antibodies against Omicron variants more than repeated vaccination with ancestral strain, which has been noted elsewhere.
私たちの研究では、オミクロンを含むブースターワクチンまたはPVIは、他の場所で指摘されている祖先株による繰り返しワクチン接種よりも、オミクロン変異体に対する中和抗体を中程度に増強することがわかりました。
In addition, we show that booster vaccination with XBB.1.5 increases neutralizing antibodies and thus the landscape even higher than prior bivalent vaccinations or PVIs.
さらに、XBB.1.5 による追加ワクチン接種により中和抗体が増加し、従って以前の二価ワクチン接種または PVI よりもさらに高い状況が得られることを示します。
Previous studies have suggested that vaccination with D614G containing vaccines may blunt antibody responses to the new antigens and that repeat antigen exposure may be needed to boost response to the new antigen.
これまでの研究では、D614G を含むワクチンを接種すると新しい抗原に対する抗体反応が鈍化する可能性があり、新しい抗原に対する反応を高めるには抗原への繰り返しの曝露が必要である可能性があることが示唆されています。
Compared to the prior booster vaccination that included two antigens, D614G and BA.4/BA.5, the XBB.1.5 booster only contained one antigen, which, taken with the higher titers, suggests that including the antigen of interest and excluding D614G may improve neutralizing antibody titers against the antigen of interest as well as antigenically related antigens.
D614G と BA.4/BA.5 の 2 つの抗原を含む以前の追加免疫ワクチン接種と比較して、 XBB.1.5ブースターには1つの抗原のみが含まれており、より高い力価を考慮すると、目的の抗原を含めてD614Gを除外すると、目的の抗原および抗原関連抗原に対する中和抗体力価が向上する可能性があることが示唆されます。
Residual titers plotted on the antibody landscape offer a glimpse of how well the landscape can predict titers against a particular variant.
抗体ランドスケープ上にプロットされた残存力価は、ランドスケープが特定の変異体に対する力価をどの程度正確に予測できるかを垣間見ることができます。
In prior antibody landscapes that use antigenic maps with BA.4/BA.5 as the latest variant, the landscapes with plotted residuals show landscapes that accurately fit antibody titers.
最新のバリアントとして BA.4/BA.5 の抗原マップを使用する以前の抗体ランドスケープでは、残差がプロットされたランドスケープは抗体力価に正確に適合するランドスケープを示します。
However, in landscapes that include XBB and BQ variants in their antigenic map, the measured titers against BA.4/BA.5 have generally been greater than what the landscape predicts.
ただし、抗原マップに XBB および BQ 変異体が含まれるランドスケープでは、BA.4/BA.5 に対する測定された力価は一般にランドスケープが予測するものよりも高くなっています。
In our study, titers for BA.4/BA.5 are also consistently higher than the fitted landscape, including in individuals without a history of BA.4/BA.5 infection.
私たちの研究では、BA.4/BA.5 感染歴のない個人を含め、BA.4/BA.5 の力価も当てはめられた状況よりも一貫して高くなっています。
This finding could reflect boosting or affinity maturation of cross-neutralizing antibodies to shared epitopes in BA.4/BA.5 or better exposure of such epitopes in BA.4/BA.5 than XBB variants, even for those only receiving the ancestral vaccine.
この発見は、先祖株ワクチンしか受けていない人でも、BA.4/BA.5 の共有エピトープに対する交差中和抗体のブースティングまたは親和性成熟、または BA.4/BA.5 におけるそのようなエピトープの露出が XBB 変異体よりも優れていることを反映している可能性があります。
Our multiple antigenic exposure results are consistent with previous findings of decreased distance between D614G and Omicron variants in sera from individuals boosted with various Omicron variants (BA.1, BA.5 or XBB.1.5).
我々の複数の抗原曝露の結果は、さまざまな Omicron 変異体(BA.1、BA.5 または XBB.1.5)で追加免疫された個人の血清における D614G と Omicron 変異体の間の距離が減少したというこれまでの知見と一致しています。
The visualizations of multi-antigen exposure sera in this study show that bivalent vaccination with the ancestral strain and BA.4/BA.5 variants shortens the antigenic distance between D614G and BA.4/BA.5 to a greater extent than between D614G and XBB.2.3 and XBB.1.16, as expected given that individuals were exposed to both D614G and BA.4/BA.5.
この研究における多抗原曝露血清の視覚化は、祖先株およびBA.4/BA.5変異体による二価ワクチン接種が、 D614G と XBB.2.3 および XBB.1.16 の間よりもD614GとBA.4/BA.5の間の抗原距離を大幅に短縮することを示している。個人が D614G と BA.4/BA.5 の両方に曝露されたことを考えると、予想どおりでした。
However, we also observe that four doses with the ancestral strain also reduces the distance between D614G and BA.4/BA.5 more than D614G and XBB.2.3 and XBB.1.16, which was not expected, because these individuals had not been exposed to BA.4/BA.5.
ただし、祖先株の 4 回の投与では、D614G と XBB.2.3 および XBB.1.16 よりも D614G と BA.4/BA.5 の間の距離が短縮されることも観察されています。 これらの個人は BA.4/BA.5 に曝露されていなかったため、これは予想外でした。
This is a continuation from Wang et. al. that shows decreased distance between D614G and BA.4/BA.5 when ancestral strain vaccination increases from two to three doses.
これは、祖先株ワクチン接種が 2 回から 3 回に増加すると、D614G と BA.4/BA.5 の間の距離が減少することを示す Wang らの報告の続きです。
This observation suggests there may be shared antigenic epitopes between D614G and BA.4/BA.5 that are not detectable using primary infection sera but are present following repeat exposure to the ancestral variant due to vaccination.
この観察は、D614G と BA.4/BA.5 の間に共通の抗原エピトープが存在する可能性を示唆しており、これは初感染血清では検出できないが、ワクチン接種による祖先変異体への繰り返し曝露後には存在する。
These results, along with differences in variant neutralization by primary infection sera compared to multi-antigen exposure sera, highlight how past antigenic exposures from infections, vaccines, or both can impact subsequent vaccine responses.
これらの結果は、多抗原曝露血清と比較した一次感染血清による変異中和の違いとともに、感染症、ワクチン、またはその両方による過去の抗原曝露がその後のワクチン反応にどのような影響を与える可能性があるかを浮き彫りにしています。
Importantly, in sequential exposures to multiple SARS-CoV-2 antigens, the latest new antigen not only elicits titers against the most recently exposed antigen, it also back-boosts titers to previously exposed antigens, as shown here by the boosted titers against BA.4/BA.5 after the XBB.1.5 vaccine booster (Fig. 4B and S7B).
重要なのは、複数の SARS-CoV-2 抗原への連続曝露では、最新の新しい抗原が、最も最近曝露された抗原に対する力価を誘発するだけでなく、以前に曝露された抗原に対する力価をバックブーストすることです。 ここでは、XBB.1.5ワクチン追加免疫後のBA.4/BA.5に対する追加力価によって示されているように(図4BおよびS7B)。
Such back-boosting from a new antigen, which has been shown previously by others, reflects expansion of memory B cells, leading to the production of cross neutralizing antibodies.
新しい抗原からのこのようなバックブーストは、他の研究者によって以前に示されており、記憶B細胞の拡大を反映しており、交差中和抗体の産生につながります。
In addition, the XBB.1.5 booster increased neutralization titers against the advanced JN.1 variant, though titers were 5-fold lower than against XBB.1.5.
さらに、XBB.1.5 ブースターは、高度な JN.1 変異体に対する中和力価を増加させましたが、力価は XBB.1.5 に対するものより 5 分の 1 でした。
This result suggests that the boosted antibodies had lower affinity or titers to cross-neutralizing determinants in JN.1.
この結果は、追加免疫された抗体の、JN.1 の交差中和決定基に対する親和性または力価が低いことを示唆しています。
These findings are consistent with previous reports showing that JN.1 substantially evades post-vaccination sera and that the XBB.1.5 booster can provide moderate protection against JN.1.
これらの発見は、JN.1 がワクチン接種後の血清を実質的に回避し、XBB.1.5 ブースターが JN.1 に対して中程度の防御を提供できることを示す以前の報告と一致しています。
Together, the neutralization titer data and extensive cartography analyses suggest that JN.1 may be sufficiently different from XBB.1.5 that antibody coverage could be improved with a matched vaccine antigen.
まとめると、 中和力価データと広範な地図作成分析は、JN.1 が XBB.1.5 とは十分に異なる可能性があることを示唆しており、対応するワクチン抗原によって抗体適用率が向上する可能性があります。
Limitations of this study include the variable time points in which convalescent sera was obtained post-COVID-19 diagnosis (between 2-59 days).
この研究の限界としては、COVID-19診断後、回復期血清が得られた時点(2~59日)が変動することが挙げられる。
The lack of human primary infection sera for Omicron variants did not allow us to confirm whether human and hamster have similar responses to these variants.
オミクロン変異株に対するヒトの一次感染血清が不足しているため、ヒトとハムスターがこれらの変異株に対して同様の反応を示すかどうかを確認することはできませんでした。
In addition, cone landscapes may not be an ideal tool to predict antibody titers when newer variants such as XBB are added to the antigenic map, and we do not offer a better model.
さらに、XBB などの新しい変異体が抗原マップに追加された場合、錐体ランドスケープは抗体力価を予測する理想的なツールではない可能性があります。 また、これより優れたモデルは提供していません。
However, we bring this issue to light for further consideration of how antibody landscapes may be used to predict titers for emerging SARS-CoV-2 variants.
しかし、我々は、抗体ランドスケープを使用して新興の SARS-CoV-2 変異株の力価を予測する方法をさらに検討するために、この問題を明らかにします。
A key strength of this study is the enhanced resolution of SARS-CoV-2 antigenic maps by including additional primary hamster sera for BA.1, BA.4/BA.5, XBB-lineage, and JN.1 variants together with a cartography approach that focuses on one antigenic cluster.
この研究の主な強みは、1 つの抗原クラスターに焦点を当てた地図作成アプローチとともに、BA.1、BA.4/BA.5、XBB 系統、および JN.1 変異体の追加の初代ハムスター血清を含めることによって、SARS-CoV-2 抗原マップの解像度が向上していることです。
以下省略。
この記事が気に入ったらサポートをしてみませんか?
