
新型コロナ - 都道府県別 Rt (1)
rt.liveを参考に、都道府県別の実行生産数を求めた。行程の説明を試みるが、私は素人(何度も書くが)で、プログラムも論文も全てきちんとフォローしたわけではありません。私の理解の範囲内で概要を書きます。
1. Introduction
Rt(R_t)とは 「実効再生産数 (Effective Reproduction Number)」で感染者一人が平均して何人に感染させるか、という値。R0(R_0)という基本再生産数(Basic Reproduction Number)という値があって、その時間(t)変動なのでtなのだろう。
Rtが1以上になると感染者数は増える。日毎の感染者数よりもRtの変動をみる方が、我々も危機感を持てるのでは。
東洋経済のDashboardではRt(実効再生産数)の図が更新されている。都道府県ごとのRtもある。GitHubでスクリプトも公開されていているので、どのような計算で値が出ているのかわかる。西浦教授のRt会議のスライドも参考に。
アメリカでは上のようなサイトが最近(6/26)立ち上がった。こちらもGitHubで計算が公開されている。ちなみにk-sysとmikeykというcontributers2名はInstagramの創始者で、自らプログラムを書いているところが流石。
今回は後者を参考にRtを求めてみる。前者はR言語であること、後者ではPyMC3を使っているので。
他にもRであればEpiEstimというpackageも公開されていて、こちらが山中教授がRtを求めるのに使ったエクセルのupdate版となる。
2. GenerativeModel
rt.liveの中で何をしているかを大まかに説明してみる(理解の範囲内で)。tutorialに説明がある。Modelの詳細については元の論文等を参考に。
What we want : まず、求めたいのはRt。rt.liveでの表記 R_e(t) がわかりやすい。effective (実行)な reproduction number (再生産数) でこれは時間によって変動する値。ロックダウンやsocial distancing によって変わるので、ある時間 t での R_e なので R_e(t) となる。t はここでは日。
- Aside -
ちなみに日本では social distance と言うが、これは社会学で使われる言葉で、社会的な集団(性別・人種・階級)間に存在する距離を指す。公衆衛生では、social distancing が正しい。もっと正しい言葉は physical distancing と言われている。
How : Rt は、普通は SIR (Susceptible, Infectious, or Recovered) やSEIR (Susceptible, Exposed, Infectious, or Recovered)というモデル(参考)を使って求めるが、a)仮定も結構ある、b)モデル中の ODE (ordinary differential equations, 常微分方程式) は時間をくう。なので、rt.liveではもう少し簡単な logic を使っているらしい。
ここで必要なparameterは Serial interval と Generation time の二つ。下の西浦教授のスライドに詳しい(プログラムの中身をみていると _si とか _gt とか出てくるのに相当する)。

同じスライドに発病時刻がわからない場合は、診断時刻を発病時刻にする、とある。上の図でいうと、グレーの縦線 onset (発病) になる。 なので、基本的に必要なデータは日毎の陽性患者の数で、あとは発病期間は西浦教授の論文や、世代時間を求めるための Outbreak のモデルはこのサイトから。しかし、陽性患者の数は当然検査を行った数によって変わってくるので、補正を入れるため日毎の検査数もデータとして必要になる。
その後、発病から遡って感染(上の図ではinfection)の日を求めるのにはconvolution(畳込み)をする。下の西浦教授のスライドで説明があるが、rt.liveとの違いは詳しい比較をしないとわからない。

週末に検査が少ないなど、元のデータは discrete になる。これをPyMC3で 各種 parameter に分布を持たせ、smooth にする。また、Outbreak の simulation をここから参照しているが ここでMC(モンテカルロ法) が使われる。
3. Preparation
準備に以下を実行。
・Install pymc3
・Download rtcovidlive/cvid-model
Install pymc3
conda install -c conda-forge pymc3
pymc3と他にも必要なarviz、theanoもinstallされている。

covid-modelのrequirementには、他にも色々書いてあるが、私の環境ではこのinstallで十分だった。
Download rtcovidlive/covid-model
rtcovidliveのGitHubにいってcovid-modelをclick。

CodeをclickするとDesktopをinstallするか、と聞かれたのでしてみたが、zipのdownloadだけでよかったはず。
3. Data
jupyterでtutorial.ipynbを開けて、dfをprintしてみる。##はオリジナルではなくmy comments。

上記2でも書いたように必要なデータは毎日のPCR検査数 (total) と陽性者数 (positive)。これを都道府県ごとに、上のような表にしてみる。
4. Procedure
[1] 必要 package を読む。get_and_process_covidtracking_data は data_us.py で定義されている (私はいつも grep で探す) が、州によって細かいadjustmentをしているので、特にこれは必要ない。
[2] いつも使っている東洋経済のデータを読む。年・月・日と別々のコラムにある日付を一つにし、必要のないデータは消す。PCR検査数は testedPositive → total へ、陽性者数 はtestedPositive → positiveへと名前を変える。
ちなみに、検査数の名前は rt.live が読んでいる元データではPCR検査の結果には positive と negative があるので合わせて total と読んでいるので。

[3] 日付でグループされているデータを都道府県ごとにする。GenerativeModelが読めるように、set_indexでindex を 'prefecture' と 'date' とする。
このデータは累積で日毎の数ではないため、diffでrowごとの差をとる。defaultのaxisは0(row)のため、指定する必要はない。
dfをprintするとわかるが、diffの結果NaNになるデータがある(初日とか)のでdropnaで取り除く。6251-6172=79のデータが除かれた。

[4] df2のデータをset_optionで全部書き出し、眺めてみるとわかるが累積のはずのデータが差分をとるとnegativeになる時がある。これは、行政で時々訂正が入るためらしい(toyokseizai dashboardのQ&A参考)。
printしてそのようなデータは72あること、dropでcondition(yes-or-no)を設定して取り除くと残ったデータは6100で、上の6172に合うことを確認。

[5] 京都のデータをみてみる。locでindexが"kyoto"のデータを取り出す。
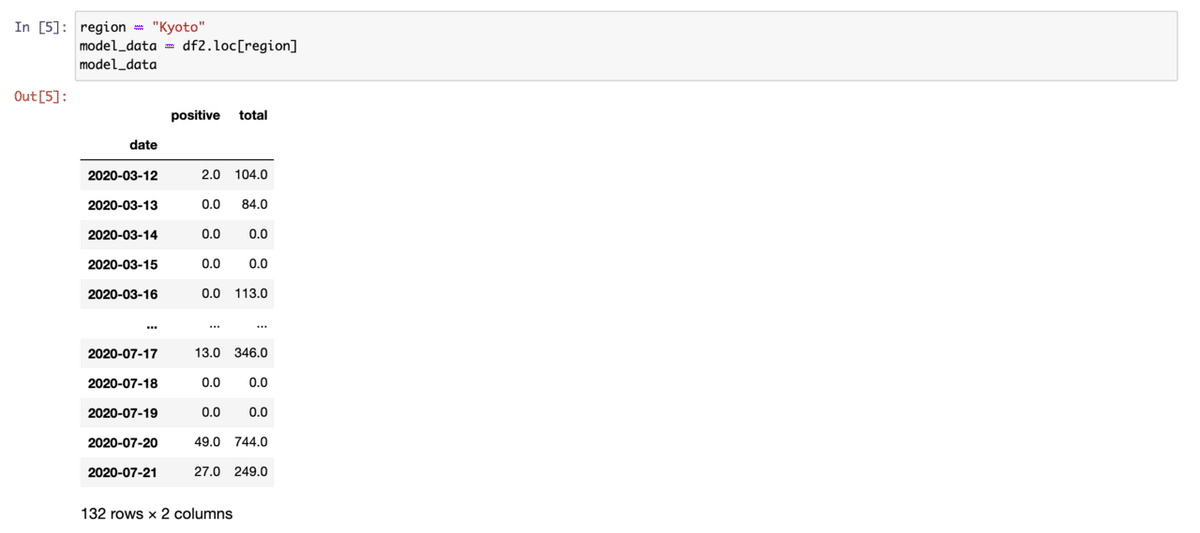
[6] GenerativeModelを走らせる。ここではMCが走っているので時間がかかり、%%timeでexecutionの時間を書き出してみる。ほぼ12分かかった。他の都道府県でもだいたい10分ちょっと。
Outbreak simulation の convolution に theano package が使われている。
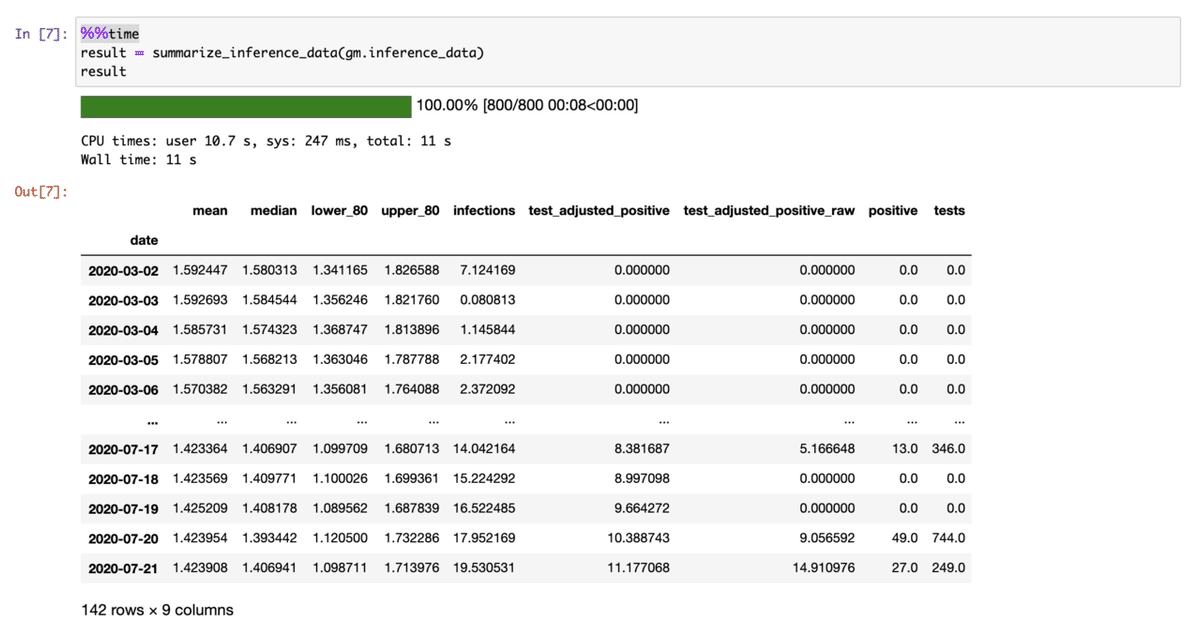
[7] data.py中にあるsummerize_inference_dataでMCで導かれたデータをinference(推定)するのに使った色々な値を書き出す。ここでArviZ packageが使われている。
debugをするときは、ここの値を例えばhistogramやscatter plotやつくってみながらおかしな値を探してみる?
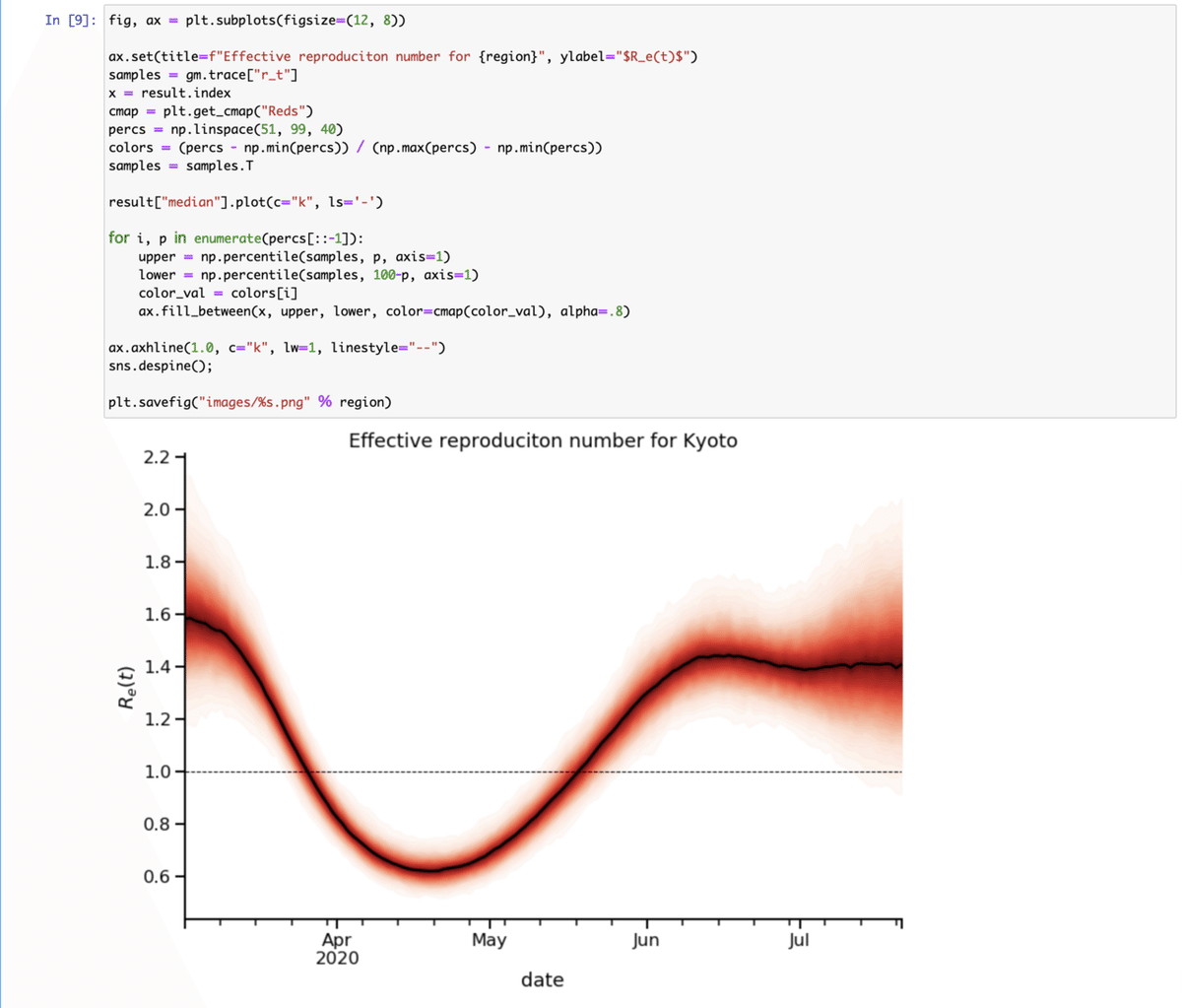
[8] ここではtutuorialに基づく説明を。まず、legendは下から読む。その順序で各ステップのモデルのoutputが図になっている。
グレー:陽性者数の元データ →
オレンジ:モデルがグレーを再現しようとしていることを表す →
青:検査数が一定だとした場合に導かれる陽性者数(日毎の検査数による補正が入る) →
緑:感染から発病までのdelay(時間差)に分布を持たせて、畳み込みし求めた感染者数。これにまた発生時間の分布を持たせ、感染が起こった時刻を求める。

[9] Rtは、陽性と診断された日ではなく、感染がいつ発生したかに基づいて導かれる。
5. Outputs
他にもいくつか。



6. Summary
rt.live の open source を元に、東洋経済のデータを使って都道府県毎の実行生産数を求めた。国民に行動を促すのであれば、感染が拡大するのか収束するのかが一目でわかるこちらの図をもっと広めるべき。
時間があれば、東洋経済の実行生産数との比較や、プログラムの中をみながらモデルをきちんとフォローしたい。
疑問もある。lockdown の前後に例えば、parameterの値は変わるのかとか。
この記事が気に入ったらサポートをしてみませんか?
