コロナワクチンの汚染DNAと有害事象
Kevin McKernan先生がコロナワクチンのDNA汚染を2023年の2月に初めて報告して以来、世界中の複数のラボから追試実験の報告が続いています。そして先日、カナダと米国の共同研究による追試実験の結果がプレプリントとして発表されました。McKernan先生も共同研究者として名を連ねています。
DNA fragments detected in monovalent and bivalent Pfizer/BioNTech and Moderna modRNA COVID-19 vaccines from Ontario, Canada: Exploratory dose response relationship with serious adverse events.
Speicher et al. (2023) OSF Preprints
https://osf.io/mjc97/
また、McKernan先生はこのプレプリントを自身のブログ上でも紹介しています。
カナダ・オンタリオ州の一価および二価のファイザーおよびモデルナmodRNA COVID-19ワクチンで検出されたDNA断片: 重篤な有害事象との用量反応関係を探る
ここで解析されたのは以下の12ロットのmRNAコロナワクチンから合計27バイアルです。
• モデルナ小児/成人用一価ワクチン (5ロット)
• モデルナ成人用BA4/5対応二価ワクチン (1ロット)
• モデルナ小児/成人用BA1対応二価ワクチン (1ロット)
• モデルナXBB1.5対応一価ワクチン (1ロット)
• ファイザー成人用一価ワクチン (3ロット)
• ファイザー成人用BA4/5対応二価ワクチン (1ロット)
解析方法は蛍光測定、qPCR、ナノポアによる三角測量です。蛍光光度系の解析では全てのバイアルで汚染DNA量は基準値を桁違いに超えていました。汚染DNA量はqPCRでは基準値を下回りましたが、これは1回の接種あたりの量ですので、2回以上の接種で即ち基準値をオーバーします。ファイザー、モデルナ、一価、二価、BA1、BA4/5対応といった種類に関係無く、DNA汚染はmRNAコロナワクチンに普遍的な問題である事が分かります。
今回はKevin McKernan先生のこのブログ記事とプレプリントの内容を紹介していきたいと思います。
蛍光光度系の原理

Qubit蛍光光度系は標的物質に結合した場合に強い蛍光を発する蛍光試薬を利用します (図1)。蛍光試薬を使い分ける事で、Qubitは二本鎖DNA、一本鎖DNA、RNA、タンパクの濃度を測定できます。例えば、Qubit二本鎖DNA検出キット (Quant-iT™ PicoGreen™ dsDNA Assay Kits and dsDNA Reagents) では蛍光試薬にピコグリーンを使用しています。使い方は簡単で、蛍光試薬と実験材料を混ぜてプラスチックの容器に入れ、Qubitにセットしてボタンを押すだけです。プロセスとしては、まずキットに付属の濃度既知のDNA (またはRNA、タンパク) を用いて、機械の内部で検量線を作成します。その後に実験材料を測定すると、瞬時に濃度を表示してくれます。
ゲノムシークエンサーでの実験にかかるコストはずいぶん下がってきたとは言え、今でも実験次第では一回機械を動かすあたりの消耗品代だけでも30万円〜50万円程かかる事も多いです。また、たとえそれだけのコストを払っても、スタート時点の実験材料の品質が悪ければ実験結果が「全く使い物にならない」場合もあるのです。ゲノム解析の現場での核酸定量においてQubit蛍光光度系は現在最も信頼され、普及している機器の一つです。
ナノポアシークエンサー

オックスフォード・ナノポア (ONT) はDNA (またはRNA) をナノポアに通過させ、特徴的な電流の乱れを読み取る事で塩基配列を決定します。PCR増幅も必要無く、長鎖の単一分子のDNAの塩基配列を解析できます。ナノポアシークエンサーは新世代ゲノムシークエンサーですが、その開発から実用化までには25年程かかっています。
ワクチン汚染DNAのリード (ディープシークエンサーで検出される塩基配列) の中央値は214 bpで、3.5 kbのリードも見つかりました (図2)。解析されたリード数は比較的少ないので (865リード)、シークエンシングの規模を拡大すればより大きなリードが見つかる可能性があります。

ゲノムシークエンサー用の高品質のDNAの調整にはSPRI、Ampureなどの磁気ビーズがよく用いられます。図3は異なるサイズのDNAのミックスをSPRI磁気ビーズによって精製したものです。SPRIはサンプルに加える試薬の量を変えるだけで、磁気ビーズに捕捉される断片のサイズ分布をコントロールする事ができます。こうした磁気ビーズによるDNAの精製技術は次世代シークエンサーなどのゲノム解析の現場で汎用されており、McKernan先生もその開発に携わっています。
イルミナなどのディープシークエンサーを使用する際には一度DNAを断片化し、塩基配列を決定した後に、コンピュータ解析によって断片DNAを繋ぎ合わせる作業が必要です。これに対し、ナノポアの長所は長鎖のDNAの塩基配列を断片化せずに直接決定できる事です。ナノポアシークエンサーを利用する事により、汚染DNAの配列だけではなく、長さも解析できます。ただし、この際には注意すべき点があります。それは実験におけるバイアスです。
ほとんどの研究者は長鎖のDNA配列を得るためにナノポアを利用しています。そのため長鎖のDNAを濃縮するのに適した0.7倍濃度の磁気ビーズを使用するのがデフォルトのプロトコルとなっているのですが、このプロトコルは150bp以下の断片を除くので、シークエンスの結果から短鎖のDNAが減少します。このように、ナノポアがDNAの配列長を解析できると言っても、結果にはDNA精製プロトコルのバイアスも反映されますので、注意が必要になります。Phillip Buckhaults博士は短鎖DNAの汚染を予期したため、ONTを実行する前に2.5倍濃度の磁気ビーズを使用しました。このため、彼が解析した平均リード長は比較的短くなっています。

FDAは200bp以下のDNAを懸念事項とはしていないようですが、これには疑問があります。例えば、DNA組換えのシグナル配列や転写因子の結合配列、スプライシング部位など、機能的な小さな配列は数多く存在します。図4で「7 bpという短いDNA断片が、組み込みや組換えの速度に影響を与える可能性がある」と記されているように、機能的な配列の最小単位は小さいのです。200bp以下なら「配列自体に意味が無い」という考えは生物学的に正しくありません。
汚染DNAと有害事象

DNA量と有害事象をグラフにすると、汚染DNA量が多いロットでは明らかに有害事象の総数が多くなる傾向が見えます (図5)。有害事象の発生が多く報告されているFN7934b、FM7380、FN7934aはファイザーのワクチンです。また、Buckhaults博士のロットやMcKernan先生が解析したFL8095、FL0007についても、汚染DNAが多く、有害事象の報告数が多いというデータが出ています。

有害事象のうち、重篤な有害事象の割合を計算するとやはり汚染DNA量と相関関係があります (図6)。ファイザーワクチンではスパイク、オリとも汚染DNA量が多いのですが、DNA量が多いほど重篤な有害事象が起きる割合も高くなります (オリ(Ori) はベクター内の配列)。また、モデルナではスパイクの汚染DNA量が多いのですが、同様にDNA量が多いほど重篤な有害事象の割合も高くなる傾向があります。
現時点で登録されている有害事象の多くは短期及び中期の副作用と考えられます。しかし、本来汚染DNAから予測されるのは長期の後遺症です。汚染DNA量と長期の後遺症の関係は今後明らかになってくるでしょう。
qPCRとQubit

図7の横軸はQubit、縦軸はqPCRによるDNA量です。QubitとqPCRでは数百倍〜千倍程度の違いが見られます。蛍光試薬による検出でもDNA、RNA間での干渉がありますが、それだけがDNA量の違いの原因でしょうか?
グラフの傾き (QubitとqPCRの量比) はファイザーとモデルナ、スパイクとオリでも異なります。ファイザーと比べて、モデルナのスパイクでは傾きがより緩やかになっており、モデルナのオリはqPCRの値が低いほどQubitの値が高くなっています。このようにQubitとqPCRの値は単純に比例しているわけではありません。モデルナの汚染DNAはqPCRでは少なく見えるのに対し、Qubitではより多く見えます。DNAがQubitでは検出しやすいのに対して、qPCRでは検出しにくくなっているのです。では、これにはどういった理由が考えられるでしょうか?
qPCRには25bpずつの2つのプライマーと25bpのプローブが必要ですので、75bp以下のアンプリコンを得るのは難しく、実際にはアンプリコンは100bp以上となります。しかしQubitはqPCRのようなサイズ制限がありません。実際モデルナワクチンではDNAの分解が進み、小さな断片になっている可能性があります。そして、それらのDNAはワクチン内に残されており、Qubitで検出されているのではないでしょうか。そうした場合、qPCRでは汚染DNA量が低い値を示しても、短い断片のDNA数は莫大に増えています。つまり、DNase Iによる分解をより丁寧に行った結果「散弾銃」の装弾数を増やしてしまった事になります。
なぜ膨大な汚染DNAが混入したのか?: 1つの仮説
mRNAの鋳型に使われたDNAがDNase I分解耐性になるのは、シュードウリジン化RNAがDNAに強固に結合して、DNAを分解から保護するためです。この強固な結合の理由はシュードウリジン化RNA自体の性質に加えて、スパイク遺伝子の配列においてGとCの含有量が高いからです。このRNAとDNAの強い結合は様々な局面にも影響するでしょう。
さて、ここから先は私自身の仮説となります。T7 RNAポリメラーゼによる転写は本来効率が良く、1つの鋳型DNAからたくさんのmRNAを転写します。ポリメラーゼがRNAを転写する際には、以前に合成したmRNAを鋳型から剥がさなければいけません。DNAから合成したmRNAが鋳型に強固に結合していた場合、次の転写を止めるようなブレーキの働きをします。mRNAの合成量が少ないならば、mRNAに対する鋳型DNAの比率も元々高かった事になります。また、途中で転写が止まった場合には中途半端なmRNAを生じます。DNase Iによる分解も不十分であれば、DNA断片は相補的なmRNAにまた強く結合してしまいます。その場合、mRNAを精製する際に小さなDNA断片だけを取り除こうとしても、mRNAにトラップされたDNAを除去する事は不可能です。
整理すると、シュードウリジン化RNAとDNAの強固な結合は、1) mRNA合成を低下させ、2) mRNA品質を低下させ、3) DNaseIによるDNA分解を阻害し、4) RNA精製の際のDNA除去を阻害し、5) 細胞内 (細胞質) での汚染DNAの分解を阻害し得ます。言い換えると、シュードウリジン化RNAはDNAと強固に結びつき過ぎているために、mRNA合成もうまくいかず、DNA分解もうまくいかず、DNA除去もうまくいかず、細胞内に取り込まれても危険なままです。つまる所、DNA汚染はLNP/mRNA製剤における根本的な技術的欠陥を示しているという事です。
qPCRとQubitの特異性の意味の違い
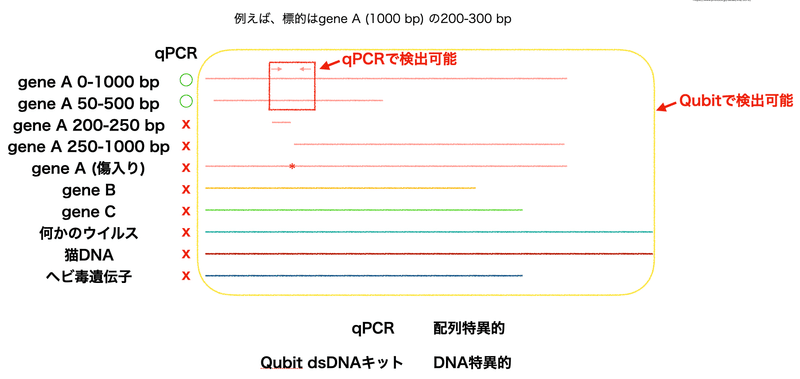
図8は私が描いたものです。QubitはどのDNAもまとめて定量できますが、qPCRで定量できるのはプライマーで増幅できる遺伝子のみです。
QubitとqPCRは測定原理が大きく異なります。最も大きな違いはその特異性です。Qubitの特異性は二本鎖DNA、一本鎖DNA、RNAなどという異なったタイプの核酸です。それに対して、qPCRや一般のPCRの特異性は遺伝子の配列に対してのものです。
qPCRは特定のDNAを増幅し、増幅のしやすさでDNA量を推定する技術です。研究の現場ではqPCRは多くの種類の遺伝子の中から特定の遺伝子だけを定量するために使われる事が多いです。例えば、RT-qPCRにより多くの遺伝子の中で、興味の対象となる遺伝子の発現レベルはどのくらいか。染色体免疫沈降qPCRで、興味の対象となる遺伝子配列はどのくらい濃縮できたかなど。コロナ騒動において市中のあちこちで行われていたPCR検査ですら、細胞内の多くのRNAの中からコロナウイルスのRNAだけを限定的に増やすための技術です。
qPCRは含まれている総DNA量を測る目的には通常使われません。qPCRでDNAの総量を測定するためには、含まれているDNAが一種類であり、しかも傷の付いていない全長のDNAだけであるという前提が必要となります。傷の付いたDNAや断片化されたDNAのような増幅できないDNAは定量できません。また、もしもコロナワクチンとは無関係なDNAが混入していた場合、コロナワクチン用のqPCRでは一切検出できません。こうした技術的限界が、qPCRが汚染DNAの定量に本来不向きな理由なのです。
ファクトチェックは正しかったか?
彼らはまず、DNAは腕から離れないと主張した (血漿、母乳、心臓組織はそうでないと言っている)。さらにDNAはないと主張した。
再現された時、彼らはそれが細胞内に入らなかったと主張した。
LNPの中にある事が示されても、核には到達していないと主張した。
SV40エンハンサーが数時間でDNAを核内に移動させる事が示されると、彼らはそれは問題ではないと主張した。
彼らの巧みな後出しジャンケンは、次はどこへ向かうのだろうか?
McKernan先生はいわゆる「ファクトチェック」が誤りを繰り返してきた過程を指摘しています。医学において人命に関わる未知のリスクを楽観論で語るべきではないのです。
#コロナワクチン
#ワクチン
#コロナ
*記事は個人の見解であり、所属組織を代表するものではありません。
この記事が気に入ったらサポートをしてみませんか?
