UMAP(Uniform Manifold Approximation and Projection)
UMAPとは?
UMAP(Uniform Manifold Approximation and Projection)は、高次元データの次元削減のための機械学習の手法です。特に、大規模なデータセットに対して高速に実行でき、データの構造を維持しながら次元を減らすことが可能です。同じ次元削減のための手法であるt-SNEと比較して、UMAPはより速い計算時間とより良い大域的構造の維持が特徴です。
インストール
pip install umap-learnデータセット
データセットは下記を利用します。
このデータセットは、キノコが食用か有毒かを識別するための特徴が含まれているデータセットです。約8124個のキノコのサンプルがあり、それぞれのキノコが食用(edible)または有毒(poisonous)のいずれかとしてラベル付けされています。
キノコの各サンプルには以下のような特徴が含まれます:
キャップ(cap): 形状(bell, conical, convexなど)、表面(smooth, scalyなど)、色(brown, pink, whiteなど)
ひだ(gills): 配置(close, crowdedなど)、色(black, brownなど)
柄(stalk): 形状、根元の形状(bulbous, rootedなど)、表面の質感(silky, smoothなど)
匂い: almond, anise, creosoteなど
生息環境: grasses, leaves, urbanなど
その他: ひだの下に輪やベールの有無など
データセットの読み込み
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
df= pd.read_csv('./mushrooms.csv')
df.head(5)
データの前処理
X = df.drop('class', axis=1)
y = df['class']
y = y.map({'p': '有毒', 'e': '食用'})目的変数の設定をしています。また分かりやすいように有毒と食用でラベルを変更しました。
cat_cols = X.select_dtypes(include='object').columns.tolist()
for col in cat_cols:
X[col] = X[col].astype('category')
X[col] = X[col].cat.codesカテゴリカルデータに対する前処理を行っています。
UMAPの適用
UMAPを適用していきます。
from sklearn.preprocessing import StandardScaler
import umap
fit = umap.UMAP()
X_std = StandardScaler().fit_transform(X)
X_u = fit.fit_transform(X_std)
X_u_data = np.vstack((X_u.T, y)).T
df = pd.DataFrame(X_u_data, columns=['Dim1', 'Dim2', 'class'])
df.head()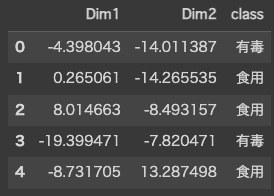
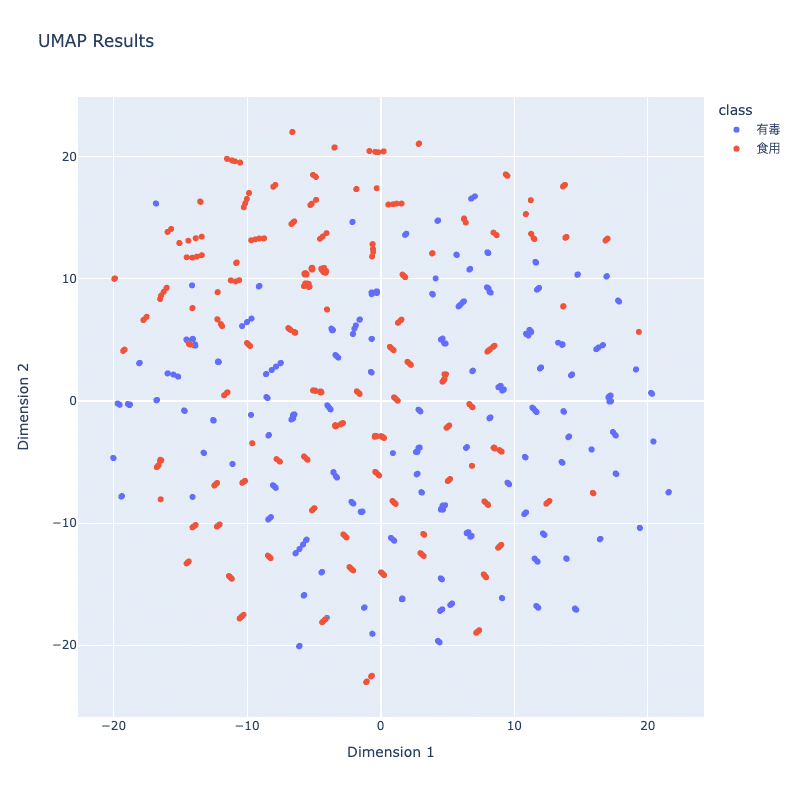
UMAPの可視化
import plotly.express as px
fig = px.scatter(df, x='Dim1', y='Dim2', color='class',
title='UMAP Results',
labels={'Dim1': 'Dimension 1', 'Dim2': 'Dimension 2'},
width=800, height=800)
fig.show()