
【DataCamp】29_Unsupervised Learning in Python①, ②
教師なし学習の前半です。
①Clustering for dataset exploration
KMeans法でクラスタリングする。ラベルがあるものはクロス集計表を作成し、精度を確認する。
-アヤメ

まずはアヤメのデータをインポート。微妙にスライドと値が違います…。

がくの長さと花びらの長さの散布図を作成し、クラスターごとに色分けします。

がくの長さは1列目、花びらの長さは3列目です。

あれ?軸ラベルが表示されない…。
scatter plotの軸ラベルがどうやっても表示されなくて、このサイト読んで、jupyter notebookをリセットしたら直りました😀matplotlibのエラー 'str' object is not callable - ましろのログ https://t.co/yeqG3fxwTk
— るじパルコ (@ruji_paruco) November 4, 2021
jupyter notebookをリセットしたら直りました!

クラスタリングの評価に、クロス集計を行います。
target_namesの0, 1, 2をそれぞれ'setosa' 'versicolor' 'virginica'に変換します。for文を使ったけれど、もっと簡単な書き方がありそう…。



スライドと順序は異なりますが、数は同じでした。
-grains
k-meansの慣性グラフ作成
今度はgrains。GrainsZIPを解凍すると、seeds, seeds-width-vs-lengthの2つのファイルがあります。今回はseedsの方を使います。seedsファイルを開くと、210行8列でした。最後の1列はラベルのようです。
DataCampの方で、samplesは210行7列であることを確認しました。
![]()

samplesの出力は、DataCampと同じみたいです。

inertiaの減少は、3から緩やかになるので、クラスターの数は3が良さそうです。
クラスター数3で、クロス集計表を作成してみましょう。しかし、ラベルの1, 2, 3が何を指すかよく分かりません。元データを見てみましょう。
UCI Machine Learning Repository seeds Data Set
Data Folder > Parent Directory, seeds_dataset.txt
Parent Directoryは上のディレクトリに移動するだけ。
seeds_dataset.txtの方は、コーストップにあるgrainsファイル内のseedsと同じ。
ラベルの説明は特にないけど、 Kama, Rosa and Canadianの順に1, 2, 3なのかな???


並びはDataCampと同じっぽいです。
クラスター数3で、クロス集計表を作成しました。

-ピエモンテ ワイン
このデータセットはヘッダーがあって、それぞれの列が何か分かりやすいです。

samplesとvarietiesを作成。ラベルの名前列があるのでラク…。

クラスター数3で、クロス集計表を作成しました。

イマイチ。データセットの特徴量の分散が大きく異なるのが原因。

分散の大きさの違いを見るため、od280とprolineの散布図を作成してみましょう。

![]()
あれ?エラー…。

![]()
今までのグラフを見直して、cで渡すのは、数値のリストでなければならないと気づきました。文字列のリストを渡すとエラーになります。

x軸とy軸のスケールを調整して

これを標準化します。27_Supervised Learning with scikit-learnの④でも出てきたStandScaler。

ワインのデータセットをクラスター化するには、StandardScalerで標準化して、KMeansする2ステップが必要。pipelineを使うと便利。
StandardScalerとKMeansオブジェクトを作成し、make_pipeline()関数を適用する。pipelineのfit_predictメソッドで、クラスターラベルを取得する。クロス集計表を作成する。

今度はクラスターラベルと品種がよく対応しています。
Pipelineとmake_pipelineの違いって何?→make_pipelineは各ステップに名前をつけなくてよい
— るじパルコ (@ruji_paruco) November 4, 2021
-fish
今度はfish.csvをインポートします。85行7列。headerがないので、それぞれの列が何を表しているか分かりません。元データを確認します。
Journal of Statistics Education
スクロールしていくと、fishcatch.dat.txtとfishcatch.txtがありました。fishcatch.dat.txtは159行9列のデータ。fish.csvと違い、1列目は数字です。
fishcatch.txtの方に説明があります。1:観察番号、2:種類、3:重量、4~6:長さ、7:高さ%、8:幅%。fish.csvには1:観察番号がありません。2:種類ですが、

fish.csvはコードをSpeciesのEnglish ver.にしてありますね。Bream, Roach, Smelt, Pikeのみ。Whitewish, ?, Perchはナシ。
DataCampのsamples.shapeは(85, 6)。fish.csvの0列目の種類を削除してsamplesにします。

まず、各列の分散を調べます。

標準化せずにクラスター化して、クロス集計表を作成すると

Breamが0と3にばらけて、RoachとSmeltはどちらもラベル1…。
StandardScalerで標準化して、KMeansでクラスター化。

クロス集計表を作成し、クラスターラベルと品種を比較。

よく対応しています。
-company-stock-movements
次は株価の動きです。終値と始値の差(ドル)。DataCampのmovementsは60行963列のnumpy array。


![]()
あれ?エラー…。
df.drop(df.columns[列番号], axis=1)でいけました。grainsはdf.drop(列番号, axis=1)でいけたのに、なぜ???


行ごとの平均値を見てみます。

分かりにくいので、棒グラフにしてみましょう。


一番小さいのはAppleで-0.3。一番大きいのはMasterCardの0.25。
Normalizerを用いて正規化します。
class sklearn.preprocessing.Normalizer(norm='l2', *, copy=True)
L2正規化がデフォルト。
https://qiita.com/panda531/items/4ca6f7e078b749cf75e8
L2正規化は、半径1の円上へのベクトルに変換しているらしいです。(StandardScalerは標準化。元のデータの平均を0、標準偏差が1のものへと変換する)


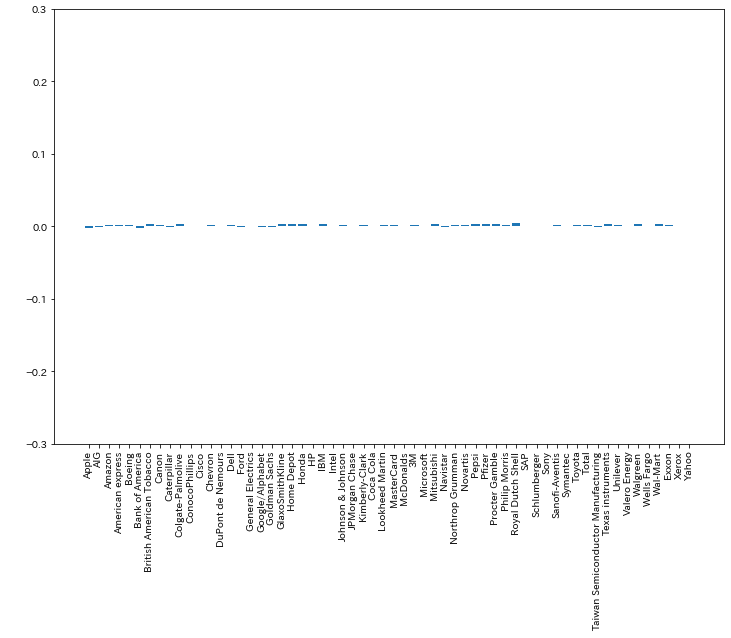
最大値と最小値の比較


10個のクラスターに分けてラベルを付けます。


②Visualization with hierarchical clustering and t-SNE
eurovisionはよく分からないのでパス。NaNあるし。
-grains
再び。
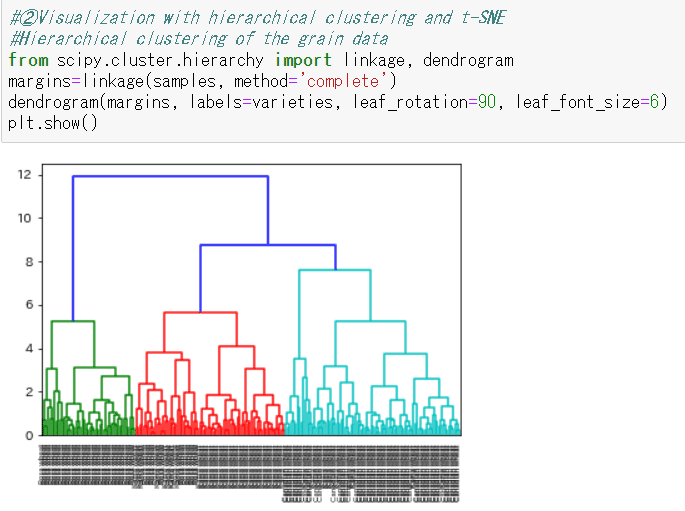
なんか気持ち悪い感じになっちゃった…と思ったら、DataCampの方のsamples、(42, 7)にデータ減らしていた…。
大体、品種ごとにクラスターに分かれています。
次はクラスターが3つになっている高さ8でクラスターラベルを抽出します。

少し混ざってますが…。
-stocks
scipy.cluster.hierarchyはpipelineに対応していない。

この記事が気に入ったらサポートをしてみませんか?
