【和訳】SARS-CoV-2の遺伝子構造は実験室由来を否定するものではない SARS-COV-2のキメラ構造とfurin切断部位は遺伝子操作の結果である可能性 (2020/09/17)
2020/09/17?の論文 これもDeepL翻訳。
(今さらですが、読みたかったので)
SARS-CoV-2の遺伝子構造は実験室由来を否定するものではない
SARS-COV-2のキメラ構造とfurin切断部位は遺伝子操作の結果である可能性
ロサナ・セグレト、ユーリ・デイギン
関連データ
データの利用可能性に関する声明
今回の研究では、データセットの作成・解析が行われていないため、データ共有の対象とはなりません。
要旨
重症急性呼吸器症候群コロナウイルス(SARS-CoV)-2′sの起源についてはまだ議論の余地がある。ゲノム解析の結果、SARS-CoV-2はキメラである可能性が高く、その配列のほとんどはコウモリのCoV RaTG13に最も近く、一方、その受容体結合ドメイン(RBD)はセンザンコウのCoVとほぼ同じであることが判明した。キメラウイルスは、自然の組み換えや人為的な介入によって発生する可能性がある。SARS-CoV-2のスパイクタンパク質にあるfurin切断部位は、このウイルスに種や組織の壁を越える能力を与えるが、これまで他のSARS様CoVには見られなかったものである。もともとヒトの受容体に結合できないコウモリ由来のCoVの中間宿主としてパンゴリンを評価するために、遺伝子操作が行われたのかもしれない。切断部位も特異的RBDも、痕跡を残さない部位特異的変異誘発によって生じた可能性がある。SARS-CoV-2の壊滅的な影響と将来のパンデミックを防ぐことの重要性を考慮すると、研究者にはSARS-CoV-2の起源と考えられるものをすべて徹底的に分析する責任があると思われる。
キーワード BtCov/4991、furin切断部位、機能獲得研究、パンゴリンCoV、RaTG13、受容体結合ドメイン、SARS-CoV-2
概要
SARS-CoV-2のヒト細胞への完全な結合能と、SARS様コロナウイルスとしては新しいfurin切断部位の存在は、進化研究の過程で行われた遺伝子操作に由来するものである可能性がある。コウモリコロナウイルスの骨格とセンザンコウの受容体結合部位を組み合わせたキメラは、完全に自然なものに見えるだろう。
写真やイラストなどを保存しておく外部ファイル。
オブジェクト名は BIES-9999-2000240-g004.jpg です。
はじめに
中国武漢で発生した重症急性呼吸器症候群コロナウイルス2型(SARS-CoV-2)の発生から1年近くが経過したが、その起源についてはいまだに議論の余地がある。国際的な研究努力にもかかわらず、直接または中間的な自然宿主はまだ特定されていない。武漢華南海鮮卸売市場が動物-ヒトウイルス感染の最初の発生源であるという仮説は、現在では決定的に否定されておりi、採取された少数の市場サンプルはヒトに適応したSARS-CoV-2のみを示し、動物由来前駆株iiの痕跡はなかった。現在までに発表されたほとんどすべての科学論文は、SARS-CoV-2が自然起源であると主張しており、実験室起源の可能性を考慮した唯一の論文[ 1 ]は、SARS-CoV-2のヒト細胞への特殊適応を可能にする技術として、連続継代に焦点をあてている。ここでは、SARS-CoV-2の2つの大きな特徴である、(1)同じグループの他のCoVにはないフリン切断部位の存在、(2)ヒト細胞に結合するために最適化された受容体結合ドメイン(RBD)[ 2 ] が、部位特異的突然変異誘発などの実験操作技術の結果である可能性について説明します。SARS-CoV-2がこの2つの特徴をほぼ同時に獲得したことは、自然現象や細胞・動物の連続継代によってのみ引き起こされるとは考えにくい。
SARS-COV-2の最も近い親戚はコウモリとセンザンコウのコロナウイルスである。
武漢ウイルス研究所(WIV)のZhouら[ 3 ]は、新しいコロナウイルス(CoV)であるSARS-CoV-2を最初に特定し、その特徴を明らかにしました。初期の症例から得られたゲノム配列は、2002-2003年に重症急性呼吸器症候群(SARS-CoV)を引き起こしたCoVと79%の配列同一性を持ち、Rhinolophus affinisコウモリから検出されたCoV配列、RaTG13(MN996532)とは96.2%の配列同一性を有していました。RaTG13は現在見つかっているSARS-CoV-2の系統的近縁種です[ 4 ]が、その完全なゲノム配列はSARS-CoV-2の発生前に発表されておらず、元のサンプルは2013年に同じWIV研究者のグループによって雲南省(中国)で収集されました。Zhouら[ 3 ]は、SARS-CoV-2とCoVのRNA依存性RNAポリメラーゼ(RdRp)の短い領域が彼らのデータベースで一致すると述べ、その後、2013年に収集したオリジナルサンプル(彼らはRaTG13と呼んだ)の全塩基配列を決定しました。
その結果、RaTG13のRdRpは、RaTG13と同じ場所・年の2013年に雲南省のRhinolophus affinisコウモリでGeら[ 5 ]が同定した配列BtCoV/4991 (KP876546) と塩基が100%一致することが判明しました。BtCoV/4991は、雲南省莫江市Tongguanzhen付近のコウモリがコロニーを作る鉱山で採取されたものである。WIVの研究者たちは、2012年に6人の鉱山労働者が重度の肺炎にかかり、そのうち3人が死亡したことから、この鉱山に調査を依頼した[ 6 ]。鉱山労働者は、鉱山内のコウモリの糞を取り除く仕事をしており、彼らの肺炎の重症度は鉱山にさらされた期間と相関があった。 [7 ] その後、4人の鉱山労働者のサンプルがWIVで検査され、すべてのサンプルからSARSに対する免疫グロブリンG(IgG)抗体が検出された[ 8 ] 2002-2004年のSARS発生時に中国本土で感染したのは約5300人で、ほとんどが広東省に住んでいたことを考えると、雲南の4人の鉱山労働者が2002-2004年に発生したSARSの抗体を保持している確率はごく僅かである。一方、鉱山労働者に実施したSARS抗体検査が、鉱山労働者が鉱山で獲得したSARS様新型コウモリウイルスと交差反応した可能性もある。Geら[ 5 ]は鉱山で多くのCoVを同定したが,系統解析によれば,BtCoV/4991が唯一のSARS関連株であり,当時知られていたすべてのαおよびβCoVから明確に分離されていた。BtCoV/4991は、2019年にWangらが行った系統解析でも他のコウモリCoVとは異なっていた[ 9 ] Chenら[ 10 ]は、当時RaTG13がまだ発表されていなかったため、BtCoV/4991をSARS-CoV-2に最も近い配列として特定した。BtCoV/4991とRaTG13は、WIVの原著者が2つの株をDatabase of Bat-associated Viruses (DBatVir) に1つの項目として登録したため、後に同一株の異なるコード名であると主張されている[iv]。
2020年7月末、WIVのCoV研究の第一人者であるZhengli Shiは、メールインタビュー [ 11 ] でRaTG13サンプルの名称変更を主張し、Zhou et al.にあるように、RaTG13のフルシーケンスはSARS-CoV-2の発生後ではなく2018年にさかのぼったと意外にも宣言しています。 [ 3 ] RaTG13のフルシークエンスが正確にいつ行われたかについてのWIVのスタンスの逆転は、SARS-CoV-2の起源に関する独立研究者が、2020年5月19日にWIVが寄託した生のシーケンスリードのファイル名から、2017年と2018年にRaTG13に対するシーケンスが行われたようだと発見したからかもしれませんv。 vi しかし、Zhouら[ 3 ]の著者からのシーケンスの年やサンプル名の変更に関する正式なエラッタムはまだ出ていませんし、現在知られている限りでは提出されていません。
BtCoV/4991に最も近い2番目の非ヒトRdRp配列(91.89%のヌクレオチド同一性)は、2019年に中国広東省(GD)のマレーシアのセンザンコウ(Manis javanica)で分離されたCoV配列MP789(MT084071)である[12] MP789のエンベロープタンパク質は驚くべきことにRaTG13の対応タンパク質とアミノ酸同一性の100%を示し、コウモリ- SL-CoVZXC21 (MG772934である。 1)、コウモリSL-CoVZC45 (MG772933.1) 、SARS-CoV-2の初期分離株 (例: YP_009724392) の対応するタンパク質と100%のアミノ酸配列を有している[ 13 ] CoVのエンベロープタンパク質は、ウイルスの侵入、複製、病原性といった、ウイルスライフサイクルの重要な局面に関与する[ 14 ] CoVは、ウイルスが増殖する際に、その増殖に必要なタンパク質を供給する。
コウモリのCOVは徹底的に研究され、遺伝的に操作されている
多くの研究により、コウモリは病原性を持つ多様なSARS様CoVの天然リザーバーであることが報告されています[ 15 , 16 ] これらのウイルスの中には、ヒトに直接感染する可能性のあるものもあれば[ 17 ]、ヒトアンジオテンシン1変換酵素2(hACE2)受容体に効果的に結合してウイルス侵入を仲介するためにスパイクタンパク質を変異させる必要性のあるものもあります。 [新規CoVの出現可能性を評価するため、研究者は、通常はヒト細胞に感染しないコウモリCoVのスパイクタンパク質を、ヒトACE2に適合するCoVのものに置き換えたキメラCoVを多数作製した。これらのキメラは、自然界で起こりうる組換え現象を模擬したものである。機能獲得実験に賛成する主な論拠の1つは、次のパンデミックに備えて薬剤やワクチンを準備しておく必要があることである[ 21 ]。 対照的に、反対する主な論拠の1つは、実験室から逃げ出す危険性があるため、次のパンデミック自体がこれらの実験によって引き起こされる可能性があることである[ 22 , 23 ]。
近年、 冠動脈ウイルス学の分野は、過去 5 年間に行われた研究や メディアの報道から明らかなように、汎 CoV 治療とワクチンに 焦点を当てていました[ 24 , 25 , 26 , 27 ]。
新しいRBDを持つキメラCovsの作成は数十年前から行われていた
研究者たちは、現代のシークエンスや遺伝子工学の技術が出現するずっと以前から、20年以上にわたってキメラCoVを生成してきた。例えば、1999年、ユトレヒト大学のグループは、標的RNA組み換えを用いて、「ネコとネズミ」のCoVキメラを作りました。ネコとネズミのCoVのRBDを交換し、この交換によって、in vitro実験中に種の向性も交換されることを実証しました[ 28 ]。
2007年、WIVのShiグループは、CoVにある種から別の種へジャンプする能力を与えるものが一体何であるかを明らかにしようと、一連の「コウモリ男」CoVキメラスパイクタンパク質を作成した。研究者らは、ヒトSARSウイルスのスパイクタンパク質の異なるセグメントを、コウモリウイルスのスパイクタンパク質の対応するセグメントと置き換えることに成功した。その結果、スパイクタンパク質の比較的短い領域(aa 310〜518)が、「Rp3-SをhuACE2結合分子に変換するのに必要かつ十分である」、すなわちコウモリCoVスパイクタンパク質にヒトACE2受容体と結合するという新しい能力を与えることが判明したのだ。
2008年、ノースカロライナ大学(UNC)のBaricグループは、WIV研究をさらに一歩進め、コウモリCoVスパイクタンパク質にヒト免疫不全ウイルス(HIV)の疑似ウイルスを用いる代わりに、生きたキメラCoVを作成した。2007年のWIVの仲間の実験に続き、BaricグループはコウモリのSARS様CoVをバックボーンとして使用し、そのRBDをヒトSARSのRBDと置き換えた[ 30 ]。
2015年、 ShiグループとBaricグループが手を組み、おそらく最も有名な機能獲得型ウイルス学の論文を発表し、別の合成キメラウイルスの作成を説明しました[ 19 ] 今回、マウスに適応したSARSバックボーン(SARS-MA15)のRBDは、2011年にShiグループが雲南省のコウモリから以前に分離したRsSHC014のRBDと置き換えました。2016年、Baricグループは同じSARS-MA15バックボーンと、同じくWIVによって以前雲南で発見され、生培養後に「WIV1」と改名されたRsSHC014の近縁種、[ 31 ]のRBDを用いて2015年の実験を繰り返しました[ 17 ]。
おそらく、作成された新規キメラウイルスの報告数が最も多いのは、WIVのShiグループによる2017年の論文で、[ 15 ]この論文では、WIV1をバックボーンとして、コウモリSARS様ウイルスの様々なRBDをそこに移植して8個のキメラウイルスを作成したことが報告されています。これらのウイルスは、ShiグループがRs3367とRsSHC014を最初に発見した雲南省昆明近郊の同じ洞窟から5年間かけて収集されたものである。8個の生きたキメラウイルスのうち2個だけを救い出すことに成功し、その2株はヒトACE2受容体に結合する能力を持つことが、hACE2発現HeLa細胞での実験とウイルスRNAのRT-PCR定量化で確認された。
SARS-COV-2はセンザンコウのCOVとRBDを共有している
SARS-CoV-2の中間宿主がパンゴリンである可能性は、以前から議論されていた。SARS-CoV-2とRaTG13の最大の相違点は、スパイクタンパク質のRBDにある[ 4 ]。 ゲノム全体の類似度はRaTG13より低いが、GD pangolinsから分離したMP789 pangolin株はSARS-CoV-2とほぼ同じRBDを持っている[ 3 ]。実際、センザンコウとSARS-CoV-2は、RBDの5つの重要な残基に同一のアミノ酸を有していますが、RaTG13はSARS-CoV-2と1つのアミノ酸しか共有していません[ 35 ] ACE2の配列類似性は、ヒトとコウモリの間よりもパンゴリンの間で高いことが知られています。興味深いことに、SARS-CoV-2のスパイクタンパク質は、ヒトのACE2受容体への結合親和性が、パンゴリンやコウモリのそれよりも高いと予測されている。 SARS-CoV-2が発生する前、コウモリ以外の哺乳類でSARS-CoV-2関連のCoVを保有し感染しているのはパンゴリンのみであった[ 12 ]。パンゴリン由来のCoVのRBDとRaTG13様のバックボーンが組み合わされて、キメラ株としてSARS-CoV-2を作り出した可能性がある。このような組換えが自然に起こるには、2つのウイルスが同じ生物の同じ細胞に同時に感染したことが必要であるが、パンゴリンの人口密度が低く、自然集団にCoVがほとんど存在しないことを考えると、これはかなりありえない出来事であるx。さらに、再構成したRaTG13の受容体結合研究では、それがパンゴリンのACE2とは結合しないことが示されたxi。
フリン切断部位:SARS-COV-2とその近縁種RATG13の重要な違い
SARS-CoV-2は、その近縁種であるRaTG13といくつかの重要な特徴を持っている。最も顕著な違いは、SARS-CoV-2のスパイクタンパク質に、これまでb系統の他のβ-CoVでは確認されていなかった[ 36 ]、中東呼吸器症候群(MERS)コロナウイルスと同様の宿主細胞酵素フリンによる切断部位があることである[ 35 ] 宿主タンパク質分解酵素は種や組織の障壁として重要な役割を担い、CoVスパイクタンパク質の切断部位を工夫することによりウイルス向性と病原性が変更されてしまうのである。 SARS-CoV-2は、臓器や組織でfurinが普遍的に発現しているため、通常他のCoVには感染しない臓器にも感染し、全身感染する能力を有している[ 37 ]。 [38 ] 上記切断部位を欠損した細胞培養SARS-CoV-2は、感染したハムスターの症状を軽減させ、 [ 39 ] 突然変異誘発研究により、SARS-CoV-2のヒト肺細胞への感染能に多塩基性furin部位が必須であることが確認されている [ 40 ]。
SARS-CoV-2のpolybasic furin部位は、S1/S2接合部にPRRAアミノ酸配列をコードする12ヌクレオチド挿入体TCCTCGGCGGCによって作られた(図1)。興味深いことに、2つの結合アルギニンは、これらのウイルスではまれな2つのCGGCGGコドンによってコードされている:SARS-CoV-2またはRaTG13ではアルギニンのわずか5%がCGGによってコードされ、新しい挿入物のCGGCGはSARS-CoV-2でこのコドンの唯一の2倍の例である。CGGCGGの挿入部分にはFauI制限部位があり、SARS-CoV-2には6箇所、RaTG13には4箇所ある(MP789には2箇所ある)。FauI部位がセレンディピティに配置されていることから、制限酵素切断長多型(RFLP)技術[ 41 ]を用いたクローニング[ 42 ]や変異のスクリーニングが可能になると考えられるが、新しいfurin部位はin vitroでは欠損しやすい[ 39 , 44 ]ため。
図1
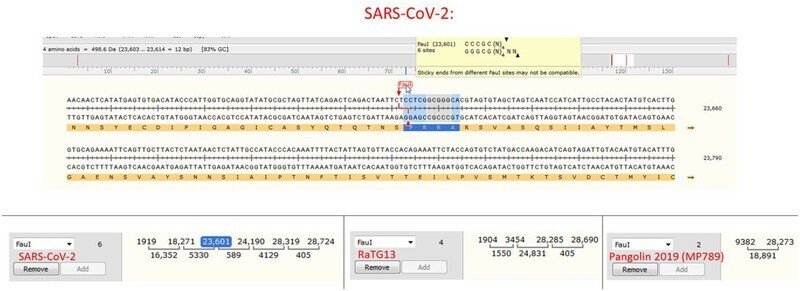
SARS-CoV-2(NC045512.2)のS1/S2接合部のSタンパク質の塩基配列。FauI酵素の制限部位を含むfurin切断部位(青色)を示す。
Zhouらによる研究[ 45 ]は、新しいCoV株RmYN02の発見を報告し、著者らは、SARS-CoV-2がPRRA挿入を持つS1/S2切断部位に天然のPAAアミノ酸挿入を示すと主張した。しかし、RmYN02の基本的なヌクレオチド配列を、その最も近い祖先であるbat-SL-CoVZC45およびbat-SL-CoVZXC21と比較して詳細に調べたところ、挿入は見られず、ヌクレオチドが変異しているだけであった(図2)。
図2
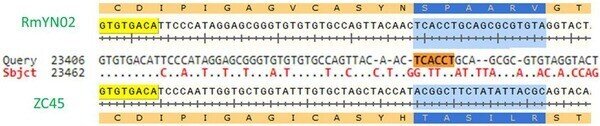
bat-SL-CoVZC45 (MG772933.1) とRmYN02のSタンパク質のS1/S2接合部における塩基配列とアミノ酸配列のアラインメント...。
したがって、SARS-CoV-2は、S1/S2接合部の多塩基性フリン部位だけでなく、それを生み出した4アミノ酸の挿入物PRRAによって、ベータCoVの親族の中でユニークな存在であり続けているのだ。この挿入物によって、MP789やRaTG13のセリンに対するオリジナルのコドン(TCA)が分裂し、SARS-CoV-2のセリンに対する新しいコドン(TCT)の一部とアミノ酸アラニン(GCA)の一部を与えることになる(図3)。
図3

RaTG13(MN996532)、MP789(...)由来のSタンパク質の塩基配列とアミノ酸配列のアラインメント。
SARS-CoV-2のfurin切断部位の挿入は、MP789やRaTG13の配列と比較すると、残りの配列とフレームがずれている(図3)。SARS-CoV-2のスパイクタンパク質は、他のCoVの組換えモデルとは異なり、それ以上の組換え事象がないように見えるため、Furin切断部位が組換えによって獲得されたという可能性は、最近Seyranらによって疑問視されている[ 47 ]。
SARS-COV-2の近接起源」に対する批判
動物からヒトへ伝播する可能性を正当化するコウモリのSARS-CoVに関する約20年にわたる広範な研究[ 48 ]から、SARS-CoV-2の実験室工学による合成由来の可能性を排除することは不可能である。広く引用されているAndersenらの論文[ 2 ]は、SARS-CoV-2が自然起源である可能性が最も高いと述べている。著者らの主な主張は、SARS-CoV-2のスパイクタンパク質とhACE2の高親和性結合は、SARS-CoVのRBDに基づくモデルでは予測できなかったというものである。Wanらの構造解析によると、[ 49 ] SARS-CoV-2は2002年に出現したSARS-CoVよりも効率的にhACE2を認識する可能性がある。さらに、CoVキメラ株の作製により、コウモリCoVのスパイクが従来の予測よりも可塑的にhACE2受容体に結合することが最近明らかになった[ 15 ] RBDのすべてのアミノ酸が広範囲に解析され、ACE2親和性の新しい予測モデルが利用可能となった[ 50 ] この点について、BatCoV Rs3367(WIV1との同一性99.9%)はRBD内の6つの重要残基中4つをSARS-CoV-2と共有していることが明らかになった[ 51 ]。WIV1がhACE2と直接結合することが示されたことを考慮すると、SARS-CoV-2のRBDについても同じ仮定が容易にできたと思われる[ 51 ]。
このように、コウモリCoVのヒトに対する病原性を研究する目的で、キメラウイルスの作製が長年行われてきた。そこで,SARS-CoV-2は,RaTG13に似た骨格と,最近パンゴリンから分離されたCoVのRBDを組み合わせて合成されたと考えられる[ 12 ]後者は,hACE2受容体に高い親和性を持っているという特徴があるからである。このような研究は、ヒトに病原性を持つ可能性のあるコウモリCoVの中間宿主として、パンゴリンを特定することを目的としていたかもしれません。Sirotkin & Sirotkin [ 1 ]が述べたように、細胞や動物の連続継代により、RBDをhACE2に完全に適合させることができたかもしれません。
furin切断部位に関して、Andersenら[2]は、「SARS-CoV-2における多塩基性切断部位の機能的な結果は不明である」と述べている。最近、いくつかのグループによる新しい研究により、この活性化部位が、ウイルスがヒトの間で効率的に広がり、複数の臓器を攻撃することを可能にする可能性があることが明らかになった[ 52 ]。 CoVスパイクタンパク質のタンパク質分解切断に関する実験が、異なる宿主におけるウイルス感染性を理解するための将来の重要な研究として最近示唆されている[ 50 ]。
Andersenら[ 2 ]も、Almazanら[ 53 ]の研究に基づいて、"遺伝子データは、SARS-CoV-2が以前使われていたどのウイルスバックボーンにも由来しないことを反駁可能に示している "と述べています。SARS-CoV-2の発生前の過去6年間、いくつかのコウモリのCoVスクリーニングによって、潜在的なコウモリのバックボーンの数は紛れもなく増加し、最後に、2020年1月にRaTG13が科学的に注目されるようになった。他の可能性のあるバックボーンも同様に、まだ公表を待っている可能性がある。
Andersenら[ 2 ]は、"多塩基性切断部位と予測されるO-結合型糖鎖の両方の獲得は、培養ベースのシナリオに対しても論証する "と断言している。感染性気管支炎CoVにおける多塩基性切断部位の挿入方法は、Chengら[ 54 ]に示されており、病原性の増加をもたらした。新しく挿入された多塩基性部位周辺の予測されたO-結合型糖鎖に関して、この予測はSARS-CoV-2スパイク糖タンパク質のCryo-EM調査によって確認されていないことに注意すべきである。 [しかしながら、O-結合型糖鎖は、免疫淘汰の下で生じる可能性が高いのは事実であるが、実験室で部位特異的突然変異誘発によって付加することもできるし[ 56 ]、例えば、ヒト肺移植と自己ヒト免疫システムを持つBLT-LマウスやhACE2受容体の発現マウスなど、in vivo実験の過程で生じる可能性がある[57 ]。 [コウモリCoVの分離の問題を克服するために、乳飲み子ラットにコウモリCoVを直接接種する実験が実施されている[ 31 ]。 ヒト化マウス、フェレット、霊長類、および/または同様のACE2構造を持つ他の動物はすべて、SirotkinとSirotkinによって詳細に説明されているように連続通過実験に使用できる[1 ]。
Andersenら[ 2 ]はまた、"その後の多塩基性切断部位の生成には、その後、ヒトと同様のACE2受容体を持つ細胞培養または動物で繰り返し継代する必要があったが、そのような作業もこれまで記述されていない "と述べている。このような実験は、結果の発表の可能性がある前に、SARS-CoV-2の発生により中止された可能性や、結果が発表される予定ではなかった可能性を排除してはならない。
2019年3月にGD州で押収され、SARS-CoV-2の自然起源を支持する発表論文のほとんどが言及している密輸されたセンザンコウのRaTG13とセンザンコウの配列[ 2 ] は、最近その組み立てデータの正確さが疑問視されており、その正確さを証明するにはさらなる解析が必要であることも言及しておく必要があります[ xiii , xiv ] 再構築したRaTG13のin vitro受容体結合研究ではいくつかの奇妙な結果を得たことも注目すべき点でしょう。[最も驚くべきことは,RaTG13はSARS-CoV-2とは異なり,RaTG13の宿主とされるR. affinisの近縁種であるR. macrotisコウモリのACE2(そのACE2受容体はまだテストされていない)とは結合できないことである[59].同時に、RaTG13はhACE2と結合することが観察されているが[ 60 ]、SARS-CoV-2が全く結合しないラットやマウスのACE2ほどには結合していない。SARS-MA15がSARSのマウス適応株であるように、RaTG13もコウモリの糞便から採取した株ではなく、実はMojiang洞窟から抽出したCoVのマウス適応版であるという可能性はないだろうか?残念ながら、RaTG13のサンプルは使い果たされ、外部での検査ができなくなってしまいました[ 11 ]。これは、その配列の生データに多くの矛盾があることを考えると、残念なことです。また,莫干鉱夫のサンプルの状態や入手可能性についても,未解決かつ非常に重要な問題として残されている。鉱山労働者からいくつかのサンプルが収集され[ 7 , 8 ]、おそらく保管されており、SARS-CoV-2様CoVの存在を調べることは大きな価値があるはずである。
もう一つの未解決の疑問は、WIV自身のウイルスデータベースを修正し、その後削除した理由である。2020年5月、複数のメディアが、WIVの内部データベースの変更追跡システムで、データベースの名称が「野生動物由来ウイルス病原体データベース」から「コウモリおよびネズミ由来ウイルス病原体データベース」に変更され、その説明が編集されて「野生動物」のインスタンスが「コウモリおよびネズミ」に置き換えられ、さらに「節足動物ベクター」の言及が削除されたと報じている。 xv その後、データベースのページは完全に削除されたが、そのスナップショットはまだWeb Archiveで見ることができるxvii 他の国際的なCoV研究所が削除される前にWIVデータベースのSQLアーカイブをダウンロードした可能性があり、その場合、それらのデータは一般に公開されるべきである。
どうしてウイルスが実験室から逃げ出したのか?
研究室から危険性の高い病原体が流出することは珍しいことではなく、いくつかの国で発生が記録されています。知られている最も顕著な実験室からの漏洩は、世界的なパンデミックを引き起こした1977年の中国からのH1N1実験室からの漏洩です[ 61 ] 最近のものは、2019年11月に中国の蘭州の2つの研究センターで発生したブルセラ症の集団発生で、100人以上の学生や職員が感染しました[ 62 ] 最初のSARSウイルスのいくつかの実験室の漏洩も報告されています:2003年の夏にシンガポール、その後2003年12月に台湾、xviii、2004年の春に中国で2回xiviです。
WIVの研究所の安全性についての懸念は、2018年に米国大使館職員が研究所を訪問し、Zhengli Shiと面談した後、提起されました。研究所の監査官は、その後のワシントンへの外交文書でその心配をまとめたxx。中国の専門家も自国の研究所の安全性について懸念を示し、「研究所のゴミには人工のウイルスや細菌、微生物が含まれていることがある」、「特定の生物処理メカニズムなしに実験後の研究材料を下水に流す研究者がいる」と嘆いているxxi。
アメリカの研究所もまた、安全性の問題を抱え込んでいる。最近では、フォート・デトリックにあるバイオセーフティ・レベル(BSL)-4の米国陸軍感染症医学研究所(USAMRIID)施設での研究業務が、特に感染性物質の廃棄に関する安全違反を受けて2019年8月に中断したxxii。 他の米国の研究所でも、安全上の問題が指摘されている。22 ]
SARS-CoV-2が研究室から流出する原因には、さまざまなシナリオが想定される。例えば、感染動物が実験室から逃げ出したか、それが作業員を引っ掻いたり噛んだりした可能性(2017年に雲南省昆明にBSL-4の霊長類ワクチン試験施設が設立されたことについて懸念された[ 64 ] )、あるいは研究者が誤って自分に接種物を付着させてしまった可能性(ロシアで2例起きたようにxxiii)などがあります。2020年まで、CoVは特に致死性、毒性が強いとは考えられていなかった。SARSに似たCoVはBSL-4を必要とせず,BSL-2やBSL-3[ 42 ]の条件下で操作できたため,誤って漏出する可能性が高くなったのである。CoVsを用いたエアロゾル実験[ 65 ]でも、使用した機器の不具合があっても、実験従事者の感染が発見されるまで長い間気づかれない可能性があるため、実験室の漏洩につながる可能性があります。さらに、適切な廃棄物処理や汚染除去の手順を踏まなかった場合、ウイルスが下水道を通じて漏れる可能性もある。
結論と展望
我々の分析に基づけば、SARS-CoV-2の人為的起源は非難されるべき根拠のない陰謀論ではなく[ 66 ]、研究者にはSARS-CoV-2出現のあらゆる可能な原因を検討する責任がある。細胞/動物の連続継代とfurin切断部位によって得られたヒトに適応したセンザンコウのRBDの挿入は、進化研究または汎CoVワクチンや薬剤の開発という文脈で、部位特異的突然変異誘発実験から生じる可能性もある。Nature誌の最近の記事[ 67 ]は、研究者が偶然に感染した可能性があるので、SARS-CoV-2の実験室起源を否定できず、SARS-CoV-2をもたらした機能獲得実験がWIVで行われた可能性があると断言している。SARS-CoV-2の遺伝子操作は、バックボーン配列を入手でき、必要な設備を備えた世界中のどの研究室でも行うことができ、何の痕跡も残さないだろう。合成遺伝学プラットフォームに基づく現代の技術では、天然の分離株を必要とせず、そのゲノム配列に基づいてウイルスを再構築することができる[ 68 ]。
SARS-CoV-2発生以前にCoV研究に携わっていたすべての研究室における株の収集と研究記録の徹底的な調査が緊急に必要である。特に、削除されたWIVデータベースに記載されている可能性のある、ウイルス研究所で作出されたが未発表のCoVの株には注意を払う必要がある。自然宿主を見つけるには、最初のSARSのように何年もかかるか[ 67 ]、あるいは成功しないかもしれないので、SARS-CoV-2の自然起源と実験室起源を等しく調査することが優先されるべきであろう。
バークレー校の研究者であるXiao Qiangは、最近こう述べている。「このウイルスがどのように発生したかを正確に理解することは、将来このようなことが起こらないようにするための重要な知識である"。[xxi] と述べている。
利益相反
Rossana SegretoとYuri Deiginは、利益相反はありません。
謝辞
原稿の校正、貴重なコメント、論争の的となる仮説に対してオープンマインドでいてくれたAllan Krill教授(NTNU)、サポートしてくれたHeribert Insam教授(インスブルック大学微生物学部長)、すべての有益な情報を提供してくれたLawrence Sellin博士に感謝します。Fernando Castro-Chavez (元New York Medical College Post-Doc)には、Research Gateでお世話になった。BtCoV4991とRaTG13が同一サンプルであることを確認するためのデータベースを発見してくれたRené Bergelt氏に感謝する。最後に、SARS-CoV-2とその関連株に関する多くの未発表の事実を発見してくれたD.R.A.S.T.I.C. (Decentralised Radical Autonomous Search Team Investigating COVID-19) Twitterグループのメンバーに非常に感謝する。特に、2012年のMojiang肺炎の発生とWIVおよびSARS-CoV-2との関連性を継続的に探ってくれたLuigi Warren、中国語の2013 Xu MScおよび2016 Huang PhDの論文を見つけてくれた@TheSeeker268に感謝します。また、Francisco de Asis de Ribera Martinには、2つの論文の英訳を提供していただき、さらにRaTG13アンプリコンデータを発見していただきました。
ノート
Segreto, R. , & Deigin, Y. (2020). SARS-CoV-2の遺伝子構造は実験室由来を否定していない。BioEssays, 00, e2000240 10.1002/bies.202000240. [PMC free article] [PubMed] [CrossRef] [Google Scholar].
本研究は、外部資金を一切受けていない。
Rossana SegretoとYuri Deiginはこの研究に等しく貢献した。
脚注
iAreddy, J. T. (2020). 中国、コロナウイルス起源として動物市場と実験室を除外。The Wall Street Journal. https://www.wsj.com/articles/china-rules-out-animal-market-and-lab-as-coronavirus-origin-11590517508 (last accessed on Oct 15, 2020).
iiZhan, S. H., Deverman, B. E., Chan, Y. A. (2020年). SARS-CoV-2はヒトによく適応している。これは再出現に何を意味するのか?BioRxiv. https://doi.org/10.1101/2020.05.01.073262 (last accessed on Oct 15, 2020).
iiiQiu, J. (2020). 中国の「コウモリ女」は、SARSから新型コロナウイルスまで、どのようにウイルスを追い詰めたのか。Sci. Am. https://www.scientificamerican.com/article/how-chinas-bat-woman-hunted-down-viruses-from-sars-to-the-new-coronavirus1/ (last accessed on Oct 15, 2020).
ivDBatVir - The Database of Bat-Associated Viruses. http://www.mgc.ac.cn/cgi-bin/DBatVir/main.cgi?func=accession&acc=MN996532 (last accessed on Oct 15, 2020).コウモリに関連するウイルスのデータベース。
vSRX8357956: RaTG13のアンプリコン配列。https://www.ncbi.nlm.nih.gov/sra/SRX8357956 (最終アクセス: 2020年10月15日).
viAnon. (2020). RaTG13のアンプリコン配列の名称。https://web.archive.org/web/20200918174030/https://graph.org/RaTG13-Amplicon-Names-07-03 (last accessed on Oct 15, 2020)。
viiKahn, J. (2020). How scientists could stop the next pandemic before it starts. NYT Magazine. https://www.nytimes.com/2020/04/21/magazine/pandemic-vaccine.html (last accessed on Oct 15, 2020)。
viiiProject Number 2R01AI110964-06, ECOHEALTH ALLIANCE, INC., https://projectreporter.nih.gov/project_info_description.cfm?aid=9819304&icde=49645421&ddparam=&ddvalue=&ddsub=&cr=1&csb=default&cs=ASC&pball= (last accessed on Oct 15, 2020)。
ixPiplani, S., Singh, P. K., Winkler, D. A., Petrovsky, N. (2020年). In silico comparison of spike protein-ACE2 binding affinities across species; significance for the possible origin of the SARS-CoV-2 virus.arXiv. http://arxiv.org/abs/2005.06199 (last accessed on Oct 15, 2020).スパイクタンパク質とACE2の結合親和性を種間で比較した。
xLee, J., Hughes, T., Lee, M.-H., Field, H., Rovie-Ryan, J. J., Sitam, F. T., ... Daszak, P. (2020). マレーシア経由で野生動物取引に入るスンダパンゴリン(Manis javanica)にコロナウイルスやその他の人獣共通感染症になりうるウイルスが存在する証拠はない。BioRxiv. https://doi.org/10.1101/2020.06.19.158717 (last accessed on Oct 15, 2020)。
xiMou, H., Quinlan, B. D., Peng, H., Guo, Y., Peng, S., Zhang, L., ... Farzan, M. (2020).。コウモリのACE2オルソログの変異は、SARS-CoV-2のACE2-Fc中和を顕著に増強する。BioRxiv. https://doi.org/10.1101/2020.06.29.178459 (last accessed on Oct 15, 2020).
xiiZhang, D. (2020). BatCoV/RaTG13 のシークエンスと出所の異常。Zenodo. https://zenodo.org/record/3969272 (last accessed on Oct 15, 2020)。
xiiiSingla, M., Ahmad, S., Gupta, C., Sethi, T. (2020年). RaTG13ゲノムのデノボアセンブリは、SARS-CoV-2の起源をさらに不明瞭にする矛盾を明らかにした。Preprints. https://doi.org/10.20944/preprints202008.0595.v1 (last accessed on Oct 12, 2020).
xivChan, Y. A., Zhan, S. H. (2020年). SARS-CoV-2とほぼ同一のスパイクRBDを持つセンザンコウのCoVの単一ソース。BioRxiv. https://doi.org/10.1101/2020.07.07.184374 (last accessed on Oct 15, 2020)。
xvDevine, M. (2020). 中国はコロナウイルスについて何を隠蔽しているのか?NYT Magazine. https://nypost.com/2020/05/06/what-is-china-covering-up-about-the-coronavirus-devine/ (last accessed on Oct 12, 2020).
xvi https://twitter.com/ydeigin/status/1259891518468427776 (last accessed on Oct 15, 2020)。
xviiBat and rodent-borne viral pathogen database. https://web.archive.org/web/20200529174243/http://csdata.org/p/308/ (last accessed on Oct 15, 2020).(最終アクセス日:2020年10月15日)。
xviii: Reuters (2003). 台湾でSARS患者を確認。Wired. https://www.wired.com/2003/12/sars-case-confirmed-in-taiwan/ (last accessed on Oct 13, 2020)。
xixWalgate, R. (2004). SARSは北京の研究所から2度脱出した。The Scientist Magazine. https://www.the-scientist.com/news-analysis/sars-escaped-beijing-lab-twice-50137 (last accessed on Oct 15, 2020).
xxRogin, J. (2020). 国務省の電報は、コウモリコロナウイルスを研究する武漢の研究所の安全性に問題があると警告していた。The Washington Post. https://www.washingtonpost.com/opinions/2020/04/14/state-department-cables-warned-safety-issues-wuhan-lab-studying-bat-coronaviruses/ (last accessed on Oct 15, 2020)。
xxiCaiyu, L., Shumei, L. (2020). ウイルス研究所の慢性的な管理の抜け穴を修正するために発行されたバイオセーフティガイドライン。Global Times. https://www.globaltimes.cn/content/1179747.shtml (last accessed on Oct 15, 2020)。
xxiiGrady, D. (2020). 致命的な細菌研究は、安全性の懸念のために軍の研究所で停止されます。NYT Magazine. https://www.nytimes.com/2019/08/05/health/germs-fort-detrick-biohazard.html (last accessed on Oct 15, 2020).
xxiii: ミラー、J. (2004). ロシア人科学者、旧兵器研究所のエボラ出血熱の事故で死亡。NYT Magazine. https://www.nytimes.com/2004/05/25/world/russian-scientist-dies-in-ebola-accident-at-former-weapons-lab.html (last accessed on Oct 15, 2020).
脚注
xxiv https://twitter.com/ydeigin/status/1262686286898397189 (最終アクセス: 2020年10月15日).
データアベイラビリティステートメント
今回の研究では、データセットの生成や分析が行われていないため、データ共有は適用されない。
論文情報
バイオエッセイ 2020 Nov 17 : 2000240.
doi: 10.1002/bies.202000240 [Epub ahead of print] (英語版のみ。
PMCID: PMC7744920
PMID: 33200842
ロサナ・セグレット通信員1、ユーリ・デイギン2
1 インスブルック大学微生物学部、インスブルック、オーストリア。
2 Youthereum Genetics Inc.、カナダ、オンタリオ州トロント。
Rossana Segreto, Email: ta.ca.kbiu@otergeS.anassoR.
corresponding authorCorresponding author.
*Correspondence
Rossana Segreto, University of Innsbruck, Technikerstraße 25, 6020 Innsbruck, Austria, Department of Microbiology.
電子メール:ta.ca.kbiu@otergeS.anassoR,
2020年9月2日受領、2020年10月16日改訂
著作権 © 2020 The Authors. ワイリーピリオディカルズ合同会社発行のBioEssays
本論文は、http://creativecommons.org/licenses/by/4.0/ ライセンスに基づくオープンアクセス論文であり、原著を適切に引用することを条件に、あらゆる媒体での使用、配布、複製を許可するものです。
この論文は、PMCの他の論文によって引用されています。
参考文献
1. Sirotkin, K. , & Sirotkin, D. (2020). SARS-CoV-2は、動物宿主または細胞培養を連続的に通過することによって生じたのかもしれない?新型コロナウイルスの特徴的なゲノムの多くに対する潜在的な説明。BioEssays, 42, 1-7. 10.1002/bies.202000091 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
2. Andersen, K. G. , Rambaut, A. , Lipkin, W. I. , Holmes, E. C. , & Garry, R. F. (2020). SARS-CoV-2の近位起源。Nat. Med., 26, 450-452. 10.1038/s41591-020-0820-9 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
3. Zhou, P. , Yang, X. L. , Wang, X. G. , Hu, B. , Zhang, L. , Zhang, W. , ... Shi, Z. L. (2020). コウモリ由来と思われる新型コロナウイルスに関連する肺炎アウトブレイク。Nature, 579, 270-273. 10.1038/s41586-020-2012-7 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
4. Cagliani, R. , Forni, D. , Clerici, M. , & Sironi, M. (2020). 新規コロナウイルス、重症急性呼吸器症候群コロナウイルス2の進化の基礎となる選択の計算論的推論。J. Virol., 94, 1-11. 10.1128/jvi.00411-20 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
5. Ge, X. Y. , Wang, N. , Zhang, W. , Hu, B. , Li, B. , Zhang, Y. Z. , ... Shi, Z. L. (2016). 廃坑内の複数のコウモリコロニーにおける複数のコロナウイルスの共存。Virol. Sin., 31, 31-40. 10.1007/s12250-016-3713-9 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
6. Wu, Z. , Yang, L. , Yang, F. , Ren, X. , Jiang, J. , Dong, J. , ... Jin, Q. (2014). ラットにおける新規のヘニパ様ウイルス、モジャン・パラミクソウイルス、中国、2012年。Emerg. Infect. Dis., 20, 1064-1066. 10.3201/eid2006.131022 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
7. Xu, L. (2013). 未知のウイルスによる重症肺炎患者6名の解析(修士論文)。昆明医科大学 救急医学(専門職学位)http://eng.oversea.cnki.net/Kcms/detail/detail.aspx?filename=1013327523.nh&dbcode=CMFD&dbname=CMFD2014。
8. ホァン、C. (2016). コウモリの新規ウイルス発見とコウモリコロナウイルス HKU9 の受容体の探索(博士論文).中国疾病管理予防センター http://eng.oversea.cnki.net/kcms/detail/detail.aspx?dbcode=CDFD&dbname=CDFDLAST2018&filename=1017118517.nh.
9. Wang, N. , Luo, C. , Liu, H. , Yang, X. , Hu, B. , Zhang, W. , ... Shi, Z. (2019). Rhinolophusコウモリのユニークなゲノム特徴を持つアルファコロナウイルスの新メンバーの特性評価。Viruses, 11(4), 379 10.3390/v11040379 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
10. Chen, L. , Liu, W. , Zhang, Q. , Xu, K. , Ye, G. , Wu, W. , ... Liu, Y. (2020). RNAベースのmNGSアプローチにより、2019年武漢アウトブレイクにおける2人の個人肺炎症例から新規ヒトコロナウィルスを同定した。エマージェンシー。Microbes Infect.、9、313-319. 10.1080/22221751.2020.1725399 [PMC無料記事] [PubMed] [CrossRef] [Google Scholar].
11. コーエン、J.武漢のコロナウイルスハンター、Shi Zhengliが語る。サイエンス, 369, 487-488. 10.1126/science.369.6503.487 [PubMed] [CrossRef] [Google Scholar].
12. Liu, P. , Chen, W. , & Chen, J. P. (2019). ウイルスメタゲノミクスにより、マラヤパンゴリン(Manis javanica)のセンダイウイルスとコロナウイルスの感染が明らかになった。Viruses, 11(11), 979 10.3390/v11110979 [PMCフリー記事] [PubMed] [CrossRef] [Google Scholar].
13. Bianchi, M. , Benvenuto, D. , Giovanetti, M. , Angeletti, S. , Ciccozzi, M. , & Pascarella, S. (2020). Sars-CoV-2のエンベロープタンパク質と膜タンパク質:構造的な違いはウイルスの特徴に関連するか?Biomed. Res. Int., 2020 10.1155/2020/4389089 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
14. Schoeman, D. , & Fielding, B. C. (2019). コロナウイルスのエンベロープタンパク質。現在の知見。Virol. J., 16, 1-22. 10.1186/s12985-019-1182-0 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
15. Hu, B. , Zeng, L. P. , Yang, X. Lou , Ge, X. Y. , Zhang, W. , Li, B. , ... Shi, Z. L. (2017). コウモリSARS関連コロナウイルスの豊富な遺伝子プールの発見により、SARSコロナウイルスの起源について新たな知見が得られた。PLoS Pathog., 13, 1-27. 10.1371/journal.ppat.1006698 [PMCフリー記事] [PubMed] [CrossRef] [Google Scholar].
16. Fan, Y. , Zhao, K. , Shi, Z. L. , & Zhou, P. (2019). 中国におけるコウモリコロナウィルス。Viruses, 11(3), 210-. 10.3390/v11030210 [PMCフリー記事] [PubMed] [CrossRef] [Google Scholar].
17. Ge, X. Y. , Li, J. L. , Yang, X. Lou , Chmura, A. A. , Zhu, G. , Epstein, J. H. , ... Shi, Z. L. (2013). ACE2受容体を利用するコウモリSARS様コロナウイルスの単離と特徴づけ。Nature, 503, 535-538. 10.1038/nature12711 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
18. Graham, R. L. , & Baric, R. S. (2010). 組換え、リザーバー、およびモジュラー スパイク。コロナウイルスの異種間伝播のメカニズム. J. Virol., 84, 3134-3146. 10.1128/jvi.01394-09 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
19. Menachery, V. D. , Yount, B. L. , Debbink, K. , Agnihothram, S. , Gralinski, L. E. , Plante, J. A. , ... Baric, R. S. (2015). 循環するコウモリコロナウイルスのSARS様クラスターは、ヒトに出現する可能性を示している。Nat. Med., 21, 1508-1513. 10.1038/nm.3985 [PMCフリー記事] [PubMed] [CrossRef] [Google Scholar].
20. Johnson, B. A. , Graham, R. L. , & Menachery, V. D. (2018). ウイルスメタゲノミクス、タンパク質構造、およびリバースジェネティクス。コロナウイルスを調査するための重要な戦略。Virology, 517, 30-37. 10.1016/j.virol.2017.12.009 [PMCフリー記事] [PubMed] [CrossRef] [Google Scholar].
21. ラカニエッロ、V. (2016). 次のパンデミックウイルスを見つけるためのメタゲノム解析の先にあるもの。PNAS, 113, 2812-2814. 10.1073/pnas.1601512113. [PMC フリーアーティクル] [PubMed] [CrossRef] [Google Scholar].
22. Weiss, S. , Yitzhaki, S. , & Shapira, S. C. (2015). 米国における最近のバイオセーフティ事件から学ぶべき教訓。Isr. Med. Assoc. J., 17, 269-273. 10.1073/pnas.1601512113 [PubMed] [CrossRef] [Google Scholar].
23. Casadevall, A. , & Imperiale, M. J. (2014). インフルエンザウイルスなど、パンデミックの可能性がある病原体を用いた機能獲得実験のリスクと利点。科学的根拠に基づく議論の呼びかけ。MBio, 5, 1-5. 10.1128/mBio.01730-14 [PMCフリー記事] [PubMed] [CrossRef] [Google Scholar].
24. Agostini, M. L. , Andres, E. L. , Sims, A. C. , Graham, R. L. , Sheahan, T. P. , Lu, X. , ... Denison, M. R. (2018). 抗ウイルス剤レムデシビル(GS-5734)に対するコロナウイルスの感受性は、ウイルスポリメラーゼと校正エクソリボヌクレアーゼによって媒介される。MBio, 9, 1-15. 10.1128/mBio.00221-18 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
25. Xia, S. , Liu, M. , Wang, C. , Xu, W. , Lan, Q. , Feng, S. , ... Lu, L. (2020). SARS-CoV-2(以前は2019-nCoV)の感染阻害は、膜融合を媒介する高い能力を保有するそのスパイクタンパク質を標的とした高活性汎コロナウイルス融合阻害剤によるものである。Cell Res., 30, 343-355. 10.1038/s41422-020-0305-x [PMC free article] [PubMed] [CrossRef] [Google Scholar].
26. Totura, A. L. , & Bavari, S. (2019). ブロードスペクトラムコロナウイルス抗ウイルス薬の発見。Expert Opin. Drug Discov., 14, 397-412. 10.1080/17460441.2019.1581171 [PMC無料記事] [PubMed] [CrossRef] [Google Scholar].
27. Wang, Y. , Sun, Y. , Wu, A. , Xu, S. , Pan, R. , Zeng, C. , ... Guo, D. (2015). コロナウイルスnsp10/nsp16メチルトランスフェラーゼは、in vitroおよびin vivoでnsp10由来のペプチドによって標的とされ、複製と病原性を低下させることができる。J. Virol., 89, 8416-8427. 10.1128/jvi.00948-15 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
28. Kuo, L. , Godeke, G. J. , Raamsman, M. J. B. , Masters, P. S. , & Rottier, P. J. M. (2000). スパイク糖タンパク質エクトドメインの置換によるコロナウイルスの再標的化。宿主細胞の種の壁を越える。J. Virol., 74, 1393-1406. 10.1128/jvi.74.3.1393-1406.2000 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
29. Maier, H. J. , Bickerton, E. , & Britton, P. (2015). コロナウイルス - メソッドとプロトコール. London: Humana Press. [Google Scholar]
30. Becker, M. M. , Graham, R. L. , Donaldson, E. F. , Rockx, B. , Sims, A. C. , Sheahan, T. , ... Denison, M. R. (2008). 合成組換えコウモリSARS様コロナウイルスは、培養細胞およびマウスで感染性を示す。PNA, 105, 19944-19949. 10.1073/pnas.0808116105 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
31. Menachery, V. D. , Yount, B. L. , Sims, A. C. , Debbink, K. , Agnihothram, S. S. , Gralinski, L. E. , ... Baric, R. S. (2016). SARS様WIV1-CoVはヒトへの出現の態勢を整えている。PNA, 113, 3048-3053. 10.1073/pnas.1517719113 [PMCフリー記事] [PubMed] [CrossRef] [Google Scholar].
32. Li, X. , Zai, J. , Zhao, Q. , Nie, Q. , Li, Y. , Foley, B. T. , & Chaillon, A. (2020). SARS-CoV-2の進化史、潜在的中間宿主動物、および種間解析。J. Med. Virol., 92, 602-611. 10.1002/jmv.25731 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
33. Lam, T. T. Y. , Jia, N. , Zhang, Y. W. , Shum, M. H. H. , Jiang, J. F. , Zhu, H. C. , ... Cao, W. C. (2020). マラヤのパンゴリンにおけるSARS-CoV-2関連コロナウイルスの特定。Nature, 583, 282-285. 10.1038/s41586-020-2169-0 [PubMed] [CrossRef] [Google Scholar].
34. Xiao, K. , Zhai, J. , Feng, Y. , Zhou, N. , Zhang, X. , Zou, J. J. , ... Shen, Y. (2020). マレー半島のパンゴリンからSARS-CoV-2関連コロナウイルスを分離した。Nature, 583, 286-289. 10.1038/s41586-020-2313-x [PubMed] [CrossRef] [Google Scholar].
35. Zhang, T. , Wu, Q. , & Zhang, Z. (2020). COVID-19アウトブレイクに関連するSARS-CoV-2の推定センザンコウ由来。Curr. Biol.、30、1346-1351.E2. 10.1016/j.cub.2020.03.022 [PMCフリー記事] [PubMed] [CrossRef] [Google Scholar].
36. Coutard, B. , Valle, C. , de Lamballerie, X. , Canard, B. , Seidah, N. G. , & Decroly, E . (2020). 新型コロナウイルス 2019-nCoV のスパイク糖タンパク質は、同じクレードの CoV には存在しないフリン様切断部位を含んでいる。Antivir. Res., 176, 104742 10.1016/j.antiviral.2020.104742 [PMC free article] [PubMed] [CrossRef] [Google Scholar] (PMC フリーアーティクル)。
37. Letko, M. , Marzi, A. , & Munster, V. (2020). SARS-CoV-2および他の系統Bベータコロナウイルスの細胞侵入と受容体利用の機能的評価。Nat. Microbiol., 5, 562-569. 10.1038/s41564-020-0688-y [PMC free article] [PubMed] [CrossRef] [Google Scholar].
38. Wang, Q. , Qiu, Y. , Li, J. Y. , Zhou, Z. J. , Liao, C. H. , & Ge, X. Y. (2020). 新型肺炎コロナウイルス(2019-nCoV)のスパイクタンパク質に予測されるユニークなプロテアーゼ切断部位は、ウイルスの感染性に潜在的に関連している。Virol. Sin., 35, 337-339. 10.1007/s12250-020-00212-7 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
39. Lau, S. , Wang, P. , Mok, B. W. , Zhang, A. J. , Chu, H. , Lee, A. C. , ... Chen, H. (2020). S1 / S2 ジャンクションに欠失を持つ弱毒性 SARS-CoV-2 変異体。Emerg. Microbes Infect., 9, 837-842. 10.1080/22221751.2020.1756700 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
40. Hoffmann, M. , & Kleine-Weber, H. (2020). SARS-CoV-2のスパイクタンパク質の多塩基性切断部位は、ヒト肺細胞の感染に必須である。Mol. Cell., 78, 779-784.E5. 10.1016/j.molcel.2020.04.022 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
41. Kaundun, S. S. , Marchegiani, E. , Hutchings, S. J. , & Baker, K. (2019). Derived polymorphic amplified cleaved sequence (dPACS): 既知の一塩基および欠失-挿入多型を検出するための新規PCR-RFLP手順。を発表した。J. Mol. Sci., 20(13), 3193 10.3390/ijms20133193 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
42. Zeng, L. P. , Gao, Y. T. , Ge, X. Y. , Zhang, Q. , Peng, C. , Yang, X. L. , ... Shi, Z. L. (2016). コウモリ重症急性呼吸器症候群様コロナウイルスWIV1は、調節に関与する余分なアクセサリータンパク質、ORFXをコードしている。J. Virol., 90, 6573-6582. 10.1128/JVI.03079-15 [PMCフリー記事] [PubMed] [CrossRef] [Google Scholar].
43. Khan, S. G. , Muniz-Medina, V. , Shahlavi, T. , Baker, C. C. , Inui, H. , Ueda, T. , ... Kraemer, K. H. (2002). ヒトXPC DNA修復遺伝子。スプライスアクセプターサイトの一塩基多型が代替スプライシングと機能に与える影響。Nucleic Acids Res., 30, 3624-3631. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC134237/. [PMC free article] [PubMed] [Google Scholar].
44. Liu, Z. , Zheng, H. , Lin, H. , Li, M. , Yuan, R. , Peng, J. , ... Lu, J. (2020). 重症急性呼吸器症候群コロナウイルス2のスパイクタンパク質における共通欠失の同定。J. Virol., 94, e00790-20. 10.1128/JVI.00790-20 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
45. Zhou, H. , Chen, X. , Hu, T. , Li, J. , Song, H. , Liu, Y. , ... Shi, W. (2020). SARS-CoV-2 に近縁な新規コウモリコロナウイルスは、スパイクタンパク質の S1/S2 切断部位に天然の挿入物を含む。Curr. Biol., 30, 2196-2203.E3. 10.1016/j.cub.2020.05.023 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
46. スタインハウアー、D.A.(1999)。インフルエンザウイルスの病原性に対するヘマグルチニン開裂の役割。Virology, 258, 1-20. 10.1006/viro.1999.9716 [PubMed] [CrossRef] [Google Scholar].
47. Seyran, M. , Pizzol, D. , Adadi, P. , El-Aziz, T. M. A. , Hassan, S. S. , Soares, A. , ... Brufsky, A. M. (2020). SARS-CoV-2の近接型起源に関する疑問。J. Med. Virol., 03. 10.1002/jmv.26478 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
48. Wang, L. F. , & Anderson, D. E. (2019). コウモリのウイルスと動物やヒトへの潜在的な波及効果。Curr. Opin. Virol., 34, 79-89. 10.1016/j.coviro.2018.12.007 [PMC無料記事] [PubMed] [CrossRef] [Google Scholar].
49. Wan, Y. , Shang, J. , Graham, R. , Baric, R. S. , & Li, F. (2020). 武漢発の新型コロナウイルスによるレセプター認識。SARSコロナウイルスの10年にわたる構造研究に基づく解析。J. Virol., 94(7), 1-9. 10.1128/jvi.00127-20 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
50. Cui, J. , Li, F. , & Shi, Z. L. (2019). 病原性コロナウィルスの起源と進化. Nat. Rev. Microbiol., 17, 181-192. 10.1038/s41579-018-0118-9 [PMC無料記事] [PubMed] [CrossRef] [Google Scholar].
51. Fraguas Bringas, C. , & Booth, D. (2020). SARS-CoV-2のスパイク糖タンパク質受容体結合ドメインと構造的特徴を共有するSARS様コウモリコロナウイルスの同定(Identification of a SARS-like bat Coronavirus that share structural features with the spike glycoprotein receptor-binding domain of SARS-CoV-2. Access Microbiol., 10-17. 10.1099/acmi.0.000166. [PMC フリーアーティクル] [PubMed] [CrossRef] [Google Scholar].
52. Mallapati, S. (2020). なぜコロナウイルスは人の間で簡単に広がるのか?Nature, 579, 183 https://www.nature.com/articles/d41586-020-00660-x。[PubMed][Google Scholar].
53. Almazán, F. , Sola, I. , Zuñiga, S. , Marquez-Jurado, S. , Morales, L. , Becares, M. , & Enjuanes, L. (2014). コロナウイルスの逆遺伝システム。感染性クローンとレプリコン。Virus Res., 189, 262-270. 10.1016/j.virusres.2014.05.026 [PMCフリー記事] [PubMed] [CrossRef] [Google Scholar].
54. Cheng, J. , Zhao, Y. , Xu, G. , Zhang, K. , Jia, W. , Sun, Y. , ... Zhang, G. (2019). QX型感染症のS2サブユニット
54. Cheng, J. , Zhao, Y. , Xu, G. , Zhang, K. , Jia, W. , Sun, Y. , ... Zhang, G. (2019). QX型感染性気管支炎コロナウイルスのスパイクタンパク質のS2サブユニットは、ニュートロプロピズムの必須な決定因子である。Viruses, 11(10), 972 10.3390/v11100972 [PMCフリー記事] [PubMed] [CrossRef] [Google Scholar].
55. Wrapp, D. , Wang, N. , Corbett, K. S. , Goldsmith, J. A. , Hsieh, C. L. , Abiona, O. , ... McLellan, J. S. (2020). プレフュージョンコンフォメーションにおける2019-nCoVスパイクのクライオ電子顕微鏡構造。Science, 367, 1260-1263. 10.1126/science.abb2507 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
56. Du, L. , Tai, W. , Yang, Y. , Zhao, G. , Zhu, Q. , Sun, S. , ... Li, F. (2016). MERSコロナウイルスサブユニットワクチンの合理的設計への中和免疫原性インデックスの導入。Nat. Commun., 7, 1-9. 10.1038/ncomms13473 [PMCフリー記事] [PubMed] [CrossRef] [Google Scholar].
57. Wahl, A. , De, C. , Abad Fernandez, M. , Lenarcic, E. M. , Xu, Y. , Cockrell, A. S. , ... Garcia, J. V. (2019). ヒト病原体に対する拡張されたトロピズムを持つ精密マウスモデル。Nat. Biotechnol., 37, 1163-1173. 10.1038/s41587-019-0225-9 [PMC無料記事] [PubMed] [CrossRef] [Google Scholar].
58. Hu, D. , Zhu, C. , Ai, L. , He, T. , Wang, Y. , Ye, F. , ... Wang, C. (2018). 中国のコウモリにおける新規 SARS 様コロナウイルスのゲノム特性および感染性.エマージェンシー.Microbes Infect., 7, 1-10. 10.1038/s41426-018-0155-5 [PMC無料記事] [PubMed] [CrossRef] [Google Scholar].
59. Hron, T. , Farkašová, H. , Gifford, R. J. , Benda, P. , Hulva, P. , Görföl, T. , ... Elleder, D. (2018). カブトコウモリ(Rhinolophidae)のゲノムにおける古代のデルタレトロウイルスの残骸。Viruses, 10(4), 185-. 10.3390/v10040185 [PMCフリー記事] [PubMed] [CrossRef] [Google Scholar].
60. Shang, J. , Ye, G. , Shi, K. , Wan, Y. , Luo, C. , Aihara, H. , ... Li, F. (2020). SARS-CoV-2による受容体認識の構造的基盤. Nature, 581, 221-224. 10.1038/s41586-020-2179-y [PMC free article] [PubMed] [CrossRef] [Google Scholar].
61. ヴェルトハイム、J.1977年のH1N1インフルエンザウイルスの再出現。生物学的に非現実的なサンプリング日を用いて分岐時間を推定する際の注意点。PLoS ONE, 5, 2-5. 10.1371/journal.pone.0011184 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
62. Cyranoski, D. (2019). 中国の研究機関は、実験従事者の病原体発生を調査している。Nature, https://www.nature.com/articles/d41586-019-03863-z [PubMed] [Google Scholar].
63. Lim, P. L. , Kurup, A. , Gopalakrishna, G. , Chan, K. P. , Wong, C. W. , & Leo, Y. S. (2004). 実験室取得型重症急性呼吸器症候群。N. Engl. J. Med., 350, 1740-1745. 10.1056/NEJMoa032565 [PubMed] [CrossRef] [Google Scholar].
64. Cyranoski D. (2017). 世界で最も危険な病原体を研究する態勢を整えた中国の研究所の内部。Nature, 542, 399-401 10.1038/nature.2017.21487 [PubMed] [CrossRef] [Google Scholar].
65. Totura, A. , Livingston, V. , Frick, O. , Dyer, D. , Nichols, D. , & Nalca, A. (2020). 高病原性コロナウイルス感染モデルとしてのアフリカグリーンモンキーのMERS-CoVへの小粒子エアロゾル曝露。Emerg. Infect. Dis., 26 10.3201/eid2612.201664. [PMC フリーアーティクル] [PubMed] [CrossRef] [Google Scholar].
66. Calisher, C. , Carroll, D. , Colwell, R. , Corley, R. B. , Daszak, P. , Drosten, C. , ... Turner, M. (2019). COVID-19と闘う中国の科学者、公衆衛生専門家、医療専門家を支持する声明。The Lancet, 395, E42-E43. 10.1016/S0140-6736(20)30418-9 [PMC無料記事] [PubMed] [CrossRef] [Google Scholar].
67. Cyranoski, D. (2020). 最大の謎。コロナウイルスの発生源を追跡するために必要なこと。Nature, 10.1038/d41586-020-01541-z. [PubMed] [CrossRef] [Google Scholar].
68. Thao, T. T. N. , Labroussaa, F. , Ebert, N. , V'kovski, P. , Stalder, H. , Portmann, J. , ... Thiel, V. (2020). 合成ゲノミクスプラットフォームを用いたSARS-CoV-2の迅速な再構築。ネイチャー, 582, 561-565. 10.1038/s41586-020-2294-9 [PubMed] [CrossRef] [Google Scholar].
この記事が気に入ったらサポートをしてみませんか?
