【和訳】SARS-CoV-2のフリン切断部位とMSH3の相同性および組換え可能性 (2022/02/21)
話題の下記論文の全文自動翻訳を記載します。
(文中のリンクは再現していませんので元文献から辿ってください。)
また、文中のモデルナ社のパテントのリンクは↓
がん関連物質生産用修飾ポリヌクレオチド
タンパク質およびペプチド
出願人:モデルナ・セラピューティクス,インコーポレイテッド,ケンブリッジ,マサチューセッツ州(米国)
アサイニー ModernatX,Inc.、Cambridge,MA (US)
https://patents.google.com/patent/US9587003B2/en
PDF版
https://patentimages.storage.googleapis.com/01/6e/60/8951ab8f4118b5/US9587003.pdf
-----------以下、タイトルの論文----------
SARS-CoV-2のフリン切断部位とMSH3の相同性および組換え可能性
Balamurali K. Ambati1、Akhil Varshney2、Kenneth Lundstrom3*、Giorgio Palú4、Bruce D. Uhal5、Vladimir N. Uversky6 および Adam M. Brufsky7
1科学的インパクトを加速するためのナイトキャンパス、オレゴン大学、ユージーン、オレゴン州、アメリカ合衆国
2シュロフ・チャリティー・アイ・ホスピタル(インド、ニューデリー
3パン・セラピューティック社(スイス、ルトリー
4パドバ大学分子医学部(イタリア・パドバ市
5ミシガン州立大学生理学教室(米国ミシガン州イーストランシング市
6南フロリダ大学モルサニ医学部分子医学科、南フロリダ大学ヘルスバードアルツハイマー研究所、南フロリダ大学、フロリダ州タンパ、アメリカ合衆国
7ピッツバーグ大学医学部、ピッツバーグ大学医療センター(UPMC)ヒルマンがんセンター、医学部、血液学/腫瘍学部門、アメリカ合衆国ペンシルベニア州ピッツバーグ市
SARS-CoV-2とコウモリRaTG13コロナウイルスとの間には多くの点変異の違いがあるが、12ヌクレオチドのフリン切断部位(FCS)のみが3ヌクレオチドを超えている。BLAST検索の結果、SARS.Cov2ゲノムのフリン切断部位を含む19ヌクレオチド部分は、ヒトmutSホモログ(MSH3)の逆相補体であるコドン最適化独自配列に100%相補的に一致することが判明した。SARS-CoV-2に存在する逆相補配列はランダムに発生する可能性があるが、他の可能性も考慮しなければならない。中間宿主での組換えは考えにくい説明である。SARS-CoV-2のような一本鎖RNAウイルスは、感染細胞内で負鎖RNAを鋳型としており、負センスSARS-CoV-2 RNAとのコピー選択組み換えにより、FCSを含むMSH3負鎖がウイルスゲノムに組み込まれる可能性がある。いずれにせよ、MSH3 mRNAの逆相補体と100%一致するFCSを含む19ヌクレオチド長のRNA配列の存在は非常に珍しく、さらなる調査が必要である。
はじめに
SARS-CoV-2の挿入変異体に関する最近の発表(1)に基づき、SARS-CoV-2スパイク(S)タンパク質のフリン切断部位(FCS)の配列に関する我々の最近の知見に注目したい。COVID-19パンデミック(2)を起こしたSARS-CoV-2は、コウモリコロナウイルスSL-CoVZC45と82.3%、SARS-CoVと77.2%、コウモリコロナウイルスRaTG13と96.2%のゲノム配列同一性を有している。SARS-CoV-2とRaTG13の間には多くの点突然変異の相違が存在するが、3ヌクレオチド(nt)を超える挿入・非類似は1つだけである:SARS-CoV-2のSタンパク質に4アミノ酸(aa 681-684, PRRA)をコードする12ヌクレオチドの挿入が発見された。この多塩基性FCSは、SARS-CoV-2と他のb系統のベータコロナウイルスや他のどのサルベコウイルスとも区別される(3)。FCSの添加により、2019年のSARS Co-V-2の感染性が向上した(4)。このFCSがない場合、動物ワクチン接種に有用なSARS-CoV-2変種が減弱するため、ヒトへの感染との関連性が強調される(5)。このFCSはヒトとフェレットの感染に不可欠であり(6)、ヒト細胞へのウイルストロピズムを拡大し(7)、SARS-CoV-2の2つの動物モデルで重症化に必須である(8)。
SARS-CoV-2 スパイクタンパク質とMSH3
SARS-CoV-2 Sタンパク質のPRRA furin切断部位をコードする塩基配列の特徴は、CGGコドンが2つ連続していることである。このアルギニンコドンはコロナウイルスでは珍しく、センザンコウのCoVのCGGの相対同義性コドン使用率(RSCU)は0、コウモリCoVでは0.08、SARS-CoVでは0.19、MERS-CoVでは0.25、そしてSARS-CoV-2では0.299である(9)。
12塩基の挿入をBLAST検索したところ、2016年2月4日に出願された米国特許9,587,003(10)に見られる独自配列(SEQ ID11652, nt 2751-2733)に100%逆マッチしました(図1)。SEQ ID11652を調べると、一致は12ヌクレオチドの挿入を越えて19ヌクレオチドの配列に及んでいることが明らかになりました。5′-CTACGTGCCCGCCGAGGAG-3′(SEQ ID11652のnt 2733-2751)、その結果mRNAが3′-GAUGCACGGCGCUCCUC-5′を有することになるようにである。または同等に5′- CU CCU CGG CGG GCA CGU AG-3′(SARS-CoV-2ゲノムのヌクレオチド23547-23565、この中の4つの太字コドンによってPRRA、そのスパイクタンパク質のアミノ酸681-684が得られる)である。これはNCBI BLASTデータベースでは非常に稀である。
図1. SARS-CoV-2におけるフリン配列の起源。
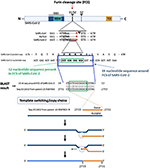
SARS-CoV、RaTG13、SARS-CoV-2のS1/S2接合部のタンパク質配列を比較すると、SARS-CoV-2にのみフリン切断部位(FCS)PRRAが存在することがわかる。FCS PRRAをコードする12ヌクレオチドストレッチのBLAST検索に基づき、19ヌクレオチド長の同一配列が特許(US 958 7003)配列Seq ID11652に同定された。SEQ ID11652は、ヒトに対してコドン最適化されていると思われるMSH3 mRNAに転写される。ヒトMSH3遺伝子に存在するFCS PRRAをコードする12ヌクレオチドを含むこの19ヌクレオチド配列は、MSH3遺伝子を過剰発現しているSARS-CoV-2感染ヒト細胞において図示のコピー選択組み換え機構によりSARS-CoV-2ゲノムに導入されたのかもしれない。
このSARS-CoV-2配列と独自に開発したmRNA配列の逆相補体との相関は、その由来が不明である。従来の生物統計学的解析では、この配列が3万塩基のウイルスゲノムの中にランダムに存在する確率は3.21×10-11である(図2)。
図2
www.frontiersin.org
図2. 研究対象である19nt配列の自然発生確率の計算結果。

SARS-CoV-2ゲノムの長さは約30,000ヌクレオチド(P1)である。特許配列の長さは約3,300ヌクレオチド(P2)。特許ライブラリは、中央値が3,300ヌクレオチドの範囲である、様々な長さの24'712配列を包含する。ヒトゲノムと特許ライブラリの配列のいずれかに19塩基の配列が存在する確率を従来の確率計算で算出した。
独自に開発した配列SEQ ID11652を順方向に読むと、ヒトmut S homolog 3(MSH3)(9)とアミノ酸が100%一致するようにコードしています。MSH3は、DNAミスマッチ修復タンパク質(MutSβ複合体の一部)である(11)。SEQ ID11652は、ヒト用にコドン最適化されていると思われるMSH3 mRNAに転写される(12)。19塩基の配列CTCCTCGGCGGCACGTAGは、BLASTデータベースで100%のカバー率と同一性を持つSARS-CoV-2以外の真核生物やウイルスゲノムでは見つからなかった(補足表1-3)。
考察
ヒトの発現にコドン最適化されたmRNA配列によるMSH3の置換は、ミスマッチ修復欠損のある癌に応用できそうである。SARS-CoV-2に存在する逆相補配列の一部は偶然の一致かもしれないが、他の可能性も考慮する価値がある。
MSH3の過剰発現はミスマッチ修復を妨害することが知られており(MSH2とMSH6からなるMutSα複合体からのMSH2の隔離はMSH6の分解とMutSαの枯渇をもたらす)(13)、これはウイルス学的に重要なことである。DNAミスマッチ修復欠損の誘導は、A型インフルエンザウイルス(IAV)のヒト呼吸器細胞への感染を許容し、病原性を増大させる(14)。ミスマッチ修復欠損は、SARS-CoV-2の排出を拡大する可能性がある(15, 16)。
BLASTデータベースでCTCCTCGGCGGCACGTAGが真核生物やウイルスのゲノムに存在しないことから、中間宿主での組み換えがSARS-CoV-2での存在を説明することはまず不可能である。ヒトMSH3と100%相同なタンパク質をコードするヒトコドン最適化mRNAは、ウイルス研究の過程で、不注意にあるいは意図的にヒト細胞株のミスマッチ修復欠損を誘発し、SARS様ウイルス感染に対する感受性を高める可能性がある。SEQ ID11652-MSH3が導入されたヒト細胞にSARS様ウイルスが感染すると、コピーチョイス組換えが可能になる可能性がある(15)。SARS-CoV-2をはじめとする正極性のRNAゲノムを持つ一本鎖RNAウイルスの複製は、感染細胞の細胞質で負鎖RNAが合成されることにより開始される(17)(図1)。負鎖RNAは、非構造タンパク質、複製・転写複合体、新しいビリオン・キャプシドの翻訳に利用される正鎖RNAの合成の鋳型となる。コロナウイルスは、感染初期にゲノム複製とmRNAの転写により二本鎖RNAを生成する(18)。
過剰発現したポジティブセンスMSH3 mRNAから逆補体FCS配列を獲得することは、ネガティブセンスSARS-CoV-2 RNA中間体とのコピーチョイス組み換えによって起こる可能性があり(15)、あるテンプレートから別のテンプレートへのジャンプを伴う(19)(図1). SARS-CoV-2と他の既知のコロナウイルスとの相同性は途絶え、ほとんどのSARS-CoV-2配列は、コウモリRaTG13と比較的最近の共通祖先に由来している。さらに、類似性プロット(SimPlots)により、SARS-CoV-2とRaTG13の間の配列同一性の急激な変化が確認され、潜在的な組み換え事象を示唆しており、これは、RaTG13 RBDにはない、そのRBDを介してACE2に結合するSARS-CoV-2の能力を説明できると考えられる(15)。
この仮説に対する批判は、同定された配列がSEQ ID11652のオープンリーディングフレームの反対側の鎖にあることである。しかし、ミスマッチ修復欠損を誘発するMSH3でトランスフェクトされた細胞は、SEQ ID11652をコードする二本鎖cDNAを標的にしている可能性がある。RdRpを発現するSARS様ウイルスと共導入した細胞は、この19塩基配列に結合し(15)、オープンリーディングフレームの反対鎖にあるにもかかわらず、FCSを含むウイルスゲノムに負鎖からの断片の統合を可能にすることができる。ミスマッチ修復機構は、実験モデルにおいてアンチセンス鎖からの短い断片の統合を可能にした(20, 21)。マイクロホモロジーはMSH3とSARS様ウイルスとの間の組換えを誘導することができ、それは関心のある19塩基配列で起こる可能性がある。
SARS-CoV-2において、そのスパイクタンパク質のアミノ酸681にFCSをコードする19ヌクレオチドRNA配列が、独自のMSH3 mRNA配列の逆相補体と100%同一であることは、非常に珍しいことである。この相関関係を説明できる可能性があるため、さらに調査する必要がある。
データの利用可能性に関する声明
本研究では、一般に公開されているデータセットを分析した。このデータはこちらでご覧いただけます。SEQ ID11652。
著者による貢献
調査および原案作成はBA、AV、ABが担当した。可視化はBA, AV, AB, GPが行った。執筆と編集は、BA、AV、KL、GP、BU、VU、ABが行った。全著者が論文に貢献し、提出されたバージョンを承認した。
利益相反について
KLはPanTherapeuticsに雇用されていた。
残りの著者は、本研究が利益相反の可能性があると解釈される商業的または金銭的関係がない状態で実施されたことを宣言する。
出版社からのコメント
本論文で述べられたすべての主張は、著者個人のものであり、必ずしも所属団体、出版社、編集者、査読者の主張を代表するものではありません。この記事で評価される可能性のある製品,またはそのメーカーが行う可能性のある主張は,出版社によって保証または承認されるものではありません.
謝辞
米国ユタ大学Jian Ying博士の確率論的解析への貢献に感謝します。
補足資料
本論文の補足資料は、https://www.frontiersin.org/articles/10.3389/fviro.2022.834808/full#supplementary-material でご覧いただけます。
参考文献
1. Garushyants SK, Rogozin IB, Koonin EV. SARS-CoV-2ゲノムのテンプレートスイッチングと重複は、監視に値する挿入変種を生じさせる。Commun Biol. (2021) 4:1343. doi: 10.1038/s42003-021-02858-9
PubMed Abstract|CrossRef Full Text|Google Scholar
2. Wu F, Zhao S, Yu B, Chen YM, Wang W, Song ZG, et al. A new coronavirus associated with human respiratory disease in China.中国におけるヒト呼吸器疾患に関連する新しいコロナウイルス。Nature. (2020) 579:265-9. doi: 10.1038/s41586-020-2008-3
PubMed Abstract|クロスリファレンス全文|Google Scholar
3. Coutard B, Valle C, de Lamballerie X, Canard B, Seidah NG, Decroly E. The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade. Antiviral Res. (2020) 176:104742. doi: 10.1016/j.antiviral.2020.104742.
PubMed Abstract|CrossRef Full Text|Google Scholar
4. Walls AC, Park YJ, Tortorici MA, Wall A, McGuire AT, Veesler D. Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein. Cell. (2020) 181:281-92.e286. doi: 10.1016/j.cell.2020.02.058
PubMed Abstract|CrossRefフルテキスト|Google Scholar
5. Lau SY, Wang P, Mok BWY, Zhang AJ, Chu H, Lee ACY, et al. S1/S2 ジャンクションに欠失を持つ弱毒化 SARS-CoV-2 変異体。Emerg Microbes Infect. (2020) 9:837-42. doi: 10.1080/22221751.2020.1756700
PubMed Abstract|CrossRef Full Text|Google Scholar
6. Peacock TP, Goldhill DH, Zhou J, Baillon L, Frise R, Swann OC, et al. SARS-CoV-2 スパイクタンパク質のフリン切断部位はフェレットでの感染に必要である。Nat Microbiol. (2020) 6:899-909. doi: 10.1038/s41564-021-00908-w
PubMed Abstract|CrossRef Full Text|Google Scholar
7. Xia S, Lan Q, Su S, Wang X, Xu W, Liu Z, et al. SARS-CoV-2 スパイクタンパク質を介した膜融合におけるトリプシン存在下または非存在下のフリン開裂部位の役割。Signal Transd. Targeted Ther. (2020) 5:92. doi: 10.1038/s41392-020-0184-0
PubMed Abstract|CrossRef Full Text|Google Scholar
8. Hou W. SARS-CoV-2 のコドン使用パターンの特徴づけ。Virology J. (2020) 17:138. doi: 10.1186/s12985-020-01395-x
PubMed Abstract|CrossRef Full Text|Google Scholar
9. 9. Kandeel M, Ibrahim A, Fayez M, Al-Nazawi M. From SARS and MERS CoVs to SARS-CoV-2: Moving towards more biased codon usage in viral structural and nonstructural genes. J Med Virol. (2020) 92:660-6. doi: 10.1002/jmv.25754.
PubMed Abstract|CrossRef Full Text|Google Scholar
10. Bancel S, Chakraborty T, De Fougerolles A, Elbashir SM, John M, Roy A, et al. Modified Polynucleotides for the Production of Oncology-Related Proteins and Peptides.(がん関連タンパク質およびペプチドの生産のための改変ポリヌクレオチド)。Cambridge, MA: United States Patent. (2016).
グーグル スカラー
11. MacRae SL, McKnight Croken M, Calder RB, Aliper A, Milholland B, White RR, et al. 寿命の差が激しい種におけるDNA修復。Aging. (2015) 7:1171-84. doi: 10.18632/aging.100866.
PubMed Abstract|クロスリファレンス全文|Google Scholar
12. マウロVP、チャペルSA。A critical analysis of codon optimization in human therapeutics(ヒト治療薬におけるコドン最適化の批判的分析)。Trends Mol Med. (2014) 20:604-13. doi: 10.1016/j.molmed.2014.09.003
PubMed Abstract|CrossRef Full Text|Google Scholar
13. Marra G, Iaccarino I, Lettieri T, Roscilli G, Delmastro P, Jiricny J. Mismatch repair deficiency associated with overxpression of MSH3 gene(MSH3遺伝子の過剰発現に伴うミスマッチ修復欠損)。Proc Natl Acad Sci USA. (1998) 95:8568.
PubMed Abstract|CrossRef Full Text|Google Scholar
14. DNAミスマッチ修復は宿主の自然応答に必要であり、インフルエンザウイルス感染後の細胞の運命を制御する。Nat Microbiol. (2019) 4:1964-77. doi: 10.1038/s41564-019-0509-3.
PubMed Abstract|CrossRef Full Text|Google Scholar
15. ギャラハーWR.SARS-COV-2におけるコピーチョイス組換えの共通ブレークポイント寄与因子としての回文状RNA配列。Arch Virol. (2020) 165:2341-8. doi: 10.1007/s00705-020-04750-z
PubMed Abstract|CrossRef Full Text|Google Scholar
16. Haque F, Lillie P, Haque F, Maraveyas A. Deficient DNA mismatch repair and persistence of SARS-CoV-2 RNA shedding: a case report of Hereditary Nonpolyposis Colorectal Cancer with COVID-19 infection.リリー・P・ハック、マラベイヤス、ハック F、マラベイヤス A. Deficient DNA mismatch repair and persistence of SARS-CoV-2 RNA shedding. BMC Infect Dis. (2020) 21:854. doi: 10.1186/s12879-021-06500-1
PubMed Abstract|CrossRef Full Text|Google Scholar
17. V'kovski P, Kratzel A, Steiner S, Stalder H, Thiel V. Coronavirus biology and replication: implications for SARS-CoV-2. Nat Rev. (2021) 19:155-70. doi: 10.1038/s41579-020-00468-6
PubMed Abstract|CrossRef Full Text|Google Scholar
18. グラハムRL、バリックRS。Recombination, reservoirs, and the modular spike: mechanisms of coronavirus cross-species transmission. J Virol. (2010) 84:3146.論文番号: 10.1128/JVI.01394-09
PubMed Abstract|CrossRef Full Text|Google Scholar
19. Sola I, Almazan F, Zuniga S, Enjuanes L. Coronaviruses, Continuous and discontinuous RNA synthesis in coronaviruses. Annu Rev Virol. (2015) 2:265-88. doi: 10.1146/annurev-virology-100114-055218
PubMed Abstract|クロスリファレンス全文|Google Scholar
20. Rakosy-Tican E, Lörincz-Besenyei E, Molnár I, Thieme R, Hartung F, Sprink T, et al. New Phenotypes of potato co-induced by mismatch repair deficiency and somatic hybridization(ミスマッチ修復欠損と体細胞交雑が誘発するジャガイモの新しい表現型)。Front Plant Sci. (2019) 10:3. doi: 10.3389/fpls.2019.00003
PubMed Abstract|CrossRef Full Text|Google Scholar
21. Harmsen T, Klaasen S, van de Vrugt H, te Riele H. DNA mismatch repair and oligonucleotide end-protection promote base-pair substitution distal from a CRISPR/Cas9- induced DNA break. Nucl Acids Res. (2018) 46:2945-55. doi: 10.1093/nar/gky076
PubMed Abstract|CrossRef Full Text|Google Scholar
キーワード SARS-CoV-2スパイク、フリン切断部位、MSH3遺伝子、配列相同性、組換え性
引用元 Ambati BK, Varshney A, Lundstrom K, Palú G, Uhal BD, Uversky VN and Brufsky AM (2022) MSH3 Homology and Potential Recombination Link to SARS-CoV-2 Furin Cleavage Site. Front. Virol. 2:834808. doi: 10.3389/fviro.2022.834808.
Received: 2021年12月13日; Accepted: 2022年1月19日
公開:2022年2月21日
編集者
Xin Yin、中国農業科学院(CAAS)ハルビン獣医学研究所、中国
査読者
中国農業科学院(CAAS)ハルビン獣医学研究所 張潔涛
Copyright © 2022 Ambati, Varshney, Lundstrom, Palú, Uhal, Uversky and Brufsky. これは、クリエイティブ・コモンズ表示ライセンス(CC BY)の条件の下で配布されるオープンアクセス論文である。原著者および著作権者のクレジットを表示し、本誌の原著を引用することを条件に、他のフォーラムでの使用、配布、複製を許可する。これらの条件を満たさない使用、配布、複製は許可されない。
*Correspondence: Kenneth Lundstrom, lundstromkenneth@gmail.com
免責事項:本論文で述べられているすべての主張は、著者個人のものであり、必ずしも所属団体、出版社、編集者、査読者のものを代表するものではありません。この記事で評価される可能性のある製品やそのメーカーによる主張は,出版社によって保証または承認されたものではありません。
-------以上、ここまで論文和訳---------
以上で本文全訳でしたが、これについて概要を解説した記事↓
の全文和訳をAlzhackerさんが作成して下さり↓、これがとても読みやすく非常に分かりやすいので一読をお勧めです!
