分泌されたAkkermansia muciniphilaのスレオニルtRNA合成酵素が免疫恒常性を監視・調節する機能を発揮する
分泌されたAkkermansia muciniphilaのスレオニルtRNA合成酵素が免疫恒常性を監視・調節する機能を発揮する
https://www.cell.com/cell-host-microbe/fulltext/S1931-3128(23)00200-7
キム・スマン 18
パク・シンヒ 18
ファン・スンホ(Seung-Ho Hwang
キム・ソンフン
イ・ウォンジェ
キム・ミョンヒ 20
すべての著者を表示する
脚注を表示する
オープンアクセス公開日:2023年06月02日DOI:https://doi.org/10.1016/j.chom.2023.05.007
PlumX メトリクス
ハイライト
腸内細菌のAkkermansia muciniphilaは、スレオニルtRNA合成酵素(AmTARS)を分泌する。
分泌されたAmTARSはマクロファージを標的とし、抗炎症性のTLR2-CREB軸を活性化する。
AmTARSの進化的に獲得された領域がTLR2との特異的な相互作用を媒介する。
AmTARSはマクロファージの恒常性を回復させ、IL-10を増加させ、マウスの大腸炎を減衰させる。
まとめ
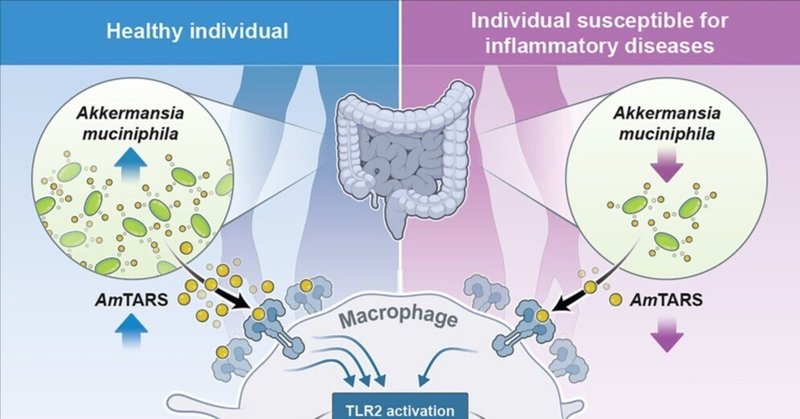
日和見菌は、炎症性疾患に対する耐性の確立に重要な役割を担っており、その分子機構は解明されつつある。生命界はすべてアミノアシルtRNA合成酵素(ARS)を産生する。これまで、ARSの非翻訳的な役割は、真核生物で多く報告されてきた。今回、我々は、腸内細菌Akkermansia muciniphilaのスレオニルtRNA合成酵素(AmTARS)が分泌され、免疫恒常性の監視と調節に機能していることを報告する。分泌されたAmTARSは、M2マクロファージの極性化を引き起こし、TLR2との特異的な相互作用を媒介する進化的に獲得された独自の領域を介して、抗炎症性IL-10の産生を組織化させる。この相互作用により、MAPKおよびPI3K/AKTシグナル経路が活性化され、CREBに収束することで、IL-10の効率的な産生と炎症メディエーターのNF-κBの抑制につながります。AmTARSはIL-10陽性マクロファージを回復させ、血清中のIL-10濃度を上昇させ、大腸炎マウスの病的作用を減弱させる。このように、常在型tRNA合成酵素は、恒常性を維持する内在性メディエーターとして機能することができる。

図解抄録
大きな画像を見る
高解像度画像をダウンロードする
キーワード
腸内細菌叢
常在菌
アッカーマンシア・ムチニフィラ
スレオニルtRNAシンテターゼ
抗炎症性免疫メディエーター
炎症
免疫ホメオスタシス
はじめに
宿主と腸内細菌叢の共進化により、腸内細菌叢は宿主のホメオスタシス維持に基本的な役割を果たす高度な相互依存関係が形成されています。
1
,
2
,
3
,
4
腸内細菌叢の乱れ(すなわち、ディスバイオシス)は、代謝および免疫プロセスに著しい影響を及ぼし、最終的には複数の代謝および炎症性疾患に関連する宿主生理に影響を及ぼすことを示す証拠が増えてきている。
5
,
6
,
7
腸内細菌叢とヒトの健康や疾患との関連、特に免疫系の形成との関連は明らかになりつつあるが、免疫の恒常性を制御・調整する分子決定因子やその基礎メカニズムはまだほとんどわかっていない。宿主の免疫を制御する常在細菌由来の分子に関する最近の包括的な研究では、短鎖脂肪酸やインドール誘導体などの食事誘導代謝物に焦点が当てられており、これらはシグナル伝達分子として作用し、宿主受容体やその他の標的分子を介して免疫恒常性を維持する。
8
,
9
,
10
,
11
常在菌の生物学的システムが宿主の免疫や生理とどのように関連しているのかについては、ほとんど知られていない。
すべての生命界では、アミノアシルtRNA合成酵素(ARS)と呼ばれる酵素が生成され、適切なアミノ酸を同種のtRNAに結合させる。
12
ARSは、遺伝情報を解読するために不可欠な酵素であり、タンパク質合成の際に、リボソーム内でペプチド鎖にアミノ酸を結合させる。しかし、過去数十年の間に蓄積された証拠から、これらの酵素は翻訳以外の生物学的役割を担っていることが示唆されている。
13
,
14
,
15
,
16
真核生物の細胞質ARSには、機能の多様性とシステムの複雑性に関連するドメインとモチーフが追加されている。
13
,
14
,
15
,
16
,
17
特に、トリプトファニルtRNA合成酵素1(WARS1[真核生物のARSは、対応するアミノ酸の1文字コードに続いて、細胞質酵素は「ARS1」、ミトコンドリア酵素は「ARS2」で略記])などの哺乳類のARSは分泌され、免疫制御などの生理的活動に関与していると考えられるが、その分泌機構はまだ不明である。
18
,
19
,
20
,
21
ARSは、その多面的な役割から、他の生物学的役割と同様に、翻訳を調整することによって恒常性を維持するメディエーターと考えられている。
13
,
15
,
16
,
17
これまで、ARSの非翻訳的役割に関する研究は、主に真核生物に焦点が当てられてきた。これまで、宿主のホメオスタシスを制御する常在菌のARSの役割については、ほとんど注目されてこなかった。
我々は、腸内常在菌のARSが免疫制御を介して宿主のホメオスタシスの維持に寄与している可能性があると仮定した。A. muciniphilaは、Verrucomicrobia門の中で、生後1年以内にヒトの腸内に安定的に定着する唯一の種として知られています。
22
多くの研究が、A. muciniphilaの欠如または存在量の減少と複数の疾患との間に相関関係があることを示している23。
23
したがって、この細菌は、炎症や代謝障害に伴う代謝や免疫の障害を持つ人々の健康増進に役立つという理由で、関心が高まっている。
24
,
25
,
26
,
27
,
28
,
29
,
30
そこで、本研究では、この生物が産生するARSが果たす恒常的な役割について検討することを目的とした。その結果、A. muciniphilaは通常の定常状態においてスレオニルtRNA合成酵素(以下、AmTARS)を分泌し、分泌されたAmTARSはToll様受容体2(TLR2)-cAMP応答要素結合タンパク質(CREB)抗炎症免疫軸を活性化して免疫恒常性メディエーターとして機能することがわかった。これらの結果から、常在性ARSは内在性免疫メディエーターとして共進化した可能性があり、宿主と腸内細菌叢の相互作用の進化に新たな光を与えることが期待されます。
研究成果
A. muciniphilaはAmTARSを分泌し、抗炎症性免疫応答を促進する。
常在菌のARSの免疫制御への関与を調べるため、A. muciniphilaのARS(AmARS)を精製し、骨髄由来マクロファージ(BMDM)に対する抗炎症活性を試験しました。その結果、AmTARSは他のAmARSと異なり、定常状態においてBMDMsによる抗炎症性IL-10の産生を有意に増加させることを見出した(図1A)。さらに、Bacteroides fragilis、Escherichia coli、Ruminococcus bromiiなど他の代表的な腸内常在菌やヒト由来のTARSで処理したBMDMsは、IL-10を生成しなかったことから、免疫制御におけるAmTARSの特定の役割が示唆されます(図1B)。次に、AmTARSが細胞外に分泌され、免疫調節に関与しているのかどうかを検討した。AmARS特異的抗体を用いた免疫測定により、A. muciniphilaは通常の定常状態において、チロシルtRNA合成酵素(AmYARSをコントロールとして使用)ではなく、AmTARSを分泌することがわかった(図1C)。グラム陰性菌同士のコミュニケーションは、可溶性メディエーターや細胞外小胞(EV)の分泌を介することが多い、
31
であり、A. muciniphilaも同様である。
32
そこで、AmTARSを調べたところ、培養上清から精製したEVではなく、可溶性画分に多く検出された(図1D、S1A-S1C)。
図1A. ムチニフィラは細胞外からAmTARSを分泌し、抗炎症性免疫制御を行う
キャプションの全文を表示する
大きな画像を表示する
高解像度画像をダウンロードする
注目すべきは、AmTARSは正常な条件下でBMDMsによる抗炎症性IL-10の産生を誘導したものの、炎症性サイトカインIL-6とTNF-αの産生には影響を与えなかったことです(図1E-1G)。さらに、AmTARSは、リポポリサッカライド(LPS)によるBMDMsの刺激後、IL-10レベルを増加させ、IL-6とTNF-αをさらに大きく抑制しました(図1H-1J)。同様の結果は、正常および炎症(LPS)条件下でAmTARS処理したTHP1細胞でも観察された(図S1D-S1I)。以上のことから、常在菌であるA. muciniphilaは、AmTARSを分泌して宿主の免疫恒常性を維持(正常な条件下)、誘導(炎症条件下)していることが示唆されました。
AmTARSはIL-10陽性マクロファージの分極を活性化することで抗炎症作用を発揮する
消化管の慢性疾患である炎症性腸疾患(IBD)は、遺伝的要因、微生物、環境要因の複雑な相互作用から生じ、異常な免疫反応と炎症につながるものである。
33
,
34
,
35
統合ヒトマイクロバイオームプロジェクト(iHMP)内の炎症性腸疾患マルチオミクスデータベース(IBDMDB)を用いた最近のメタアナリシスにより、IBD患者と非IBDコントロールからの縦断的なマルチオミクスデータセットが得られた。IBDにおける腸内細菌生態系の包括的な概要を提供し、疾患活動中に義務的嫌気性菌を犠牲にして通性嫌気性菌が特徴的に増加することを同定しています。
36
また、嫌気性常在菌であるA. muciniphilaの存在量とIBDを含む炎症および代謝異常との間に逆相関があることがいくつかの研究で示されている、
37
,
38
,
39
,
40
我々は、IBDMDBを利用して、A. muciniphilaの存在量とIBDとの相関を再評価した。これまでの報告と同様に、IBDMDBメタゲノム解析の結果、非IBDコントロールと比較して、潰瘍性大腸炎(UC)またはクローン病(CD)の被験者では、A. muciniphilaの存在量が著しく低いことが判明した(図S2A)。さらに機能プロファイリングを行った結果、総菌体TARSの存在量はIBDの被験者で有意に低いわけではないことが判明しました(図S2B)。しかし、個々の種で層別化すると、AmTARSのレベルは、UCまたはCDの患者では、非IBDのコントロールよりも有意に低いことが判明した(図S2C)。他のAmARSのレベルもUCまたはCDの被験者で低く(図S2D)、AmARSのレベルはA. muciniphilaの存在量と相関していることが示唆された。さらにメタトランスクリプトーム解析を行ったところ、IBDの有無にかかわらず、すべての細菌からのTARSの存在量に差はなかったが、UC被験者のAmTARSレベルは非IBD被験者のそれよりも有意に低かったこともわかった(図S2EおよびS2F)。上位10貢献者のTARSの発現レベルは、IBD被験者と非IBD被験者の間で有意な差はなかった(図S2G)。メタゲノミクスの結果と一致して、その他のAmARSの発現量は、IBD対象者では非IBD対象者よりも低いことがわかった(図S2H)。全体として、これらのデータは、A. muciniphilaの存在量の減少がAmTARSの低レベルと関連しており、IBD患者においてAmTARSを介した抗炎症免疫応答の調節不全をもたらすことを示唆している。
この仮説を検証するため、デキストラン硫酸ナトリウム(DSS)誘発大腸炎モデルマウスにおいて、AmTARSの抗炎症機能を評価しました。まず、腹腔内投与されたAmTARSの生体内での安定性を評価した。AmTARSは投与後3日間観察された(図S3AおよびS3B)。その後、AmTARS(0.05~0.4 mg kg-1)を1日おきに10日間i.p.投与した後の用量依存性反応(すなわち、体重および結腸長の変化)を評価しました(図2A)。全体として、AmTARSの投与は、体重減少および結腸短縮を用量依存的に(0.2 mg kg-1まで)減衰させた(図S3CおよびS3D)。0.4mgkg-1の用量でAmTARSを投与すると、最大7日間体重減少が有意に減衰したが、10日目にはリン酸緩衝生理食塩水(PBS)で処理した大腸炎マウスと同様の結果が得られたことから、高用量のAmTARSは副作用がある可能性が示唆された(図S3CとS3D)。そこで、AmTARSの抗炎症免疫効果のさらなるin vivo評価に0.2 mg kg-1の用量を用いた。AmTARSのi.p.投与は、体重減少および結腸短縮に加え、大腸炎マウスの疾患活動スコアを改善した(図2B、S3E、およびS3F)。特に近位結腸における上皮内壁の破壊と損傷スコアは、AmTARSを投与した大腸炎マウスでコントロールマウスより有意に低かった(図2Cおよび2D)。
図2AmTARSはIL-10陽性マクロファージの活性化により、DSS誘発大腸炎マウスを改善する
キャプションの全文を表示する
大きな画像を表示する
高解像度画像をダウンロードする
AmTARSが標的とする細胞を特定するため、マウスに蛍光標識したAmTARSをi.p.-注射し、腹膜滲出液細胞(PECs)の蛍光陽性細胞をフローサイトメトリーで解析しました。蛍光標識Alexa568-AmTARSは、T細胞(CD3+)やB細胞(CD19+)よりもむしろマクロファージ(F4/80+)に優位に検出されました(図2E)。次に、Mac2免疫蛍光抗体を用いて、i.p.注射されたAmTARSが腸管マクロファージを標的とするかどうかを検証した。まず、抗6X Hisタグ抗体が投与されたHisタグ付きAmTARS(抗AmTARS抗体を用いて検出)と特異的に相互作用するかどうかを検証しました。HisタグとAmTARSは明確に共局在しており、抗6X Hisタグ抗体は投与されたAmTARSを特異的に検出することが示された(図S3G)。Mac2の強い発現は、大腸上皮と、前膜に位置するマクロファージで検出された。投与したAmTARSは主に前膜のマクロファージと共局在し、AmTARSが腸のマクロファージを標的としていることが示された(図2F)。また、全身投与されたAmTARSが、滞在型F4/80陽性マクロファージに直接作用するか、全身刺激された腹膜マクロファージ集団から遊走した細胞に作用するかについても疑問を呈した。そこで、CD45.1マウスとCD45.2マウスをそれぞれレシピエントとドナーとして用い、養子縁組実験を実施した。CD45.2マウスのマクロファージ(F4/80+CD45.2+)を含むPECを、CD45.1マウスに腹腔内移植した。PBSまたはAmTARSを投与したCD45.1マウスの腹腔内には、5日目および10日目にそれぞれ、移植したPECs集団のマクロファージが観察された。しかし、CD45.1マウスの大腸では、移入されたマクロファージは検出されなかった(図S3H)。これらのデータから、全身投与されたAmTARSは、滞在型マクロファージに直接作用するようであることが示唆された。腹腔と腸からマクロファージを単離し、AmTARSを介した抗炎症性IL-10の産生をさらに検証した(図2G、2H、S3IおよびS3J)。
M2マクロファージの極性化はIL-10のような抗炎症性サイトカインの産生に先行するため、マクロファージの極性化に対するAmTARSの影響を評価した。BMDMをAmTARSの非存在下および存在下でPBS(ネガティブコントロール)、LPS(10 ng mL-1;炎症を誘発)、またはIL-4/IL-13(M2極性を誘発;ポジティブコントロール)で処理し、得られたM2マクロファージ集団をM2マクロファージ表面マーカーCD206に対する特異的抗体を用いたフローサイトメーターで測定した。注目すべきことに、AmTARSはPBS処理した細胞でもM2極性を活性化し、LPS誘発炎症条件下で細胞の著しいM2極性を誘導した(図2Iおよび2J)。M2偏光は、既報の通り、IL-4/IL-13によって完全に誘導された、
41
,
42
は、AmTARS の非存在下および存在下で、IL-4/IL-13 によって完全に誘導された(図 2I および 2J)。その結果、AmTARSは大腸炎による炎症性単球(CD11b+F4/80-)の浸潤を有意に抑制し、大腸細胞内のIL-10陽性マクロファージ(CD11b+F4/80+IL-10+)数を回復させ、血清中のIL-10レベルを増加した(図2K - 2N)。
AmTARSのマクロファージ依存的な大腸炎改善機能をさらに検証するため、マクロファージが減少した大腸炎マウスでAmTARSの抗炎症作用を検討しました。生理学的データと一致して、AmTARSはマクロファージ枯渇マウスの炎症性単球やIL-10陽性マクロファージの数に影響を与えなかった(図S4A-S4C)。したがって、AmTARSは、マクロファージ欠失マウスにおいて、大腸炎による体重減少、大腸の短縮、損傷スコア、疾患活動性スコア、上皮内層の破壊に影響を与えず(図2O、2P、S4D-S4F)、AmTARSのマクロファージ特異的機能がさらに確認された。これらの結果を総合すると、AmTARSはIL-10陽性マクロファージの分極を活性化することにより、抗炎症サイトカイン的な役割を担っていることがわかります。
AmTARSはユニークな領域を同化して免疫調節機能を発揮する
AmTARSの抗炎症免疫機能の分子メカニズムを明らかにするため、ヒトTARS1および腸内細菌叢を代表する細菌種のTARSのアミノ酸配列と比較した。AmTARSは、N末端(99-113残基、以下U1)および触媒(290-312残基、以下U2)ドメイン内に、他のTARSにはない独自の領域を2つ追加している(図S4G)。また、Blastベースの配列検索(https://blast.ncbi.nlm.nih.gov/Blast.cgi)では、固有領域と有意な相同性を持つ配列は検出されなかったことから、これらの固有領域はA. muciniphilaが免疫制御に特化して進化的に同化させた可能性がある。
次に、AlphaFoldを用いてAmTARSの構造をモデル化しました。
43
を用いた構造モデルを作成したところ、ユニークな領域を除き、Staphylococcus aureus TARS (PDB: 1NYQ) やヒトTARS1 (PDB: 4HWT) など他のTARSと大きな構造類似性を示した(図S4Hおよび図S4I)。この構造モデルから、AmTARS内のU1およびU2はともに表面に露出しており、シグナル受容体などの宿主ターゲットとの相互作用に関与している可能性が示唆された(図3A)。そこで、これらのユニークな領域を欠損させた変異タンパク質(ΔU1、ΔU2、ΔU1ΔU2)を作製し、U1およびU2の機能性を評価しました。いずれの変異タンパク質も、酵素活性に関しては野生型(WT)AmTARSと異なるが(図3B)、BMDMsにおけるIL-10産生を消失させた(図3C)。その結果、いずれの変異タンパク質も、大腸炎誘発マウスにおいて、体重減少、疾患活動性スコア、大腸の短縮、または上皮内層の破壊および損傷スコアの回復を促進しなかった(図3-3F、S4J、およびS4K)。大腸炎誘発性炎症性単球を減少させ、大腸のIL-10陽性マクロファージを増強するAmTARSの正の免疫制御機能は、変異AmTARSを大腸炎マウスに投与すると消失した(図3GおよびS4L)。WT AmTARSと同様に、ΔU1ΔU2変異体は、大腸炎誘発体重減少、疾患活動性スコア、またはマクロファージ枯渇マウスの結腸の短縮に影響を与えなかった(図S4M-S4O)。このように、これらのデータは、AmTARS内のユニークな領域U1およびU2が、その抗炎症機能に必須であることを明確に示している。
図3AmTARSのユニークな領域は抗炎症性免疫制御に必須である
キャプションを表示する
大きな画像を見る
高解像度画像をダウンロードする
AmTARSはTLR2リガンドとして、抗炎症性免疫応答を活性化する
AmTARSの宿主標的を同定するために、THP1細胞を用いて膜タンパク質インタラクトーム解析を行いました。自然免疫に関わる受容体であるいくつかの相互作用候補が同定され(図S5A)、プルダウンアッセイを用いてAmTARSとの相互作用が評価された。その結果、試験した候補の中で、AmTARSはTLR2のみと相互作用することが示唆された(図4A、S5B、S5C);したがって、TLR2はIL-10を介した抗炎症免疫調節を引き起こすためにAmTARSが標的とする受容体である可能性がある。対照的に、プルダウンアッセイは、AmTARSとTLR4との間の相互作用を明らかに示さなかった(図S5D)。AmTARSとTLR2との間の相互作用をさらに検証するために、精製TLR2およびAmTARSタンパク質を用いてin vitroプルダウンアッセイを実施した(図4B)。その結果、AmTARSがU2を含む触媒ドメイン(CD)を介してTLR2と直接相互作用することが明確に示された。U2を欠失したCD(ΔU2CD)タンパク質とTLR2との相互作用は著しく弱かった。U1を含むN末端ドメイン(NTD)とTLR2との間には直接的な相互作用は見られなかった。TLR2は、TLR1またはTLR6とホモ二量体またはヘテロ二量体を形成することで機能する。
44
したがって、U1はヘテロ二量体との相互作用に関与している可能性がある。
図4AmTARSはTLR2依存の抗炎症性免疫メディエーターである
キャプションの全文を表示する
大きな画像を見る
高解像度画像をダウンロードする
AmTARSのTLR2依存性免疫機能は、TLR2ノックアウト(KO)マウスを用いてさらに確証された。AmTARSはTLR2-KOマウスのBMDMにおいてIL-10産生を誘導せず、AmTARSとTLR2の相互作用が抗炎症免疫活性化に不可欠であることが示された(図4C)。さらに、BMDMsにおいて抗炎症免疫機能を持たない大腸菌TARS(EcTARS)をエンジニアリングすることにより、AmTARSのTLR2依存性免疫機能に対するユニーク領域の必要性を確認しました(図1B)。AmTARSのU1およびU2配列をEcTARSの対応する領域に組み込むこと(EcTARS(U1/U2))で、TLR2との相互作用を介したEcTARSでのIL-10産生を誘発する能力を付与した(図4Dおよび図4E)。しかし、EcTARS(U1/U2)によるIL-10産生は、TLR2-KOマウスのBMDMでは消失しており、AmTARSがTLR2依存的に固有の領域を介して宿主免疫細胞を制御することをさらに示す証拠となりました(図4D)。さらに、EcTARS(U1/U2)を用いて、in vivoでのユニーク領域の機能性を確認した。DSS誘発大腸炎マウスに、BMDMにおけるAmTARSと比較してIL-10を誘導する能力に基づいて、0.4 mg kg-1の用量でEcTARS(U1/U2)をi.p.-注射した(図4D)。EcTARS(U1/U2)のi.p.投与により、体重減少、コロンの短縮およびコロンの上皮破壊は大腸炎マウスのAmTARS投与と同様に改善されました(図S5E-S5G)。EcTARS(U1/U2)は、大腸のIL-10陽性マクロファージを有意に回復させ、血清中のIL-10レベルを増加させた(図S5HおよびS5I)。これらのデータを総合すると、AmTARS内のユニークな領域U1およびU2が、その抗炎症免疫機能に必須の前提条件であることが強く証明された。
続いて、AmTARSの機能におけるTLR2の役割をin vivoで評価しました。TLR2-KOマウスは、既報の通り、DSS誘発大腸炎に対してWTマウスよりもわずかに感受性が高かった。
45
それにもかかわらず、TLR2-KOマウスの大腸炎誘発体重減少および疾患活動性スコアは、AmTARS処理後に改善しなかった(図4FおよびS5J)。大腸の長さの短縮、上皮内壁の破壊、および損傷スコアについても同様であった(図4G、4H、およびS5K)。これらの結果と一致して、AmTARSは、大腸炎TLR2-KOマウスにおいて、炎症性単球数の減少もIL-10分泌マクロファージ数の増加も誘発しなかった(図4Iおよび図4J)。さらに、AmTARSは、非炎症条件下で、TLR2-KOマウスの腹膜マクロファージのM2マクロファージ分極を引き起こさなかった(図4K)。同様に、TLR2-KOマウスから単離したPECでは、M2マクロファージは観察されなかった(図4L)。これらの結果は、AmTARSが宿主の免疫恒常性の維持に必要な常在菌由来のTLR2リガンドであることを強く示唆している。
AmTARS/TLR2はCREBを活性化することで効率的に抗炎症応答を誘導する
TLRは微生物感染から宿主を守るために炎症を刺激する重要な役割を担っていますが、これらの受容体は、過剰な炎症性サイトカイン産生を防ぎ、炎症を解決するために重要な抗炎症シグナルも制御しています。
46
,
47
TLRは、マクロファージにおいて、マイトジェン活性化プロテインキナーゼ(MAPK)またはホスホイノシチド3キナーゼ(PI3K)/プロテインキナーゼB(AKT)経路を介して抗炎症シグナルを引き起こす、
48
が、CREBを活性化する経路に集約される。
49
この転写因子CREBは、マクロファージによる抗炎症性IL-10の産生を含む多くの細胞応答を制御している。
50
,
51
,
52
AmTARSによって引き起こされるシグナル伝達経路を特定するために、まずキナーゼとCREBを評価した。細胞外シグナル制御キナーゼ(ERK;PD98059)、マイトジェン活性化プロテインキナーゼp38(p38;SB203580)、マイトジェン・ストレス活性化キナーゼ(MSK)1/2(SB747651A)、CREB(666-15)、PI3K(wortmannin)、AKT(AKT1/2)の阻害剤で処理したBMDMsで、続いてAmTARSで刺激する前にIL-10の生産とM2極性を測定した.これらの分子のそれぞれを阻害すると、AmTARSによるIL-10産生(図5A)およびマクロファージ極性化(CD206陽性)が有意に抑制され(図5B)、AmTARSがMAPKおよびPI3K/AKTシグナル経路をトリガーして抗炎症応答を誘導することが示された。
図5AmTARSはCREBを活性化することで効率的に抗炎症応答を促進する
キャプションの全文を表示する
大きな画像を表示する
ハイレゾ画像のダウンロード
続いて、BMDMsとTHP1細胞の両方において、AmTARS/TLR2が誘導するシグナル分子の時間依存的な活性化を測定するために、イムノブロッティングアッセイを実施しました。AmTARSは15分以内にERK、p38、MSK1/2を急速に活性化し、その後、CREBのSer133でのリン酸化(IL-10産生に必須)が起こった。
52
BMDMs(図5C)およびTHP1細胞(図5D)において、30分以内にERK、p38、MSK1/2を急速に活性化した。次に、30分間刺激したBMDMsにおいて、AmTARSがトリガーするTLR2シグナルを、Pam3CSK4(合成カノニカルTLR2/1アゴニスト、以下Pam3という)を用いた典型的なリガンドトリガーシグナルと比較しました。全体として、AmTARSをトリガーとする経路のシグナル伝達分子のTLR2依存的な活性化パターンは、Pam3によってトリガーされた分子のそれと同様であり、これはTLR2抗体によって完全にブロックされた(図5E)。しかし、CREBの活性化は、Pam3処理したBMDMよりもAmTARS処理したBMDMで有意に大きく観察された(図5E)。対照的に、NF-κBの活性化は、AmTARS処理BMDMよりもPam3処理BMDMでより大きかった(図5E)。Ser133でリン酸化されたCREBは、共有のコアクチベーターであるCREB結合タンパク質(CBP)/p300の限られた量を競合することによってNF-κB活性化を阻害し、それによって炎症反応を抑制する。
49
,
53
,
54
我々の結果は、AmTARSが炎症条件下のマクロファージにおいて、IL-10の産生を誘導し、さらには炎症性サイトカインIL-6とTNF-αの産生を抑制することを示しています(図1H-1J、S1G-S1I)。これらのデータは、典型的なTLR2リガンドとは異なり、AmTARSが腸内常在菌由来のTLR2リガンドであり、炎症性シグナルを制限することで免疫恒常性を維持することを示しています。
AmTARSは、BMDM(図5F)およびTHP1細胞(図5G)において、15分以内にPI3KおよびAKTを活性化し、その後、AKTによるSer9でのリン酸化を介してGSK3β活性を阻害した。TLR刺激によるGSK3βの阻害は、NF-κBではなく、コアクチベーターCBPとCREBの間の結合も増加させ、それによってIL-10産生を増加させ、炎症性サイトカインの放出を抑制する。
55
したがって、これらのデータは、AmTARSとTLR2の相互作用が、MAPKおよびPI3K/AKTシグナル伝達経路をトリガーしてCREBを効率的に活性化することを示しています。
TLR2は活性化時にTLR1またはTLR6とヘテロ二量体を形成するため、AmTARSがどちらの形態を標的とするかを評価した。イムノアッセイでは、AmTARSが誘導するTLR2シグナルは、TLR1、TLR6、およびTLR2を標的とする抗体によって遮断されることが示され、AmTARSがTLR2/1とTLR2/6ヘテロダイマーとTLR2ホモダイマーの両方を標的にして抗炎症シグナルを活性化していることが示されました(図5Hと図5I)。
次に、AmTARSまたはPam3で1時間または3時間処理したBMDMのグローバル遺伝子発現プロファイリングを実施しました(図6A)。主成分分析(PCA)により、1時間ではAmTARSとPam3で同様の遺伝子発現パターンを示したが、3時間では明らかに異なるプロフィールを示した(図6B)。AmTARSによって発現が影響を受けた遺伝子の数およびその(アップまたはダウン)調節の程度は、Pam3によって影響を受けた遺伝子と大きく異なっており、AmTARSが典型的なTLR2リガンドとは異なる免疫調節因子として作用することがさらに示された(図S6A)。AmTARSとPam3の間で異なる発現パターンを示す遺伝子のサブセットは、階層的クラスタリング分析によって6つのクラスターに分類された(図S6AおよびS6B)。特に3時間後の遺伝子発現パターンは、クラスター3と4で異なっていた(図6C、S6A、およびS6B)。一般に、クラスター3および4に属する遺伝子は、それぞれ炎症および抗炎症に関連している(図6C)。
図6AmTARSは定常的な、免疫恒常性メディエーターである
キャプションの全文を表示する
大きな画像を見る
高解像度画像をダウンロードする
遺伝子セット濃縮解析により、Pam3処理したBMDMでは炎症反応関連遺伝子が有意に誘導され、過去の報告結果と一致した
51
,
56
(図6D)。しかし、AmTARS処理細胞では、炎症反応関連遺伝子の誘導は見られなかった(図6D)。注目すべきは、Pam3によるBMDMの処理は、NF-κB、RELA、TFAP2Aなどの炎症関連転写因子のアップレギュレーションと有意に関連していたが、AmTARS処理は、抗炎症関連転写因子CREBのアップレギュレーションと関連していた(図6E)。これらの遺伝子発現プロファイルは、IL-10の定常産生を介して免疫恒常性を維持するAmTARS-TLR2-CREB軸媒介の抗炎症シグナルと正の相関を示した。
我々は、インビボでNF-κB活性をモニタリングできるTg(NFκB:EGFP)ゼブラフィッシュトランスジェニックラインを用いてAmTARSの抗炎症機能を検証した。
57
遺伝子セット濃縮解析パターン(図 6D と 6E)およびシグナル伝達経路の結果(図 5C-5G) と一致し、DSS の反復投与により誘発されたゼブラフィッシュ幼虫の遠位腸における NF-κB 活性の増加は、AmTARS により有意に減衰した(図 6F-6H) ことから、AmTARS が生体内の炎症反応に対して抑制機能を持つことが示された。
EcNを分泌するAmTARSは、大腸炎マウスの炎症を有意に改善する。
大腸菌ニッスル1917(EcN)は、本質的に抗菌・抗炎症作用を持つプロバイオティクスとして知られている細菌である。
58
EcNは、治療用の人工細菌として開発される候補として急速に浮上しています。
59
そこで、AmTARSの生体内における生理機能をさらに検証するために、この細菌を操作した。まず、染色体上でEcNのTARSをAmTARSに置換することで、AmTARSを分泌する細菌を作製することを試みた。しかし、アミノアシル化に必要なEcN TARSの同族tRNAがAmTARSと適合しない可能性があるため、この置換はEcNにとって致死的であった。なお、ARSは認識するアミノ酸やtRNA基質の選択に関して非常に特異的であり、生物によって異なる認識様式を持つ。そこで、我々は次に、染色体の異なる位置に異所的に発現する追加のTARS(すなわち、AmTARS)を含む染色体工学的EcNを開発した。AmTARS遺伝子とlacプロモーターは、他の研究で以前から利用されているexo/cea収束遺伝子間領域に挿入された
60
,
61
したがって、隣接する遺伝子の発現を妨害することはない(図S6C)。AmTARS の C 末端には、さらなる解析に使用するために 6X His タグを付加した。これは、オリジナルのAmTARSにもシグナルペプチドがないためである。実際、非翻訳的な機能を持つARSの分泌はヒトでも観察されているが、そのメカニズムは不明である。
18
,
19
,
21
,
62
人工EcN(EcN(AmTARS))はA. muciniphilaよりも有意に低いレベルでAmTARSを細胞外に分泌した。実際、EcN(AmTARS)の40倍濃縮培養上清中のAmTARS量はA. muciniphilaが分泌したものと同じだった(図 7A).その後、Hisタグを用いて、EcN(AmTARS)を経口投与したDSS誘発大腸炎マウスの大腸において、本工作細菌が分泌したAmTARSを評価した。免疫蛍光染色により、人工EcNを分泌したAmTARS(His)の多くが大腸の固有層に存在するマクロファージ(F4/80陽性)と共局在することが明らかになり(図7B、7C)、AmTARSが腸内のマクロファージを標的としていることが確認されました。
図7EcN(AmTARS)の経口投与はDSS誘発大腸炎を緩和する
キャプションの全文を表示する
大きな画像を見る
高解像度画像をダウンロードする
EcN(AmTARS)の経口投与は、大腸炎誘発体重減少、大腸長短縮、疾患活動性スコア、上皮内壁の破壊、損傷スコアを有意に改善した(図7D-7F、S6D、およびS6E)。さらに、炎症性単球による大腸炎誘発浸潤を有意に減少させ、IL-10を分泌するマクロファージを回復させ、大腸炎マウスの血清中のIL-10レベルを顕著に増加させた(図7G-7I)。
これらのデータを総合すると、常在菌A. muciniphilaが分泌するAmTARSは、典型的なTLR2リガンドとは異なる定常免疫恒常性メディエーターであり、炎症チャレンジに応答して腸内の居住マクロファージの調節を介して免疫恒常性を維持・誘導するというさらなる証拠が得られた(図7J)。
考察
ヒトの腸内には1013以上の細菌が生息しており、宿主との相互関係を形成することで、特に腸の生理学に関連した多くの利益をもたらしている。
63
,
64
腸管免疫活性の形成に失敗すると、IBDなどの慢性炎症性疾患と関連する炎症反応が制御不能になり、過剰な炎症反応が起こる。
5
,
6
,
7
食事に依存する栄養素や常在菌由来の短鎖脂肪酸などの代謝産物が宿主免疫の重要な制御因子であることはよく知られているが、
8
,
9
は、宿主免疫の重要な制御因子であることはよく知られているが、宿主の生体システムそのものの関与は未解明である。
免疫系は、有益な常在菌と病原体を識別し、食物抗原を認識することができ、免疫応答と耐性のバランスをとりながら腸の恒常性を維持する。
65
,
66
通常の定常状態では、TLRは常在菌を感知することで、これらの事象に重要な役割を果たします。
45
抗炎症サイトカインIL-10とその主要生産者であるマクロファージは、腸のホメオスタシス維持に重要な役割を担っています。
67
,
68
,
69
,
70
,
71
このように、常在菌、TLR、IL-10の相互作用は、常に炎症にさらされる環境において、恒常的なバランスを保つために必要な免疫反応を誘発し、形成する重要な軸となる可能性があります。さらに、この複雑な相互作用における重要な調節因子は、まだ特定されていない。
一般的に、TLR2は、リポペプチドやリポテイコ酸などの古典的な細菌成分を認識し、宿主保護に必要なNF-κBを介した炎症などの自然免疫応答を開始する。
72
最近の研究では、TLR2は、マウスの腸内細菌叢の一員であるHelicobacter hepaticusが産生する大型の可溶性多糖類も認識することが示されている。
51
多糖類を認識したTLR2は、腸管マクロファージにおいてMSK/CREBシグナルを誘発し、それによりIL-10の早期産生を誘導し、炎症性チャレンジに対する耐性を促進する。TLR2リガンド多糖体は、典型的なリガンドであるPam3とは異なる機能を持つ。実際、Tlr2-/-BMDMsではIL-10の産生を誘導できなかったが、WT、Tlr1-/-およびTlr6-/-細胞ではできなかった。TLR2を介して引き起こされる腸内常在菌関連の免疫寛容イベントのもう一つの代表的な例は、Bacteroides fragilisが分泌する多糖類A(PSA)を介するものである。Pam3のような典型的なTLR2リガンドとは異なり、分泌されたPSAは、TLR2を通じてCD4+ T細胞を活性化することによりIL-10の産生を促進する。
65
B. fragilis分泌のPSAは、WT、Tlr1-/-、Tlr6-/- CD4+T細胞で大量のIL-10産生を誘導するが、Tlr2-/-細胞では誘導しない。
65
A. muciniphilaは、TLR2特異的なリガンドとして作用し、炎症性チャレンジから保護するために免疫恒常性を監視・誘導する免疫メディエーター、AmTARSの定常分泌を示すことをここに報告する。PSAや可溶性多糖類と同様に、AmTARSは抗炎症性免疫シグナルを引き起こすが、それらのリガンドとは異なる機能を持つようである。TLR欠損細胞実験から、B. fragilisが分泌するPSAとH. hepaticusが生産する可溶性多糖類は、ホモ二量体のTLR2のみを標的とすることが示唆されているが、AmTARSはTLR2/2、TLR2/1、TLR2/6を標的にして抗炎症シグナルを引き起こす(図5Hおよび5I)。TLR2に対する特異性に関するAmTARSの特徴は、MAPKとPI3K/AKTの両方のシグナル伝達経路を通じてCREBを活性化することにより、免疫恒常性を効率的に誘導することができると考えられます。このように、これらの実験から、宿主との相互作用を積極的に刺激し、抗炎症性TLRシグナルを介して腸のホメオスタシスを維持する腸内生息生物の存在が明らかになりました。さらに、これらの研究は、TLR2が常在菌とのコミュニケーションによって腸のホメオスタシスを維持する主要な受容体として働く可能性を示唆しています。
嫌気性細菌であるA. muciniphilaは、ヒトや動物の消化管に生息するVerrucomicrobia門の唯一の既知の種である、
73
この菌の多さは、代謝障害や炎症性疾患と逆相関している。
37
,
38
,
39
,
40
一般に、疣贅菌の仲間はホモセリンからスレオニンの合成に必要な酵素をコードしているが、A. muciniphilaのゲノムには、L-スレオニンが増殖に必須であるにもかかわらず、これらの酵素をコードする遺伝子は存在しない。
74
,
75
スレオニンはムチンに多く含まれるため、この細菌の粘膜環境への適応は、スレオニン補助栄養の結果である可能性がある。
76
A. muciniphilaは、ムチン分解酵素の広範なレパートリーを産生することができることは、注目に値する。
74
AmTARSの標準的な機能は、タンパク質合成のためにスレオニンを対応するtRNAに細胞内で共有結合させる触媒である。我々は、AmTARSの定常的な分泌は、生物のスレオニン補助栄養に関連していると考えられるが、同時に腸の免疫状態を監視することができるのではないかと推測している。この仮説は、まだ決定的な証明には至っていない。しかし、今回の発見は、A. muciniphilaと腸内環境との絶妙な相互依存関係を浮き彫りにし、宿主の免疫恒常性に寄与している。
IBDの発症率は世界中で急速に増加しており、IBDは世界的な新興疾患となっています。
77
,
78
IBDの発症には、腸内細菌叢の変化、あるいは細菌叢に対する免疫応答の異常が重要な役割を果たすことが明らかになっています。
36
,
79
IL-10の働きは、腸のホメオスタシスの維持に重要な役割を担っている
69
,
80
,
81
このように、IBDの治療薬として期待されています。
82
AmTARSはIL-10の産生につながる抗炎症性免疫メディエーターとして機能することから、IBDのバイオマーカーおよび治療薬としての可能性が示唆されました。実際、大腸炎を発症したマウスにおいて、体重減少、疾患活動性スコア、大腸長短縮、上皮内壁の破壊、損傷スコアなどの大腸炎病態に対するAmTARSの治療効果は、IL-10と同等だった(図S7A~S7E)。AmTARSはIL-10と同様に大腸炎による炎症性単球の浸潤を抑制した(図S7FおよびS7G)。また、全身投与されたAmTARSは腸の炎症を調節することから、関節リウマチや全身性エリテマトーデスなどの疾患における全身性の炎症も制御できる可能性があります。
以上のことから、本研究は、常在性ARSが、定常状態のIL-10を介した抗炎症性免疫制御を誘発することにより、免疫恒常性の維持に重要な役割を果たすことを明らかにした。同時に、本研究は、腸内細菌と宿主の相互作用というユニークな視点を提供し、宿主ARSがホロビオントの恒常性を形成する内在性メディエーターとして機能する可能性を示した。したがって、本研究で得られた知見は、IBDを含む様々な炎症性疾患の予防・治療戦略につながる可能性がある。
STAR★メソッド
キーリソース表
REAGENT or RESOURCEIDENTIFIRAntibodiesMouse monoclonal anti-StrepIBACat# 2-1509-001Mouse monoclonal anti-FLAGCell signaling technologyCat# 8146; RRID: AB_10950495Rabbit monoclonal anti-β-ActinCell signaling technologyCat# 12620; RRID: AB_2797972Rabbit polyclonal anti-phospho-ERK1/2 (T202/Y204)Cell signaling technologyCat# 9101; RRID: AB_331646ウサギモノクローナル抗ERK1/2Cell signaling technologyCat# 4695; RRID: AB_390779ウサギモノクローナル抗ホスホ-p38 MAPK (T180/Y182)Cell signaling technologyCat# 4511; RRID: AB_2139682Rabbit monoclonal anti-p38 MAPKCell signaling technologyCat# 8690; RRID: AB_10999090Rabbit polyclonal anti-phospho-MSK1 (T581)Cell signaling technologyCat# 9595; RRID: RRid: AB_2181783Rabbit monoclonal anti-phospho-NF-κB p65 (S536)Cell signaling technologyCat# 3033; RRID: AB_331284Rabbit monoclonal anti-NF-κB p65Cell signaling technologyCat# 8242; RRID: AB_10859369Rabbitモノクローナル抗ホスホ-CREB(S133)Cell Signaling TechnologyCat# 9198; RRID:AB_2561044Rabbitモノクローナル抗CREBCell Signaling TechnologyCat# 9197; RRID.AB_331277Rabbit monoclonal anti-phospho-GSK-3β (S9)Cell signaling technologyCat# 5558; RRID: AB_10013750Rabbit monoclonal anti-GSK-3βCell signaling technologyCat# 12456; RRID: AB_2636978Rabbit monoclonal anti-phospho-PI3 Kinase (Y458/Y199)Cell signaling technologyCat# 17366; RRID: AB_2895293Rabbit monoclonal anti-PI3 Kinase p55Cell signaling technologyCat# 11889; RRID: AB_2797756Rabbit monoclonal anti-phospho-AKT (S473)Cell signaling technologyCat# 4060; RRID: AB_2315049Rabbit monoclonal anti-AKT (pan)Cell signaling technologyCat# 4691; RRID: AB_915783Horse anti-mouse IgG, HRP-conjugatedCell signaling technologyCat# 7076; RRID: AB_330924Goat anti-rabbit, HRP-conjugatedCell signaling technologyCat# 7074; RRID: AB_2099233Goat polyclonal anti-MSK1R&D systemsCat# AF2518; RRID: AB_2301227Rabbit anti-goat IgG, HRP-conjugatedR&D systemsCat# HAF017; RRID: AB_562588Mouse monoclonal anti-MSK2Santa Cruz BiotechnologyCat# sc-377151Rabbit polyclonal anti-MSK2 (T568)Assay BiotechCat# A8149; RRID: AB_10684516Armenian Hamster Anti-CD3e, FITC-conjugatedBD BiosciencesCat# 553062; RRID: AB_394595Rat monoclonal anti-CD19, FITC-conjugatedBD BiosciencesCat# 553785; RRID: AB_395049Rat monoclonal anti-CD4, PE-conjugatedBD BiosciencesCat# 553730; RRID: AB_395014Rat monoclonal anti-IL10, PE-conjugatedBD BiosciencesCat# 554467; RRID: AB_395412Rat monoclonal anti-CD11b, PE-Cy7-conjugatedBD BiosciencesCat# 552850; RRID: AB_394491Rat monoclonal anti-F4/80, APC-conjugatedBD BiosciencesCat# 565853; RRID: AB_2744474Hamster monoclonal anti-CD11c, APC-Cy7-conjugatedBD BiosciencesCat# 561241; RRID: AB_10611727Rat monoclonal anti-CD8, APC-conjugatedBD BiosciencesCat# 553035; RRID: AB_398527Rat monoclonal anti-CD206 (MMR), Alexa Fluor 488-conjugatedThermoFisherCat# 53-2061-82;
RRID:AB_2784749ラットモノクローナル抗マウスMac2CEDARLANECat# CL8942AP;
RRID: AB_10060357Rabbit polyclonal anti-rat IgG, FITC-conjugatedabcamCat# ab6730; RRID: AB_955327Mouse monoclonal anti-6X His tag antibodyInvitrogenCat# MA1-135; RRID: AB_2536841Rabbit monoclonal anti-His tagCell signaling technologyCat# 12698; RRID: AB_2744546Ratモノクローナル抗F4/80AbcamCat# ab6640;RRID:AB_1140040Rabbitポリクローナル抗IgGSanta Cruz BiotechnologyCat# sc-66931;RRID:AB_1125055Mouse monoclonal anti-hTLR1InvivogenCat# mabg-htlr1.
RRID:AB_11124907マウスモノクローナル抗mTLR2InvivogenCat# mabg-mtlr2;
RRID:AB_11125339ハムスターモノクローナル抗hTLR2InvivogenCat# maba2-htlr2;
RRID:AB_11142484マウスモノクローナル抗hTLR6InvivogenCat# mabg-htlr6;
RRID:AB_11142485ウサギポリクローナル抗AmTARSThis paperN/ARabbit polyclonal anti-AmYARSThis paperN/ARabbit polyclonal anti-AmGAPDHThis paperN/ABacterial strainsA. muciniphila MucTGerman Collection of Microorganisms and Cell Cultures GmbHCat# DSM 22959E. coli: NiCo21(DE3) CompetentNEBCat# C2529HClearColi® BL21(DE3) Electrocompetent CellsLucigenCat# 60810E. coli: Nissle 1917 (EcN)Yim et al.
83
N/AE. coli: Nissle 1917::AmTARS (EcN(AmTARS))本論文N/AC化学物質、ペプチド、組み換えタンパク質Dextran sulfate sodium salt, colitis gradeMP biomedicalCat# 160110Lipopolysaccharides from E. coli O111::Nissle 1917,:AmTARS (EcN(AmTARS)) coli O111: B4(LPS)SigmaCat# L2630Phorbol 12-myristate 13-acetate (PMA)SigmaCat# P8139Pam3CSK4InvivogenCat# tlrl-pmsRecombinant IL-4PeprotechCat# 200-04Recombinant IL- 13PeprotechCat# 200-04 13PeprotechCat#200-13組み換えマウスIL-10BioLegendCat#575806EncapsomeEncapsula Nano SciencesCat#CLD-8910ClodrosomeEncapsula Nano SciencesCat# CLD- 8909SCREEN-WELL® キナーゼ阻害剤ライブラリーEnzo LifesciencesCat#BML-2832ポリエチレンイミンPolybiosciencesCat#23966Triton X-? 114SigmaCat# X1143X FLAG peptideSigmaCat# F4799Porcine gastric mucin (Type III)SigmaCat# M17788% Paraformaldehyde in 2X PBSBiosesangCat# PC2184-050-00SucroseSigmaCat# S7903; CAS# 57-50-1Magnesium sulfate heptahydrateSigmaCat# 63138; CAS# 10034-99-8Potassium chlorideSigmaCat# P5405; CAS# 7447-40-7Sodium chlorideSigmaCat# S7653; CAS# 7647-14-5Calcium chloride dehydrateSigmaCat# C7902; CAS# 10035-04-8Ethyl 3-aminobenzoate methanesulfonate saltSigmaCat# S5040; CAS# 886-86-2Sodium citrateSigmaCat# S464; CAS# 6132-04-3Agarose, Low Melting Point, Analytical GradePromegaCat# V2111Mounting Medium with DAPIabcamCat# ab104139Optip solution (60% (w/v))SigmaCat# D1556; CAS#
92339-11-2Critical commercial assaysBD OptEIA™ Mouse IL-6 ELISA setBD BiosciencesCat# 555240; RRID: AB_2869049BD OptEIA™ Mouse IL-10 ELISA SetBD BiosciencesCat# 555252; RRID: AB_2869052BD OptEIA™ Mouse TNF (Mono/Mono) ELISA SetBD BiosciencesCat# 555268; RRID: AB_2869055BD OptEIA™ Human IL-6 ELISA SetBD BiosciencesCat# 555220; RRID: AB_2869045BD OptEIA™ Human IL-10 ELISA SetBD BiosciencesCat# 555157; RRID: AB_2869031BD OptEIA™ Human TNF ELISA SetBD BiosciencesCat# 555212; RRID: AB_2869042Experimental models: cell linesHuman: HEK 293T cellsATCCCat# CRL-11268;
RRID:CVCL_1926ヒト:THP-1細胞KCLBCat# 40202; RRID:CVCL_0006ヒト:HEKブルーTLR4細胞InvivogenCat# 293-htlr4a;
RRID:CVCL_Y393実験モデル:生物/系統マウス: C57BL/6JThe Jackson LaboratoryJAX 000664Mouse: Tlr2 KO (B6.129-Tlr2tm1Kir/J)The Jackson LaboratoryJAX 004650Mouse: B6-CD45.1The Jackson LaboratoryJAX 002014Zebrafish: AB株(野生型)ZIRCZDB-GENO-960809-7Zebrafish: Tg(NFκB:EGFP)Lee et al.
57
N/Aオリゴヌクレオチド:バクテリアエンジニアリングに使用するプライマーexo/ceaの上流領域を標的とするプライマー:
CTCAGTACCCGCATGAAACCAGA
CTGAAGC(フォワード)、
CGAGCCGATGATAATGTCAACGT
AAATGATTGCGG(リバース)本紙N/APプライマーexo/ceaの下流域をターゲットとする:
CTTGTCTGTAAGCGATGCCATTACG
TTATCCAGACG(フォワード)、
CTCGAGTACGCGTCAGGGTTGACAG
GGAAAAC(リバース)本紙N/APrimersは、exo/cea-targeting配列をpTargetに挿入するために使用します:
CTGATAACGTAAATGATGTTTTA
GAGCTAGAAATAG(フォワード)、
CAATCATTACGTTATCCAGACTAGT(前進)、CAATCATTACGTTATCCAGACTAGT
ATTATACCTAGGAC(リバース)本紙N/AR組換えDNApET21aベクターNovagenCat# 69740pET21a-AmAARSThis paperN/ApET21a-AmCARSThis paperN/ApET21a- AmDARSThis論文N/ApET21a-AmEARSThis論文N/ApET21a-AmFARSaThis論文N/ApET21a-AmFARSbThis論文N/ApET21a-AmGARSThis論文N/ApET21a-AmFARSb AmHARST 本紙N/ApET21a-AmIARST 本紙N/ApET21a-AmKARST 本紙N/ApET21a-AmLARST 本紙N/ApET21a-AmMARST 本紙N/ApET21a-AmMARST AmNARST:紙N/ApET21a-AmPARST:紙N/ApET21a-AmQARST:紙N/ApET21a-AmRARST:紙N/ApET21a-AmTARST:紙N/ApET21a-AmTARST:紙N/ApET21a-AmTARST AmVARST紙N/ApET21a-AmWARST紙N/ApET21a-AmYARST紙N/ApET21a-AmGAPDHT紙N/ApET21a- ΔU1AmTARST紙N/ApET21a- ΔU2AmTARST紙N/ApET21a- ΔU3AmTARST紙N/ApET21a- ΔU4AmTARST紙N/Apet21a ΔU2AmTARST本紙N/ApET21a- ΔU1ΔU2AmTARST本紙N/ApET21a-AmTARS_NTDT本紙N/ApET21a-AmTARS_CD本紙N/ApET21a- ΔU2AmTARST 本文 AmTARS_ΔU2CD本紙N/ApET21a-AmTARS_ABDT本紙N/ApET21a-BfTARST本紙N/ApET21a-EcTARST本紙N/ApET21a-EcTARST EcTARS (U1/U2)本論文N/ApET21a-RbTARSThis paperN/ApET22b(+) vectorNovagenCat# 69744-3pET22b(+)-HsTARSThis paperN/ApIRES-FLAG vectorGack et al.
84
N/ApIRES-NLRX1-FLAG本論文N/ApIRES-CD163-FLAG本論文N/ApIRES-TLR4-FLAG本論文N/ApCMV3-TLR2-FLAGSino BiologicalCat# HG10061-CFpEXPR-. IBA105ベクターIBACat# 2-3505-000pEXPR-IBA105-AmTARShis paperN/ApCasAddgeneCat# 62225pTargetAddgeneCat# 62226Software and algorithmsPrism(v6. 0)N/Ahttp://www.graphpad.comCellQuest proBD biosciencesN/AAlphaFold2_advancedJumper et al.
43
https://colab.research.google.com/github/sokrypton/ColabFold/blob/main/beta/AlphaFold2_advanced.ipynbMMseqs2Mirdita et al.
85
https://github.com/soedinglab/MMseqs2PyMOL (v1.8.6.0)Schrödinger, LLChttps://pymol.org/2/MaxQuant (v1.6.10.43 and v2.1.1.0)Tyanova et al.
86
https://www.maxquant.orgPerseus (v1.5.8)Tyanova et al.
87
https://www.maxquant.org/perseus/STAR (v2.5.1)Dobin et al.
88
N/AedgeR (v3.12.1)Goldman et al.
89
N/AMeVHowe et al.
90
https://mev.tm4.org/R pheatmap package (v1.0.12)Kolde et al.
91
https://cran.r-project.org/web/packages/pheatmap/index.htmlGSEA (v4.0.0)Subramanian et al.
92
https://www.gsea-msigdb.org/gsea/index.jspEnrichrKuleshov et al.
93
https://maayanlab.cloud/Enrichr/MaAsLin2 (v1.2.0)Mallick et al.
94
N/ADepositedデータA. muciniphilaのEVおよびEVフリー上清のLC-MS/MS生データ、インタラクトーム解析結果、プロテオーム解析結果本論文PRIDE Archive: PXD041790OtherNi-NTA AgaroseQIAGENCat# 30230Strep-Tactin® Sepharose® resinIBACat# 2-1201-002SnakeSkin, Dialysis tubing, 10K MWCOThermoFisherCat# 68100Slide-A-Lyzer Dialysis Cassette G2, 10K MWCOThermoFisherCat# 87730Bolt 4-12%, Bis-Tris, 1. 0mm、ミニプロテインゲルInvitrogenCat# NW04125BOXiBlot 2 Transfer Stacks、 PVDFInvitrogenCat#IB24002Mem-PER Plus Membrane Protein Extraction KitThermoFisherCat# 89842HEK Blue selection (250X concentrate)InvivogenCat# hb-selHEK Blue detectionInvivogenCat# hb-. detProtease inhibitor cocktailGenDEPOTCat# P3100Phosphatase inhibitor cocktailGenDEPOTCat# P3200Alexa Fluor 568 Protein Labeling KitInvitrogenCat# A102238
新しいタブで表を開く
リソースの有無
リード連絡先
本研究で使用したリソースや試薬に関する情報やリクエストは、リードコンタクトのMyung Hee Kim (mhk8n@kribb.re.kr)にご連絡ください。
材料の入手方法
材料は、リクエストに応じて、リードコンタクトから入手可能です。
実験モデルおよび被験者の詳細
マウス
C57BL/6JマウスはDamul Science社(Daejeon, Korea)から購入した。Tlr2-KO(B6.129-Tlr2tm1Kir/J)およびB6-CD45.1(B6.SJL-Ptprca Pepcb/BoyJ)マウスはJackson Laboratory(Bar Harbor, ME)より購入した。すべてのマウスは、22±1℃で12時間の明暗サイクルで特定の病原体を含まない条件下で維持され、すべての子孫は、実験に使用する前にRT-PCRを用いて遺伝子型判定された。マウスを用いたすべての実験は、Korea Research Institute of Bioscience and Biotechnology (KRIBB-AEC-20030) のInstitutional Animal Use and Care Committeeのガイドラインに従って行われた。
初代細胞の単離
マウスから大腸を取り出し、0.5-1cmの長さに縦に切り、リン酸緩衝生理食塩水(PBS)緩衝液(137mM NaCl、2.7mM KCl、10mM Na2HPO4、1.8mM KH2PO4 )で軽く洗浄した。洗浄した組織を、5mlのRPMI-1640(Gibco)+1mg ml-1のIV型コラゲナーゼ(Sigma, St.Louis, MO)を含む50mlコニカルチューブに移し、250rpmの振盪インキュベーターで30分間インキュベートした。消化後、組織誘導体をメッシュ(100ミクロン)で濾過し、500×g、4℃で5分間遠心分離し、細胞ペレットをPBSに再懸濁した。腹膜滲出液細胞(PEC)は、2%(v/v)ウシ胎児血清(FBS)(Gibco)を含む冷RPMI1640を10ml腹腔内に送り込み、液体を静かに吸引することにより収集した。洗浄液を500×g、4℃で3分間遠心分離し、ペレットをPBSに再懸濁した。PECは、10%(v/v)FBSを含むダルベッコ変法イーグル培地(DMEM;Hyclone)中で培養した。マクロファージを精製するため、分離した腸管および腹膜細胞をCD11b-PEおよびF4/80-APC抗体で染色し、CD11b+F4/80+二重陽性細胞をFACSAriaTM III(BD)を用いて選別した。BMDMは7-8週齢のマウスから分離し、赤血球はACK溶解バッファー(Gibco社製)で溶解させた。BMDMは、10%(v/v)FBSおよび50ng ml-1 GM-CSF(R&D systems)を含むDMEM中で5日間培養した;培地は1日おきに新鮮な完全培地に交換された。次に、BMDMを10 ng ml-1 LPS(Sigma)で24時間刺激して炎症反応を誘導し、または20 ng ml-1 IL-4(Peprotech)および20 ng ml-1 IL-13(Peprotech)で刺激してM2偏光を誘導した。
HEK293T細胞株
ヒト腎臓上皮細胞株HEK293T(ATCC)は、10%(v/v)FBS(Gibco)及び1%(v/v)Antibiotic-Antimycotic(Gibco)を含むDMEM中で維持した。HEK Blue-TLR4細胞(InvivoGen)は、10%(v/v)FBS、1%(v/v)Antibiotic-Antimycotic、100μg ml-1 Normocin(InvivoGen)、および1X HEK Blue selection(InvivoGen)を含むDMEM中で増殖した。細胞株は5%(v/v)CO2中、37℃でインキュベートした。
THP-1細胞株
ヒト白血病単球細胞株THP-1(ATCC)は、10%(v/v)FBSおよび1%(v/v)Antibiotic-Antimycoticを含むRPMI1640培地(Gibco)中、37℃/5%(v/v)CO2で培養した。THP1細胞は、10ng ml-1 Phorbol 12-myristate 13-acetate (PMA, Sigma)で48時間処理することにより、付着性マクロファージ様表現型に分化させ、培養液は毎日新鮮な培地に交換した。
A. muciniphilaの培養
A. muciniphila MucT(=DSM 22959T)を37℃の基礎培地で嫌気的に培養した。
96
16gl-1大豆ペプトン、25mMグルコース、0.05%豚胃ムチン(III型、Sigma)を添加した基礎培地96で嫌気培養した。微量元素およびビタミン溶液は、前述の通りであった。
97
すべての化合物はオートクレーブ滅菌したが、ビタミンはフィルター滅菌した。培地調製のためのすべての手順は、以前に確立された方法に従って嫌気的条件下で行われた。
98
ゼブラフィッシュの飼育
ゼブラフィッシュ(Danio rerio)AB(野生型、WT)株およびTg(NFκB:EGFP)成魚を標準条件下で28.5℃に維持した。Tg(NFκB:EGFP)株は、腸のNF-κB活性を測定するためのNF-κB活性レポーターとして使用した。
57
魚は毎日乾燥した餌を与え、14時間、10時間暗黒の明暗スケジュールで維持した。ゼブラフィッシュの飼育および動物の世話は、Korea Research Institute of Bioscience and Biotechnology (KRIBB)のガイドラインに従って行い、すべての実験はKRIBB-AEC-22179の承認を得ている。
方法の詳細
DSS誘発大腸炎の誘発と疾患活動性の測定
マウスに2%DSS(分子量36-50kDa;MP Biomedicals, Santa Ana, CA)を10日間、飲料水から投与した。AmTARS、AmTARS欠失変異体、マウスIL-10(BioLegend)などのタンパク質を1日おきにi.p.-injectした。マウスに投与したタンパク質の量は、図の説明文に記載されている。マウスからマクロファージを枯渇させるために、DSS投与の4日前およびDSS投与中のDay0、2、4、6に、200μlのClodrosome(クロドロネート内包リポソーム;Encapsula Nano Sciences)またはEncapsome(コントロールリポソーム)をi.p.-injectされた。DSS投与中の人工EcN(AmTARS)またはEcNの投与は、107CFUの細菌を含む100μlの懸濁液(25%グリセロールを含むPBS)を経口投与(毎日)することにより行った。大腸炎症状の毎日の臨床評価は、体重測定、便の硬さの分析、直腸出血の有無の確認により行い、以下のようにスコア化した: 体重減少(0, なし; 1, 1-5%; 2, 6-10%; 3, 11-15%; 4, >15%), 便の硬さ(0, 形の整ったペレット; 2, ゆるい便; 4, 下痢), 便の血液(0, 血便検査陰性; 1, 血便検査陽性; 2, 便に目に見える血がある; 3, 目に見える血があり肛門で血が固まった; 4, 総出血)、これらは以前に記述されているように、次のように評価した。
99
H&E 染色および免疫蛍光染色
大腸組織は、10%(v/v)ホルムアルデヒドで固定した室温(RT)で一晩固定し、水道水で6時間以上洗浄し、Tissue Embedding Centerモジュール(Tissue-Tek、Sakura Finetek株式会社、日本)を使用して埋め込んだ。組織切片(厚さ7μm)は、標準プロトコールに従ってキシレンで脱パラフィンし、エタノールで再水和した。組織学的分析のために、組織切片(厚さ7μm)をH&E(Sigma)で染色し、光学顕微鏡(BX51、Olympus Corp、東京、日本)で画像化した。組織学的スコアは、前述したように決定した。
100
免疫蛍光染色では、スライドを抗原回収バッファー(0.1 M クエン酸ナトリウム pH 6.0 および 0.05% Tween-20)で 15 分間加熱インキュベートし、3%ウシ血清アルブミン含有 PBS で 1 時間 RT でブロッキングした。スライスを抗マウスMac2(CEDARLANE)一次抗体で染色し、続いてFITC標識抗ラットIgG(abcam)および抗6X Hisタグ抗体(Invitrogen)でそれぞれ2時間、RTで染色した。0.1% Tween-20を含むPBSで3回洗浄した後、DAPI(abcam)を含むマウントメディウムでスライドをマウントした。画像はNikonレーザー走査型共焦点顕微鏡(C2plus)で取得し、NIS-Elementsソフトウェアで解析した。
フローサイトメトリー
一次細胞を、FACSバッファー(2%(v/v)FBSおよび0.05%NaN3を含むPBS)中で4℃、30分間、個々の蛍光プローブ標識抗体で染色した。FACS バッファーで 2 回洗浄後、染色した細胞を FACSCaliburⓇ で解析し、CellQuest ソフトウェア(BD)を用いてデータを解析した。フローサイトメトリーには、以下の抗体を使用した: FITC-CD3 (553062), FITC-CD19 (553785), PE-CD4 (553730), PE-IL-10 (554467), Cy5.5-CD11b (552850), APC-F4/80 (565853), APC-CD11c (561241), APC-CD8 (553035) 及び FITC-CD206 (53-2061-82), 全て BD Biosciences (San Diego, CA) から。
遺伝子クローニング
Akkermansia muciniphila (Am) alanyl-tRNA synthetase (AARS), cysteinyl-tRNA synthetase (CARS), aspartyl-tRNA synthetase (DARS), glutamyl-tRNA synthetase (EARS), phenylalanyl-tRNA synthetase alpha subunit (FARSa).GENES (GN)をコードする、 フェニルアラニルtRNA合成酵素βサブユニット(FARSb)、グリシルtRNA合成酵素(GARS)、ヒスチジルtRNA合成酵素(HARS)、イソロイシルtRNA合成酵素(IARS)、リシルtRNA合成酵素(KARS).ロイシルtRNA合成酵素(LARS)、メチオニルtRNA合成酵素(MARS)、アスパラギニルtRNA合成酵素(NARS)、プロリルtRNA合成酵素(PARS)、グルタミニルtRNA合成酵素(QARS)、アルギニルtRNA合成酵素(RARS)、 トリプトファニルtRNA合成酵素(WARS)、チロシルtRNA合成酵素(YARS)、グリセルアルデヒド3リン酸デヒドロゲナーゼ(GAPDH)は、A.Muciniphilaを用いたPCR増幅により得られた。muciniphilaのMucTゲノムDNAを鋳型としてPCR増幅し、C-terminal hexa-HisタグをコードするpET21aベクターにクローニングした。AmTARS欠失変異体(ΔU1AmTARS、残基99-113欠失;ΔU2AmTARS、残基290-312欠失;およびΔU1ΔU2AmTARS、残基99-113および290-312二重欠失)は、配列およびライゲーション非依存クローニング法(SLIC)により pET21aベクターへクローニングした。
101
Bacteroides fragilis、Escherichia coli、Ruminococcus bromiiから分離したゲノムDNAからTARS遺伝子を増幅し、pET21aにクローン化した。Gly152とGlu153の間、およびGlu323とAsn324の間にそれぞれAmTARS U1およびU2領域[EcTARS(U1/U2)]を挿入した大腸菌TARS遺伝子を合成し(Macrogen、韓国)、pET21aベクター内にクローニングした。ヒトの遺伝子はすべてHEK 293T cDNAから増幅した。ヒトTARS1はpET22b(+)ベクターに、ヒトNLRX1、CD163、およびTLR4遺伝子はpIRES-FLAGベクターに、前述のようにクローニングされた。
84
インビトロプルダウンアッセイのために、N末端ドメイン(NTD、残基1-184)、触媒ドメイン(CD、残基184-523)、ΔU2触媒ドメイン(ΔU2CD、CDの残基290-312を削除)、アンチコドン結合ドメイン(ABD、残基524-630)含むAmTARSドメインをpET21aベクターにサブクローンしました。C末端Flag®タグ付き-ヒトTLR2は、Sino Biological社から購入した。免疫沈降解析のために、AmTARSをpEXPR-IBA105ベクター(IBA)にサブクローニングした。
イムノブロット
AmTARSとPam3CSK4(InvivoGen)によるTLR2媒介シグナルを比較するために、WTおよびTLR2-KOマウス由来のBMDMを0.5μM AmTARSまたは0.5μM Pam3CSK4と30分間インキュベートした。抗体ブロック実験では、BMDMをTLR抗体(1μg ml-1, InvivoGen)で1時間前処理し、TLR2をブロックした後、0.5μM AmTARSまたは0.5μM Pam3CSK4で37℃、30分間の処理後にシグナルを分析した。AmTARSが標的とするTLR2ヘテロダイマーの選好性を評価するために、分化THP1細胞を1μgの抗体(ヒトIgG、抗hTLR1、抗hTLR2、および抗hTLR6)で1時間前処理し、0.5μM AmTARSに37℃で30分間曝露した。処理した細胞を溶解バッファー(50 mM Tris-HCl, pH 7.5, 100 mM NaCl, 1% (v/v) NP-40, 1 mM EDTA, 0.25% (w/v) sodium-deoxycholate, protease inhibitor cocktail (GenDEPOT), and phosphatase inhibitor cocktail (GenDEPOT) )により溶解し、抽出したサンプルを免疫ブロックにより解析しました。A muciniphilaの培養サンプルを5,000×gで30分間遠心分離し、細胞を1X PBSバッファーで再懸濁させた。その後、細胞を超音波処理(Vibra cell, SONICS)により溶解した。溶解物および上清からのAmTARSおよびAmYARSタンパク質は、イムノブロッティングにより分析した。
タンパク質の発現と精製
A. muciniphila由来のHisタグ付きARSおよびGAPDH;B. fragilis, E. coli, R. bromii;およびヒト由来のTARS;およびEcTARS(U1/U2)は、E.で過剰発現させた。大腸菌 Clear coli BL21 (DE3)細胞(Lucigen)において、0.5 mM IPTGで18℃、30時間誘導することにより過剰発現させた。Hisタグ付きAmTARSドメインをコードするプラスミドは、大腸菌 NiCo21 (DE3) 細胞(NEB)において、0.5 mM IPTGで18℃、20時間誘導することにより発現した。 5 mM IPTGで18℃、18時間誘導し、細胞を50 mM Tris-HCl, pH 8.0, 300 mM NaCl, および10 mM imidazole, pH 8を含む溶解バッファーで再懸濁させた。 Ni-NTAアフィニティークロマトグラフィーを用いて精製した組換えタンパク質は、50 mM Tris-HCl, pH 8.0, 150 mM NaCl, 150 mM imidazole, pH 8.0で溶出させた。溶出したタンパク質をPBS緩衝液に対して透析し、前述のようにLPSを除去するためにTX-114洗剤を用いてさらに精製した、
102
に記載されているように、LPSを除去するためにTX-114洗浄剤を用いてさらに精製した。簡単に言うと、タンパク質を10%(v/v)TX-114と混合して2%混合物を作り、4℃で30分間撹拌しながらインキュベートし、次に37℃で10分間インキュベートした。混合物からTX-114を分離するために、20,000×g、25℃で3分間、遠心分離を行った。この手順を2回または3回繰り返し、タンパク質中の残留LPSをHEK Blue-TLR4細胞を用いたSEAPレポーターアッセイシステムにより検出した。精製したAmTARSをAlexa Fluor™ 568 Protein Labeling Kit (Invitrogen)を用いてAlexa 568色素で標識した。150mm培養皿で増殖したHEK293T細胞に、PEIを用いてTLR2-FLAG構築物をトランスフェクションした。24時間後、収穫した細胞を溶解バッファー(50 mM Tris-HCl, pH 7.5, 100 mM NaCl, 1% (v/v) NP-40, 1 mM EDTA, 0.25% (w/v) sodium-deoxycholate, protease inhibitor cocktail, and phosphatase inhibitor cocktail)を用いて溶解し、4℃で16000 × g, 1時間遠心をかけることで透明化した。細胞溶解液を抗FLAG M2アフィニティゲル(シグマ社製)にロードして重力溶出を行い、1%(v/v)NP-40を含むTBSバッファ(50 mM Tris-HCl, pH 7.4, and 137 mM NaCl)により洗浄した。結合したタンパク質は、150μg ml-1の3X FLAGペプチド(Sigma)を含むTBSバッファーで溶出した。タンパク質の純度は、SDS-PAGEに続いてクマシーブリリアントブルー染色を行うことで評価した(図S7H)。
インタラクトーム解析
分化したTHP1細胞を溶解し、膜タンパク質抽出キット(ThermoFisher)を用いて、膜タンパク質と細胞質タンパク質に分画した。膜タンパク質と細胞質タンパク質(各150μg)を、AmTARS-His(15μg)と共に4℃で一晩、撹拌しながらインキュベートした。Ni-NTA樹脂(QIAGEN)を添加し、4℃で30分間、撹拌しながらインキュベートした。Ni-NTA樹脂を混合物に加え、攪拌しながら4℃で30分間インキュベートした。樹脂を洗浄バッファー(50 mM Tris-HCl, pH 8.0, 300 mM NaCl, and 30 mM imidazole)で5回十分に洗浄し、1Xタンパク質サンプルバッファーに再懸濁し、10分間煮沸した。サンプルはSDS-PAGEで分析した。ゲルからタンパク質バンドを切り出し、50mM重炭酸アンモニウム/50%アセトニトリル(ACN)で脱染した。ゲルストリップに存在するタンパク質は、ジチオスレイトール(DTT)で還元し、ヨードアセトアミドでアルキル化した。ゲル内消化は、トリプシン(Promega)を用いて37℃で一晩行い、得られたペプチドを抽出し、真空乾燥した。ペプチドを0.1%ギ酸/2%ACN溶液に再懸濁し、LC/MS/MSシステムへ注入した。ペプチドは、Easy-nanoLC1000とLTQ-Orbitrap elite(ThermoFisher)を用いて分離・分析した。トラップカラムはAcclaim PepMap 100 (ThermoFisher, 75 μm × 2 cm, C18, 3 μm, 100 Å) カラム、分析カラムはPepMap RSLC C18 (ThermoFisher, 2 μm, 100 Å, 75 μm × 50 cm) カラムでした。カラム温度は50℃に設定した。移動相は、100%水/0.1%ギ酸(バッファA)および100%ACN/0.1%ギ酸(バッファB)であった。カラムグラジエントは、流速300nl min-1で、2% Bから32% Bまで120分間で直線的に流す方法で開発した。サーベイスキャンの設定は以下の通りである: Resolution = 60,000、Max IT = 200ms、AGC Target = 1E6、質量範囲 ∼350-2000 Th. データ依存解析(DDA)MS/MSスキャン設定は以下の通り: Analyzer = orbitrap; Fragmentation = HCD Top 15 double play; Resolution = 15,000; max IT = 100 ms; AGC target = 5E4; Threshold, 200,000; 正規化衝突エネルギー = 27%; isolation width = 2.0; dynamic exclusion parameter repeat count 1; repeat duration time for 10 s; exclusion list size = 500; exclusion duration time = 60 sec. LC/MS/MS データは、MaxQuant92 v1.6.10.43 および Perseus93 v1.5.8 で解析しました。MaxQuantのパラメータは以下の通り:データベース = uniprot homo sapiens、酵素 = trypsin/P、可変修飾 = Oxidation (M), Acetyl (protein N-term), 固定修飾 = carbamidomethyl (C), LFQ and match between runs. Perseusで「razor+unique peptide >=2」のときにタンパク質群を解析対象として選択し、そのLFQ強度を比較しています。
プルダウンアッセイ
クローン化したStrep-AmTARS遺伝子を、FLAGタグ付きTLR2、NLRX1、CD163およびTLR4遺伝子とともに、PEIを用いてHEK 293T細胞に共トランスフェクトした。細胞を、50 mM Tris-HCl, pH 7.5, 100 mM NaCl, 1% (v/v) NP-40, 1 mM EDTA, 0.25% (w/v) sodium-deoxycholate, protease inhibitor cocktail, and phosphatase inhibitor cocktail を含む溶解バッファに溶解し、溶解液を 16,000 × g, 20 minで遠心分離した。抽出したタンパク質は、SDS-PAGEで分離した。Strepタグ付きタンパク質を沈殿させるために、細胞を溶解バッファーで溶解し、Strep-Tactinビーズ(IBA)を4℃で1時間(撹拌しながら)添加した。ビーズを溶解バッファーで洗浄し、1Xタンパク質サンプルバッファー(LPS溶液)中で煮沸して溶出した。沈殿したサンプルは、SDS-PAGEで分析した。
インビトロ結合アッセイ
TLR2-FLAGと、全長、NTD、CD、ΔU2 CD、およびABDを含むHisタグ付きAmTARSを2:1の比で含む精製タンパク質混合物を、4℃で5時間、撹拌しながらインキュベートした。U1/U2を介したTLR2との相互作用を解析するために、精製TLR2-FLAGタンパク質をHisタグAmTARS、EcTARS、EcTARS(U1/U2)と2:1の割合で混合し、4℃で5時間攪拌しながらインキュベーションした。これを4℃で30分間Ni-NTA樹脂にロードし、氷冷した洗浄バッファー(50 mM Tris-HCl, pH 7.5, 300 mM NaCl, 40 mM imidazole)で10回洗浄し、1Xタンパク質サンプルバッファー(LPS液)を溶出した。溶出したサンプルはSDS-PAGEで分離し、Coomassie Brilliant Blueで染色した。
酵素結合免疫吸着法 (ELISA)
炎症性サイトカインを検出するために、BMDM細胞をPam3CSK4またはタンパク質(すなわち、AmARS、微生物叢由来TARS、EcTARS(U1/U2)、AmTARS+LPS)で24時間刺激し、その後BMDM上清を回収した。阻害剤研究のために、BMDMを阻害剤で1時間前処理した(すなわち、、 PD98,059(ERK阻害剤、10μM)、SB203580(p38阻害剤、10μM)、SB747651A(MSK阻害剤、1μM)、666-15(CREB阻害剤、10μM)、Wortmannin(PI3K阻害剤、10μM)およびAKT1/2(AKT阻害剤、10μM)))で処理してからAmTARSと共に24時間インキュベートした。分化したTHP1細胞は、通常およびLPS誘発の炎症条件下で、AmTARSと24時間処理した。PECおよび腸に由来するマクロファージは、AmTARSで24時間処理した。マウス血清を得るために、眼窩内静脈から血液を採取し、RTで4時間インキュベートした。採取した血液を1,500×gで30分間遠心分離し、上層の血清層を採取した。細胞上清およびマウス血清中のサイトカイン濃度は、製造者の指示に従ってELISA法により測定した;マウスIL-6(555240)、マウスIL-10(555252)、マウスTNF(555268)、ヒトIL-6(555220)、ヒトIL-10(555157)およびヒトTNF(555212)(いずれもBD Biosciences)。
A. muciniphila EVの単離
A. muciniphilaが分泌するタンパク質を解析するために、1Lの容積で24時間培養した。各サンプルは、2.68, 3.89, 4.3 × 109 cells ml-1の密度になるように増殖した。ボトルで培養した細胞から上清を回収した。まず、上澄みから5,000×g、4℃で15分間、細胞をスピンダウンさせた。上清を回収し、10,000×g、さらに20,000×gで4℃、15分間紡糸した。20,000 × gでスピンした後に回収した上清を、100KカットオフのAmicon遠心分離フィルター(Merck Millipore)を用いて40倍に濃縮した。濃縮液は200,000で4℃、2時間遠心分離した。濃縮液からEVを分離するために、Optiprep密度勾配超遠心法を用いて限外濾過を実施した。
32
,
103
Optiprep溶液(60% w/v iodixanol; Sigma Aldrich)は、50%から10%の濃度で層を形成し、50%の混合物は濃縮物を含んでいた。簡単に説明すると、50% 3 ml、40% 3 ml、20% 3 ml、10% Optiprep 3 ml、および PBS 1 ml を順次重ね、Himac CP100WX 超遠心機 (P40ST-2561) で 100,000 × g、4℃、18 時間遠心分離をした。
A. muciniphila由来の可溶性画分およびEVのLC-MS/MS分析
サンプルは、Strap micro(PROTIFI)を用いて、メーカーのプロトコル(v4.7)に従い消化した。このプロトコルには、トリス(2-カルボキシエチル)ホスフィン(TCEP)およびメチルメタンスルホン酸(MMTS)をそれぞれ還元およびアルキル化のために使用することが含まれています。簡単に説明すると、1μgのトリプシン/lysC(ピアス、部品番号A40009)を各サンプルに添加し、そして37℃で一晩インキュベートした。溶出したサンプルを自作のステージチップ(C18)で脱塩し、スピードバキュームで乾燥させた。ペプチドを2%アセトニトリル/0.1%ギ酸溶液に再懸濁し、RSLCnano u3000/Orbitrap Exploris 240 (ThermoFisher) システムに注入した。トラップカラムはAcclaim PepMap 100(75μm×2cm、C18)、分析カラムはBEH C18(75μm×25cm、1.7μm、130Å)である。両カラムの温度は50℃に設定した。移動相は、水中0.1%ギ酸(バッファA)およびアセトニトリル中0.1%ギ酸(バッファB)であった。流速300nl/minで以下のグラジエントを使用した:100分で2-20%B、20分で20-32%B。サーベイスキャンの設定は以下の通りである: Resolution = 120,000; Max IT = 50 ms; AGC 300%; mass range 375-1200 Th. 選択したプリカーサーはHCDでフラグメント化し、オービトラップで分析した。MS/MSスキャンの他のパラメータは以下の通りである: Top15ダブルプレイ、解像度=30,000、最大IT=200ms、AGC標準、閾値=1E5、正規化衝突エネルギー=30%、分離幅=2、動的排除パラメータ exclude after n times=1、排除継続時間=20s、質量許容度 low/hy=10 ppm. LCMSMSの生データは、MaxQuant v2.1.0.0とPerseus v1.5.8で解析しました。MaxQuantのパラメータは以下の通り:データベース = uniprot akkermansia muciniphila; 酵素 = trypsin/P; 可変修飾 = Oxidation(M), Acetyl(protein N-term); 固定修飾 = methylthio (C); LFQとラン間のマッチ = selected. proteingroups.txtファイルをPerseusで読み込んだ後、「razor+unique peptide >=2」基準でタンパク質グループをフィルタリングした。AmTARSとAmOmats(外膜オートトランスポーターバレルドメイン蛋白)の相対量は、各サンプルにおける総蛋白に対する各蛋白の強度比として算出した。
DSS誘発ゼブラフィッシュ大腸炎モデル
ゼブラフィッシュに腸炎を誘発するために、Chuangらの反復DSS-injuryプロトコルを用いた。
104
のプロトコルを採用し、変更した。簡単に説明すると、胚は、Tg(NFκB:EGFP)をAB系統にアウトクロスさせ、その後、E3卵水(5 mM NaCl, 0.17 mM KCl, 0.33 mM CaCl2, 0.33 mM MgSO4)中で維持することによって生成した。NF-κB 依存性 EGFP 発現を示す正常発生胚は、受精後 2 日目(dpf)に SZX16 顕微鏡(オリンパス)を用いて選別された。卵黄嚢注入のため、3 dpf 幼虫を 3-aminobenzoate methanesulfonate ethyl salt で麻酔し、1% low-melting-point agarose (LMA) に埋め込んだ。LMA固化後、PV830マイクロインジェクター(World Precision Instruments)を用いて、2nlの1×PBSまたはAmTARS(0.5μg/μl)を卵黄嚢に注入した。注入後、幼虫を新鮮なE3卵水で3回洗浄し、0.5%DSSで24時間処理した。翌日、幼虫を新鮮なE3卵水で24時間洗浄/回復し、腸の炎症を増幅するために0.5%DSSで24時間再処置した。最後に、幼虫を4%PFA溶液(4%パラホルムアルデヒド(PFA)、4%スクロース、0.15mM CaCl2、1×PBS)中で4℃で一晩固定し、6dpf幼虫の腸内NF-kB活性を高解像度共焦点顕微鏡で撮像した(詳細は下記参照)。すべての動物実験は、1群あたり最低10匹のゼブラフィッシュ幼虫で少なくとも2回繰り返し、28.5℃で実施した。
腸管NF-κB活性の共焦点顕微鏡による解析
固定した幼虫を1×PBST(0.1% Tween 20を含む1×PBS)で短時間洗浄し、ガラス底のイメージングディッシュ上で1% LMAに埋め込んだ。肛門から250μmの領域にまたがる腸の中遠位部を、FV1000共焦点レーザー走査顕微鏡(オリンパス)を用いて画像化した。共焦点z-プロジェクションは、9〜10個の切片(各5μm厚)を積み重ねることによって作成した。Z投影画像におけるNF-kB依存性EGFPシグナルの相対強度は、Image JおよびROIマネージャによって測定した。各群のNF-kB活性は、PBSを注射したWT腸の平均蛍光強度に対する割合として表示した。
抗体の作製
AmTARS、AmYARS、およびAmGAPDHに特異的なウサギポリクローナル抗体は、Abclon Inc.(韓国、ソウル)で製造した。ウサギは、完全フロイントアジュバント(Sigma)中、200μgのタンパク質抗原を4回腹腔内注射(0、4、6、8週)して免疫した。抗体はIgG精製により血清から精製した。
アミノアシル化アッセイ
精製AmTARS、ΔU1AmTARS、ΔU2AmTARS、ΔU1ΔU2AmTARS、AmYARSを用いて、スレオニン依存性アミノアシル化アッセイを実施した。アッセイは、1mM DTT、250μM ATP、2.5mM L-スレオニン、1U ml-1 inorganic pyrophosphatase、および2μMのタンパク質を含む反応バッファ(50mM Tris-HCl, pH7.5, 10mM MgCl2, 100mM NaCl, 100 mM KCl)中で37℃で30分間行い、マラカイトグリーンリン酸測定キット (BioAssay Systems) により620nmでフリー無機リン酸を検出しました。
遺伝子組換えEcN株の構築
C末端Hisタグ付きAmTARSを染色体発現する人工EcN株は、既報の通りラムダレッドを介したCRISPR-Cas9システムにより構築した。
105
EcN染色体のexo/cea遺伝子間領域は、収束したORFで構成されている。この特性を利用して、隣接する遺伝子の発現への影響を最小限に抑えながら、異種配列の挿入を可能にした研究が他に行われている。
60
,
61
exo/cea間遺伝領域の20 ntの配列をpTargetベクターにクローニングした。Hisタグ付きAmTARSとlacプロモーターをexo/cea遺伝子間領域の両側に相同な500bpのアームでアセンブルし、修復テンプレートDNAとして使用した。pCasを保有するEcNを100ngのpTargetと修復テンプレートDNAで形質転換した。得られた工学的EcNを選択し、染色体上のexo/cea遺伝子間領域を包含するPCRにより確認した。EcN(AmTARS)]によるHisタグ付きAmTARSの発現を検証するために、菌株を37℃のLB中でインキュベートした(振盪を伴う)。その後、10,000×gで15分間、4℃で培養から細胞をスピンダウンさせた。上清を0.45μm孔のMinisartシリンジフィルター(Sartorius)でろ過し、30KカットオフのAmicon遠心フィルター(Merck Millipore)を用いて40倍濃縮した。この細胞溶解液と培養上清をイムノブロッティングで解析した。比較のため、EcN(AmTARS)またはA. muciniphilaの全タンパク質と同量(15μg)相当の細胞溶解液を負荷し、EcN(AmTARS)またはA. muciniphilaの濃縮または元の培養上清をそれぞれ同容量(40μl)負荷した。
養子縁組による細胞移植
養子縁組のために、B6-CD45.2マウスから腹腔細胞を分離し、ドナー細胞として使用した。単離した細胞(1匹あたり5×106個)を6Gy照射したB6-CD45.1マウスに腹腔内移植した(レシピエント)。その後、マウスにAmTARS(0.2 mg kg-1)またはPBSを1日おきに10日間投与した。細胞移植後5日目と10日目に、それぞれ腹腔と結腸のCD45.2-APCとF4/80-PE陽性細胞数をフローサイトメトリーで測定した。
定量化および統計解析
AmTARSの構造モデリング
AlphaFold2_advancedを用いて、単量体AmTARSの構造モデルを作成した、
43
これは https://colab.research.google.com/github/sokrypton /ColabFold/blob/main/beta/ AlphaFold2_advanced.ipynb で入手可能である。AmTARSのアミノ酸配列は、MMseqs2によって生成された複数の配列とアライメントされた。
85
pLDDTスコアが最も高いモデルから上位5つのモデル構造を得た。pLDDTスコアが最も高い92.53のAmTARSの構造モデルが選択された。すべての分子グラフィックスはPyMOL version 1.8.6.0 (Schrödinger, LLC)を用いて作成した。
RNA配列決定
PureLink RNA mini kit(ThermoFisher)を用いて、0.5μM AmTARSまたは10ng ml-1 Pam3CSK4で1時間または3時間処理したBMDMからRNAを精製した。 TruSeq RNA Sample Prep Kit(イルミナ、サンディエゴ、カリフォルニア、米国)を用いてmRNAシーケンスライブラリを作成し、イルミナ HiSeq2000プラットフォームを用いてシーケンスを行って100 bpペアエンドリードを作成した。参照用ヒトゲノムはNCBIゲノム(https://www.ncbi.nlm.nih.gov/genome/)から入手し、STARアライメントツール(v2.5.1)を用いてゲノムのインデックス付けを実施した。
88
配列決定されたリードはヒトゲノム(mm10)STARにマッピングされ、STARアライナーのcountモジュールを用いて遺伝子発現量が定量された。また、edgeR(v3.12.1)を使用した。
89
パッケージを用いて、RNA-seq countデータから条件間で差のある発現遺伝子を選択した(-fold change > 1.5, FDR < 0.05)。各遺伝子のTMM(M値正規化のトリミング平均)正規化CPM(counts per million)値を1に加算し、さらなる解析のためにlog2変換をした。ヒートマップは、MeV
90
とR (v3.5.0, https://www.r-project.org/) のpheatmapパッケージ (v1.0.12, https://cran.r-project.org/web/packages/pheatmap/index.html) を用いて作成した。
遺伝子セット濃縮解析(GSEA)
GSEAソフトウェア
92
は、RNA-seqデータのGSEAに使用されました(v4.0.0、https://www.gsea-msigdb.org/gsea/index.jsp)。発現データセットは、ホールマーク、KEGGパスウェイ、GO遺伝子セット(MSigDB v7.0のH、C2、C5.gmtファイル)に対して分析された。濃縮スコア(ES)の統計的有意性(公称p値)は、1000遺伝子セットの順列を実行することで算出した。遺伝子セットの大きさを考慮し、正規化濃縮スコア(NES)を使用した。濃縮された転写因子(TRANSFACおよびJASPAR PWM)は、Enrichrを使用して同定した。
93
iHMPデータセットに含まれる細菌性tRNA合成酵素の統計解析
公開データセットであるIBDMDB
36
iHMP は、non-IBD n = 27; CD (Crohn's disease) n = 67; または UC (Ulcerative colitis) n = 38 という 3 つの臨床診断を受けた 132 名の参加者からなる縦断コホートである。IBDMDBは、メタゲノム解析(https://ibdmdb.org/tunnel/public/HMP2/WGS/1818/ 製品)およびメタトランスクリプトーム解析(https://ibdmdb.org/tunnel/public/HMP2/MTX/ 1750/製品)から加工された分類学的および機能的プロファイルを提供しています。Microbiome Multivariable Association with Linear Models(MaAsLin2)パッケージ(v1.2.0)を用いて、線形モデルフィッティング、
94
を使用して、交絡変数を調整しながら、細菌の特徴とIBDの状態との間の関連性を決定した。モデルフィッティングの前に、有病率が低い特徴(カットオフ10%)は表から削除した。診断、抗生物質、ディスバイオシスの状態はカテゴリー変数またはバイナリー変数として、年齢は連続変数として使用した。同じ参加者の経時的な反復測定による影響を調整するため、被験者はランダム変数としてコード化した。132人の参加者から得た1638のメタゲノムサンプルのうち、46サンプルは低シーケンスリード(100万リード未満)のためにフィルタリングされ、835のメタトランスクリプトームサンプルのうち、126サンプルは低品質リードまたはサンプリング日のディスバイオシス状態が不明なために削除されました。統計的有意性はWaldの検定で計算し、Benjamini-Hochberg法(q-value)で補正した。ヒートマップは、pheatmapパッケージ(v1.0.12)を用いて特徴の存在量を可視化したものである。
91
統計解析
結果は、平均値±SEMで示した。統計解析はPrism v.6.0を用いて行い、p値は両側Student's t-testを用いて算出した。p値が<0.05のとき、データは統計的に有意であるとみなされた。
データおよびコードの入手方法
本研究で生成された質量分析プロテオミクスデータは、PRIDEを介してProteomeXchange Consortiumに寄託されています。
95
データベース(データセット識別子:PXD041790)に寄託した。
本論文では、オリジナルコードは報告していません。
本論文で報告されたデータを再解析するために必要な追加情報は、要求に応じてリードコンタクトから入手できます。
謝辞
この仕事は、科学とICTの省によって賄われる韓国の国立研究財団(2015M3C9A4053394 to M.H.K.; 2015R1A3A2033475 and 2022M3A9F3082326 to W.-J.L. )とKRIBB研究イニシアティブプログラム(KGM1382312 to M.H.K. )は補助金を得ています。
著者の貢献
M.H.K.は、プロジェクトの構想および監督を行った。S.-M.K.は、S.P.、J.-H.K.、T.-H.K.、C.-H.Lの協力を得て、すべての動物実験を設計し実施した。S.P.は、S.-M.K.の助けを借りて生化学、細胞生物、免疫学の分析を設計し実施した。 S.H.H.は、M.K.およびE.-K.K.の協力を得て、AmTARSを分泌する大腸菌Nissleを操作した。S.-M.K., J.-H.K., S.-Y.K. はトラクノミック解析を実施した。G.S.L.とJ.H.M.はインタラクトーム解析のためのLC/MS実験を行った。G.L.とG.K.は、iHMPデータセットを用いて、細菌のアミノアシルtRNA合成酵素のメタゲノム解析とメタトランスクリプトーム解析を行った。D.-H.C.とB.-C.K.はA.muciniphilaを用いた嫌気性培養実験を実施した。J.H.とY.L.は、AmTARSの構造モデリングを行った。J.-G.L.とJ.-S.L.がゼブラフィッシュを用いた実験を行った。S.-M.K., S.P., J.-H.R., J.-Y.K., S.K., W.-J.L., and M.H.K. がデータを解析し解釈した。S.-M.K.とS.P.が原稿を作成し、M.H.K.が全著者の意見を取り入れながら原稿を編集した。
利害関係の宣言
著者らは、競合する利害関係はないことを宣言している。
補足情報
.pdfをダウンロード (22.48 MB)
pdfファイルのヘルプ
資料S1. 図S1~S7
参考文献
オハラ・A.M.
シャナハンF.
忘れ去られた臓器としての腸内細菌叢。
EMBO Rep. 2006; 7: 688-693https://doi.org/10.1038/sj.embor.7400731
記事で見る
スコープス (1927)
PubMed
クロスフィルム
グーグル奨学生
ソマーF.
ベックヘド F.
腸内細菌叢--宿主の発生と生理の支配者。
Nat. Rev. Microbiol. 2013; 11: 227-238https://doi.org/10.1038/nrmicro2974
記事で見る
スコープス (2167)
PubMed
クロスフィルム
グーグル奨学生
ターンボー P.J.
ハマディM.
ヤツネンコT.
カンタレルB.L.
ダンカン A.
レイ・R.E.
ソギン・M.L.
ジョーンズ W.J.
ロー・B.A.
アフォーティットJ.P.
et al.
肥満と痩せの双子における中核的な腸内細菌叢。
Nature. 2009; 457: 480-484https://doi.org/10.1038/nature07540
記事で見る
スコープス (5715)
PubMed
クロスフィルム
グーグル奨学生
Zheng D.
リウィンスキーT.
エリナブE.
健康と病気における微生物叢と免疫の相互作用。
Cell Res. 2020; 30: 492-506https://doi.org/10.1038/s41422-020-0332-7
記事で見る
スコープス (1032)
PubMed
クロスフィルム
グーグル奨学生
ファン Y.
ペダーセンO.
ヒトの代謝の健康と病気における腸内細菌叢。
Nat. Rev. Microbiol. 2021; 19: 55-71https://doi.org/10.1038/s41579-020-0433-9
記事で見る
スコープス (1112)
PubMed
クロスフィルム
グーグル奨学生
レヴィ M.
コロジエジツクA.A.
タイスC.A.(Thaiss C.A.
エリナブ E.
ディスバイオシスと免疫系
Nat. Rev. Immunol. 2017; 17: 219-232https://doi.org/10.1038/nri.2017.7
記事で見る
スコープス(800)
パブコメ
クロスレフ
グーグル奨学生
ヒトマイクロバイオームプロジェクトコンソーシアム
健康なヒトのマイクロバイオームの構造、機能、多様性。
Nature. 2012; 486: 207-214https://doi.org/10.1038/nature11234
記事で見る
スコープス (7251)
PubMed
クロスフィルム
グーグル奨学生
ブレストフ J.R.
アーティスD.
宿主の代謝と免疫系の接点にある常在菌。
Nat. Immunol. 2013; 14: 676-684https://doi.org/10.1038/ni.2640
記事で見る
スコープス (624)
パブコメ
クロスフィルム
グーグル奨学生
ポストラー T.S.
ゴーシュ S.
ホロビオントを理解する:微生物の代謝物が人間の健康にどのように影響し、免疫系を形成しているか。
Cell Metab. 2017; 26: 110-130https://doi.org/10.1016/j.cmet.2017.05.008
記事で見る
スコープス (446)
PubMed
要旨
全文
全文PDF
グーグル奨学生
ルークス M.G.
ギャレットW.S.
腸内細菌叢、代謝産物、宿主免疫。
Nat. Rev. Immunol. 2016; 16: 341-352https://doi.org/10.1038/nri.2016.42
記事で見る
スコープス(1683)
PubMed
クロスフィルム
グーグル奨学生
ベルカイド Y.
ハンドT.W.
免疫と炎症における微生物叢の役割。
Cell. 2014; 157: 121-141https://doi.org/10.1016/j.cell.2014.03.011
記事で見る
スコープス (2679)
PubMed
概要
全文
全文PDF
グーグル奨学生
イバ M.
Söll D.
アミノアシルtRNA合成のルネサンス。
EMBO Rep. 2001; 2: 382-387https://doi.org/10.1093/embo-reports/kve095
記事で見る
スコープス (69)
PubMed
クロスフィルム
グーグル奨学生
クォン N.H.
フォックス P.L.
Kim S.
治療標的としてのアミノアシルtRNA合成酵素。
Nat. Rev. Drug Discov. 2019; 18: 629-650https://doi.org/10.1038/s41573-019-0026-3
記事で見る
スコープス(119)
パブコメ
クロスレフ
グーグル奨学生
ヤオ P.
フォックス P.L.
アミノアシルtRNA合成酵素の医学と疾患における役割。
EMBO Mol. Med. 2013; 5: 332-343https://doi.org/10.1002/emmm.201100626
記事で見る
スコープス (204)
パブコメ
クロスフィルム
グーグル奨学生
グオ M.
シンメルP.
tRNA合成酵素の必須非翻訳機能。
Nat. Chem. Biol. 2013; 9: 145-153https://doi.org/10.1038/nchembio.1158
記事で見る
スコープス (270)
パブコメ
クロスフィルム
グーグル奨学生
グォー M.
ヤン X.L.
シンメルP.
アミノアシルtRNA合成酵素の翻訳以外の新しい機能。
Nat. Rev. Mol. Cell Biol. 2010; 11: 668-674https://doi.org/10.1038/nrm2956
記事で見る
スコープス (247)
PubMed
クロスフィルム
グーグル奨学生
キム M.H.
キム S.
マルチtRNA合成酵素複合体の構造と機能.
Enzymes. 2020; 48: 149-173https://doi.org/10.1016/bs.enz.2020.06.008
記事で見る
スコープス(4)
PubMed
クロスフィルム
グーグル奨学生
Cho S.
キム・S.B.
リー Y.
Song E.C.
Kim U.
Kim H.Y.
Suh J.H.
ゴヌールP.C.
キム Y.
Yoon I.
他
ヒトのシステイニルtRNA合成酵素1の触媒領域に埋め込まれた内因性TLR2リガンド。
J. Immunother. Cancer. 2020; 8https://doi.org/10.1136/jitc-2019-000277
記事で見る
スコープス(13)
クロスフィルム
グーグル奨学生
アン・Y.H.
パク S.
Choi J.J.
Park B.K.
Rhee K.H.
Kang E.
Ahn S.
Lee C.H.
リーJ.S.
インK.S.
他
感染症に対する一次防御システムとしての分泌型トリプトファニルtRNA合成酵素。
Nat. Microbiol. 2016; 2: 16191https://doi.org/10.1038/nmicrobiol.2016.191
記事で見る
スコープス(49)
パブコメ
クロスフィルム
グーグル奨学生
キム・S.B.
キム・H.R.
Park M.C.
Cho S.
ゴヌールP.C.
ハン D.
ユン I.
キム Y.
カン・T.
ソン イ.
ら。
癌細胞からのエクソソームにおける炎症性リシルtRNA合成酵素の分泌をカスパーゼ8が制御している。
J. Cell Biol. 2017; 216: 2201-2216https://doi.org/10.1083/jcb.201605118
記事で見る
スコープス(59)
パブコメ
クロスフィルム
グーグル奨学生
ウィリアムズ T.F.
ミランドA.C.
ウィルキンソンB.
フランクリンC.S.
Lounsbury K.M.
分泌型スレオニルtRNA合成酵素は内皮細胞の移動と血管新生を刺激する。
Sci. Rep. 2013; 3: 1317https://doi.org/10.1038/srep01317
記事で見る
スコープス (67)
PubMed
クロスフィルム
グーグル奨学生
コラード M.C.
デリエン M.
イソラウリE.
デ・ヴォスW.M.
サルミネン S.
乳児、成人、高齢者に存在する腸内細菌叢のムチン分解メンバーであるAkkermansia muciniphilaと腸内環境の完全性。
Appl. Environ. Microbiol. 2007; 73: 7767-7770https://doi.org/10.1128/AEM.01477-07
記事で見る
スコープス (450)
パブコメ
クロスフィルム
グーグル奨学生
カニ・P.D.
デポミエC.
デリアンM.
エベラールA.
デボスW.M.
アッカーマンシア・ムチニフィラ:次世代有益微生物群のパラダイム。
Nat. Rev. Gastroenterol. Hepatol. 2022; 19: 625-637https://doi.org/10.1038/s41575-022-00631-9
記事で見る
スコープス(72)
PubMed
クロスフィルム
グーグル奨学生
シン J.
Noh J.R.
Choe D.
Lee N.
Song Y.
Cho S.
カン E.J.
ゴ M.J.
ハ S.K.
Chang D.H.
et al.
加齢・若返りモデルから、健康的な加齢に関連する主要な微生物群集の変化が明らかになった。
Microbiome. 2021; 9: 240https://doi.org/10.1186/s40168-021-01189-5
記事で見る
スコープス(22)
パブコメ
クロスフィルム
グーグル奨学生
グリアー R.L.
ドン X.
モラエスA.C.
ジールケR.A.
フェルナンデスG.R.
ペレミスロヴァE.
バスケス=ペレス S.
シェーンボーンA.A.
ゴメス E.P.
ペレイラ A.C.
他
Akkermansia muciniphilaは、グルコース代謝に対するIFNγの負の効果を媒介する。
Nat. Commun. 2016; 7: 13329https://doi.org/10.1038/ncomms13329
記事で見る
スコープス(186)
パブコメ
クロスフィルム
グーグル奨学生
Yoon H.S.
Cho C.H.
Yun M.S.
Jang S.J.
You H.J.
キム・J.H.
Han D.
Cha K.H.
ムンS.H.
Lee K.
他
アッカーマンシア・ムチニフィラが分泌するグルカゴン様ペプチド-1誘導タンパク質は、グルコースホメオスタシスを改善し、マウスのメタボリック病を改善する。
Nat. Microbiol. 2021; 6: 563-573https://doi.org/10.1038/s41564-021-00880-5
記事で見る
スコープス (146)
パブコメ
クロスフィルム
グーグル奨学生
バソロ A.
ホーヘナデルM.
アンQ.Y.
ピアッジP.
ハイニッツ S.
ウォルター M.
ウォルター P.
パリントン S.
トリニダードD.D.
フォン・シュワルツェンバーグR.J.
et al.
ヒトの腸内細菌叢と栄養吸収に対する低栄養と経口バンコマイシンの影響。
Nat. Med. 2020; 26: 589-598https://doi.org/10.1038/s41591-020-0801-z
記事で見る
スコープス(50)
パブコメ
クロスフィルム
グーグル奨学生
バーセナ C.
バルデス-マスR.
マヨラルP.
ガラバヤC.
デュランS.
ロドリゲス F.
フェルナンデス=ガルシア M.T.
サラザール N.
ノガッカ A.M.
ガラタッチャN.
他
プロジェロイドマウスへの糞便微生物叢移植による健康寿命延長効果
Nat. Med. 2019; 25: 1234-1242https://doi.org/10.1038/s41591-019-0504-5
記事で見る
スコープス(241)
パブコメ
クロスレフ
グーグル奨学生
プロヴィエ・H.
エベラールA.
ドゥルアートC.
デポミエC.
ヴァンヒュルM.
グールトL.
シルー J.
オットマン N.
デュパルク T.
リヒテンシュタインL.
他
Akkermansia muciniphilaまたは低温殺菌菌の精製膜タンパク質は、肥満および糖尿病マウスの代謝を改善する。
Nat. Med. 2017; 23: 107-113https://doi.org/10.1038/nm.4236
記事で見る
スコープス(1114)
パブコメ
クロスフィルム
グーグル奨学生
アンサルド E.
スレイデンL.C.
チング K.L.
コッホM.A.
ウルフ・N.K.
プリヒタ D.R.
ブラウン E.M.
グラハムD.B.
ザビエルR.J.
ムーンJ.J.
他
アッカーマンシア・ムチニフィラ(Akkermansia muciniphila)は、恒常性維持のために腸管適応免疫応答を誘導する。
サイエンス(Science)。2019; 364: 1179-1184https://doi.org/10.1126/science.aaw7479
記事で見る
スコープス(235)
パブコメ
クロスレフ
グーグル奨学生
キューン M.J.
ケスティN.C.
細菌外膜ベシクルと宿主・病原体相互作用。
Genes Dev. 2005; 19: 2645-2655https://doi.org/10.1101/gad.1299905
記事で見る
スコープス (662)
PubMed
クロスフィルム
グーグル奨学生
カン・C.S.
バン M.
Choi E.J.
Moon H.G.
Jeon J.S.
キムD.K.
Park S.K.
Jeon S.G.
Roh T.Y.
Myung S.J.
他
腸内細菌叢、特にAkkermansia muciniphila由来の細胞外小胞は、デキストラン硫酸ナトリウム誘発大腸炎の進行を保護する。
PLoS One. 2013; 8: e76520https://doi.org/10.1371/journal.pone.0076520
記事で見る
スコープス (330)
PubMed
クロスフィルム
グーグル奨学生
マクガバンD.P.
クガタサンS.
Cho J.H.
炎症性腸疾患の遺伝学。
Gastroenterology. 2015; 149 (1163–1176.e2)https://doi.org/10.1053/j.gastro.2015.08.001
記事で見る
スコープス(260)
アブストラクト
全文
全文PDF
Google Scholar
ピオヴァーニ D.
ダネーゼS.
ペイラン=ビルーレL.
ニコロプロスG.K.
リトラスT.
ボノヴァスS.
炎症性腸疾患の環境リスクファクター:メタアナリシスのアンブレラレビュー。
Gastroenterology. 2019; 157 (647–659.e4)https://doi.org/10.1053/j.gastro.2019.04.016
記事で見る
スコープス(263)
PubMed
要旨
全文
全文PDF
Google Scholar
パラダ・ベネガス D.
デ・ラ・フエンテ M.K.
ランズクロンG.
ゴンザレスM.J.
ケラ・R.
ダイクストラG.
ハームセン H.J.M.
フェイバー・K.N.
ヘルモソM.A.
短鎖脂肪酸(SCFA)を介した腸管上皮および免疫制御と炎症性腸疾患との関連。
Front. Immunol. 2019; 10: 277https://doi.org/10.3389/fimmu.2019.00277
記事で見る
スコープス(1363)
パブコメ
クロスフィルム
グーグル奨学生
ロイド=プライス J.
アーゼC.
Ananthakrishnan A.N.
シルマーM.
アビラ=パチェコJ.
プーン T.W.
アンドリュース E.
アジャミ N.J.
ボナム K.S.
ブリスローンC.J.
他
炎症性腸疾患における腸内細菌生態系のマルチオミクス。
ネイチャー(Nature)。2019; 569: 655-662https://doi.org/10.1038/s41586-019-1237-9
記事で見る
スコープス(1062)
パブコメ
クロスレフ
グーグル奨学生
デポミエC.
エベラールA.
ドゥルアートC.
プロヴィエH.
ヴァンヒュルM.
ヴィエイラ=シルヴァS.
ファロニーG.
レーズ J.
メーテル D.
デルゼンヌN.M.
他
過体重および肥満のヒトボランティアにおけるAkkermansia muciniphilaのサプリメント:概念実証のための探索的研究。
Nat. Med. 2019; 25: 1096-1103https://doi.org/10.1038/s41591-019-0495-2
記事で見る
スコープス(930)
パブコメ
クロスレフ
グーグル奨学生
ヤスールM.
イム M.Y.
Yun H.S.
ティックル・T.L.
ソン J.
ソン Y.M.
Lee K.
フランゾーザ E.A.
モーガンX.C.
ゲバースD.
他
肥満と2型糖尿病の腸内細菌バイオマーカーのサブクリニカル検出。
ゲノム・メッド。2016; 8: 17https://doi.org/10.1186/s13073-016-0271-6
記事で見る
スコープス(178)
パブコメ
クロスフィルム
グーグル奨学生
ダオ M.C.
エベラールA.
アロン-ウィスネフスキーJ.
ソコロフスカN.
プリフティ E.
ベルジェE.O.
カイザーB.D.
レヴェネス F.
シルー J.
ホイルズL.
他
肥満における食事介入中のAkkermansia muciniphilaと代謝健康の改善:腸内細菌叢の豊かさおよび生態との関係。
Gut. 2016; 65: 426-436https://doi.org/10.1136/gutjnl-2014-308778
記事で見る
スコープス(1122)
PubMed
クロスフィルム
グーグル奨学生
ワン L.
タン・リー(Tang L.
Feng Y.
Zhao S.
ハン M.
Zhang C.
Yuan G.
Zhu J.
Cao S.
Wu Q.
et al.
Akkermansia muciniphilaまたは低温殺菌菌の精製膜タンパク質は、マウスのCD8(+)T細胞の調節によって大腸炎関連腫瘍の発生を鈍らせる。
Gut. 2020; 69: 1988-1997https://doi.org/10.1136/gutjnl-2019-320105
記事で見る
スコープス (206)
PubMed
クロスフィルム
グーグル奨学生
マントヴァーニA.
シーカ A.
ソッツァーニS.
アラベナP.
ヴェッキA.
ロカーティ M.
マクロファージの活性化および極性化の多様な形態におけるケモカインシステム。
Trends Immunol. 2004; 25: 677-686https://doi.org/10.1016/j.it.2004.09.015
記事で見る
スコープス (4586)
PubMed
要旨
全文
全文PDF
グーグル・スカラー
マルティネス F.O.
ヘルミングL.
ゴードン S.
マクロファージの代替活性化:免疫学的機能的視点。
Annu. Rev. Immunol. 2009; 27: 451-483https://doi.org/10.1146/annurev.immunol.021908.132532
記事で見る
スコープス (2083)
PubMed
クロスフィルム
グーグル奨学生
ジャンパーJ.
エバンスR.
プリッツェルA.
グリーンT.
フィガーノフM.
ロンネバーガー O.
トゥンヤスヴナクールK.
ベイツR.
ジーデックA.
ポタペンコA.
他
AlphaFoldによる高精度なタンパク質構造予測。
Nature. 2021; 596: 583-589https://doi.org/10.1038/s41586-021-03819-2
記事で見る
スコープス(6920)
パブコメ
クロスフィルム
グーグル奨学生
ジェインウェイ・ジュニア、C.A.
メジトフ・R.
自然免疫認識。
Annu. Rev. Immunol. 2002; 20: 197-216https://doi.org/10.1146/annurev.immunol.20.083001.084359
記事で見る
スコープス (6247)
PubMed
クロスフィルム
グーグル奨学生
ラコフ・ナホウムS.
パグリーノJ.
エスラミ・ヴァルザネF.
エドバーグS.
メジトフR.
Toll様受容体による常在細菌叢の認識は、腸のホメオスタシスに必要である。
Cell. 2004; 118: 229-241https://doi.org/10.1016/j.cell.2004.07.002
記事で見る
スコープス (3339)
PubMed
要旨
全文
全文PDF
グーグル奨学生
ラング T.
Mansell A.
toll-like受容体および関連経路の負の制御。
Immunol. Cell Biol. 2007; 85: 425-434https://doi.org/10.1038/sj.icb.7100094
記事で見る
スコープス (113)
PubMed
クロスフィルム
グーグル奨学生
リュウ F.Y.
シュウD.
ブリントE.K.
O'Neill L.A.
toll様受容体を介した免疫応答の負の制御。
Nat. Rev. Immunol. 2005; 5: 446-458https://doi.org/10.1038/nri1630
記事で見る
スコープス (1264)
PubMed
クロスフィルム
グーグル奨学生
サライバ M.
オガーラ A.
免疫細胞によるIL-10産生の制御。
Nat. Rev. Immunol. 2010; 10: 170-181https://doi.org/10.1038/nri2711
記事で見る
スコープス (2075)
PubMed
クロスフィルム
グーグル奨学生
ウェン A.Y.
サカモトK.M.
ミラー L.S.
転写因子CREBの免疫機能における役割。
J. Immunol. 2010; 185: 6413-6419https://doi.org/10.4049/jimmunol.1001829
記事で見る
スコープス (506)
PubMed
クロスフィルム
グーグル奨学生
山陰D.E.
プレンダーガストC.T.
マウントフォードA.P.
マクロファージにおけるIL-10産生は、細胞代謝に関わる遺伝子と関連するTLR駆動のCREBを介したメカニズムによって制御される。
J. Immunol. 2015; 195: 1218-1232https://doi.org/10.4049/jimmunol.1500146
記事で見る
スコープス(75)
パブコメ
クロスフィルム
グーグル奨学生
ダンヌC.
リジャコフG.
マルティネス-ロペスM.
イロット・N.E.
フランキーニF.
カスキンF.
ロウ E.C.
ブラーズS.J.
アーサーJ.S.C.
パウリー F.
Helicobacter hepaticusが産生する大型多糖類は、マクロファージにおいて抗炎症性遺伝子シグネチャーを誘導する。
Cell Host Microbe. 2017; 22 (733–745.e5)https://doi.org/10.1016/j.chom.2017.11.002
記事で見る
スコープス(71)
PubMed
要旨
全文
全文PDF
グーグル奨学生
アナニーバ O.
ダラーグJ.
ヨハンセンC.
カー・J.M.
マキラスJ.
パークJ.M.
ウィングゲート A.
モンク C.E.
トートR.
サントス S.G.
他
キナーゼMSK1およびMSK2は、toll-like receptorシグナル伝達のネガティブレギュレーターとして作用する。
Nat. Immunol. 2008; 9: 1028-1036https://doi.org/10.1038/ni.1644
記事で見る
スコープス (248)
パブコメ
クロスフィルム
グーグル奨学生
オリヴィエV.
パリーG.C.
コブR.R.
デプロストD.
マックマンN.
上昇したサイクリックAMPは、ヒト単球細胞および内皮細胞におけるNF-κBを介した転写を阻害する。
J. Biol. Chem. 1996; 271: 20828-20835https://doi.org/10.1074/jbc.271.34.20828
記事で見る
スコープス (322)
パブコメ
概要
全文
全文PDF
グーグル奨学生
パリーG.C.
マックマンN.
サイクリックAMPによるNF-κBを介した転写阻害におけるサイクリックAMP応答要素結合タンパク質の役割。
J. Immunol. 1997; 159: 5450-5456
論文で見る
PubMed
クロスフィルム
グーグル奨学生
マーティン M.
レハニK.
ヨープR.S.
ミハレック S.M.
Toll様受容体を介したサイトカイン産生は、グリコーゲン合成酵素キナーゼ3によって異なる制御を受ける。
Nat. Immunol. 2005; 6: 777-784https://doi.org/10.1038/ni1221
記事で見る
スコープス (916)
PubMed
クロスフィルム
グーグル奨学生
リーW.B.
カン J.S.
Choi W.Y.
Zhang Q.
キムC.H.
Choi U.Y.
Kim-Ha J.
キム・Y.J.
Mincleを介した翻訳制御は、強い一酸化窒素の産生と炎症解消に必要である。
Nat. Commun. 2016; 7: 11322https://doi.org/10.1038/ncomms11322
記事で見る
スコープス(46)
パブコメ
クロスフィルム
グーグル奨学生
リーJ.G.
リー S.
Jeon J.
コン H.G.
Cho H.J.
キム・J.H.
Kim S.Y.
Oh M.J.
リー D.
Seo N.
他。
宿主のtp53変異は、シアル酸代謝の乱れを介して炎症を誘発する腸内細菌異常症を誘導する。
Microbiome. 2022; 10: 3https://doi.org/10.1186/s40168-021-01191-x
記事で見る
スコープ(4)
PubMed
クロスフィルム
グーグル奨学生
リンチ・J.P.
ゴアーズL.
レッサー C.F.
Escherichia coli Nissle 1917に基づく治療薬開発のための新たな戦略。
Trends Pharmacol. Sci. 2022; 43: 772-786https://doi.org/10.1016/j.tips.2022.02.002
記事で見る
スコープス (12)
PubMed
概要
全文
全文PDF
Google Scholar
Sonnenborn U.
Escherichia coli Nissle 1917株-ベンチからベッドサイドへ、そして戻る:プロバイオティクス特性を持つ特別なEscherichia coli菌株の歴史。
FEMS Microbiol. Lett. 2016; 363https://doi.org/10.1093/femsle/fnw212
記事で見る
スコープス(131)
PubMed
クロスフィルム
グーグル奨学生
イザベラ V.M.
ハB.N.
カスティーヨM.J.
ルブコビッチD.J.
ロウ S.E.
ミレー Y.A.
アンダーソン C.L.
リー N.
フィッシャー A.B.
ウェストK.A.
他
ヒト代謝疾患フェニルケトン尿症に対する合成生菌治療薬の開発。
Nat. Biotechnol. 2018; 36: 857-864https://doi.org/10.1038/nbt.4222
記事で見る
スコープス(263)
パブコメ
クロスレフ
グーグル奨学生
レヴェンタールD.S.
ソコロフスカA.
リーN.
プレシアC.
コロジイS.A.
ギャラントC.W.
クリスマス・R.
ガオ J.R.
ジェームス M.J.
アビン=フエンテスA.
ら。
抗腫瘍免疫のためのSTING経路を標的とした、人工細菌による免疫療法。
Nat. Commun. 2020; 11: 2739https://doi.org/10.1038/s41467-020-16602-0
記事で見る
スコープス(117)
パブコメ
クロスフィルム
グーグル奨学生
Zhou J.J.
Wang F.
Xu Z.
ロー・W.S.
Lau C.F.
Chiang K.P.
ナングルL.A.
アシュロックM.A.
メンドレイン・J.D.
ヤン X.L.
他
炎症性筋炎の自己抗体の主要なエピトープとなるヒスチジルtRNA合成酵素のスプライスバリアントの精緻化。
J. Biol. Chem. 2014; 289: 19269-19275https://doi.org/10.1074/jbc.C114.571026
記事で見る
スコープス(38)
PubMed
概要
全文
全文PDF
グーグル奨学生
センダーR.
フックスS.
ミロR.
私たちは本当に多勢に無勢なのだろうか?ヒトにおける細菌と宿主細胞の比率を再考する。
Cell. 2016; 164: 337-340https://doi.org/10.1016/j.cell.2016.01.013
記事で見る
スコープス(1161)
PubMed
アブストラクト
全文
全文PDF
Google Scholar
フーパー L.V.
ゴードン・J.I.
腸内における宿主と細菌の共生関係。
Science. 2001; 292: 1115-1118https://doi.org/10.1126/science.1058709
記事で見る
スコープス (1823)
PubMed
クロスフィルム
グーグル奨学生
ラウンドJ.L.
リーS.M.
リー J.
トランG.
ジャブリB.
チャティラ T.A.
Mazmanian S.K.
Toll様受容体2経路はヒト微生物叢の常在菌によるコロニー形成を確立する。
Science. 2011; 332: 974-977https://doi.org/10.1126/science.1206095
記事で見る
スコープス (1159)
PubMed
クロスフィルム
グーグル奨学生
ロドリゲス-シルケY.
ヴィセクルナA.
グラウベンR.
ジークムントB.
シュタインホフ U.
粘膜および全身性免疫系による食物抗原の認識:腸の発達とホメオスタシスへの影響。
Int. J. Med. Microbiol. 2021; 311: 151493https://doi.org/10.1016/j.ijmm.2021.151493
記事で見る
スコープス(2)
パブコメ
クロスフィルム
グーグル奨学生
マントヴァーニ A.
マルケージF.
IL-10とマクロファージは腸のホメオスタシスを司る。
Immunity. 2014; 40: 637-639https://doi.org/10.1016/j.immuni.2014.04.015
記事で見る
スコープス(28)
PubMed
概要
全文
全文PDF
グーグル奨学生
イプ・ワン・ケー・イー(Ip W.K.E.
ホシ N.
シュヴァルD.S.
スナッパーS.
メジトフR.
マクロファージの代謝再プログラミングを介したIL-10の抗炎症効果。
サイエンス(Science)。2017; 356: 513-519https://doi.org/10.1126/science.aal3535
記事で見る
スコープス(683)
パブコメ
クロスフィルム
グーグル奨学生
キューン R.
レーラーJ.
レニックD.
ラジェフスキーK.
ミュラーW.
インターロイキン10欠損マウスは慢性腸炎を発症する。
Cell. 1993; 75: 263-274https://doi.org/10.1016/0092-8674(93)80068-P
記事で見る
スコープス (3688)
PubMed
要旨
全文PDF
グーグル奨学生
デニング T.L.
ワン・Y.C.
パテルS.R.
ウィリアムズI.R.(Williams I.R.
Pulendran B.
前庭マクロファージと樹状細胞は、制御性T細胞反応とインターロイキン17産生T細胞反応を異なる形で誘導する。
Nat. Immunol. 2007; 8: 1086-1094https://doi.org/10.1038/ni1511
記事で見る
スコープス (808)
PubMed
クロスフィルム
グーグル奨学生
モルハルトT.L.
林 敦
越智 崇
クイロスM.
北本 聡
ナガオ・キタモト H.
クファ P.
アタラシ K.
ホンダ K.
Kao J.Y.
et al.
マクロファージが産生するIL-10は、小腸の上皮の完全性を制御する。
Sci. Rep. 2019; 9: 1223https://doi.org/10.1038/s41598-018-38125-x
記事で見る
スコープス(53)
パブコメ
クロスフィルム
グーグル奨学生
ヘネシーE.J.
パーカーA.E.
オニールL.A.
toll-likeレセプターを標的とする:新たな治療法?
Nat. Rev. Drug Discov. 2010; 9: 293-307https://doi.org/10.1038/nrd3203
記事で見る
スコープス (645)
PubMed
クロスフィルム
グーグル奨学生
ミラー R.S.
ホスキンスL.C.
ヒト大腸生態系におけるムチン分解。最確数」法で推定したムチン分解菌の糞便集団密度。
Gastroenterology. 1981; 81: 759-765
論文で見る
PubMed
要旨
全文PDF
グーグル奨学生
オットマン N.
ダビッドM.
スアレス=ディエス M.
ボーレンS.
シャープP.J.
マルティンス・ドス・サントスV.A.P.
スミット H.
ベルザー C.
デ・ヴォスW.M.
Akkermansia muciniphilaのゲノムスケールモデルとオミックス解析による代謝能力の解析 優先的なムチン分解ライフスタイルを明らかにする。
Appl. Environ. Microbiol. 2017; 83https://doi.org/10.1128/AEM.01014-17
記事で見る
スコープス(124)
パブコメ
クロスフィルム
グーグル奨学生
ファン・デル・アーク K.C.H.
アールビンクS.
スアレス=ディエス M.
シャープP.J.
デ・ヴォスW.M.
ベルザー C.
Akkermansia muciniphilaの最小培地のモデル駆動設計は粘液の適応を確認する。
Microb. Biotechnol. 2018; 11: 476-485https://doi.org/10.1111/1751-7915.13033
記事で見る
スコープス(30)
パブコメ
クロスフィルム
グーグル奨学生
シュレーガーJ.
消化管粘液の化学組成と機能。
Gut. 1970; 11: 450-456https://doi.org/10.1136/gut.11.5.450
記事で見る
スコープス(40)
PubMed
クロスフィルム
グーグル奨学生
エムコマA.E.
炎症性腸疾患:拡大する世界的な健康問題。
Clin. Med. インサイトガストロエンテロール。2013; 6: 33-47https://doi.org/10.4137/CGast.S12731
記事で見る
スコープス (217)
パブコメ
クロスフィルム
グーグル奨学生
Ananthakrishnan A.N.
IBDの疫学と危険因子。
Nat. Rev. Gastroenterol. Hepatol. 2015; 12: 205-217https://doi.org/10.1038/nrgastro.2015.34
記事で見る
スコープス(950)
パブコメ
クロスフィルム
グーグル奨学生
グラスナー K.L.
アブラハムB.P.
クィグリーE.M.M.
マイクロバイオームと炎症性腸疾患。
J. Allergy Clin. Immunol. 2020; 145: 16-27https://doi.org/10.1016/j.jaci.2019.11.003
記事で見る
スコープス(286)
PubMed
要旨
全文
全文PDF
グーグル奨学生
ジグモンド E.
ベルンシュテインB.
フリードランダーG.
ウォーカーC.R.
ヨナ S.
キム・K.W.
ブレンナー O.
クラウトガマーR.
ヴァロルC.
ミュラーW.
ら。
マクロファージに制限されたインターロイキン10受容体の欠損は、IL-10欠損ではなく、重度の自然発生的な大腸炎を引き起こす。
Immunity. 2014; 40: 720-733https://doi.org/10.1016/j.immuni.2014.03.012
記事で見る
スコープス(391)
PubMed
要旨
全文
全文PDF
グーグル奨学生
シュヴァル D.S.
ビスワスA.
ゲッテルJ.A.
マッキャンK.
コナウェイE.
レドゥ N.S.
マスカンフローニ I.D.
アル・アダム Z.
ラヴォア S.
イボークM.
他
自然免疫細胞におけるインターロイキン10受容体シグナルは、粘膜免疫寛容と抗炎症マクロファージ機能を制御する。
Immunity. 2014; 40: 706-719https://doi.org/10.1016/j.immuni.2014.03.011
記事で見る
スコープス(364)
PubMed
要旨
全文
全文PDF
グーグル・スカラー
マーロー G.J.
ヴァン・ジェントD.
ファーガソンL.R.
インターロイキン10の補充がクローン病患者に効かない理由。
World J. Gastroenterol. 2013; 19: 3931-3941https://doi.org/10.3748/wjg.v19.i25.3931
記事で見る
スコープス(107)
PubMed
クロスフィルム
グーグル奨学生
Yim J.
Cho S.W.
Kim B.
Park S.
ハン Y.H.
Seo S.W.
プロバイオティクスEscherichia coli Nissle 1917 Strainの模擬微小重力下における転写プロフィル。
Int. J. Mol. サイ. 2020; 21: 2666
論文で見る
スコープス (16)
パブコメ
クロスフィルム
グーグル奨学生
ガック M.U.
シン・Y.C.
Joo C.H.
ウラノ T.
Liang C.
Sun L.
竹内 O.
アキラ S.
陳 Z.
井上 聡.
他
TRIM25 RING-finger E3 ユビキチンリガーゼはRIG-Iを介した抗ウイルス活性に必須である。
Nature. 2007; 446: 916-920https://doi.org/10.1038/nature05732
記事で見る
スコープス (1222)
PubMed
クロスフィルム
グーグル奨学生
ミルディタM.
シュタイネガーM.
Söding J.
MMseqs2デスクトップとローカルウェブサーバーアプリによる高速で対話的な配列検索。
バイオインフォマティクス(Bioinformatics)。2019; 35: 2856-2858https://doi.org/10.1093/bioinformatics/bty1057
記事で見る
スコープス(125)
パブコメ
クロスレフ
グーグル奨学生
ティアノバ S.
テムT.
Cox J.
質量分析ベースのショットガンプロテオミクスのためのMaxQuant計算プラットフォーム。
Nat. Protoc. 2016; 11: 2301-2319https://doi.org/10.1038/nprot.2016.136
記事で見る
スコープス(2027)
パブコメ
クロスフィルム
グーグル奨学生
ティアノヴァ S.
テムT.
シニトシンP.
カールソンA.
ハイン M.Y.
ガイガーT.
マン M.
コックス J.
プロテオミクスデータの包括的解析のためのPerseus計算機プラットフォーム。
Nat. Methods. 2016; 13: 731-740https://doi.org/10.1038/nmeth.3901
記事で見る
スコープス (3756)
パブコメ
クロスフィルム
グーグル奨学生
ドビンA.
デイビスC.A.
シュレシンジャーF.
ドレンコウJ.
ザレスキーC.
ジャ S.
バトゥット P.
シャイソン M.
ジンゲラス T.R.J.B.
STAR:超高速ユニバーサルRNA-seqアライナー。
Bioinformatics. 2013; 29: 15-21https://doi.org/10.1093/bioinformatics/bts635
記事で見る
スコープス (20526)
PubMed
クロスフィルム
グーグル奨学生
ゴールドマン M.J.
クラフト・B.
ヘイスティ M.
レペチカK.
マクデイドF.
カマス A.
バネルジー A.
ルオ Y.
ロジャース D.
ブルックス A.N.J.N.b.
他
Xenaプラットフォームによるがんゲノムデータの可視化および解釈。
Nat. Biotechnol. 2020; 38: 675-678https://doi.org/10.1038/s41587-020-0546-8
記事で見る
スコープス (1249)
PubMed
クロスフィルム
グーグル奨学生
ハウ E.A.
シンハR.
シュラウチD.
クワッケンブッシュ J.J.B.
MeVでのRNA-Seq解析。
Bioinformatics. 2011; 27: 3209-3210https://doi.org/10.1093/bioinformatics/btr490
記事で見る
スコープス (343)
PubMed
クロスフィルム
グーグル奨学生
コルデ R.
コルデ M.R.J.R.p.
パッケージ 'pheatmap'.
2015: 790
記事で見る
グーグル スカラー
スブラマニアン A.
タマヨP.
ムータ・V.K.
ムカルジーS.
イベートB.L.
ジレットM.A.(Gillette M.A.
パウロヴィッチ A.
ポメロイS.L.
ゴルブ T.R.
ランダー E.S.
他
遺伝子セット濃縮解析:ゲノムワイド発現プロファイルを解釈するための知識ベースのアプローチ。
Proc. Natl. Acad. Sci. USA. 2005; 102: 15545-15550https://doi.org/10.1073/pnas.0506580102
記事で見る
スコープス (27801)
PubMed
クロスフィルム
グーグル奨学生
クレショフ M.V.
ジョーンズM.R.
ルイラードA.D.
フェルナンデスN.F.
Duan Q.
ワン・ズ
コプレフ S.
ジェンキンス S.L.
ジャゴドニック K.M.
ラッハマンA.
などがある。
Enrichr: a comprehensive gene set enrichment analysis web server 2016 update.
Nucleic Acids Res. 2016; 44: W90-W97https://doi.org/10.1093/nar/gkw377
記事で見る
スコープス(4248)
PubMed
Crossref
グーグル奨学生
マリック H.
ラーナバードA.
マカイバーL.J.
Ma S.
チャン・Y.
グエン・L.H.
ティックル・T.L.
ウェインガート・G.
レン B.
シュワガー E.H.
ら。
集団規模のメタオミクス研究における多変量関連発見。
PLoS Comput. Biol. 2021; 17: e1009442https://doi.org/10.1371/journal.pcbi.1009442
記事で見る
スコープス(233)
パブコメ
クロスフィルム
グーグル奨学生
ペレス・リベロル Y.
バイ・J.
バンドラC.
ガルシア-セイスデドスD.
ヘワパティラナS.
カマッチナタン S.
クンドゥ D.J.
プラカシュ A.
フレリックス=ジッパー A.
アイゼナッカーM.
et al.
2022年のPRIDEデータベースリソース:質量分析に基づくプロテオミクスエビデンスのハブ。
Nucleic Acids Res. 2022; 50: D543-D552https://doi.org/10.1093/nar/gkab1038
記事で見る
スコープス (957)
PubMed
クロスフィルム
グーグル奨学生
デリエン M.
ヴォーンE.E.
プラグ・C.M.
デボスW.M.
ヒト腸管ムチン分解菌Akkermansia muciniphila gen.nov., sp.nov.
Int. J. Syst. Evol. Microbiol. 2004; 54: 1469-1476https://doi.org/10.1099/ijs.0.02873-0
記事で見る
スコープス (1254)
PubMed
クロスフィルム
グーグル奨学生
スタムス A.J.
ヴァン・ダイクJ.B.
ダイケマC.
プラッゲC.M.
メタン生成菌非存在下におけるフマル酸を用いた合成栄養型プロピオン酸酸化細菌の増殖。
Appl. Environ. Microbiol. 1993; 59: 1114-1119https://doi.org/10.1128/aem.59.4.1114-1119.1993
記事で見る
スコープス(265)
パブコメ
クロスフィルム
グーグル奨学生
アーン S.
ジン T.E.
Chang D.H.
Rhee M.S.
Kim H.J.
イ S.J.
パクD.S.
Kim B.C.
ヒト糞便から分離された厳密な嫌気性酪酸産生腸内細菌Agathobaculum butyriciproducens gen.nov.sp.nov. およびEubacterium desmolansをAgathobaculum desmolans comb.nov.
Int. J. Syst. Evol.Microbiol. 2016; 66: 3656-3661https://doi.org/10.1099/ijsem.0.001195
記事で見る
スコープス(20)
パブコメ
クロスフィルム
グーグル奨学生
マーシー S.N.
クーパーH.S.
シム H.
シャー・R.S.
イブラヒムS.A.
Sedergran D.J.
デキストラン硫酸ナトリウム誘発マウス大腸炎に対するシクロスポリン内服による治療法
Dig. Dis. Sci. 1993; 38: 1722-1734https://doi.org/10.1007/BF01303184
記事で見る
スコープス (464)
パブコメ
クロスフィルム
グーグル奨学生
セロンR.K.
トンコノジー S.
シュルツ M.
ディールマンL.A.
グレンサー・W.
バリッシュ E.
レニック・D.M.
Sartor R.B.
インターロイキン10欠損マウスにおける自然発症の大腸炎と免疫系の活性化には常在腸内細菌が必要である。
Infect. Immun. 1998; 66: 5224-5231https://doi.org/10.1128/IAI.66.11.5224-5231.1998
記事で見る
PubMed
クロスフィルム
グーグル奨学生
リ・エムズ
エッジ S.J.
SLIC:配列・ライゲーション非依存型クローニング法
Methods Mol. Biol. 2012; 852: 51-59https://doi.org/10.1007/978-1-61779-564-0_5
記事で見る
スコープス (179)
PubMed
クロスフィルム
グーグル奨学生
テオドロヴィッチ M.
パーダイクO.
フェルフックI.
ゴーバースC.
サベルクールH.F.
タン Y.
ウィッチャーズ H.
Broersen K.
最適化されたTriton X-114アシストによるリポポリサッカライド(LPS)除去法により、食品タンパク質の免疫調節作用が明らかになった。
PLoS One. 2017; 12: e0173778https://doi.org/10.1371/journal.pone.0173778
記事で見る
スコープス(51)
PubMed
クロスフィルム
グーグル奨学生
リュウ J.H.
チェンC.Y.
Liu Z.Z.
ルオ・Z.W.
ラオ・S.S.
ジン L.
Wan T.F.
Yue T.
タン Y.J.
Yin H.
et al.
子供の腸内細菌叢からの細胞外小胞は骨に入り込み、骨量と強度を維持する。
Adv. Sci. (Weinh). 2021; 8: 2004831https://doi.org/10.1002/advs.202004831
記事で見る
スコープス(43)
PubMed
クロスフィルム
グーグル奨学生
チュアン・L.S.
モリソン J.
Hsu N.Y.
ラブリアスP.R.
ナヤル S.
チェン E.
ヴィラベルデ N.
フェイシーJ.A.
ボスケッティG.
ギリ M.
他
腸管損傷、細菌曝露、薬剤のゼブラフィッシュモデリングにより、ヒト炎症性腸疾患に関連する上皮のin vivo応答が定義された。
Dis. Model. Mech. 2019; 12https://doi.org/10.1242/dmm.037432
記事で見る
スコープス(22)
パブコメ
クロスフィルム
グーグル奨学生
姜 毅(Jiang Y.)
チェンB.
Duan C.
Sun B.
Yang J.
ヤン S.
CRISPR-Cas9システムによる大腸菌ゲノムの多遺伝子編集。
Appl. Environ. Microbiol. 2015; 81: 2506-2514https://doi.org/10.1128/AEM.04023-14
記事で見る
スコープス (683)
パブコメ
クロスレフ
グーグル奨学生
記事情報
出版履歴
掲載されました: 2023年6月2日
受理された: 2023年5月9日
改訂版受理 2023年3月23日
受理された: 2022年7月23日
出版段階
インプレス、修正プルーフ
識別
DOI: https://doi.org/10.1016/j.chom.2023.05.007
著作権について
© 2023 The Author(s). 発行:エルゼビア・インク
ユーザーライセンス
クリエイティブ・コモンズ 表示 (CC BY 4.0) |.
再利用の方法
サイエンスダイレクト
ScienceDirectでこの論文にアクセスする
図版
図解要旨
図1A. ムチニフィラは細胞外にAmTARSを分泌し、抗炎症性の免疫制御機能を発揮する
図2AmTARSはIL-10陽性マクロファージを活性化することで、マウスのDSS誘発大腸炎を改善させる
図3AmTARSのユニークな領域が抗炎症性免疫制御に必須であることがわかる
図4AmTARSはTLR2依存の抗炎症性免疫メディエーターである
図5AmTARSはCREBを活性化することで抗炎症反応を効率的に促進する
図6AmTARSは定常的な、免疫恒常性メディエーターである
図7EcN(AmTARS)の経口投与はDSS誘発大腸炎を緩和する
関連記事
本サイトのコンテンツは、あらゆる分野の医療従事者や研究者を対象としています。
研究ジャーナル
細胞
癌細胞
細胞化学生物学
細胞ゲノミクス
細胞宿主と微生物
細胞代謝
セルレポート
セルレポートメディスン
セルレポートメソッド
セルレポートフィジカルサイエンス
細胞幹細胞
細胞システム
化学
化学触媒
カレントバイオロジー
発生細胞
ヘリオン
イミュニティ
iScience(アイサイエンス
ジュール
物質
医学
分子細胞
ニューロン
一つの地球
パターン
STAR プロトコル
構造
トレンドレビュー誌
生物化学
バイオテクノロジー
癌
細胞生物学
化学
認知科学
エコロジー&エボリューション
内分泌学・代謝学
遺伝学
免疫学
微生物学
分子医学
神経科学
寄生虫学
薬理学
植物科学
パートナージャーナル
AJHG
生物物理学雑誌
生物物理学レポート
EBioMedicine
HGGアドバンス
モレキュラープラント
分子療法ファミリー
植物通信
幹細胞レポート
イノベーション
コレクション
ベスト・オブ・セルプレス
セルプレスレビュー
セルプレスセレクション
コンソーシアムハブ
Nucleus Collections
スナップショット・アーカイブ
ジャーナルを越えて
Cellキャリアネットワーク
セルメンター
細胞シンポジューム
ラボリンク
ウェビナー
記事を進化させる
コミュニティレビュー
Figure360
スニークピーク
STARメソッド
サイエンス・イン・ソサイエティ
セル画展
セルプレスポッドキャスト
セルプレスビデオ
ぬりえ・コミック
リサーチアーク
コネクト
セルプレスについて
採用情報
お問い合わせ
ヘルプ&サポート
ニュースルーム
出版物アラート
アクセス
購読する
今すぐ読む
図書館に薦める
INFORMATION
広告主様向け
採用担当者様へ
図書館員の方へ
ご利用条件
個人情報保護方針
アクセシビリティ
当社は、サービスの提供・向上およびコンテンツのカスタマイズのためにクッキーを使用しています。クッキーの設定を変更するには、本サイトのクッキー設定にアクセスしてください。
著作権 © 2023 Elsevier Inc.ただし、第三者が提供する一部のコンテンツを除く。
この記事が気に入ったらサポートをしてみませんか?
