
未知の種レベルのゲノムビンを用いたMetaPhlAn 4プロファイリングにより、マウスの食事に関連したマイクロバイオームの変化の特徴づけが改善される
メインコンテンツへスキップ
ログイン
報告書|42巻5号、112464、2023年5月30日発行
全号ダウンロード
未知の種レベルのゲノムビンを用いたMetaPhlAn 4プロファイリングにより、マウスの食事に関連したマイクロバイオームの変化の特徴づけが改善される
パオロ・マンギ 10
アイトール・ブランコ=ミゲス 10名
セレーナ・マナラ
ティル・ストローウィグ
ハリー・ソコル
ニコラ・セガッタ 11
すべての著者を表示する
脚注を表示する
オープンアクセス公開日:2023年05月03日DOI:https://doi.org/10.1016/j.celrep.2023.112464
PlumX メトリクス
ハイライト
マウスの腸内細菌叢はまだ未知の微生物分類群に支配されている
未知の分類を考慮することで、マウスの食事とマイクロバイオームの関連性が改善される
食事による脂肪摂取の違いを示す最強のバイオマーカーは未知のタクサン
MetaPhlAn 4は、マウスで未知の分類群を正確にプロファイリングすることができます。
概要
マウスモデルは、宿主とマイクロバイオームの相互作用を調べるための重要なツールである。しかし、ショットガンメタゲノミクスでは、マウス腸内細菌叢の限られた部分しかプロファイルできない。ここでは、マウスの腸内細菌叢のプロファイリングを改善するために、メタゲノム集合ゲノムの大規模カタログ(マウスの22,718個のメタゲノム集合ゲノムを含む)を利用するメタゲノムプロファイリング法、MetaPhlAn 4を採用する。我々は、8つの公開データセットと97のマウスマイクロバイオームの追加コホートから622サンプルを組み合わせ、メタアナリシス手法を使用して、宿主マイクロバイオームの食事関連変化をより良く特定するためのMetaPhlAn 4の可能性を評価しました。その結果、食事に関連する微生物バイオマーカーが複数、強力、かつ再現性よく見つかり、参照情報のみに依存する他の手法で同定可能なバイオマーカーを大幅に増加させることができました。食事に起因する変化の最も強力なドライバーは、未特定の分類群やこれまで検出されていなかった分類群であり、包括的なプロファイリングのためにメタゲノム集合体を統合したメタゲノム手法を採用することの重要性が確認された。
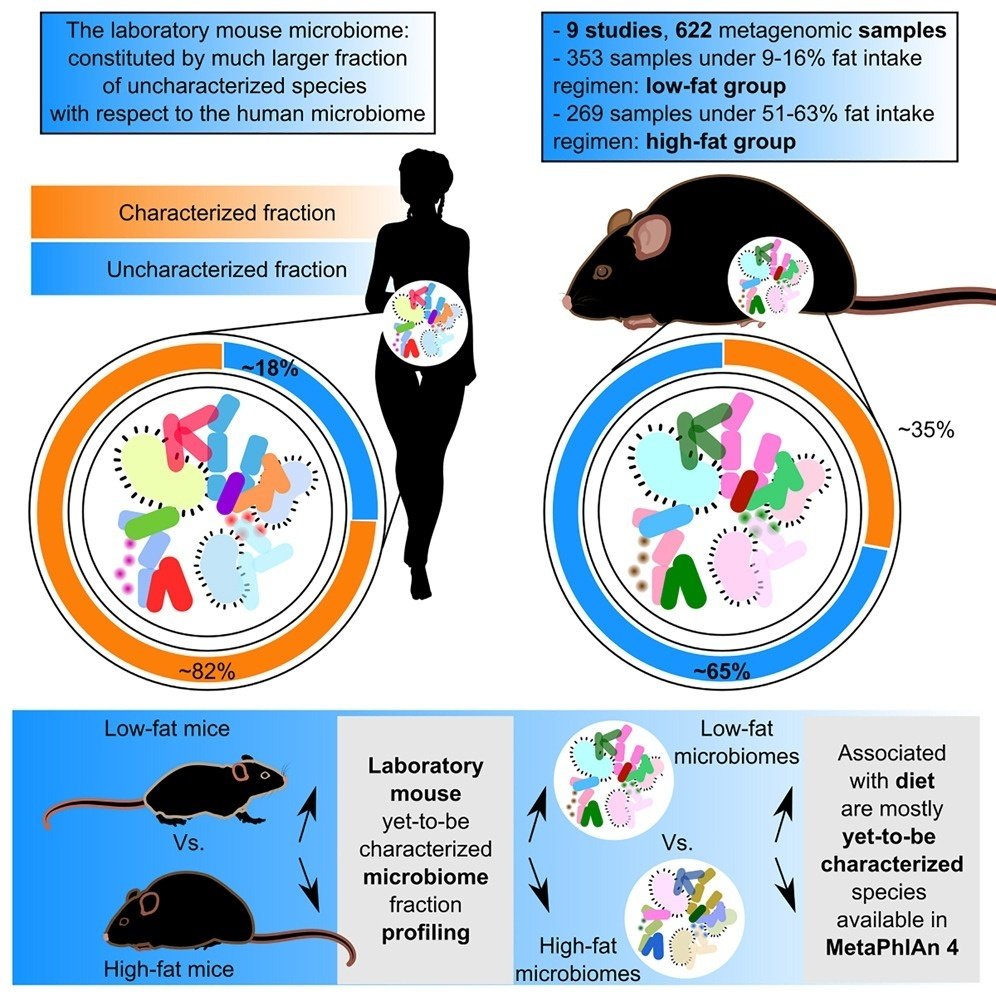
図解抄録
大きな画像を見る
高解像度画像ダウンロード
キーワード
マウスマイクロバイオーム
メタゲノミクス
ダイエット
未特定の微生物種
メタファルナン
研究テーマ
CP:マイクロビオロジー
はじめに
マウスは、進化的、解剖学的、生理学的にヒトに近いことから、生物医学研究のモデル生物として成功を収めています。飼育が容易であること、有効な疾患モデル、増殖が早いこと、さらに多世代実験や食事に関連した介入を行うことができることから、マウスはヒト腸内細菌叢の研究のための主要な前臨床モデルとして確立しました。
1
,
2
,
3
,
4
,
5
,
6
マウスでは、遺伝学、栄養学的または薬学的な曝露、およびその他の実験構成要素などのいくつかの変数を制御しながらマイクロバイオーム実験を実施することができます。
1
,
2
,
3
,
5
,
6
,
7
,
8
,
9
,
10
,
11
,
12
,
13
,
14
,
15
,
16
,
17
,
18
,
19
しかし、実験用マウスや野生マウスのマイクロバイオームの構成は、ヒトとは異なるため、
3
,
4
マウスマイクロバイオームの構造と多様性については、まだ包括的な研究が行われておらず、その結果、マウスの基礎研究やトランスレーショナルリサーチに限界がある。
マウスの食餌、疾患、表現型に関連する変化を特徴づけるマイクロバイオームの特徴の解析は、16S rRNA遺伝子アンプリコンシーケンスを用いて広範囲に行われている、
5
,
14
,
15
,
20
,
21
,
22
,
23
は、コストの削減にもかかわらず、その系統的、分類学的、機能的な解像度に限界があると考えることができる。高解像度ショットガンメタゲノムアプローチは、現在、ヒトマイクロバイオーム研究の標準となっているものである、
24
は、マウスのマイクロバイオームの大半のメンバーをカバーする参照ゲノムがないため、マウス研究ではまだあまり採用されていません。
25
マウスメタゲノムの系統的なバイオインフォマティックアセンブリにより、マウスマイクロバイオームの多様性をデノボでカタログ化する取り組みが行われています、
25
,
26
,
27
,
28
,
29
しかし、新しい研究やリファレンスベースの分類学的プロファイリングをサポートするために、それらを効率的に利用することは依然として困難である。
本研究では、MetaPhlAn 4と呼ばれる計算論的分類学的プロファイリング手法を活用し、ショットガンメタゲノミクスによってマウスの腸内細菌叢とその栄養パターンとの関連性を正確かつ包括的に調査できることを示すものである、
30
MetaPhlAnは、そのデータベースに大規模なアセンブルを統合したものである。MetaPhlAn 4は、マウス由来の糞便、セカル、回腸のメタゲノム1,906個から、種レベルのゲノムビン(SGB)戦略で取得した22,718個以上のメタゲノム集合体(MAGs)を考察しています。
31
このアプローチを大規模かつ異質なマウスマイクロバイオームカタログに適用することで、マウスモデルにおける栄養学的研究の文脈では、メタゲノム的に定義された種を考慮することが必要であることを示す。また、分離株からのゲノムに対するマッピングでは検出できない微生物種が、食事に関連したマイクロバイオームの変化の最大の割合を占めていることも明らかにした。
研究成果
高脂肪食がマウスの腸内細菌叢に与える影響を研究するためのマルチコホートデータセット
高脂肪食がマウスの腸内細菌叢に及ぼす影響を調べるために、我々は、食事中の脂肪の含有量と変動量に関してマウスの細菌叢を評価する公開メタゲノムデータセットを収集した(表S1)。収集したサンプルについて、年齢、抗生物質の使用状況、可能であれば複数の時点、遺伝的背景、サンプリング部位、食事情報などの関連するマウスの共変数を手動でキュレーションし検証した(表S2)。食事については、特に食事中の脂肪由来のカロリーの割合(以降、「脂肪率」と呼ぶ)と食事介入開始からの経過時間に注目した(Table S2)。当初は15の公開データセットから623サンプルを取得したが、少なくとも2つの食事処理(高脂肪群と低脂肪群、または複数の食事脂肪摂取群)があり、総サンプル数が20以上あるデータセットのみを保持し、8データセットから525サンプルに削減した。さらに、マウスを複数の食事レジメンや抗生物質処理など、複数種類の腸内細菌叢摂動に供したマウスマイクロバイオーム研究のセカールおよび回腸腸内細菌叢をシーケンスし、合計97個のメタゲノム(STAR Methodsを参照)を取得しました。全体として、9つのデータセットと622サンプル(高脂肪摂取レジメンに従う269サンプル、平均脂肪含量57%、95%信頼区間(CI)[0.51、0.63];および低脂肪摂取レジメンに従う353サンプル、平均13%、95%CI[0.09、0.16])を解析しました(表S1およびS2)。
マウス腸内細菌叢の再検索により、未特定の微生物種が圧倒的に存在することがわかった
利用可能な分離株とMAG(平均配列深度=4690万、95%CI [7.6, 86])で表されるすべての種について統合コホートの正確な分類学的プロファイリングを行うために、MetaPhlAn 4を適用しました。
30
MetaPhlAn 4はSGBアプローチを採用
31
は、参照ゲノムとMAGの両方を既知または未知の種にグループ化し、kSGBとuSGBとラベル付けしています。未知種(uSGB)は、このように、未培養のままであり、その存在がメタゲノム集合体からの情報に依存している微生物種の代理である。uSGBと既知のSGB(kSGB)の両方を検出・定量化することで、既存の参照ゲノムで表現されていないマイクロバイオームの一部もプロファイリングすることが可能になります。実際、複数のソースから入手した1,906個のマウス腸内メタゲノムから再構成した22,718個のMAGを組み込んだ後(表S3)、MetaPhlAn 4は、同じサンプルで行ったMAG再構成手順では捉えられなかった336個のマウス関連uSGBを特定し定量することができ、このアプローチの強さを強調しています。本研究で検討した622サンプルにおいて、703の異なる種から合計740の微生物SGB(uSGB425、kSGB316、サンプルあたりの平均SGB数142、95%CI [28.32, 255.68] )を特定し、MetaPhlAn 3による197種の検出を大幅に上回った。
次に、単一のSGBの有病率を評価し、9つのデータセットにわたる有病率の平均値として全体の有病率を推定しました。その結果、有病率が50%を超えるuSGBの数は、有病率の高いkSGBの数よりも多く(uSGB51種 vs kSGB25種)、すべての有病率パーセンタイルでuSGBがkSGBを統計的に上回った(p = 4.5×10-4, 二項検定、図1A)。さらに、サンプルごとの豊富さを考慮すると、マウスの腸内細菌叢はkSGBよりもuSGBを多く保有していることがわかりました(サンプルごとの平均数 = 95, 95% CI [10.72, 179.28], vs. 49, 95% CI [9.8, 88.2], それぞれ Wilcoxon signed-rank test, p < 2 × 10-16; 図1B)。uSGBは、個々のマイクロバイオームサンプルにおいてkSGBよりも高い相対的存在量を占めている(サンプルあたりの平均50.88%、95% CI [46.95, 54.81] for uSGB vs. 48.94%, 95% CI [45.01, 52.87] for kSGB, Wilcoxon signed-rank test, p = 0.005; Figure 1C)。これらの結果は、マウス腸内細菌叢の未知の部分にもアクセスし、その特徴を明らかにする必要があることを裏付けています。
図1マウス腸内細菌叢は、微生物叢と宿主の食事療法との関連を改善するために非常に関連性の高い、未特性の微生物種によって支配されている。
フルキャプションを表示
大きな画像を見る
高解像度画像ダウンロード
uSGBsを考慮することで、高脂肪食と低脂肪食の介入の識別が向上する
次に、高脂肪食が実験用マウスのマイクロバイオームに与える影響を、αおよびβ多様性の変化を通して評価した。シャノン多様性は、低脂肪群と比較して6つのデータセットの高脂肪群で増加したが(平均標準化平均差[aSMD]=1.16、95% CI [1.06, 1.26], p < 0.05 [Wilcoxon signed rank] で4つ有意)、これらのアルファ多様性の増加は交絡因子考慮時には有意ではなかった(STAR Methodsおよび表S2参照)。β多様性は、Bray-Curtis非類似度行列のPermanova分析を用いて各データセットで分析し、9データセット中8データセットで有意性を認めた(一変量のPermanova p < 0.05; Figure S1A)。低脂肪マウスと高脂肪マウスのペアワイズ距離の群平均差を測定したところ、低脂肪群の9つのうち6つが、対応する高脂肪群よりも変動が大きかった(全体のaSMD = 0.71, 95% CI [0.004, 1.416] )。研究ID、マウス系統、年齢、性別、サンプリングボディ部位、食事タイプ(高脂肪または低脂肪)で補正した多変量ペルマノバによる順列検定では、マイクロバイオーム組成の変動性を決定する最も重要な変数として研究ID(調整R2=36%)が特定され、次いでマウス系統(調整R2=21%)が示された。高脂肪食または低脂肪食の投与がマイクロバイオームの総変動に占める割合は、試験IDの変動に反映される実験的差異と比較して小さいため(調整済みR2=3%)(図S1Bおよび表S4)、このような試験設定の効果を明確に考慮したクロススタディおよび多変量のメタ解析モデルを用いて、食事に関連する変化を調べた。
マウスの腸内細菌叢と高脂肪食レジメンと低脂肪食レジメンとの関連をさらに評価するために、我々はランダムフォレスト
32
食事条件とサンプルに含まれるSGBの存在や量を関連付けるための分類器アプローチ。
33
,
34
,
35
2つの相互予測マトリックスを構築した
36
,
37
(1)各データセットに対して個別にクロスバリデーション(CV)で分類器を学習・評価する、(2)あるデータセットで分類器を学習し、別のデータセットで適用する、(3)あるデータセットを除くすべてのデータセットで分類器を学習し、省くデータセットで適用する(リーブオンダータセットアウト[LODO])ことにより、分類器を作成しました。
36
,
37
,
38
,
39
抗生物質の介入がないサンプルだけを考慮した場合(表S2),微生物ベースのモデルは,すべての予測設定で高脂肪食と低脂肪食を与えたマウスの間で高い識別性を達成した(図1D).すべてのSGBを組み込んだMetaPhlAn 4プロファイルを使用すると,RefSeq
40
が含まれています(RefSeqベースのSGB、STAR Methodsを参照)。CVでの性能は2つのCV設定の間で大きく変化しませんでしたが、MetaPhlAn 4データベースを組み込んだLODO設定では著しく向上し、平均曲線下面積(AUC)は0.80となりました(RefSeqベースのSGBのみではAUC 0.74 )(図 1D)。抗生物質を投与したマウスのサンプルを考慮した場合にも同様の改善が見られ、CVの平均AUCは0.92、LODOは0.79に達しました(RefSeqベースのSGBのAUCのみを用いた場合の平均はそれぞれ0.91と0.72です;図S2)。このことから、コホート内予測(CV)を正確に行うには、十分に特性化された種で十分である一方、モデルの汎化のより良い代理となるコホート間(LODO)性能は、uSGBに大きく依存するようであることがわかります。このように、まだ解明されていない生物種のMAGを組み込むことで、食餌療法など関心のある条件とのより強い再現性のある関連性を見つけることができます。
高脂肪食と低脂肪食のデータセット横断的な微生物バイオマーカーの多くは、uSGBsである。
各SGBについて、また各データセットについて、性別、年齢、抗生物質投与、マウス系統、サンプリング部位で調整した線形モデルを用いて、高脂肪食体制と低脂肪食体制での存在量の差分解析を行った。そして、各SGBの線形モデルの結果は、ランダム効果モデルによるメタ分析でプールされた。
41
,
42
(STAR Methodsを参照)。高脂肪食と有意に関連する37のSGBと低脂肪食と関連する10のSGBを同定した(偽発見率[FDR] < 0.2, 20%以上の有病率で少なくとも5つのデータセットに存在するSGBを考慮;表S5)。重要なのは、これらの47のバイオマーカーのうち32がuSGBであることです(予想される偶数頻度に関するカイ2乗p:0.01)。グループ間の差が最も大きい30個のSGB(図2A)を考慮すると、11個だけがkSGBであり、そのうちの9個はまだ十分に特徴づけられていない微生物種に属する(すなわち、Turicibacter sp. TS3、Lachnospiraceae bacterium MD308、細菌D16-50、Emergenceia sp. 1XD21-10 、Dorea sp. 5-2 、「Lachnospiraceae bacterium」と表示された3つの異なるSGBおよびLachnospiraceae bacterium 28-4 ; 図2A)。最大の効果量を持つ3つのSGBは、Lachnospiraceae bacterium 28-4(SGB7272)、Adlercreutzia caecimuris(SGB14802)、Ruminococcaceae SGB43546(aSMD = 1.05, 95% CI [0.48, 1. 63], 1.05, 95% CI [0.33, 1.76], and 0.99, 95% CI [0.13, 1.86]; Q = 0.007, 0.04, and 0.13, respectively) これは、平均存在量の 4.0, 6.6, 3.7 倍増加に相当する(STAR Methods 参照)。これらのSGB(1つのuSGBと2つのkSGBはゲノム解析が不十分である)は、一次胆汁酸を二次胆汁酸に変換することが確認されている属に属していることが注目される。
43
または、腸の高脂肪関連炎症に対する防御的な役割について説明されている。
44
A. caecimuris (SGB14802) は、さらに、高脂肪食摂取時のレプチン増加、および血清一次胆汁酸との相関がある可能性があります。
45
,
46
,
47
図2高・低脂肪食関連SGBの標準化平均値差のメタアナリシス
フルキャプションを表示
大きな画像を見る
高解像度画像ダウンロード
少なくとも5つのデータセットで低脂肪群で増加したSGBは10種のみで、最も強い関連を示したのはClostridia SGB41001(uSGB, aSMD = -0.61, 95% CI [-1.14, -0.08], Q = 0.13, fold change = 1.6, 95% CI [0.95, 2.7] )である。低脂肪関連株で属が特定された唯一のSGBはTuricibacter sp. TS3 (SGB39153) (aSMD = -0.57, 95% CI [-1.08, -0.06], Q = 0.15, fold change = 4.5, 95% CI [1.0, 19.4]) である.この結果は、肥満でTuricibacterの存在量が減少することを示した先行研究と一致するものである、
49
核因子-κB蛋白質複合体との負の相関がある。
50
は、炎症シグナル伝達に中心的な役割を果たすが、炎症性腸疾患患者ではその存在量が少ない。
51
全体として、高脂肪食または低脂肪食に関連する分類群の大部分はuSGBであり、kSGBとuSGBの両方を統合したメタゲノム分類プロファイリング(MetaPhlAn 4)が、マウス腸内メタゲノムの複雑性を克服する基本であることが示された。
食事性脂肪の摂取と微生物のシフトに相関がある
次に、研究によって脂肪摂取量が異なることがあるため、食事脂肪の割合の違いが各SGBの存在量と直接相関するかどうかを検証した(表S1およびS2;図S1A)。そこで、前回のメタ分析で考慮した共変量と同じ利用可能なセット、および投与期間について回帰モデルを当てはめ、脂肪の割合とSGBの存在量の間の部分的なスピアマン相関のメタ分析を行った(図S3およびS5;表S6)。合計で、脂肪量と相関する44のSGB(スピアマンの偏相関Q<0.2、少なくとも4つの研究で検出)を見つけ、そのうち32(73%)は正の相関があった(図S3Bおよび2B)。予想通り、これらの有意な相関と前回の二値結果関連メタ解析の間には強い一致が見られた(2つの解析の効果量間のPearson, Spearman, Kendallのrho = それぞれ0.79, 0.76, 0.36; Pearsonのp = 1×10-61, Spearmanのp = 3×10-53, Kendallのp = 9.5×10-19; Figure S3C)。
バイナリーアソシエーションメタ解析と同様に、食事中の脂肪量と正負に相関するSGBの大部分はuSGBであり(それぞれ59%、83%、合計で44件中29件がuSGB、カイ二乗p=0.03)、食事期間と相関するSGBの大部分(最低3研究で検出した合計23件中19件、カイ二乗p=0.002)にもなった。これらのuSGBはすべて既知の属にすら割り当てられておらず、その多くはLachnospiraceaeとRuminococcaceaeのファミリーに属していた(それぞれ29個のuSGBのうち9個と4個)。全体として、kSGBよりもuSGBの方が有意な相関を検出し、脂肪量と相関するSGBタイプ(uSGBとkSGB)ごとの上位15SGBを考慮すると、uSGBとの相関は、フィッシャーZ変換ベースの平均でkSGBよりも高かった(平均スピアマンのrho = 0.30 vs. 0.29; STAR Methods参照)。これらの結果は、uSGBがkSGBよりも脂肪食への反応やその期間により優位に関与していることを示唆している。
独立した縦断的データセットが、食事に関連したuSGBの一貫したダイナミクスを示す
最初の観察結果をさらに検証するために、さらに2つの縦断的データセットを分析しました。
7
,
48
において、マウス群に複数の食事介入を行った。Nobel et al.のデータセットから。
7
は、3匹のマウスから採取した18個のメタゲノム(各6時点、図2D)を取得した。
48
は、異なる複数の食事療法を受ける各3匹のマウスからなる2つの異なるグループ(図2Eおよび図2F)に内部的に細分化される。
横断的メタアナリシス(図2A)で最も高い識別係数を持つ30個のSGBのうち、ランダムフォレストアルゴリズムで最も高くランク付けされた8個のuSGBを選択しました(表S7)。これらのuSGBの時間的軌跡を評価したところ、3つのデータセットすべてにおいて、脂肪摂取量が多い場合に相対量が明らかに増加することが確認された(図2D-2F、ウィルコクソン署名順位検定p = 5.3×10-5, 5.9×10-6, 0.0003, "(L)LFD" から "HFD" それぞれ)。低脂肪摂取に切り替えると、その量は再び減少し、その変化は1日単位で観察できた(図2Eおよび2F;Wilcoxon signed-rank test p = 1.3 × 10-5 および 0.0004, "HFD" to "(L)LFD", respectively)。興味深いことに、この変化は、軽度の脂肪の増減に対する反応としても直接観察することができ、KimとBaeのマウスの両群は
48
図2Eおよび図2F)。脂肪摂取量10%の低脂肪食(「LFD」)から脂肪摂取量6.2%の低脂肪食(「LLFD」)へ移行すると、8種類のuSGBの存在量中央値がわずかに減少することがわかりました。このように、高脂肪食の最も強力で未特性の横断的な微生物バイオマーカーは、2つの介入コホートにおいて食事との関連性を確認した。
考察
我々は、MetaPhlAn 4マーカーベースアプローチの参照データベースに膨大な数のMAG(マウス由来22,718個)を統合することにより、実験用マウスの腸内細菌叢を分類学的に高解像度でプロファイリングすることに成功しました。この分解能の向上が、マウスのマイクロバイオームと宿主の状態との関連性の発見につながるかどうかを調べるために、我々は、マイクロバイオームと食事との関連性の解析に焦点を当てました。我々は、9つのマウスマイクロバイオームデータセット(そのうちの1つは本研究で利用可能になった)を収集し、手作業でキュレーションし、プロファイリングを行った。このデータはすべて、脂肪の割合が異なる複数の食事レジームが存在するという特徴を持つ。MAG-enhancedデータベースを用いてプロファイリングしたコホートについて機械学習とメタ分析を行ったところ、食事とのコホート横断的な関連性が、従来よりも強く、そのほとんどが未特性の微生物種(uSGBs)によってもたらされていることが明らかになりました。
この結果は、マウスマイクロバイオームデータの分類学的プロファイリングの過程で、未培養微生物のゲノムを含める必要性と、メタゲノム解析によってのみ得られる種が、特に実験室マウスにおける宿主とマイクロバイオームの相互作用に果たす重要な役割を明らかにした。重要なことは、MetaPhlAn 4がuSGBプロファイリングをメタゲノム解析に効率的に統合できることで、マウスモデルのマイクロバイオーム解析を大きく改善できることを示したことである。
本研究の限界
私たちの研究とツールは、前臨床モデルにおけるマイクロバイオームと宿主-マイクロバイオーム相互作用に対する栄養学的効果についてのより微妙な研究の基礎となり得るものである。例えば、飽和脂肪酸と多価不飽和脂肪酸の摂取量の違いを考慮した研究デザインを改善することができる。
52
私たちの研究では、ベースライン時のマウスの体重で分析を補正したり、多糖類の栄養成分を考慮したりすることはできませんでした、
13
が、これらの側面は、食事誘発性肥満の発症に関して、わずかな交絡因子であることが示された。
53
,
54
,
55
食事-微生物-宿主の関係は依然として複雑であり、マウスモデルはその研究に有用であるため、微生物の転写物、代謝物、タンパク質など、マイクロバイオームの未特性の側面をプロファイルする能力を拡張することも重要である。
56
STAR★メソッド
キーリソース表
REAGENT or RESOURCEIDENTIFIER生物試料CM_mice_1コホートからのツール試料本論文NACritical commercial assaysallPrep DNA/RNA Mini KitQiagen, Hilden, GermanyCatalog No 80284Nextera XT DNA Library Preparation KitIllumina, California, USAFC-131-1096Deposited dataRaw sequencing data (CM_mice_1 cohort)This paperNCBI-SRA BioProject: PRJEB52043ソフトウェアおよびアルゴリズムMetaPhlAn (version 3.0.13)
57
https://github.com/biobakery/MetaPhlAn/MetaPhlAn (バージョン4.0.0)
30
https://github.com/biobakery/MetaPhlAn/metAML
33
https://github.com/segatalab/metamlOtherSILVA データベース
58
https://www.arb-silva.de/RefSeq データベース
40
https://www.ncbi.nlm.nih.gov/refseq/
新しいタブでテーブルを開く
リソースの確保
リードコンタクト
さらに詳しい情報、リソースや試薬のリクエストは、リードコンタクトであるNicola Segata (nicola.segata@unitn.it)までお願いします。
材料の入手方法
本研究では、新規のユニークな試薬は生成していない。
実験モデルおよび被験者の詳細
CM_mice_1実験は、IERP(INRAe, Jouy-en-Josas、協定C78-720)の特定病原体フリー動物施設において、温度管理された環境下で、12時間の明暗サイクルで厳密に行われました。動物実験は、Apafis 19750-2019041014309428の協定に基づき、地元の倫理委員会およびフランス国民教育省、高等教育省、研究省に従って行われた。Janvier(フランス)産の雌のC57BL/6J 50匹を最低7日間馴化させた(ケージ/グループあたり3~5匹のマウス)。その後、各群の腸内細菌叢を以下の課題の1つで摂動させた:バンコマイシン、コリスチン、ペニシリン、コリスチン+オブロキサシン、バンコマイシン+ペニシリン、コリスチン+メトロニダゾール、高脂肪食(Envigo TD.88137; 42%脂肪)、高乳脂肪食(Envigo TD.97222、 38%脂肪)、低トリプトファン食(Ssniff S9868-E020、17%脂肪)、高トリプトファン食(Ssniff S9868-E030、17%脂肪)、および各タイプのチャレンジのコントロール群(高脂肪食はEnvigo TD.97222, 低・高トリプトファン食はSsniff S9868-E010 )。抗生物質投与マウスには、従来のチャウ食を与えた(Envigo TD.120508)。抗生物質は、以下の用量で7日間飲水投与した:バンコマイシン、0.5g/L;メトロニダゾール、1g/L;コリスチン、1g/L;ペニシリン、1g/L;オブロキサシン、0.25g/L。食事介入は、犠牲の前に5週間維持した(表S2)。摂食介入の終わりに、マウスを安楽死させ(70日齢)、解剖し、盲腸と回腸のアリコートを取り出して-80℃で保存した。
メソッドの詳細
公共データセットの収集とキュレーション
NCBIから、実験用マウスの糞便、盲腸、回腸のメタゲノム(N = 623サンプル)から得られた合計15の公開ショットガンメタゲノムデータセットをダウンロードした。さらに、マウスの年齢、遺伝的背景、抗生物質の使用状況、サンプリング部位、脂肪摂取の割合、投与期間(日数)が、コホートの出版物の記述に直接記載されているか、対応するNCBI項目で検索できることを条件とした。ヒトまたは他の動物から糞便微生物叢移植(FMT)を受けた動物からのサンプルは事前に除外した。また、単一量の脂肪を含む食事療法を含むデータセットや、20サンプル未満のデータセットも除外した。ここで使用した高脂肪・低脂肪の表示は、Campbell et al.のデータセットを除き、すべてのケースで原著作者によって割り当てられた、
59
において、脂肪摂取率13%の餌と16.3%の餌の2群のマウスの存在により、この割り当てが著者により操作された。最終的に含まれるデータセット数は8(N = 525サンプル)であった。最終的な公開リード数は256億リード(1サンプルあたり平均48.7万リード、95%CI [9.7, 87.8])となった。本研究で使用したデータセットと相対的な食事脂肪率の概要については、表S1を参照されたい。公開されているデータセットは、高脂肪マウス243サンプルと低脂肪マウス282サンプルで構成されており、詳細な情報はTable S2に掲載されている。
複数の食餌性脂肪を摂取したマウスのマイクロバイオームの配列決定
DNAは、allPrep DNA/RNA Mini Kit (Qiagen)を用いて、製造者の指示に従って抽出した。シーケンスライブラリーは、Nextera XT DNA Library Preparation Kit(Illumina)を用いて、製造者のガイドラインに従って作成した。シーケンスは、イタリア・トレント大学のシーケンス施設にて、HiSeq2500(Illumina)を用いて実施した。リードの品質フィルタリングは、trim_galore(パラメータ:--nextera --stringency 5 --length 75 --quality 20 --max_n 2 -trim-n)を用いて行い、品質が20未満で75ヌクレオチドより短いリードをすべて取り除いた。フィルタリングされたリードは、C57BL/6J実験用マウスゲノム(GCA_000001635.8)、PhiXゲノム、SILVAデータベースに割り当てられました。
58
(ver.132)を用いて、宿主、汚染物質、16S物質を除去した。97サンプル(高脂肪食26サンプル、普通食71サンプル)が得られ、3.6 Bln.リードが得られた(サンプルあたり平均37 Mln、95% CI [2.8, 70] )。
種およびSGBレベルのメタゲノム分類学的プロファイリング
選択された622個のマウス腸内メタゲノムサンプルは、MetaPhlAn 3を用いて分類学的にプロファイリングされました。
57
(バージョン3.13; デフォルトのパラメータ)とMetaPhlAn 4
30
(バージョン4.0.0; デフォルトパラメータ)。MetaPhlAn 4は、Species-level Genome Bins (SGB) システムを組み込んだマーカーデータベースに依存し、参照ゲノムとメタゲノム集合ゲノムを既知種 (kSGB) と未知種 (uSGB) に分類しています。
31
更新されたデータベースには、1,906個のマウス腸内メタゲノムから再構成された22,718個のMAGを含む、1M以上の微生物ゲノムが組み込まれており、540個のマウス関連uSGBにまたがっている(表S3)。RefSeqベースのSGBは、RefSeqアセンブリデータベースに存在する参照ゲノムがまたがるSGBのみを使用して選択された
40
(2023年2月9日にアクセス)。
定量化・統計解析
統計・機械学習ベースのアプローチ
グループの統計的差異は、両側Wilcoxon-signed rank testおよび両側二項検定(Scipy python library, ver1.4.1使用)により評価した。β多様性解析は、Bray-Curtis一対距離に基づいて、Scipy (ver. 1.4.1) と Scikit-Bio (ver. 0.5.6) を用いてSGB相対量から算出した。対距離行列の有意性は、Python(Scikit-Bio library, ver.0.5.6)の単変量Permanova解析で評価した。変数の重要度(修正R2)は、veganパッケージのcapscale関数(ver.2.5.7.)を用いて推定した。
60
(多変量ペルマノバ)のモデルでBray-Curtis非類似度行列を作成した: 「β-多様性 ∼ study-id + マウスの年齢(日) + マウスの性別 + 採取部位 + 食餌療法". 関数 ordistep (vegan, ver. 2.5.7) を実行した。
61
は、capscaleの出力に共変量の重要度によって最適なモデルを推定するものである。シャノン多様性、ジニ-シンプソン多様性、サンプルリッチ度は、カスタムPythonスクリプトで計算された。95%信頼レベルの区間は、αおよびβ多様性の標準化平均差およびSGB数平均の推定平均値の標準誤差の1.96倍として計算された。uSGB対kSGBの平均比率とその他の相対的存在量は二項分布とみなし、信頼区間はそれに従って計算した。機械学習実験は、scikit-learnのランダムフォレスト(RF)を用いて実行した。
32
)分類器の実装(ver.0.24.2)はmetAMLソフトウェアにホストされている。
33
,
36
具体的には,推定木は1,000本,各葉の最大サンプル数は5,各木の最大深さは固定せず,各木への入力は特徴空間長の平方根,不純物基準はShannon-entropyとした.Shannon-entropyはThomas et al.に従って選択した、
36
木の本数は、Behini et al.に従って選択した、
57
とし、他のパラメータはすべてデフォルト値で設定した。少なくとも1つのコントロールと1つの高脂肪食サンプルを含むデータセットに存在し、高脂肪食を受けたと分類されるベースラインマウス(複数のタイムポイントの場合)を陽性クラスとし、高脂肪食を受けていない対応するマウスをコントロールクラスとする。SGBの相対量は、アークシン平方根で変換した後、特徴量として使用された。スコアリング指標は、受信者動作特性曲線(AUC)下の面積とした。性能評価にはいくつかの手法が用いられた。クロスバリデーション(CV)は、単一データセットに対して、10回、クラスごとにバランスよく分割し、10回のランダムな繰り返しで実施された。LODO(leave-one-dataset-out)では、各コホートをテストセットとして繰り返し使用し、他のすべてのコホートでアルゴリズムを学習させた。もう1つのタイプのテストは、1つのコホートでアルゴリズムをトレーニングし、異なるコホートでそれをテストすることからなる。各実験は10回繰り返された。最終的なAUC値は、CVでは100回のテストの平均、トランスファーとLODOでは10回のテストの平均とした。AUCは漸近的に正規とみなされ、その信頼区間は自由度n-1のt分布に基づき、nは検討したデータセット数6、実験の無作為化数20とした。RF平均特徴量ランキングは、各LODOテストのランクを平均して抽出され、オーバーフィッティングを避けるためにトレーニングセットからのみ計算された。
標準化平均差のメタ分析
各SGBについて、線形モデルを用いて、コントラスト高脂肪/低脂肪に対する単一特徴量のデータセット毎の依存性を評価した。各データセットと各SGBについて、以下のようなタイプの線形モデルを評価した: 「特徴量∼年齢(日)+サンプリングボディ-サイト+マウス背景+抗生物質使用量+食事タイプ"。サンプリングボディ・サイト、遺伝的背景、抗生物質使用量、食事タイプ(高脂肪/低脂肪)はカテゴリー変数として符号化した。これらのモデルのそれぞれから、標準化された効果量とその標準誤差が、Nakagawa and Cuthillに記載されているCohenのdに相当する尺度として計算された、
62
は、相対共変量の t 値から開始した。食事と特徴の関係の有意性は、Wald-testで評価した。線形モデル、t-検定、およびそれらの有意性は、Statsmodels python libraryの普通最小二乗法(OLS)を用いて計算された(Seabold and Perktold
63
を、ver. 0.11.1). その後、Borenstein et al.の手順を実装したpythonスクリプトを使用して、ランダム効果メタ解析で効果サイズを結合した。
64
とStatsmodels(ver.0.11.1)のPaule-Mandel異質性から算出した。メタアナリシス係数(平均標準化平均差)95%信頼区間は、Borenstein et al.の記述に従って計算した。
64
第1回および第2回のメタアナリシスにおける単一データセットのWald-PsおよびSpearmanのPs、ならびにランダム効果の生のp値は、Benjamini-Yakuteli手順(Scikit-Bio、Ver 0.5.6)により偽発見率(FDR)で補正し、有意値を0.2にした。
ログフォールドチェンジのメタアナリシス
SGB相対存在量のゼロ値(0.0001)にイプシロンを付加した。そして、それらをlog-2変換して平均し、その差を取った(log fold change)。標準化平均差の効果サイズが正であれば、対数比の最初の要素を高脂肪群として選択し、逆に正であれば低脂肪群として選択した。対数変化の分散は、すべてのケースで独立した2つの対数変化の分散の合計として計算された。この分散と効果を用いて、固定効果モデルによるメタアナリシスでログフォールド変化を要約した。このステップでは、前と同じスクリプトが使用された。ログフォールド変化のメタ分析係数の標準誤差は、平均化された効果の分散の平方根を分析したデータセット数の平方根で割ったものとし、95%信頼区間は、平均効果±この標準誤差に、データセット数マイナス1の自由度を持つt分布の97.5分位をかけたものとして計算された。そして、パーセンテージの結果は、平均値と信頼区間への2つの上昇として計算された。
相関メタアナリシス
Pingouin pythonライブラリを用いて計算されたスピアマンの偏相関係数とその有意性をメタアナリシスすることで、2つの同様の手順を実施した。
65
(ver.0.3.7)を使用しました。相関係数は、Balduzzi et al.の結果を再現するカスタムpythonスクリプトを使用して、Fisher-Z変換し、要約し、元に戻した。
66
共変量全体と脂肪率で補正した食事療法の投与期間と、共変量全体と投与期間で補正した脂肪率の両方をメタ解析した。ランダム効果係数の有意性は、Borenstein et al.の記述に従って計算された。
64
第1回および第2回のメタアナリシスにおける単一データセットのWald-PsおよびSpearmanのPs、ならびにランダム効果の生のp値は、Benjamini-Yakuteli手順(Scikit-Bio、Ver 0.5.6)によりFDR(偽検出率)を補正し、有意値を0.2にした。スピアマンの相関は、Fisher-Z変換後に平均化し、その結果得られた値に対して逆のFisher-Z変換を適用した。2つの検証用データセットにおける時間的軌跡の検証
7
,
48
は、SGB相対量について実施した。ノーベルらから抗生物質とダムを引き受けたサンプル。
7
は除外した。
データおよびコードの利用可能性
本研究で作成した97種類のマウスメタゲノムは、European Nucleotide Archiveのアクセッション番号PRJEB52043で公開されています。公開コホートのアクセッションコードは表S1に、非公開コホートおよび公開コホートの手動でキュレーションしたメタデータは表S2に掲載されています。(NCBI)のアクセッション番号GenBank:CP091140-CP091142およびCP091091で公開されています。
本論文はオリジナルコードを報告するものではありません。
本作品で報告されたデータの再分析に必要な追加情報は、要求に応じてリードコンタクトから入手できます。
謝辞
本研究は、欧州研究評議会(ERC-STGプロジェクトMetaPG-716575およびERC-CoG microTOUCH-101045015)からN.S.に、ERC-CoG(プロジェクト助成契約番号101001355)からO.Kに、欧州H2020プログラム(ONCOBIOME-825410プロジェクトおよびMASTER-818368プロジェクト)からN.Sに、国立がん研究所the National Institutes of Health (1U01CA230551) to N.S., by a N、 Premio Internazionale Lombardia e Ricerca 2019からN.S.、欧州連合のHorizon 2020研究・イノベーションプログラム(ERC-2016-StG-71577)に基づく欧州研究会議からH.S.、VolkswagenStiftungのイニシアチブ「Niedersächsisches Vorab」(助成番号76251-99)からT.S.、欧州研究会議(CoG 865466)からT.S.
著者の貢献
P.M.とN.S.は本研究を考案した。A.B.-M.、F.C.、F.B.、E.P.は、本研究で使用したMetaPhlAnのバージョン開発に貢献した。P.M.とA.B.-M.は、ほとんどの解析を行った。A.N., S.M., D.G., K.D.H., A.M.T., G.P., M.P., M.V.-C., M.Z., T.R.L., F. Asnicarは方法論の設計と検証をサポートし、解析に貢献した。H.S.、M.B.、J.P.、J.G.は、新しいマウスサンプルを採取した。F. Armaniniはメタゲノムデータを作成した。P.M., A.B.-M., F. Asnicar, T.S., O.K., H.S., and N.S. は、著者全員の貢献と編集により論文を執筆した。F.AsnicarとN.S.が監修した。著者全員が最終版の原稿を読み、承認した。
利害関係の宣言
著者は競合する利害関係はないことを宣言している。
インクルージョンと多様性
私たちは、包括的で多様な、そして公平な研究実施を支援します。
補足情報
ダウンロード.pdf (1.52 MB)
pdfファイルに関するヘルプ
資料S1:図S1~S3
ダウンロード .xlsx (.01 MB)
xlsxファイルに関するヘルプ
表S1. 本研究で検討した、STAR Methodsに関連する9つのデータセット
ダウンロード .xlsx (.06 MB)
xlsxファイルに関するヘルプ
表S2. STAR Methodsに関連する、シーケンス統計情報を持つ622サンプルのキュレーションサンプルメタデータ
ダウンロード .xlsx (1.12 MB)
xlsxファイルに関するヘルプ
表S3. カタログに含まれるマウス由来MAG、STAR Methodsに関連するもの
ダウンロード .xlsx (.01 MB)
xlsxファイルに関するヘルプ
表S4. 図1に関連するマイクロバイオーム組成に関する共変量のペルマノバ解析(MetaPhlAn 4)(Permanova analysis of covariates on microbiome composition
ダウンロード .xlsx (.21 MB)
xlsxファイルに関するヘルプ
表S5.SGB高脂肪/低脂肪メタアナリシス、図2に関連するもの
ダウンロード .xlsx (.08 MB)
xlsxファイルに関するヘルプ
表S6. SGB脂肪-%スピアマン相関メタ分析、図2に関連する。
ダウンロード .xlsx (.03 MB)
xlsxファイルに関するヘルプ
表S7. 図2に関連するランダムフォレスト特徴量ランキング
参考文献
グエン・T.L.A.
ヴィエイラ・シルヴァS.
リストンA.
レイズ J.
ヒトの腸内細菌叢研究において、マウスはどの程度参考になるか?
Dis. Model. Mech. 2015; 8: 1-16
記事で見る
Google Scholar
シャプトン T.
リャリナS.
ルオン J.
ファムJ.
ディール E.M.
アーマーC.
ゴールク C.
サンジャビ S.
ポラード K.S.
炎症性腸疾患の発症は、マウスの腸内メタゲノムの縦断的再構築と関連している。
mSystems. 2017; 2e00036-17https://doi.org/10.1128/mSystems.00036-17
記事で見る
Google Scholar
ロスハート・S.P.
ヴァサロ B.G.
アンジェレッティD.
ハッチンソンD.S.
モーガン A.P.
タケダ・ケイ
ヒックマンH.D.
マッカロクJ.A.
バジャーJ.H.
アジャミ N.J.
他
野生マウスの腸内細菌叢は宿主の体力を促進し、病気への抵抗力を向上させる。
Cell. 2017; 171: 1015-1028.e13
記事で見る
Google Scholar
ロスハート S.P.
ヘルツ J.
ヴァサロ B.G.
ハンター A.
ウォールM.K.
バジャーJ.H.
マッカロクJ.A.
アナスタサキスD.G.
サルシャドA.A.
レオナルディ I.
et al.
野生マウスから生まれた実験用マウスは自然な微生物叢を持ち、ヒトの免疫応答をモデル化する。
サイエンス(Science) 2019; 365eaaw4361
記事で見る
Google Scholar
ワーブナーM.
バーシェットY.
ヴェルブナーN.
ジグドンM.
アヴェルブッフ I.
ジブ O.
ブラント B.
エリオット E.
ゲルバーグ S.
ティテルバウムM.
ら。
社会的ストレスに応答する微生物叢は、自己反応性エフェクターTヘルパー細胞の刺激を誘導する。
mSystems. 2019; 4e00292-18https://doi.org/10.1128/mSystems.00292-18
記事で見る
Google Scholar
ウザン-ユルザリ A.
モルル M.
タレフ・ナブワニ H.
ジブO.
マジッド=ネリヤD.
アルモニR.
ミュラー E.
ライボビッチ A.
ボレンシュタイン E.
ルーゾーン Y.
et al.
乳がんおよび婦人科悪性腫瘍に対する術後補助化学療法を受けた女性における腸内細菌叢、体重、代謝の変化。
BMC Med. 2020; 18: 281
記事で見る
Google Scholar
ノーベル Y.R.
コックス L.M.
キリギン・F.F.
ボクリッチ・N.A.
ヤマニシ S.
テイトラー I.
チョン J.
ソン J.
バーバー C.M.
ゴールドファーブ D.S.
他
早期のパルス抗生物質投与による代謝およびメタゲノム上の結果。
Nat. Commun. 2015; 6: 7486
記事で見る
Google Scholar
ラマスB.
リチャードM.L.
ルドゥックV.
ファム H.-P.
ミシェル・M.-L.
ダ・コスタ G.
ブリドノー C.
ジェグー S.
Hoffmann T.W.
ナティヴィダード J.M.
他
CARD9は、トリプトファンの腸内細菌叢によるアリール炭化水素受容体リガンドへの代謝を変化させることで大腸炎に影響を与える。
Nat. Med. 2016; 22: 598-605
記事で見る
Google Scholar
タイスC.A.
イタフ・S.
ロスチャイルドD.
メイジャーM.T.
レヴィ M.
モレシ C.
ドフナロヴァーL.
ブラヴァーマン S.
ロジン S.
マリツキーS.
ら。
持続的なマイクロバイオームの変化は、ダイエット後の体重回復率を調節する。
Nature. 2016; 540: 544-551
記事で見る
Google Scholar
ファビアーノ S.
スアレス=ザモラーノN.
シュバリエC.
ラザレヴィッチ V.
キーサー S.
リゴ D.
レオ S.
ヴェイラット=デュレベックスC.
ガイヤ N.
マレスカ M.
ら。
機能的な腸内細菌叢のリモデリングは、カロリー制限による代謝の改善に寄与している。
Cell Metab. 2018; 28: 907-921.e7
記事で見る
Google Scholar
ファトフリナ A.R.
ペシュコワ I.O.
Dzutsev A.
アガエフT.
マッカロクJ.A.
トヴァライV.
バジャーJ.H.
ヴァッツR.
スンドP.
タン・H.-Y.
他
インターロイキン23-インターロイキン22軸は、腸内微生物のホメオスタシスを制御し、食事誘発性アテローム性動脈硬化症から保護する。
Immunity. 2018; 49: 943-957.e9
記事で見る
グーグルシュラー
リヴァ A.
クジーク O.
フォルスベリ E.
シウズダクG.
プファンC.
ハーボールド C.
ダイムス H.
ロイ A.
ワース B.
ベリーD.
繊維不足の食事は、ネズミの大腸マイクロバイオームの微細な空間構造を乱す。
Nat. Commun. 2019; 10: 4366
記事で見る
グーグルシュラー
モリセットA.
クロップ C.
ソンパディット J.-P.
ユングス・モレイラR.
コスタ J.
マリネ-カサドR.
ピロン G.
ヴァラン T.V.
ドゥドネ S.
ブーテクラフトL.
et al.
ブルーベリープロアントシアニジンおよびアントシアニンは、食事誘発性肥満マウスの腸内細菌叢依存性メカニズムを通じて代謝の健康を改善する。
Am. J. Physiol. Endocrinol. Metab. 2020; 318: E965-E980
記事で見る
グーグルシュラー
ビニャーミンD.
ワーブナーN.
ヌリエル・オハヨンM.
ウザンA.
モル・H.
アッバスA.
ジブ O.
テペリーノR.
ガットマンR.
コレンO.
老化したマウスのマイクロバイオームには肥満誘発の特徴がある。
Genome Med. 2020; 12: 87
記事で見る
Google Scholar
ヒルトゥネン H.
ハナニ H.
ルオト・R.
トゥルジェマンS.
ジブ O.
イソラウリ E.
サルミネン S.
コレン O.
ラウタヴァ S.
早産児の糞便微生物移植は、無菌マウスにおいて成長不全、炎症活性化、代謝障害を引き起こす。
セル・リップ・メド. 2021; 2100447
記事で見る
Google Scholar
アグランヨニ O.
メニンガー・モルデカイS.
ウザンA.
ジヴ O.
サーモン=ディヴォンM.
ロダン D.
ラズ O.
コマンI.
コレンO.
ピニャソフ A.
ナボン-ベネツィアS.
腸内細菌叢はマウスの社会的行動を決定し、その脂肪組織において代謝および炎症性の変化を誘導する。
NPJ Biofilms Microbiomes. 2021; 7: 28
記事で見る
Google Scholar
ウザン=ユルザリ A.
トゥルタ O.
ベロゴロフスキー A.
ジブO.
クンツ C.
ペルシュバッハー S.
ノイマン H.
パゾッリ E.
オズ A.
ベンアムラムH.
他
新生児期の抗生物質への曝露は、腸内微生物のコロニー形成を乱すことによって、生後6年間の子供の成長を損なう。
Nat. Commun. 2021; 12: 443
記事で見る
Google Scholar
カストレーンC.
レケッキA.
ヴァン・デア・アー A.
シモエンス P.
ヴァンデンブルックW.
マウスからヒトへの経口投与変換の前提条件となるマウス腸管の表面積評価。
Lab. Anim. 2010; 44: 176-183
記事で見る
Google Scholar
トレウティング P.M.
ディンツィスS.M.
比較解剖学と組織学: マウスとヒトのアトラス.
アカデミック出版社、2012年
記事で見る
Google Scholar
リュウ・セイ
Qin P.
Wang J.
高脂肪食はストレプトゾトシン誘発2型糖尿病マウスの腸内細菌叢を変化させる。
Microorganisms. 2019; 7176https://doi.org/10.3390/microorganisms7060176
記事で見る
Google Scholar
シン・R.P.
ハラカD.A.
ハユカ Z.
ティロシュO.
高脂肪食によるマウス微生物叢の変化とフラクトオリゴ糖のエタノール生産への利用機能性の検討
Front. Cell. Infect. Microbiol. 2020; 10: 376
記事で見る
Google Scholar
ターンボーP.J.
ベックヘド F.
フルトン・L.
ゴードン・J.I.
食事誘発性肥満は、マウス遠位腸内細菌叢の顕著かつ可逆的な変化と関連している。
Cell Host Microbe. 2008; 3: 213-223
記事で見る
Google Scholar
ダニエル・H.
ゴーラミ A.M.
ベリー・D.
デスマーシュリエ C.
ハーネ H.
ロー G.
モンドット S.
ルパージュ P.
ロートバラー M.
ウォーカーA.
他
高脂肪食はマウスの腸内細菌叢の生理を変化させる。
ISME J. 2014; 8: 295-308
記事で見る
Google Scholar
カリンC.
ウォーカーA.W.
シンプソンJ.T.
ローマンN.J.
セガタ・N.
ショットガン・メタゲノミクス、サンプリングから解析まで。
Nat. Biotechnol. 2017; 35: 833-844
記事で見る
Google Scholar
レスカー T.R.
ドゥララジ A.C.
Gálvez E.J.C.
ラグクーヴァルドスI.
ベインズ J.F.
クラーベル T.
スキュルバA.
マクハーディA.C.
ストローウィグ T.
統合メタゲノムカタログから、マウス腸内細菌叢の新たな知見が明らかになった。
セル・レップ 2020; 30: 2909-2922.e6
記事で見る
Google Scholar
シャオ・L.
Feng Q.
リャン・S.
ソンネ・S.B.
Xia Z.
Qiu X.
Li X.
Long H.
Zhang J.
Zhang D.
et al.
マウス腸内メタゲノムのカタログ。
Nat. Biotechnol. 2015; 33: 1103-1108
記事で見る
Google Scholar
Zhu J.
Ren H.
Zhong H.
Li X.
Zou Y.
ハン M.
Li M.
マドセン L.
Kristiansen K.
Xiao L.
マウス腸内メタゲノムの拡張遺伝子カタログ。
mSphere. 2021; 6: e01119-e01120https://doi.org/10.1128/mSphere.01119-20
記事で見る
Google Scholar
ベレスフォード=ジョーンズ B.S.
フォースターS.C.
スターズM.D.
ノットリーG.
ヴィシャニ E.
ブラウン H.P.
ベームラーD.J.
ソダーホルム A.T.
クマール N.
ヴェルヴィエ K.
他
マウス消化管細菌カタログは、機能マッピングによるマウスとヒトの腸内細菌叢間の翻訳を可能にする。
Cell Host Microbe. 2022; 30: 124-138.e8
記事で見る
Google Scholar
キーザー S.
ズドブノフ E.M.
トラジコフスキー M.
マウス微生物群の包括的なゲノムカタログから、ヒトとの大きな違いが明らかになった。
PLoS Comput. Biol. 2022; 18e1009947
記事で見る
Google Scholar
ブランコ=ミゲス A.
ベギーニF.
クンボF.
マカイバーL.J.
トンプソン・K.N.
ゾルフォ・M.
マンギ P.
デュボアL.
ホアン・K.D.
トーマス A.M.
et al.
MetaPhlAn 4を用いた未特性種のメタゲノム分類学的プロファイリングの拡張と改善。
Nat. Biotechnol. 2023;https://doi.org/10.1038/s41587-023-01688-w
記事で見る
Google Scholar
パゾッリ E.
アスニカーF.
マナラ S.
ゾルフォ M.
カーチャーN.
アルマニーニ F.
ベギーニF.
マンギ P.
テット A.
ゲンシP.
et al.
年齢、地理、ライフスタイルを網羅するメタゲノムから15万以上のゲノムを抽出し、未踏のヒト微生物群の多様性を明らかにした。
Cell. 2019; 176: 649-662.e20
記事で見る
Google Scholar
ブレイマンL.
ランダムフォレスト
Mach. Learn. 2001; 45: 5-32
記事で見る
Google Scholar
パゾッリ E.
チュオン D.T.
マリクF.
ウォルドロンL.
セガタ・N.
大規模メタゲノムデータセットの機械学習によるメタ分析:ツールと生物学的洞察。
PLoS Comput. Biol. 2016; 12e1004977
記事で見る
Google Scholar
ハリス Z.N.
ドゥンゲルE.
モシオールM.
Ahn T.-H.
存在量ベースの機械学習を用いた大規模なメタゲノム・データ解析。
Biol. Direct. 2019; 14: 12
記事で見る
Google Scholar
Zhou Y.-H.
ギャランスP.
マイクロバイオーム宿主形質予測のための機械学習手法のレビューとチュートリアル。
Front. Genet. 2019; 10: 579https://doi.org/10.3389/fgene.2019.00579
記事で見る
Google Scholar
トーマス A.M.
マンギP.
アスニカーF.
パゾッリ E.
アルマニーニ F.
ゾルフォ M.
ベギーニ F.
マナラ S.
カーチャー N.
ポッツィ C.
他
大腸がんデータセットのメタゲノム解析により、クロスコホート微生物診断シグネチャーとコリン分解との関連性が明らかになった。
Nat. Med. 2019; 25: 667-678
記事で見る
Google Scholar
ベルナウ C.
リースターM.
ブーレステックス A.-L.
パルミジャーニG.
ハッテンハワーC.
ウォルドロンL.
Trippa L.
予測アルゴリズムの評価のためのクロススタディバリデーション。
Bioinformatics. 2014; 30: i105-i112
記事で見る
Google Scholar
ウィルベル J.
ジーチ K.
エセックス M.
カーチャー N.
カルタルE.
サラザール G.
ボーク P.
スナガワ S.
Zeller G.
SIAMCAT機械学習ツールボックスで実現したマイクロバイオームメタ解析と疾患横断比較。
ゲノム・バイオロジー 2021; 22: 93
記事で見る
Google Scholar
ウィルベル J.
ピル・P.T.
カルタル E.
ジーチ K.
カシャニA.
ミラネーゼA.
フレック J.S.
ヴォイグト A.Y.
パレジャA.
ポンヌドゥライR.
他
糞便メタゲノム解析により、大腸がんに特異的なグローバルな微生物シグネチャーが明らかになった。
Nat. Med. 2019; 25: 679-689
記事で見る
Google Scholar
オリアリーN.A.
ライトM.W.
ブリスター・J.R.
シウフォ・S.
ハダッドD.
マクビーR.
ラジプト B.
ロバートス B.
スミス-ホワイト B.
アコ-アドジェイD.
et al.
NCBIの参照配列(RefSeq)データベース:現状、分類学的拡大、機能アノテーション。
ヌクレイック・アシッズ・レズ 2016; 44: D733-D745
記事で見る
Google Scholar
デュヴァレC.
ギボンズ S.M.
ガリーT.
イリザリーR.A.
アルム E.J.
腸内細菌研究のメタアナリシスにより、疾患特異的な反応と共有される反応が明らかになった。
Nat. Commun. 2017; 8: 1784
記事で見る
Google Scholar
サナ S.
ファン・ズィーダム・N.R.
マハジャン A.
クリルシコフ A.
ヴィッチ・ヴィラ A.
ヴォンサU.
ムジャギッチ Z.
マスクリー A.A.M.
ヨンカースD.M.A.E.
オースティング M.
他
腸内細菌叢、短鎖脂肪酸、代謝性疾患との因果関係。
Nat. Genet. 2019; 51: 600-605
記事で見る
Google Scholar
柿山吾郎
パンダック・W.M.
ジルベットP.M.
ハイレモンP.B.
ヒューマンD.M.
ダイタ・K.
タケイ H.
ムトー A.
ニトノ H.
リドロンJ.M.
他
肝硬変における腸内細菌叢による糞便中胆汁酸プロファイルの変調。
J. Hepatol. 2013; 58: 949-955
記事で見る
Google Scholar
ビドル A.
スチュワートL.
ブランシャール J.
Leschine S.
多様な腸内環境におけるLachnospiraceaeとRuminococcaceaeによる繊維分解特殊化の遺伝的基盤を解明する。
Diversity. 2013; 5: 627-640https://doi.org/10.3390/d5030627
記事で見る
Google Scholar
ラフムーニ K.
モーガンD.A.
モーガンG.M.
マークA.L.
ヘインズ W.G.
食事誘発性肥満高血圧症における選択的レプチン抵抗性の役割.
Diabetes. 2005; 54: 2012-2018
記事で見る
Google Scholar
パン W.W.
マイヤーズ・ジュニア、M.G..
レプチンと体重増加の維持。
Nat. Rev. Neurosci. 2018; 19: 95-105
記事で見る
Google Scholar
デッカー・ニタート・M.
ムーサA.
バレットH.L.
ナデルプールN.
ド・クールテンB.
腸内細菌叢の組成の変化は、過体重および肥満の人の腰痛と関連している。
Front. Endocrinol. 2020; 11: 605
記事で見る
Google Scholar
Kim M.-S.
Bae J.-W.
ライソジニーはネズミの腸内細菌叢に蔓延し、広く分布している。
ISME J. 2018; 12: 1127-1141
記事で見る
Google Scholar
ジャオ・N.
ベイカー S.S.
ニュージェントC.A.
ツォンパナM.
Cai L.
Wang Y.
バック・M.J.
ジェンコ・R.J.
ベイカー・R.D.
朱 R.
チュー・エル.
腸内細菌は肥満齧歯類のインスリン抵抗性と全身性炎症に寄与する可能性がある:メタアナリシス.
Physiol. Genomics. 2018; 50: 244-254
記事で見る
Google Scholar
リュー・ウー
クロット J.W.
Lyu L.
ファルツァーA.C.(Pfalzer A.C.
Li J.
Choi S.-W.
Yang Y.
Mason J.B.
Liu Z.
食事および遺伝的に誘導された肥満は、apcマウスの腸内でマイクロバイオーム、炎症および経路に変化をもたらす:比較と対照。
J. Cancer. 2016; 7: 1780-1790
記事で見る
Google Scholar
ジョーンズ・ホール Y.L.
コージック A.
Nakatsu C.
炎症性腸疾患モデルマウスにおいて、腫瘍壊死因子の切除は炎症の減少および微生物叢の変化と関連している。
PLoS One. 2015; 10e0119441https://doi.org/10.1371/journal.pone.0119441
記事で見る
Google Scholar
チャダイドK.S.
カーモディ R.N.
異なる食餌性脂肪の代謝における宿主-微生物間の相互作用。
Cell Metab. 2021; 33: 857-872https://doi.org/10.1016/j.cmet.2021.04.011
記事で見る
Google Scholar
シャオ・L.
ソンネ・S.B.
フェン Q.
Chen N.
Xia Z.
Li X.
Fang Z.
Zhang D.
Fjære E.
Midtbø L.K.
et al.
肥満よりも高脂肪食がマウスの腸内細菌叢の分類学的・機能的変化を促進する。
Microbiome. 2017; 5: 43
記事で見る
Google Scholar
ラボット S.
メンブレスM.
ブランチャーF.
ベルジェB.
モワンD.
クラウスL.
ビビローニR.
ブリュノー A.
ジェラールP.
シッダールJ.
他
高脂肪食はマウスの腸内細菌叢の構成に関係なく肥満を促進する。
Sci. Rep. 2016; 632484
記事で見る
Google Scholar
スリアーノF.
ヴィエイラ=シルヴァ S.
ファロニーG.
デ・ウータース・ドプリンターA.
パオーン P.
デルゼンヌ N.M.
エベラール A.
レーズ J.
ヴァンヒュルM.
カニ・P.D.
マウスにおける腸内細菌叢の変化、肥満および関連する代謝異常の決定要因として、糖分ではなく脂肪が挙げられる。
Am. J. Physiol.Endocrinol. Metab. 2023; 324: E85-E96https://doi.org/10.1152/ajpendo.00141.2022
記事で見る
Google Scholar
バジェス・コロマーM.
メンニ C.
ベリー・S.E.
バルデスA.M.
スペクターT.D.
Segata N.
心代謝系の健康、食事、腸内細菌叢:メタオミクスの観点から。
Nat. Med. 2023; 29: 551-561
記事で見る
Google Scholar
ベギーニ F.
マカイバーL.J.
ブランコ=ミゲス A.
デュボアL.
アスニカーF.
マハルジャンS.
メイリャンA.
マンギP.
ショルツM.
トーマスA.M.
他
bioBakery 3による多様な微生物群集の分類学的、機能的、および菌株レベルのプロファイリングの統合.
Elife. 2021; 10e65088https://doi.org/10.7554/eLife.65088
記事で見る
グーグルシュラー
クアスト C.
プルエッセE.
ユルマズP.
ゲルケン J.
シュヴェール T.
ヤルザ P.
ペプリーズ J.
Glöckner F.O.
SILVAリボソームRNA遺伝子データベースプロジェクト:データ処理とウェブベースツールの改善。
ヌクレイック・アシッズ・レズ 2013; 41: D590-D596
記事で見る
グーグルシュラー
キャンベル S.C.
ウィスニエフスキーP.J.
野地真理
マクギネス・L.R.
ヘッグブロムM.M.
ライトフット S.A.
ジョセフ・L.B.
ケルクホフL.J.
食事と運動がマウスの腸内環境と微生物多様性に及ぼす影響。
PLoS One. 2016; 11e0150502
記事で見る
グーグルシュラー
ブランシェ F.G.
レジャンドルP.
ボルカードD.
説明変数の前方選択
Ecology. 2008; 89: 2623-2632
記事で見る
グーグルシュラー
ホフマンG.E.
Schadt E.E.
variancePartition:複雑な遺伝子発現研究における変動のドライバーを解釈する。
BMC Bioinf. 2016; 17: 483
記事で見る
グーグルシュラー
中川 聡
カティル I.C.
効果量、信頼区間、統計的有意性:生物学者のための実践ガイド。
Biol. Rev. Camb. Philos. Soc. 2007; 82: 591-605
記事で見る
グーグルシュラー
シーボルド S.
パークトールド J.
Statsmodels: Pythonによるエコノメトリックおよび統計モデリング。
2010https://doi.org/10.25080/majora-92bf1922-011(Proceedings of the 9th Python in Science Conference)
記事で見る
グーグルシュラー
ボーレンシュタイン M.
ヘッジス L.V.
ヒギンズ J.P.T.
ロススタイン H.R.
メタアナリシス入門.
ジョン・ワイリー&サンズ社、2021年
記事で見る
Google Scholar
Vallat R.
Pingouin:Pythonで統計学。
J. Open Source Softw. 2018; 3: 1026
記事で見る
Google Scholar
バルデュッツィ S.
リュッカーG.
シュヴァルツァーG.
Rでメタアナリシスを行う方法:実践的なチュートリアル。
Evid. Based. Ment. Health. 2019; 22: 153-160https://doi.org/10.1136/ebmental-2019-300117
記事で見る
Google Scholar
記事情報
出版履歴
オンラインで公開されました: 2023年5月3日
受理されました: 2023年4月17日
改訂版として受理された: 2023年3月10日
受領しました: 2022年12月28日
アイデンティフィケーション
DOI: https://doi.org/10.1016/j.celrep.2023.112464
著作権について
© 2023 The Authors.
ユーザーライセンス
クリエイティブ・コモンズ 表示(CC BY 4.0)|日本経済新聞社
再利用の方法
サイエンスダイレクト
ScienceDirectでこの記事にアクセスする
数値
グラフィカルアブストラクト
図1マウスの腸内細菌叢は、微生物叢と宿主の食餌療法との関連を改善するために非常に関連性の高い未特定の微生物種によって支配されている
図2高・低脂肪食関連SGBの標準化平均値差のメタアナリシス
関連記事
本サイトのコンテンツは、あらゆる分野の医療従事者や研究者を対象としています。
研究雑誌
セル
がん細胞
セルケミカルバイオロジー
セルゲノミクス
細胞宿主と微生物
細胞メタボリズム
セルレポート
セルレポート医学
Cell Reports Methods
セルレポート 物理科学
セルがサステナビリティを報告
細胞幹細胞
セルシステム
ケム
ケム・キャタリシス
カレントバイオロジー
発生細胞
デバイス
ヘリヨン
イミュニティ
アイサイエンス
ジュール
マター
メド
分子細胞
ニューロン
ワンアース
パターン
STARプロトコル
構造
トレンドレビュー ジャーナル
生物化学系
バイオテクノロジー
がん(Cancer
細胞生物学
ケミストリー
コグニティブサイエンス
エコロジー&エボリューション
内分泌・代謝内科
遺伝学
免疫学
微生物学
分子医学
ニューロサイエンス
寄生虫学
薬理科学
植物科学
パートナージャーナル
AJHG
バイオフィジカルジャーナル
バイオフィジカルレポート
HGGアドバンス
モレキュラープラント
分子療法ファミリー
プラントコミュニケーション
ステムセルレポート
ザ・イノベーション
コレクションズ
ベスト・オブ・セルプレス
セルプレスのレビュー
セルプレスセレクション
コンソーシアムハブ
Nucleus Collections
SnapShotアーカイブ
誌面を越えて
セルキャリアネットワーク
セルメンター
細胞シンポジューム
ラボリンクス
ウェビナーズ
奉加帳
コミュニティレビュー
Figure360
スニークピーク
STARメソッド
社会科学
セル画展
セルプレスポッドキャスト
セルプレスの動画
カラーリングとコミック
リサーチアーク
コネクト
セルプレスについて
採用情報
連絡先
ヘルプ&サポート
ニュースルーム
パブリケーションアラート
ACCESS
サブスクライブ
リード・イット・ナウ
ライブラリアンへの推薦
INFORMATION
広告主様向け
採用担当者様へ
ライブラリアン向け
ご利用条件
プライバシーポリシー
アクセシビリティ
当社は、サービスの提供や強化、コンテンツのカスタマイズのためにクッキーを使用しています。クッキーの設定を変更するには、本サイトのクッキー設定にアクセスしてください。
Copyright © 2023 Elsevier Inc. 第三者から提供された一部のコンテンツを除く。
この記事が気に入ったらサポートをしてみませんか?
