
コンタミDNAのPCR定量は過剰評価?
前回、定量PCRはDNaseでランダムに分断したDNAを定量する場合にはあてにならないと書きましたが$${^{1)}}$$、実はぶちぶちに切れた標的部分のDNAを自己修復して存外正確な値を示す可能性があることを東京大学の新田剛氏から指摘されました。
McKernan氏が検出したファイザーワクチンのコンタミDNAは7,810塩基対(bp)の発現プラスミドですが、これをDNaseで平均200bpの断片に分解するとスパイク蛋白をコードする遺伝子上にある定量化PCRのための標的遺伝子もバラバラに壊されます。
バラバラに壊れるといっても、ワクチンの中にRNAの1/10、すなわち3μg/dose入っていれば分子数は4.67x10$${^{11}}$$になりそのうちのいくつかは一つも切れ目が入っていないものがでてきます。これがPCRで増やす時の鋳型になります。その確率はポアソン分布$${^{2)}}$$に従い、
Exp(-114/200)=0.5684 (Exp(a)=e$${^a}$$, e: ネイピア数)
プラスミドに存在する鋳型DNAの56.9%がちゃんと鋳型として機能し、残りは壊れて鋳型として機能しなくなるのです。ですから、これを定量PCRで定量しても3μgの56.8%の1.7μgしか検出されないはずです。逆にいえば、実測値を存在確率で割ると真の値が出るはずです。
しかし、必ずしもこうはならないというのが新田氏の指摘でした。PCRの反応過程で分断化したDNAの断片がお互いにプライマーと鋳型になって機能を失ったDNA断片を修復して再び鋳型として機能できるようにするからです。
PCRというのはDNAの特定の区間をプラス鎖の一端と同じ配列をもつプライマー(赤矢印:センスプライマー)とマイナス鎖のもう一端と同じ配列をもつプライマー(黄矢印:アンチセンスプライマー)で増幅する手法です。

まず、DNAを95℃に熱し、二本の鎖をはがします。次に、温度を下げてこれにプライマーをくっつけ(アニーリング)、DNAポリメラーゼによりそれぞれのプライマーの3’末端に鋳型DNAを元に新しいDNAを合成します(Ct=1: 緑点線矢印)。この時点では標的とする長さのDNAはできません(図1A)。
次に再び、95℃に熱して二本の鎖をはがしてから温度を下げてそれぞれのDNAにプライマーをくっつけてDNAを合成させます (Ct=2)。この時点で標的とする長さの1本鎖のDNAが2つできあがります。
さらに同じ操作をもう一度繰り返すと標的とする長さの1本鎖のDNAが6つできあがり(Ct=3)、これをn回繰り返すと(Ct=n)標的DNAは2$${^{n-1}}$$+2個に増幅します。10回くり返すと514本、20回繰り返す52万本になり電気泳動しても微量で見えなかったDNAが見えてくるという寸法です。
定量PCRは蛍光強度をリアルタイムで測定することにより増幅度を追跡する手法で、一定の蛍光強度に達したCt値からもとのDNAの量を推定できます。サンプルAが20回目で、サンプルBが10回目で同じ蛍光強度に達したならサンプルBのDNAはサンプルAよりも520,000/514=1000倍のDNAが存在していたことがわかります。
ところがもともとのDNAがランダムにちぎれているとDNAの標的となる部分が分断され鋳型としての機能を失います。McKernan氏は標的DNAとしてスパイク遺伝子内の114bpの配列を選んでいます。DNaseで平均200bpに分断すると114bpのDNAには平均114/200=0.57の切れ目が生じることになります。
DNAに平均zの切れ目が生じたときにk個の切れ目が生じる確率pはポアソン分布に従います。
p=e$${^{-z}}$$z$${^k}$$/k!
k!は階乗を表す記号で1x2x3x4x・・・xkです。eは自然対数の底でネイピア数(約2.72)です。この数字がでてくる時点でこの式を導きだすのは義務教育の範囲を超えているのでここでは割愛しますが、エクセルを使えばパソコンが計算してくれます。
p=poisson(k,z,false)
これを利用するとDNaseで分解した時にできる断片の数を推定できます。例えば5つの切れ目が生じれば環状DNAは5つのDNAに分解されますし、10の切れ目が生じれば10断片が生じます。k個切れ目が生じるとk個の断片が生じます。平均200bpで切った場合には切れ目の数の平均は39ですから、この確率は、
p$${_5}$$=poisson(5,39,false)=8.31x10$${^{-12}}$$
p$${_{10}}$$= poisson(10,39,false)=2.50x10$${^{-8}}$$
p$${_n}$$=poisson(n,39,false)
コンタミプラスミドの数は4.65x10$${^{11}}$$/doseですから、断片数Nはこの確率に断片の数をかけて、
N$${_5}$$=4.67x10$${^{11}}$$x5x poisson(5,39,false)= 3.87
N$${_{10}}$$=4.67x10$${^{11}}$$x10xpoisson(10,39,false)= 2.32x10$${^5}$$
N$${_k}$$=4.67x10$${^{11}}$$xkxpoisson(k,39,false)
図2の左はこのポアソン確率と断片数の分布を示したものです。横軸は切れ目の数つまり断片の数です。

これを全てたせばDNase処理によって生じる断片の数が計算できます。DNAは7,810bpですからk=0からk=7,810までを計算してたせばいいのですが、実際には平均塩基数200、50、10で切ったときの存在確率は、95個、259個、982個で0になりますからエクセルの計算もそれほど手間ではありません。
この操作で生じる断片数は1doseあたり、3.64x10$${^{13}}$$、1.45x10$${^{14}}$$、7.27x10$${^{14}}$$となります。
k=0の時、すなわちnbpのDNA断片に一つも切れ目が生じない確率は断片の平均塩基数をmとすると
poisson(0, n/m, false)=Exp(-n/m)
平均塩基数200bpで114bpの標的DNAを切ったときに切れ目がはいらず鋳型として機能する確率は
Exp(-114/200)=0.5655
ワクチンに3μg/doseのDNAが入っているなら、そのうち鋳型として機能するのは56.5%で、これをPCRで定量化した場合には補正値として0.565で割れば真の値がでてくるというのが前回示した推定値です$${^{1)}}$$。
nを0から7,810まで変化させるとそれぞれの塩基数をもつ断片の存在確率の分布となりますが、これは確率の比率の分布となるので合計は1とはならず、m=10、50、200の時には10、50、200となります。ですから確率分布はExp(-n/m)/mとなり、これと断片数との積が塩基数nの分布になります (図3下)。

新田氏の指摘はDNaseでプラスミドを切ったとき生じたDNA断片同士が互いにプライマーと鋳型になり自己修復する可能性です。標的遺伝子の領域内を全くカバーしていないプラス鎖の断片(黒矢印: 図1B1)はマイナス差の断片とくっつくのですがその断片には反応液のセンスプライマーにもつきます。
センスプライマーやアンチセンスプライマーは鋳型DNAに比べて大過剰に存在しますから、マイナス鎖を鋳型としたDNAの伸長反応はほとんど全てがセンスプライマーから伸長し、黒矢印のDNA断片から伸長することはありません。
ですからこの場合に断片が修復されて標的DNAの新たな鋳型となることはないと考えられます。自己修復するのは標的領域内に末端をもつDNA断片ということになります。
このような断片には3種類があります。標的領域内に3’末端をもつ断片、標的領域内に5’末端をもつ断片、標的領域内に3’末端と5’末端の両方をもつ断片で、これをそれぞれタイプ1,2,3の断片と呼ぶことにします。
プラス鎖のタイプ1の断片がマイナス鎖のタイプ1とくっついた場合、この2本の断片には外から入れたプライマーはつきませんから、それぞれの末端をプライマーとしてDNAが伸長し、PCRの標的DNAの鋳型に変換されます(図1C1)。PCR反応により壊れた鋳型が修復されるわけです。
プラス鎖のタイプ1の断片がマイナス鎖のタイプ2とくっついた場合(図1C2)にはタイプ2にはセンスプライマーがつきますからタイプ1からの伸長反応はおきません。しかし、センスプライマーからのタイプ2を鋳型とした伸長反応は新たなタイプ1断片を作り出します(緑点線矢印)。
タイプ1がタイプ3とくっついた場合(図1C3)にもプラス鎖にもマイナス鎖にもプライマーはつかないので、お互いをプライマーとした伸長反応がおきますがタイプ1から生じた伸長反応は長さが少し長くなるだけで新たな鋳型として修復されることはありません。これに対しタイプ3から生じたDNAは新たなタイプ2断片を生じます(緑点線矢印)。
タイプ2とタイプ2がくっついた場合にはそれぞれプライマーがつきますから断片からの伸長反応は起きず、それぞれのプライマーから生じた新たなタイプ1断片を生じます(図1D3, E2)。タイプ2とタイプ3がついた場合にもタイプ2から新たなタイプ1断片が生じ、プライマーのつかないタイプ3はそのまま残ります。
タイプ3同士がくっついた場合にはお互いをプライマーとした伸長反応はおきますが結果的にはタイプ3の断片が残ることになります(図1DE)。
要するに、鋳型が修復されるのはタイプ1同士がくっついたときだけなのですが、タイプ2は常に新たなタイプ1を生じ、タイプ3はタイプ1とくっついたときに新たなタイプ2を生じることになります。
もともとワクチンに存在するプラスミドは4.65x10$${^{11}}$$です。平均塩基対数200bpで切ったときに鋳型として機能するDNA断片の存在確率はExp(-114/200)ですから残りに機能しない断片が含まれることになります。タイプ1およびタイプ2はこの領域に切れ目を生じた断片です。
しかし、領域内のぎりぎりのところに末端をもったのでは相補鎖の断片と相互作用できません。プライマーの長さの半分の9塩基(塩基: b)あれば相互作用できると仮定するとこのような断片の数は
4.67x10$${^{11}}$$x(1- Exp(-96/200))x2=3.55x10$${^{11}}$$
最後に2倍してあるのはそれぞれの断片はプラス、マイナス両方のDNA鎖から生じるからです。この中でタイプ1およびタイプ2となる断片はのりしろ部分の9bを持つものです。その存在確率はExp(-9/200)となります。
以上を想定して平均塩基対数200bpで切った時のタイプ1,2の分子数を計算すると
4.67x10$${^{11}}$$x(1- Exp(-96/200))x2xExp(-9/200)=3.39x10$${^{11}}$$
50bp、10bpで切ったときには6.64x10$${^{11}}$$、3.78x10$${^{11}}$$となります (表1)。
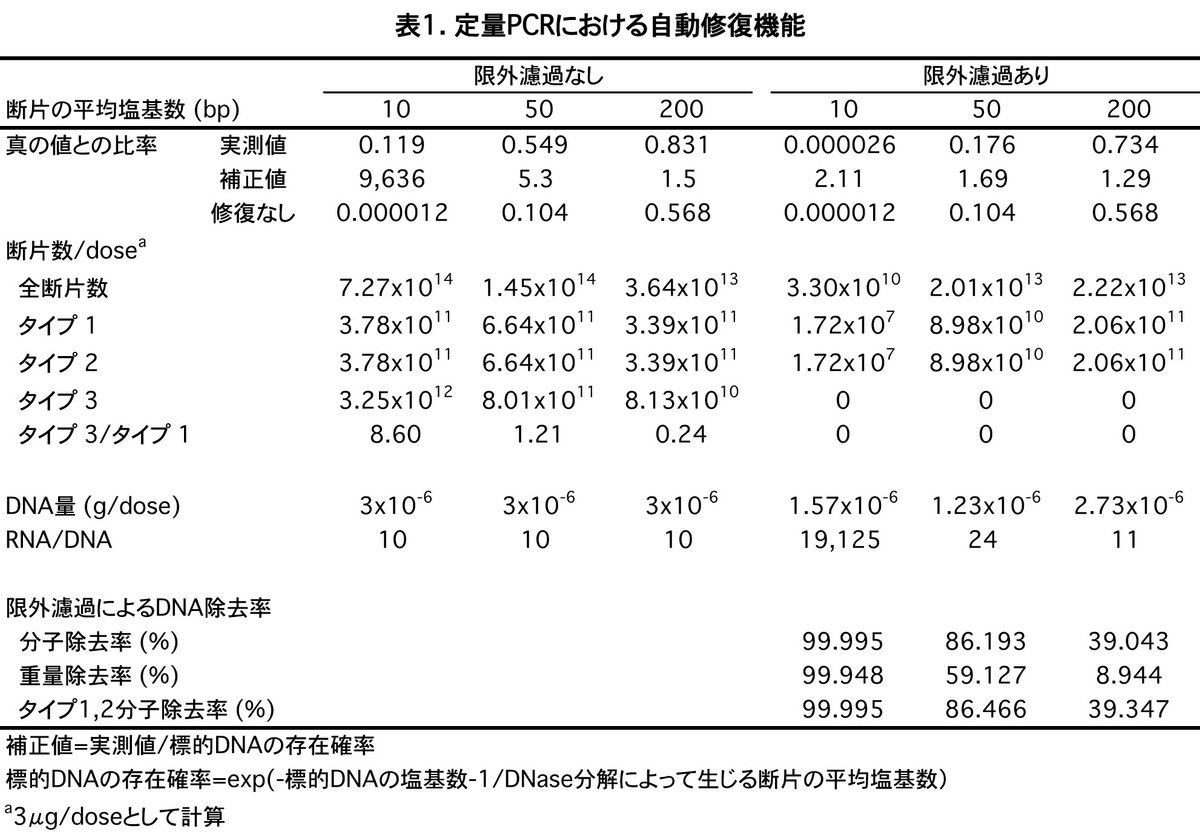
タイプ3の場合にはのりしろ部分を除いた標的領域は96bですから、これをランダムに切った時の断片数がタイプ3の全断片数ということになります。平均200bpで切った場合平均断片数は96/200=0.49となりますからn個の断片が生じる確率はpoisson(n,0.49, false)となり(図2右)、これをn=0から96までたせば総分子数が計算できます。
この場合にも9b以下の短い断片は相互作用できずにタイプ3として機能できないと想定しますからこの部分を差し引き続き、n=10から96までの合計となり、8.13x10$${^{10}}$$、同様に50bpおよび10bpで切った場合には、8.01x10$${^{11}}$$、3.25x10$${^{12}}$$となります。
プライマーがついていないタイプ1とタイプ3の結合は確率的に起きると考えられます。プラス鎖とマイナス鎖にそれぞれタイプ1がa、タイプ3がbあるなら、プラス鎖のタイプ1あるいはタイプ3にマイナス鎖のタイプ1がつく確率はa/(a+b)、タイプ3がつく確率はb/(a+b)になると考えられます。
このように考えると、Ct=nのときのタイプ1、タイプ2、タイプ3、修復された新規鋳型の数を、N1$${_n}$$、N2$${_n}$$、N3$${_n}$$、Nt$${_n}$$とすると次の連立漸化式が成立することになります。
1. N1$${_n}$$=N3$${_{n-1}}$$/(N1$${_{n-1}}$$+N3$${_{n-1}}$$)+N2$${_{n-1}}$$
2. N2$${_n}$$=N3$${_{n-1}}$$1xN1$${_{n-1}}$$/(N1$${_{n-1}}$$+N3$${_{n-1}}$$)
3. N3$${_n}$$=N3$${_{n-1}}$$xN3$${_{n-1}}$$/(N1$${_{n-1}}$$+N3$${_{n-1}}$$)
4. Nt$${_n}$$=N1$${_{n-1}}$$xN1$${_{n-1}}$$/(N1$${_{n-1}}$$+N3$${_{n-1}}$$)
これを平均断片塩基数m=10、50、100bpについて計算したものが表2と図4左になります。

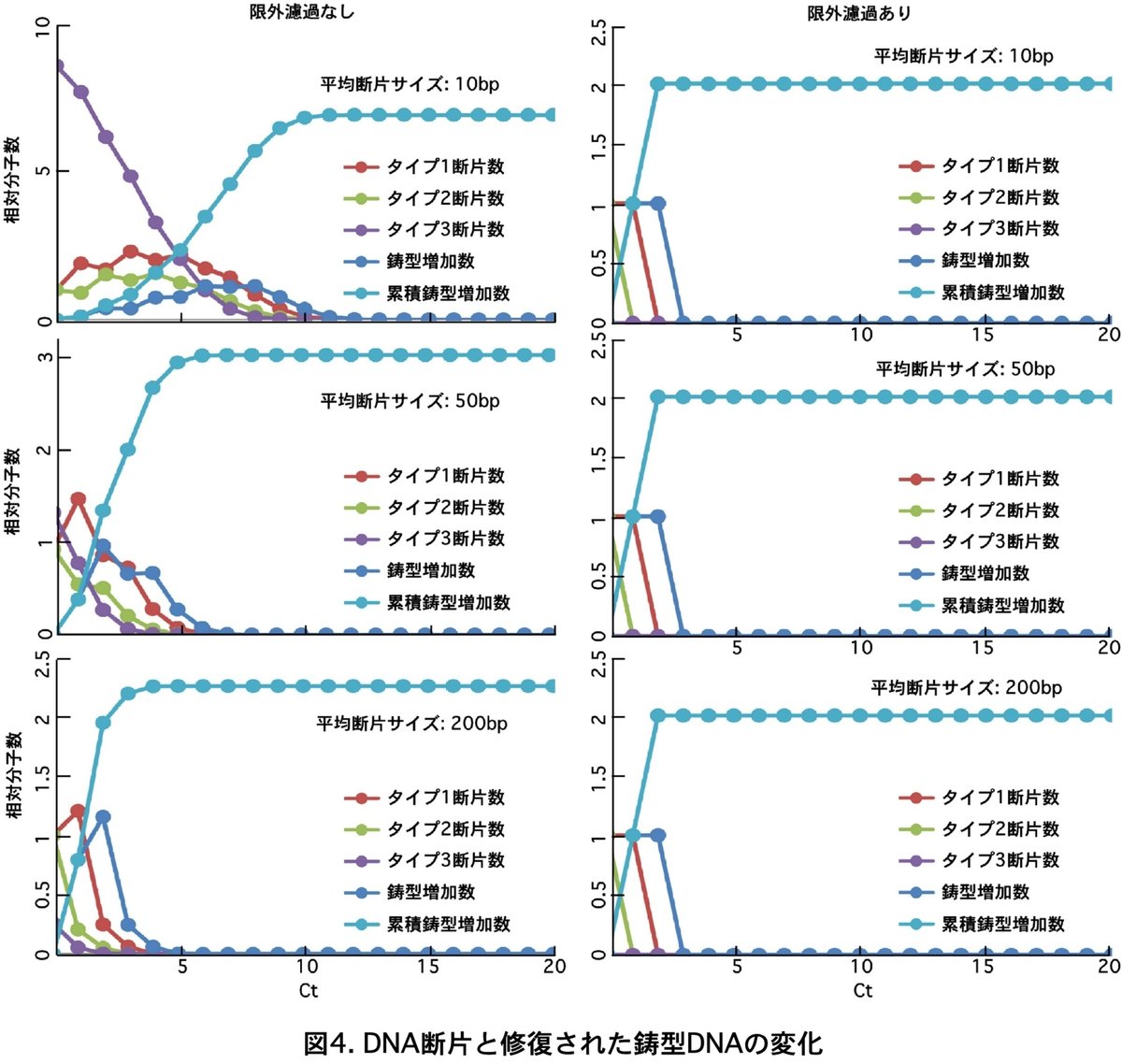
タイプ3/タイプ1の比はm=10、50、100のとき8.60、1.21、0.24になりますが、タイプ1はCt=12、7、5でゼロになりこれ以上機能のある鋳型は修復できなくなります。修復された鋳型の累積個数はDNase処理する前の鋳型の数の6.9、3.1、2.2倍になり、元の数より増えることになります。
これらの修復された鋳型は順次PCRで増幅されることになりますからCt=15ではNt$${_n}$$x2$${^{(14-n)}}$$となります。定量PCRを行ったときのCt=15の実測値は、断片化の後残った傷のない鋳型とn=1から15までの新たにできた鋳型からの増幅値の合計になります。このPCR生成物の増殖曲線を示したものが図5左のグラフになります。

青い点は断片化の後残った傷のない鋳型からの増殖曲線で、緑の点は断片化しない場合の鋳型からの増殖曲線です。これらはm=200ではほとんど重なりあっていますが、mが小さくなるにつれ、つまり細かく断片化すると大きく乖離するようになります。
これに対して実測値はPCRによる断片同士の自動修復効果により青の値から緑の値に近づいて行くことがわかります。m=10、50、200のときのCt=15の時の実測値は真の値の12%、55%、83%となり、ほぼ真の値を反映していることになります(表1)。
この修復効果を無視して実測値を鋳型DNAの存在確率で割ることで測定値を補正すると、その値は実測値の9,636、5.3、1.5倍となり、細かく断片化した場合には真の値から大きく乖離した過剰評価をすることになります (表1)。
実際のワクチン精製過程では限外濾過により細かい断片を取り除きます$${^{1)}}$$。プラスミドの分子量は3,867kDa、塩基数は7,810bpですから、限外ろ過で50kDa以下の分子を除くと
50,000/(3,866,512/7,810)=100.6
およそ100bp以下の断片が取り除かれることになります。図3の断片の確率分布から除去率が計算でき、m=10、50、200では分子数で99.995、86.2、39.0%、塩基数すなわち重量では99.95、59.1、8.94%となります (表1)。
同様にタイプ1,2の断片も100以下の断片が除かれ、除去率はm=10、50、200の分子数除去率は99.995、86.5、39.3%、となります。タイプ3の断片は96bp以下ですから全て除かれます。一方、断片化後に存在した傷のない鋳型断片は114bp以上ありますから除去されることはありません (表1)。
この条件でPCR生成物の増殖曲線 (図5右)とCt=15のときの実測値を測定し、実際の存在量との比率を計算すると、m=10、50、200では0.000026、0.176、0.734と過小評価になります。存在確率で補正した場合には、2.11、1.69、1.29倍の過剰評価になります (表1)。
限外濾過で精製した場合にはタイプ3の断片が除かれるので新田氏の指摘した修復効果はほとんど現れないのです。この場合には、小さく断片化した場合にも存在確率で補正したほうが実際の値に近い値を示します。
McKernan氏の報告したファイザーの2価ワクチンのDNA量を彼の示した検量線を使ってQPCRで定量化すると9.6ng/doseになります$${^{1,3)}}$$。
m=50bpで断片化していたなら、実測値は真の値の17.6%ですからコンタミDNA量は9.6ng/0.176=54.6ng/dose、RNA/DNA=550
で基準値の10ng/dose$${^{4)}}$$は満たしていません。しかし、限外濾過の除去率は59%ですから鋳型DNAは133ng/dose使ったことになり生成物の0.445%と整合性のある数値となります。
しかし、もしm=10なら実測値は真の値の0.0026%でコンタミDNA量は9.6ng/0.000026=0.37mg/doseとmRNAの12倍も存在することになり非現実的な数値となります。このことから平均50bp程度のDNase処理が施されていたことが予想されます。
ここで予測した修復効果のシミュレーションではいくつか単純化するための仮定を設けています。プライマーとのアニーリングの閾値が9塩基であること、断片化したタイプ1、2、3の断片が効率よく全てくっつきあうことなどです。
さらに全ての配列との結合が同じ親和性をもつことも実際にはあり得ないことでしょう。Mckernan氏が指摘するようにワクチンに存在するmRNAと鋳型DNAの相互作用も考慮する必要があるかもしれません$${^{5)}}$$。
これらのことを考えると理論のみで真の値を予測するのは不可能で、検証実験を行ってシミュレーションがどこまで正しいのか確かめる必要があります。自然科学では実証実験がなく理論的シミュレーションだけで世の中に受け入れられることは、たとえ物理であってもあり得ません。
常に未知の可能性が残されていて完全な証明等できないからです。前提がまちがっていれば理論的考察は砂上の楼閣となりもろくも崩れ去ることは常に念頭においておく必要があります。
1) mbi, note (2023/5/16)
2) ポアソン分布、Wikipedia
3) Anandamide, Substack (2023/3/30)
4) Yang H, PDA J Pharm Sci Tech, 67:155 (2013)
5) Anandamide, Substack (2023/4/15)
