BA.2.86・JN.1・EG.5・EG.5.1・HV.1のスパイク構造、受容体結合、免疫回避(2024年6月)
Spike structures, receptor binding, and immune escape of recently circulating SARS-CoV-2 Omicron BA.2.86, JN.1, EG.5, EG.5.1, and HV.1 sub-variants
元→https://linkinghub.elsevier.com/retrieve/pii/S0969212624002302
SUMMARY
最近出現した BA.2.86、JN.1、EG.5、EG.5.1、および HV.1株は、成長に有利です。この研究では、BA.2.86、JN.1、EG.5、EG.5.1、および HV.1株の受容体結合と免疫回避の構造的基礎を調査します。
私たちの調査結果から、BA.2.86 は強力な受容体結合を示すのに対し、その JN.1サブ系統はヒト ACE2 (hACE2) に対する結合親和性が低下していることが明らかになりました。
複雑な構造解析により、BA.2.86 受容体結合ドメイン (RBD) の R493Q の復帰が受容体結合を促進する役割を果たし、JN.1 RBD の L455S 置換が最適な親和性を回復することが分かりました。
さらに、BA.2.86 RBDと複合したモノクローナル抗体(mAb)S309の構造は、K356T変異の重要性を強調しています。K356T変異は新しいNグリコシル化モチーフをもたらし、S309に代表されるRBD-5に属するmAbの結合パターンを変えます。
これらの発見は、SARS-CoV-2感染の新たな波を防ぐために、BA.2.86とそのサブ系統を注意深く監視することの重要性を強調しています。
INTRODUCTION
SARS-CoV-2は集団免疫の背景に適応し続け、多数の亜種に進化しています。XBB.1.9.2に由来するオミクロンEG.5は、2023年2月17日に最初に特定され、急速に世界中に広がりました。2023年8月9日までに、EG.5とその亜系統は、世界保健機関(WHO)によって関心対象の変異体(VOI)に分類されました。2023年第44週時点で、EG.5とその亜系統 HV.1・HK.3・EG.5.1.1・JG.3は、世界のSARS-CoV-2配列の33.2%を占めていました。
XBB.1.5と比較すると、EG.5スパイク(S)タンパク質には追加のF456L置換があり、EG.5.1には追加のQ52H変異があります。米国では、2023年11月12日から25日までの間に、EG.5とHV.1がそれぞれ新規症例の31.7%と13.1%を占めました。
2023年8月13日、別の亜変異体であるBA.2.86が、GISAIDに報告されました。BA.2.86 の S タンパク質には、その祖先である BA.2 と比較して、最大 29 個の置換、4 個の AA 挿入、4 個の非連続欠失 (2 個の AA 欠失が 1 箇所) が含まれています。
このような多数の変異により、潜在的な免疫回避と受容体結合に関する懸念が直ちに高まり、WHO はわずか 4 日後に BA.2.86 を監視対象変異 (VUM) に指定しました。2023 年 11 月 21 日、BA.2.86 とそのサブ系統 (BA.2.86.1 および JN.1 を含む) は VOI として再分類されました。
2024年1月26日現在、15,971のBA.2.86とその亜系統の配列がGISAIDに提出されており、2024年1月8日から15日までに世界中で入手可能なほぼすべての配列が含まれています。
BA.2、XBB、EG.5/EG5.1 の S タンパク質のヒト ACE2 (hACE2) に対する結合親和性は類似していますが、BA.2.86 S は hACE2 に対して 2 倍高い結合親和性を示しました。それでも、擬似ウイルス侵入アッセイでは、BA.2.86はXBB.1.5やEG.5よりも感染性が低いことが示されました。一方、他の2つの研究では、TMPRSS2依存的にBA.2.86の肺細胞侵入効率が高いことが報告されています。いくつかの研究により、BA.2.86 は、現在優勢な XBB.1.5 や EG.5.1 よりもヒト抗血清に対する耐性が高くないことが示されています。
一方、BA.2.86 は、B.1、BQ.1.1、BA.5、および XBB S mRNA ワクチンによって誘導される抗体を著しく回避しており、BA.2.86 はこれらのサブバリアントとは抗原的に異なることが示唆されています。当然のことながら、BA.2.86 は、ベブテロビマブ、ソトロビマブ、シルガビマブ、チクサゲビマブなどの多くの治療用モノクローナル抗体 (mAbs) も回避します。ただし、受容体結合ドメイン (RBD) の内面を標的とする抗体の中和活性は有効のままでした。BA.2.86、JN.1、EG.5、EG.5.1、および HV.1 サブバリアントの受容体結合と免疫回避の構造的基礎はまだ解明されていません。
本研究では、BA.2.86、JN.1、EG.5、EG.5.1、および HV.1 の S タンパク質 (アポおよび hACE2 と複合体を形成したもの) を特徴付け、これらのサブバリアントの体系的な比較分析を実施しました。BA.2.86 S タンパク質の変異の程度が高いにもかかわらず、受容体結合を促進する R493Q の復帰を除いて、結合インターフェースにはほとんど変化が見られませんでした。
しかし、JN.1 では、L455S 置換により結合親和性が最適な 2 桁 nM 範囲に回復し、免疫回避がさらに強化されました。これは、以前の提案と一致しています。
8 つの抗体エプトープのうち 6 つに変異残基が見られ、BA.2.86 RBD ではその祖先である BA.2 と比較して大規模な静電的変化が観察されました。特に、K356T 変異は新しい N グリコシル化モチーフをもたらし、RBD-5 mAb S309 の結合パターンを変えます。これらの発見は、BA.2.86、JN.1、EG.5、EG.5.1、および HV.1 の受容体結合および免疫回避メカニズムに関する貴重な分子的洞察を提供します。
RESULTS
BA.2.86・JN.1・EG.5・EG.5.1・HV.1 の受容体結合及び擬似ウイルス感染性
祖先の BA.2 と比較すると、BA.2.86 の S タンパク質には、4-AA の挿入と 4 つの非連続欠失 (2-AA 欠失が 1 箇所) に加えて、29 もの置換が含まれています (図 S1A)。

現在流通している Omicron サブバリアントからの BA.2.86 の変異パターンは大きく異なっており、BA.2.86 が独自の経路に沿って進化していることを示しています。BA.2.86 と比較すると、JN.1 は RBD に L455S の変異がさらに 1 つあります (図 S1C)。

Omicron EG.5 とそのサブ系統である EG.5.1、EG.5.1.1、および HV.1 は急速に世界中に広がりました (図 1A)。最近では、BA.2.86とその亜系統JN.1派生株が多くの地域で増加しています(図1A)。

BA.2.86、JN.1、EG.5、EG.5.1、および HV.1 の感染性を評価するために、これらのサブバリアントの受容体結合能力と擬似ウイルス感染性を、代表的なバリアントおよびサブバリアント (BA.1、BA.2、BA.2.75、BA.4/5、および XBB.1.5) を比較対象としてテストしました (図 1B および 1C)。
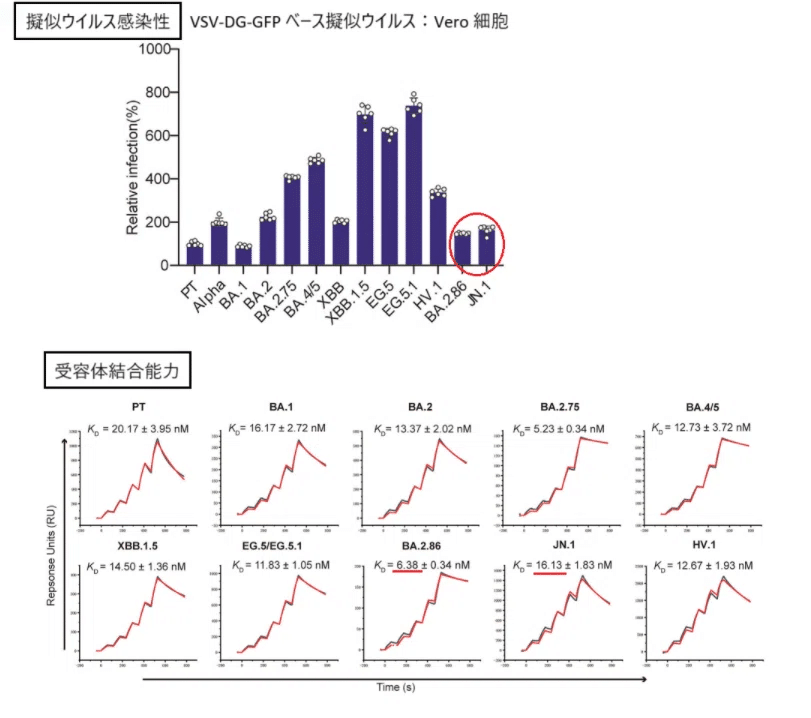
BA.2.86 は BA.2.75 と同等の結合親和性を示し、XBB.1.5 より 2.3 倍高かったのに対し、JN.1 は 2 つの数字の平均親和性に戻りました (図 1C)。一方、EG.5 (EG.5.1 は EG.5 と同一の RBD 配列を共有) は、BA.2.75 を除く他のサブバリアントと有意な差を示しませんでした (図 1C)。
擬似ウイルスアッセイでは、BA.2.86 は Vero 細胞侵入に ACE2 を利用するのに最も効果のないサブバリアントの 1 つであることが示されました。対照的に、EG.5 と EG.5.1 は、プロトタイプ (PT) と比較して約 2 倍と 4 倍の感染性を示し、EG.5.1 は最も効果的なサブ変異体です (図 1B)。
BA.2.86、JN.1、EG.5、EG.5.1、および HV.1 S タンパク質の構造解析と hACE2 との複合体構造
構造を得るために、BA.2.86・JN.1・EG.5・EG.5.1・HV.1 S タンパク質の融合前安定化三量体エクトドメインを前回のように発現および精製しました。BA.2.86、JN.1、EG.5、EG.5.1、および HV.1 アポ S タンパク質のクライオ電子顕微鏡構造は、それぞれ 3.07 A˚、2.94 A˚、2.61 A˚、3.31 A˚、および 2.90 A˚ の解像度で決定されました (図 S2 ~ S6、データ S1)。

以前の研究では、BA.1 のアポ S タンパク質は 1 つの RBD が上向きの構造を優先的に採用するのに対し、BA.2 のアポ S は 3 つの RBD がすべて下向き、または 1 つの RBD が上向きのオープン構造の 2 つの構造状態を示すことが報告されています (図 S7)。この研究では、BA.2.86 の apo S が 1 つの RBD が上向きの構造を示すことがわかりました (図 2A)。ただし、BA.2.86 S の開口度は、BA.2 S (PDB コード: 7XIW、8D56) と比較して低くなっています (図 2B)。


祖先ウイルス XBB.1 (図 S7) と同様に、EG.5、EG.5.1、および HV.1 の apo S タンパク質は、3 つの RBD がすべて下向きの構造 (閉じた RBD) を示します (図 2A)。公開されている S アポ構造を分析すると、BA.2 とその直接の誘導体 (BA.2.12.1、BA.2.13、BA.2.75、BA.2.86 など) はよりオープンな構造を採用しており、 アポ構造は主にクローズド構造または 1-RBD アップ構造を示しています。一方、XBB 誘導体 (XBB.1、XBB.1.5、EG.5、EG.5.1、および HV.1) はより密にパッケージ化されており、 アポ構造は主にクローズド構造を採用しています。
変異を S タンパク質の構造にマッピングしたところ (図 S1)、BA.2.86 の変異は主に N 末端ドメイン (NTD) (13) と S プロトマーの RBD (14) に位置していることが示されました。特に、N481K と V483 の欠失 (Del483) は、優勢なサブバリアントで初めて観察されました。その他のユニークな変異の中で、Ins 16 (MPLF) はシグナルペプチド S タンパク質に隣接して位置していますが、S939F と P1143L は S2 ドメインにあります。NTD では、サイト 21 (R21T)、50 (S50L)、158 (R158G)、216 (L216F)、および 264 (A264D) は、他の代表的なオミクロン株では変異が観察されていません (図 S1A)。

EG.5 S と EG.5.1 S の唯一の違いは、1 つのアミノ酸置換 Q52H です (図 S1B)。全体的に、EG.5 S の立体構造は EG.5 S および HV.1 S の立体構造に非常に似ており、すべての Ca 原子の二乗平均平方根偏差 (RMSD) はそれぞれ 0.762 A˚ および 0.648 A˚ です (図 2C)。
詳細な分析により、Q52もH52の側鎖も他の残基と接触していないことが明らかになりました (図 2C)。HV.1 S にはさらに 2 つの置換 (F157L および L452R) がありますが、これらも S タンパク質の三量体構造に影響を与えません (図 2C)。

以前に報告したように、hACE2 の結合により RBD は上向きコンフォメーションを採用します。
hACE2 とのインキュベーション後、それぞれ 3.26 A˚、2.98 A˚、2.87 A˚、2.78 A˚ の解像度で hACE2 と複合した BA.2.86、JN.1、EG.5.1、HV.1 S タンパク質のクライオ電子顕微鏡構造が得られました (図 2D~2G および S8~S11、データ S1)。

これらの構造を分析すると、BA.2.86 と JN.1 は 3 つの RBD が上向きの構造を示すことが明らかになりました (図 2D と 2E)。 一方、EG.5.1 と HV.1 S タンパク質は 2 つの RBD が上向きの構造を示し、hACE2 に結合した RBD は 1 つだけであることが明確に観察されました (図 2F と 2G)。
BA.2.86、JN.1、EG.5、EG.5.1、HV.1 の RBD と複合体を形成した hACE2 の構造
BA.2.86、JN.1、EG.5、EG.5.1、およびHV.1の受容体認識の構造的基礎を解明するために、BA.2.86 RBD/hACE2のクライオ電子顕微鏡構造が2.89Åの解像度で決定されました。 一方、hACE2に結合したJN.1、EG.5/EG.5.1、およびHV.1 RBDのローカルマップは、それぞれ3.04Å、2.79Å、および2.64Åの解像度で解像されました(図2D~2G、データS1)。

特に、以前の SARS-CoV-2 変異体では、N331 と N343 に 2 つの N グリコシル化モチーフがあり、以前の構造ではグリコシル化されていることが観察されました (図 3A)。BA.2.86 と JN.1 RBD の K356T 変異により、N354 (NRT) に新しい N グリコシル化モチーフがもたらされます (図 3A)。BA.2.86 および JN.1 RBD の構造を調べたところ、新しいモチーフがグリコシル化できることが分かりました (図 3B)。

全体的に、複合体の構造は他の系統のものと似ています (図 3C~3F)。
EG.5/EG.5.1 RBD と HV.1 の残基の唯一の明確な違いは、結合界面に位置していない L452R 残基です。したがって、EG.5/EG.5.1 RBD と hACE2 の相互作用は、HV.1 RBD と hACE2 の相互作用に匹敵します。
BA.2.86 と JN.1 RBD の重大な変異にもかかわらず、相互作用ネットワークは他の オミクロン系統と同様です。以前に報告されたように、相互作用する RBD 残基は 2 つのパッチに集まりました。
パッチ 1 では、RBD の N487 は、これら複合体すべてで hACE2 の Y83 と水素結合 (H 結合) を形成し、 4 つの複合体のうちJN.1 を除く 3 つでは hACE2 の Q24 と H 結合を形成します (図 3C ~ 3F、表 S1)。さらに、BA.2.86 および JN.1 RBD の Q493 は、hACE2 E35 と H 結合を形成します (図 3C および 3D、表 S1)。EG.5/EG.5.1 RBD および HV.1 RBD の両方の N477 は、hACE2 の S19 と H 結合を形成します (図 3E および 3F、表 S1)。さらに、hACE2 の S19 も EG.5/EG.5.1 RBD の N477 と H 結合を形成します (図 3E、表 S1)。
hACE2 の K31 は、EG.5/EG.5.1 RBD と HV.1 RBD の両方の Q493 と水素結合を形成し (図 3E および 3F、表 S1)、HV.1 RBD の S490 と追加の水素結合を形成します (図 3F、表 S1)。hACE2 の N90 の N グリカンは、BA.2.86 RBD/hACE2、JN.1 RBD/hACE2、および EG.5/EG.5.1 RBD/hACE2 複合体において、RBD の T415 と水素結合を形成します (図 3C~3E、表 S1)。この相互作用は、BA.2 RBD/hACE2 複合体でも観察されました。


パッチ 2 では、BA.2.86、EG.5/EG.5.1、および HV.1 RBD の Y449 が水素結合を介して D38 および Q42 に結合し、RBD の G502 は 4 つの複合体すべてにおいて hACE2 の K353 と水素結合を形成します。
BA.2.86 RBD/hACE2 および JN.1 RBD/hACE2 複合体では、T500 は hACE2 の R357 に結合しますが、他の 2 つの複合体では、T500 は水素結合を介して Y41 と相互作用します (図 3C~3F、表 S1)。
さらに、JN.1 および EG.5/EG.5.1 RBD の R498 は hACE2 の D38 と塩橋を形成し、HV.1 RBD の R498 は hACE2 Q42 と水素結合を形成します (図 3D~3F、表 S1)。

hACE2に結合するBA.2.86およびJN.1 RBDの重要な残基
BA.2.86 RBD では、R493Q 逆変異と F486P が ACE2 との結合界面にあります。受容体結合への影響を評価するために、逆置換である Q493R と P486F を個別に導入した 2 つの BA.2.86 RBD 変異体を作成し、hACE2 との親和性を測定しました。
結果から、Q493R は ACE2 結合親和性を 10.7 倍低下させることが示され、BA.2.86 RBD の R493Q 逆変異により、Q493R 変異によって失われた hACE2 に対する親和性が回復することが示されました (図 4A)。

さらに、2 つの置換の構造の詳細を比較しました (図 4B ~ 4D)。BA.2 RBD の R493 は hACE2 の E35 と 1 つの水素結合のみを形成しますが、BA.2.86 RBD の Q493 はそれぞれ H34 および E35 と 2 つの水素結合を形成します (図 4C)。一方、F486P 置換により、F28、L79、M82、および Y83 で構成される疎水性パッチが弱まりました (図 4D)。

F486P と同じループに 3 つの変異 (N481K、Del483、および A484K) がありますが、ACE2 相互作用残基の構成に大きな変化は見られませんでしたが、Del483 によりループがわずかに短くなりました (図 4B)。
JN.1 RBDのL455S置換はS進化における最適な受容体結合の維持を強調
受容体認識と免疫回避の絡み合った効果により、S タンパク質の進化が促進されます。以前の研究では、最適な ACE2 結合親和性範囲 5~40 nM と、「2 段階前進、1 段階後退」モデルを提案し、最適な受容体結合を維持しながら免疫回避を強化することを説明しました。具体的にいうと、SARS-CoV-2 の継続的な進化において、RBD にいくつかの変異が発生し、免疫回避が強化されましたが、受容体結合親和性は低下しました。 一方、受容体結合を維持する復帰変異も発生しましたが、抗体との相性は低下しました。
全体として、以前の変異体に関連する抗体と血清はますます多くが新たに出現した変異体によって回避され、受容体結合親和性は最適な範囲にとどまりました。
競合アッセイに基づいて、RBD を標的とする抗体は 8 つのカテゴリに分類されました。JN.1 RBD で観察された L455S 置換は、RBD-1 エピトープにあります (図 5D)。

我々の提案を検証するために、BA.2.86 と JN.1 の免疫回避をさらに評価しました。代表的な RBD-1 に属する 6 つの mAbs (BD-604、BD-629、P2C-1F11、S2K-146、CB6、CT-P59) を選択し、BA.2、BA.2.86、JN.1 RBD との結合親和性をテストしました。
結果は、CB6 と CT-P59 が 3 つの RBD のいずれにも結合しなかったことを示しました。BA.2.86 RBD は BD-604 および P2C-1F11 との結合が減少しましたが、JN.1 RBD は S2K146 のみとの結合を示しました (図 1D)。一方、JN.1 RBD は BA.2.86 と比較して hACE2 への結合親和性が低く、最適な親和性範囲に戻ることがわかりました (図 1C)。したがって、L455S 置換は「2 段階前進、1 段階後退」モデルにも当てはまります。

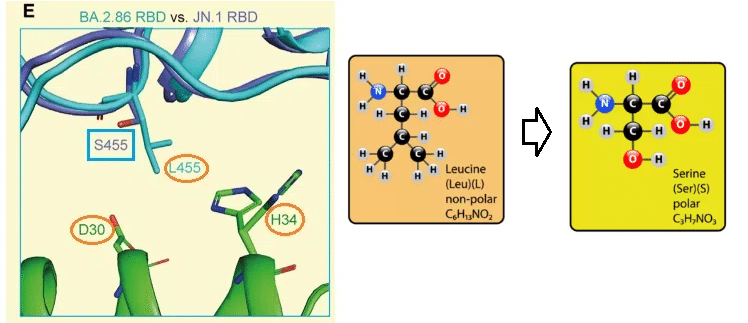
さらに、hACE2 と相互作用する L455S 置換の構造詳細を分析しました。
BA.2.86 RBD では、L455 は hACE2 の D30 および H34 とファンデルワールス力を形成し、受容体の認識に寄与しています。ただし、L455 が S455 に置換されると、側鎖が短くなり、hACE2 の D30 および H34 との接触が失われ、hACE2 の相互作用が損なわれます (図 4E)。
BA.2.86 RBD の免疫逃避の構造的基礎
RBD 表面には BA.2 から BA.2.86 までの 15 箇所の変異部位があります。免疫逃避の分子メカニズムを解明するために、これらの変異部位を RBD 表面にマッピングしました (密度不足のため I332V 変異は除く) (図 5A~5C)。これらの変異のほとんどは RBD の外側と上部に位置していることがわかりました (図 5A および 5B)。一方、RBD が上向きの位置にある場合にのみ露出する隠れたエピトープを表す内側には、変異残基がわずかしか見つかりませんでした (図 5C)。

各抗原部位の変異残基をまとめました。
RBDの内側にあるRBD-6とRBD-7を除き、8つのエピトープのうち6つに変異残基があることがわかりました。
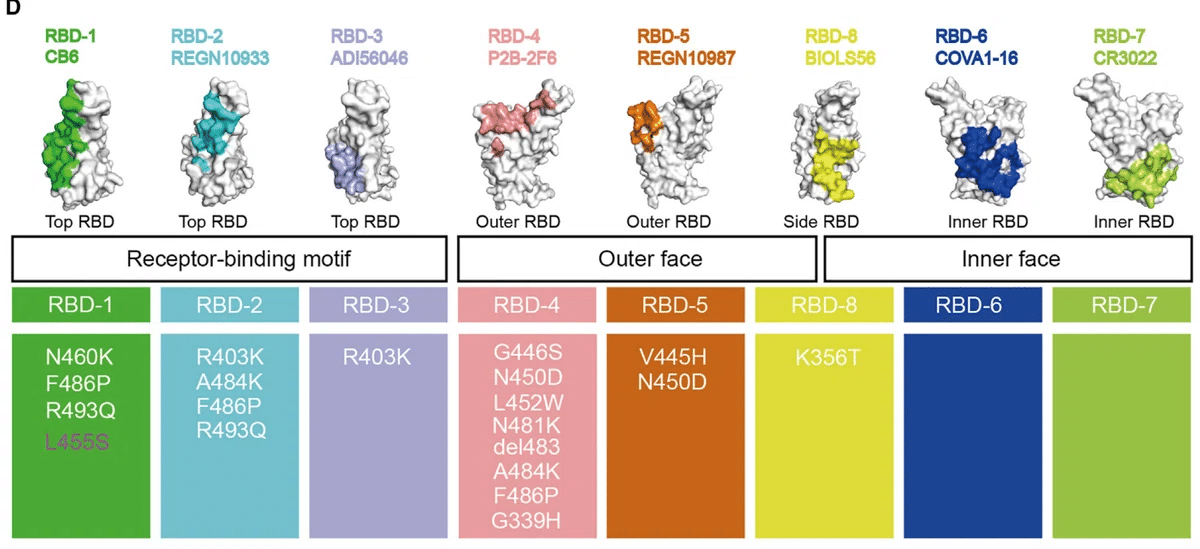
RBD-1、RBD-2、RBD-4には3つ以上の変異部位があります(図5D)。
RBD-3、RBD-5、RBD-8には1つまたは2つの変異残基があります(図5D)。
その中で、RBD-4には最も多くの変異残基があります(図5D)。
さらに、変異した15残基のうち9残基が静電的に変化していることが分かりました。

外側では、非荷電アミノ酸V445、N450、N481、A484がそれぞれH455、D450、K481、K484に変異しました。塩基性アミノ酸K356が中性アミノ酸T356に変異し、酸性アミノ酸D339が塩基性アミノ酸H339に変異しました(図5E)。
上側では、塩基性アミノ酸R493が非荷電アミノ酸Q493に変異し、塩基性アミノ酸R403がK403に変異しました(図5F)。
内側では、非荷電アミノ酸 N460 が塩基性アミノ酸 K460 に変異しました (図 5G)。
静電の変化は、BA.2.86 における免疫回避の主な要因であることが判明しました。
K356Tによってもたらされる新しいNグリコシル化はS309の結合パターンを変化
前述のように、BA.2.86 および JN.1 の K356T は、RBD-5 および RBD-8 に属する mAb のエピトープ付近に位置する新しい N グリコシル化をもたらしました (図 5D)。一貫して、Qu らは、RBD-5 mAb S309 は BA.2.86 を中和できないと報告しており、これは D339H および K356T 置換によるものだとしています。このような推測を検証するために、単一サイクルと複数サイクルの両方の SPR アッセイを使用して、S309 と BA.2、BA.2.86、および JN.1 の RBD 間の結合親和性テストを実施しました。
単一サイクルと複数サイクルの両方のアッセイで測定された S309 の BA.2.86 および JN.1 RBD への結合親和性は、正確な測定値を示しませんでした (図 6A および S14)。

S309 の結合増加の構造的根拠を探るため、S309 Fab および hACE2 と複合体を形成した BA.2.86 RBD の構造をさらに決定しました。以前に公開された構造と一致して、S309 Fab は RBD の外面に結合し、hACE2 の結合と競合しません (図 6B~6F)。

N343 に結合したグリカンは、S309 の重鎖 (H) と軽鎖 (L) の両方と相互作用し、mAb の標的化において重要な役割を果たします (図 6C および 6D)。
注目すべきは、立体障害を引き起こす代わりに、BA.2.86 RBD の H339 は S309 重鎖の S31 および Y100 と 2 つの水素結合を形成することです (図 6C)。一方、K356T の効果に関する分析では、K356 と E108 の間の塩橋は妨害されるものの、S309-H の F106 と N354 の新しい N グリコシル化との相互作用によって補償されることが実証されました (図 6G、6H、および S15)。

N グリコシル化が S309 の結合に与える影響をさらに調べるために、BA.2、BA.2.86、および JN.1 バリアントからの PNGase F 処理済み RBD への S309 の結合を調べ、未処理のタンパク質をコントロールとして使用しました。PNGase F 処理済み BA.2.86 および JN.1 RBD への S309 の結合親和性は十分にテストされています (図 1A)。
N354 のグリカンは不均一であり、この部位のグリカンの一部は S309 の結合を促進し、他のグリカンは結合を妨げる可能性があることを示唆しています。このグリコシル化の不均一性により、SPR で測定される結合にばらつきが生じ、結果の精度に影響する可能性があります。
これらの結果は、BA.2.86 および JN.1 RBD の N グリコシル化が S309 の結合に重要な役割を果たしており、D339H 変異は S309 の結合に悪影響を与えないことを示唆しています。
DISCUSSION
EG.5.1 S には EG.5 S と比較して Q52H 変異が含まれており、その後の HV.1 S には F157S と L452R という追加の変異があることは注目に値します。
以前の研究では、S タンパク質の D614G 変異によりプロトマー間の接触が妨げられ、ACE2 結合能の高い状態への構造シフトが起こり、ウイルスの感染力が強化されることが示されています。
EG.5.1 と HV.1 の感染力は EG.5 と比較して増加しましたが、それらの S タンパク質の構造は非常に類似しており、変異した残基 Q52H は他の残基と直接接触しません。したがって、これらのウイルスの感染性の向上に寄与する他の要因があるに違いありません。
たとえば、真正ウイルスまたは擬似ウイルスの表面にある S タンパク質コピーの数の変動は、ウイルス感染の親和性に影響を与える可能性があります。
現在優勢な XBB サブバリアントとは異なる系統である BA.2.86 の出現は、SARS-CoV-2 S 進化の根本原理を調査する貴重な機会を提供します。
主流の系統とは大きく異なるにもかかわらず、RBD のほとんどの変異部位はさまざまなオミクロン株で以前に観察されています(N481K と Del483 を除く)。さらに、BA.2.86 には、hACE2 結合インターフェースでは R493Q および F486P 変異のみが含まれていますが、これらは XBB株に以前から存在しています。
これらの発見は、RBD 変異とそれに関連する免疫回避が受容体結合の制約下で一定レベルの安定性に達し、選択圧が他の場所で作用している可能性があることを示唆しています。
抗原ドリフトは、S タンパク質の変異が新しいバリアントの出現につながるときに発生します。以前の株に基づいて開発されたワクチンと治療用抗体は、これらの新しいバリアントによって回避される可能性があります。
抗原関係に基づき、SARS-CoV、SARS-CoV-2 PT、およびSARS-CoV-2 オミクロン株は、3つの異なる血清型に分類されます。さらに、当研究グループは、RBD抗原性の体系的な評価を通じて、SARS-CoV-2とその亜種を5つの血清型に分類しました。BA.2.86 RBDの場合、8つの抗体エピトープのうち6つが変異し、RBDの大部分が静電的変化を経験しており、BA.2.86の抗原性が大幅に変化していることを示しています。我々は最近、RBD 抗原性に基づいて BA.2.86 を新しい血清型として提案しました。
我々は以前、XBB 誘導体に基づく最適な受容体結合親和性範囲を提案しましたが、RBD の進化は免疫回避を強化するため、その範囲に制限されます。さらに、受容体結合には生物学的に関連する上限があり、それを超えて親和性が高くなるとウイルスの伝染に悪影響を与える可能性があります。BA.2.86 の hACE2 結合が上限に近づき、その子孫である JN.1 にすぐに置き換えられることを考慮すると、これらの変異体は XBB 誘導体とは異なる系統であるため、このような仮説にも当てはまります。ただし、hACE2 結合を高める代わりに免疫回避が損なわれる以前の「2 歩前進、1 歩後退」モデルとは異なり、L455S は免疫回避と引き換えに hACE2 結合が減少する「1 歩後退、2 歩前進」に近い。
このような観察は私たちの以前の仮説をさらに洗練させますが、なぜ受容体結合にそのような上限が存在するのかはまだ答えが出ておらず、より重要視されるべきです。
我々のグループと他のグループによるこれまでの研究では、S309 は BA.2.86 と JN.1 の中和能力を低下させることが示されています。構造解析の結果、S309-H の F106 が N354 の N グリコシル化部位と直接相互作用することがわかりました。考えられる説明の 1 つは、N354 のグリカンが不均一性を示し、N 糖鎖が非常に柔軟であるということです。一部のグリカンは S309 の中和効果を高める可能性がある一方、他のグリカンはそれを妨げる可能性があります。
要約すると、最近出現した主要なサブバリアント BA.2.86、EG.5、EG.5.1、JN.1、および HV.1 の受容体結合と免疫回避の構造的基礎について包括的な評価を実施しました。私たちの研究結果は、他の SARS-CoV-2 サブバリアントと比較して BA.2.86 RBD の抗原性に大きな違いがあることを明らかにし、K356T 置換による新しい N グリコシル化の役割を強調しています。これらの結果は、現在流行しているサブバリアントのタイムリーなリスク評価を提供し、BA.2.86 で観察される免疫回避と受容体結合のメカニズムのより深い理解に貢献します。
以下省略。
この記事が気に入ったらサポートをしてみませんか?
