SARS-CoV-2 ウイルスの分子構造(2020年10月)
Molecular Architecture of the SARS-CoV-2 Virus
SARS-CoV-2 ウイルスの分子構造
元→https://www.cell.com/action/showPdf?pii=S0092-8674%2820%2931159-4

SUMMARY
SARS-CoV-2は、COVID-19パンデミックの原因となるエンベロープウイルスです。
SARS-CoV-2タンパク質の構造解明における最近の進歩にもかかわらず、無傷のウイルスの詳細なアーキテクチャはまだ明らかにされていません。
ここでは、クライオ電子線トモグラフィー(cryo-ET)とサブトモグラム平均化(STA)を使用して、本物のSARS-CoV-2ウイルスの分子集合を報告します。
融合前および融合後のコンフォメーションにおけるSタンパク質のネイティブ構造は、8.7〜11A°の平均解像度で決定されました。
ネイティブスパイクからのN結合型グリカンの組成を質量分析によって分析したところ、組換え糖タンパク質グリカンと非常に類似したネイティブグリカンの全体的な処理状態が明らかになりました。
リボ核タンパク質(RNP)のネイティブコンフォメーションとそれらの高次アセンブリが明らかになりました。
全体として、これらの特性は、SARS-CoV-2ウイルスのアーキテクチャを非常に詳細に明らかにし、ウイルスが直径約80nmの内腔に長さ約30kbの単一セグメントRNAをどのようにパックするかを明らかにしました。
INTRODUCTION
2020年9月21日の時点で、3,100万件を超えるCOVID-19の症例が報告されており、世界中で960,000人以上の命が奪われています。
原因となる病原体である重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)は、新しいβ-コロナウイルスです。
SARS-CoV-2は、(+)RNAゲノムに少なくとも29のタンパク質をコードしており、そのうち4つは構造タンパク質です。
その4つとは、スパイク(S)・膜(M)・エンベロープ(E)・およびヌクレオカプシド(N)タンパク質です。
既知の最大のクラスI融合タンパク質の1つである約600kDaの三量体Sタンパク質は、66個のN結合型グリカンで高度にグリコシル化されています。
※グリコシル化=タンパク質もしくは脂質へ糖類が付加する反応。実験では、グリコシル化は適切な折りたたみの厳格な必要条件ではないが、グリコシル化しないタンパク質は敏速に退化することが証明されている。グリコシル化は細胞間の接着のような役割も持つ。(wikiより)
各Sプロトマーは、S1サブユニットとS2サブユニット、および単一の膜貫通(TM;transmembrane)アンカーで構成されます。
Sタンパク質は、膜融合に不可欠なステップである受容体結合ドメイン(RBD)を介して、細胞表面受容体アンジオテンシン変換酵素2(ACE2)に結合します。
Sの活性化には、フューリン様プロテアーゼによるS1 / S2の切断が必要であり、融合前から融合後へのコンフォメーション変化が起こります。
3つのRBDが上または下の異なる方向を示すSタンパク質について、いくつかの融合前コンフォメーションが解決されました。
受容体結合部位は、RBDがアップコンフォメーションを採用した場合にのみ露出します。
「RBDダウン」、「1 RBDアップ」、および「2 RBDアップ」のコンフォメーションは、SARSCoV-2の組換え発現Sタンパク質で観察されています。
活性化されると、SはクラスI融合タンパク質間の古典的な経路をたどります:S1サブユニットを脱落させ、融合ペプチド(FP:fusion peptide)を標的細胞膜に挿入するなど、劇的な構造の再配列が行われます。
膜融合後、Sは針状の融合後の形態に変化し、3つのらせんが同軸に絡み合っています。
組換えタンパク質を使用して、SARS-CoV-2ウイルスの宿主認識と侵入メカニズムを原子に近い解像度で解明する努力にもかかわらず、本物のウイルスのin situ構造と景観に関する高解像度の情報が求められています。
コロナウイルスは、すべてのRNAウイルスの中で最大のゲノムを持っています。Nタンパク質がどのようにオリゴマー化、組織化、および長さ約30kbの一本鎖RNAをウイルス内腔に詰め込むかは謎めいたものです。コロナウイルスの初期のネガティブ染色電子顕微鏡検査では、直径が約15nmの一本鎖らせん状RNPが示されました。SARS-CoVのクライオ電子線トモグラフィー(cryo-ET)により、RNPがエンベロープの下で約4〜5nmの解像度で格子状に組織化されていることが明らかになりました。ただし、このような超微細構造は、プロトタイプのβ-コロナウイルスであるマウス肝炎ウイルス(MHV)では観察されません。これまでのところ、コロナウイルスRNPの分子モデルは存在せず、他の(+)RNAウイルスのRNPのアーキテクチャ、アセンブリ、およびRNAパッケージングについてはほとんど知られていません。これらの質問に対処するために、初期のウイルス株から増殖した2,294個の無傷のビリオンの画像解析のためにクライオETとサブトモグラム平均化(STA)を組み合わせました。私たちの知る限り、これはSARSCoV-2ウイルスのこれまでで最大のcryo-ETデータセットです。ここでは、本物のSARS-CoV-2ウイルスのアーキテクチャとアセンブリについて報告します。
RESULTS AND DISCUSSION
The Molecular Landscape of the SARS-CoV-2 Virus
SARS-CoV-2ウイルスの分子的展望
SARS-CoV-2ビリオン(ID:ZJU_5)は、2020年1月22日に重度の症状のある患者から収集され、Vero細胞で増殖しました。武漢からの参加者との会議中に患者が感染しました。極低温電子顕微鏡(cryo-EM)分析では、ウイルスサンプルをパラホルムアルデヒドで固定しました。これは、7〜20Åの分解能でタンパク質構造にわずかな影響を及ぼします。無傷のビリオンと濃縮されていないビリオンは、クライオEMによって上澄みから直接視覚化され、楕円形と球形のエンベロープ粒子が示されました(図S1A)。これは、ショ糖クッションを介した超遠心分離によって濃縮されたビリオンの観察と一致しています(図S1B)。
脂質エンベロープをメッシュ化することにより、2,294個のビリオンを楕円体としてモデル化し、エンベロープの短軸、中軸、および長軸の平均直径をそれぞれ64.8±11.8、85.9±9.4、および96.6±11.8 nm(平均±SD)と測定しました。
Sタンパク質とRNP(図1A、S1B、およびS5A)は、ウイルスの最も特徴的な機能を表しています。

包括的なデータ分析が実施されました。
合計で56,832のスパイクがビリオンから手動で識別され、そのうちの約97%が融合前のコンフォメーションで、3%が融合後のコンフォメーションでした(メソッドの詳細)。
平均26±15の融合前Ssが、各ビリオンにランダムに分布していることがわかりました(図1Bおよび1C)。
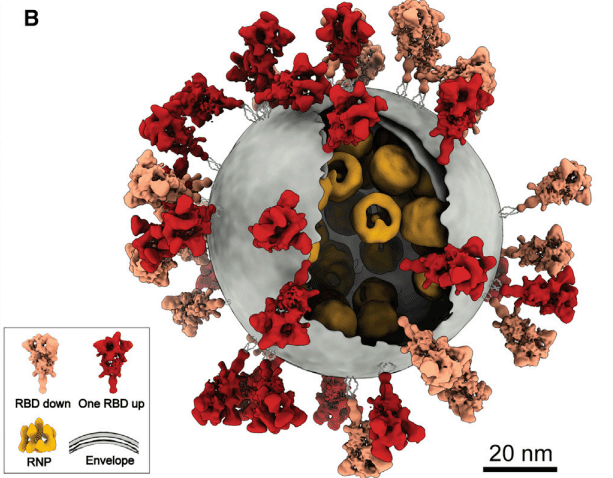

ビリオンあたりのスパイクコピー数は、ヒト免疫不全ウイルス(HIV)に匹敵しますが、ラッサウイルス(LASV)の約5分の1、インフルエンザウイルスの約10分の1です。
18,500のRNPがウイルス内腔で手動で識別され(表S1)、ビリオンあたり平均26±11のRNPが得られました。ただし、ウイルスの内腔にはRNPが密集しており、電子が不透明であるため、ビリオンあたりのRNPの実際の数は20%〜30%多いと推定されました。 つまり、ビリオンあたり30〜35のRNPです。規則的に順序付けられたRNP超構造が時折観察され(図S5A)、RNPがローカルアセンブリを形成できることを示しています。
不活化されたSARS-CoV-2ビリオンからのプレフュージョンSの2つのコンフォメーション、「RBDダウン」と「1つのRBDアップ」が分類され、STAによって8.7および10.9Åの解像度に再構築されました。ローカル解像度は7.8A°に達します(図S3A–S3C)。

S2サブユニットのヘプタッドリピート1(HR1)ドメインとセントラルヘリックス(CH)ドメインは、最もよく解決されたドメインです(図S3D)。
すべての融合前Ssに占める「RBDダウン」コンフォメーションの割合は、ビリオンあたり54%と推定されました(図1C)。
Sの膜近位ステムは、最も解像度の低い領域であり、局所解像度は約20 A°であり、構造内にTMまたは膜の痕跡は見られませんでした(図1D)。

トモグラムスライスを精査すると、エンベロープに対して垂直に立っているか、エンベロープに向かって傾いているスパイク集団が観察されました。これは、スパイクの外部ドメインがステムの周りを回転できることを示唆しています。プレフュージョンSsの洗練された向きは、エンベロープの外側で茎の周りをほぼ自由に回転し、エンベロープの法線軸に対して平均40±20度の角度で傾いていることを示しています(図1Bおよび1D)。
スパイクの回転の自由度は、密度が低いことで可能になります。これは、クラスIの融合タンパク質を持つ他のエンベロープウイルスとは著しく異なります。興味深いことに、2つの頭と1つの結合された茎を持つY字型のスパイクペアの少数の集団が観察されました(図S2AおよびS2C)。これは、茎と絡み合ったスパイクを表している可能性があります。
これらの観察結果は、SARSCoV-2スパイクがウイルスエンベロープに異常な自由を持っていることを示唆しています。
このような独自の機能により、ウイルスが周囲の環境を探索し、細胞受容体ACE2との関係を強化し、複数のスパイクを1つのACE2に結合させたり、1つのスパイクを複数のACE2に同時に結合させたりすることができます。
ただし、ウイルスエンベロープにまばらに詰め込まれたスパイクは、他の方法ではアクセスしにくいドメインやグリカンホールに結合する中和抗体に対しても脆弱です。
無傷のSARS-CoV-2の構造と景観に関する私たちの観察結果は、同時に発表された他の2つのクライオET研究と一致しています。
Native Structures of S in the Prefusion Conformation
融合前コンフォメーションにおけるSのネイティブ構造
「RBDdown」および「oneRBDup」コンフォメーションのSのネイティブ構造は、N末端ドメイン(NTD)を除いて、厳密に適合した組換えタンパク質構造と同様でした。
堅く柔軟にフィットするPDB(蛋白質構造データバンク)の比較:6XR8は、ネイティブスパイク構造のNTDがS2から9A°(重心距離)シフトしたことを示唆しています(図S3E)。組換えスパイク構造に対するネイティブスパイクでのNTDのわずかな拡張と低い局所分解能(図S3AおよびS3B)は、他のクライオET構造でも観察されました。
NTDは剛体として一定の可動性を示すことが知られています。
大規模な日付セットと分類により、スパイクの原子分解能に近いクライオEM再構成は準安定コンフォメーションを表す可能性がありますが、クライオET再構成はNTDのさまざまな動的状態の平均を表します。
10個のN結合型グリカンが「RBDダウン」で表示され、7個が「1個のRBDアップ」コンフォメーションで表示されます。そのうち、N61、N282、N801、N1098、およびN1134が最もよく解決されました。興味深いことに、糖鎖N1158およびN1173 / N1194の密度は、スパイクのステムに表示されます(図2BおよびS4C)。

一般に、ネイティブスパイクで観察されたグリカン密度は、完全長の組換え構造によく適合します。ただし、TMトランケートされた組換えスパイクで観察されたものよりもかさばります。同様の観察結果が、同時に公開されたネイティブスパイク構造の2つの研究によって報告されています。さらに、質量分析(MS)を使用してウイルスサンプルを分析することにより、ネイティブのグリカンの同一性を決定しました。ペプチド-N-グリコシダーゼF(PNGase F)消化の有無にかかわらず、ウイルス粒子はSDS-PAGEで分離されました。PNGase F処理後、S1サブユニットとS2サブユニットの重量はそれぞれ30kDaと20kDa減少しました(図2C)。

PNGase F処理前のS1およびS2に対応するバンドを、MSで分析して、22のグリコシル化部位のそれぞれのグリカン組成を明らかにしました(図2DおよびS4B)。
ネイティブグリカンの全体的な処理状態は、中東呼吸器症候群(MERS)およびSARS-CoVと共通の機能である、組換え糖タンパク質グリカンの処理状態(図S4AおよびS4B)と非常によく似ています。
未処理のオリゴマンノース型グリカンの集団は、グリカンが構造的役割を果たしていることが示唆されているN234を含め、組換え材料に見られるのと同じ部位に見られます。ただし、HIVの多くの部位で観察されるように、ウイルスは、組換え型の可溶性模倣物と比較して、オリゴマンノース型のグリコシル化のレベルがやや低くなっています。
全体として、実質的な複合型グリコシル化の存在は次の2つのことを示唆しています。つまり、小胞体の内腔へのSARS-CoV-2の出芽経路-ゴルジ装置中間コンパートメント(ERGIC)は、①グリカン成熟の障害にはならないこと、②SARS-CoVグリカンの分析、およびSARS-CoV-2のN343グリカンのフコースを標的とする中和抗体の同定と一致している、ということです。
さらに、HIVやLASVと比較してオリゴマンノース型グリカンのレベルが低いことも、グリカン密度が低いことと一致しています。
ネイティブプレフュージョンSと組換えプレフュージョンSの構造を比較すると、次のように結論付けられます。(1)ネイティブスパイクに存在するN結合型グリカンはかさばり、高レベルの複合型グリカンを含みます。(2)可溶性の三量体組換えS糖タンパク質によるネイティブウイルスのグリコシル化の主な特徴の要約は、組換えSタンパク質を利用したワクチン戦略に有望です。
The Native Structure of S in the Postfusion
融合後のSの本来の構造
Conformation
三角形のプレフュージョンSとは別に、ウイルスエンベロープに針状の密度が時折観察されました(図S2B)。15.3-A°の解像度の構造が解決され、融合後の形態のS(PDB:6XRA)にうまく適合し、Sタンパク質の融合後のコンフォメーションが示唆されました。外部ドメインの長さは22.5nm、幅は6 nmで、ウイルスエンベロープに対して垂直に立っています。
5つのN結合型グリカン(N1098、N1074、N1158、N1173、およびN1194)の密度がスパイクに表示され(図3D)、組換え後融合構造を連想させます。

融合前のコンフォメーションと比較して、エンベロープへの配向の固定は、融合後のコンフォメーションを達成するためのステム領域の劇的なコンフォメーションの再配列を示唆しています。融合後のSは、ウイルスのごく一部(すべてのウイルスの18%、それぞれが平均5つの融合後のSを持っている)でのみ検出されたため、より高い解像度に到達することができませんでした。興味深いことに、融合後のSを運ぶビリオンの中で、スパイクの総数が少ないビリオンは、融合後のSの割合が高くなる傾向がありました(図3C)。
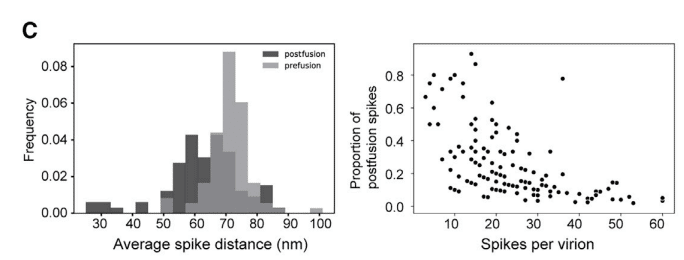
洗練された座標の統計(図3A〜3C)は、融合後のSsがウイルス表面に束ねる傾向があることを示唆しています。

そのようなバンドルの1つが再構築され、ウイルス表面上で約10 nmの間隔で配置された4つの融合後Sが示されました(図3B)。これに対して、最も近い融合前S間の平均距離は約15nmです。
SARS-CoVウイルスは、ビリオンあたり平均50〜100のスパイクを持っていると推定されていましたが、SARS-CoV-2ウイルスは、約半数の融合前Sを、たまに融合後のSを持っています。
SARS-CoV-2ウイルスで観察された融合後のSsは、次の3つの要因から来る可能性があります。
①ホストプロテイナーゼによって切断されたS1の時折の自発的な解離の産物。
②出芽した子孫ビリオンが細胞表面からいくつかの残留融合後Sを運んだときに、感染細胞上に自然に形成されたシンシチウム。
③β-プロピオラクトン固定ウイルスのクライオEM画像は、ウイルスに存在するほとんどのスパイクが融合後のようであることを示すという、サンプル準備手順。
融合前のSsのこのような不安定性は、他のβ-コロナウイルスで報告されています。さらに、分布グラフ(図3C)は、速度論的にトラップされた融合前Sが融合後Sよりも壊れやすく、ウイルスから解離する可能性さえあることを示しています。
推測は、細胞内ビリオンが平均して、私たちや他の人によって報告された細胞外ビリオンよりも多くのスパイクを持っているという事実と、私たちのデータでスパイクのない「ハゲ」ウイルスが時折観察されたことに基づいています。
要約すると、SARS-CoV-2プレフュージョンSsは不安定であると結論付けています。これは、ビリオン上の溶媒に曝されたエピトープの分布が、組換えタンパク質の観察よりも複雑であることを示しています。
私たちの観察は、十分な数の安定した抗原を好む効率的なワクチン設計と中和抗体開発に影響を及ぼします。
Architecture and Assembly of RNPs in Intact Virions
無傷のビリオンにおけるRNPのアーキテクチャとアセンブリ
コロナウイルスが直径約80nmのウイルス内腔に約30kbのRNAをどのように詰め込むかは謎のままです。
RNPは、RNAの絡み合い、結び目、さらには損傷を避けるために相互に関連して順序付けられていますか、それともウイルスの組み立てに関与していますか?
生の断層撮影スライスを検査したところ、密集したバケツのような密度がウイルスの内腔全体で識別でき、その一部は局所的に順序付けられているように見えました(図S5A)。
コロナウイルスの以前のクライオET観察とSDS-PAGE / MS分析(図2C)を組み合わせると、密度はRNPを表す可能性が最も高くなります。合計で18,500のRNPがウイルスの内腔で選択され、最初は球をテンプレートとして使用し、大きな球形のマスクを使用して位置合わせされました。
脂質二重層の密度に隣接して出現した構造的特徴がほとんどないバケツのようなコンフォメーション(図S5D)は、かなりの数のRNPが膜近位にあることを示唆しています。
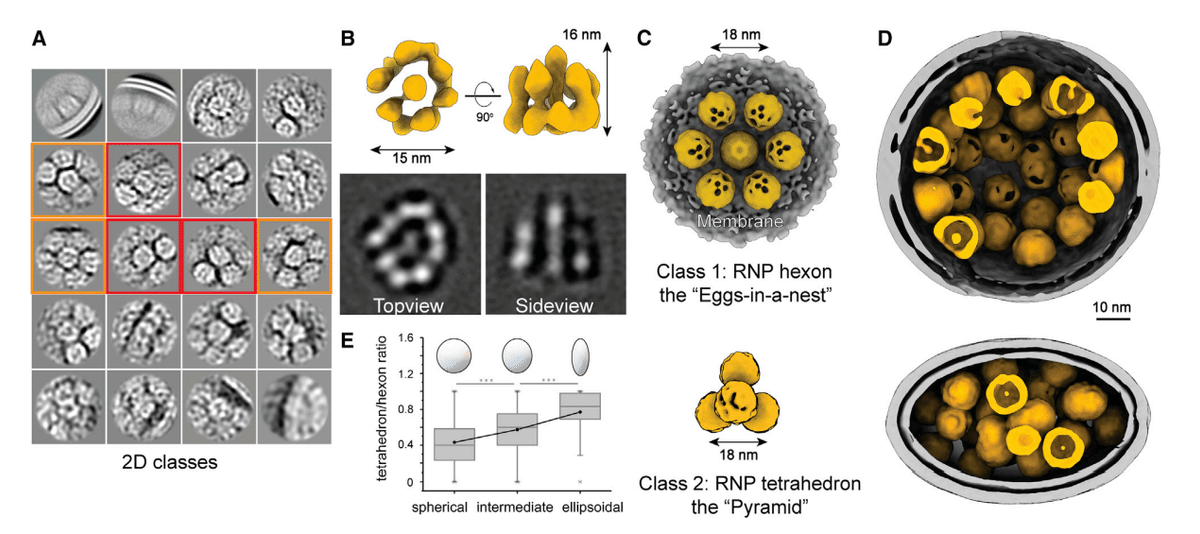
小さな球形マスクを使用した位置合わせにより、直径15 nm、高さ16 nmのRNPの13.1Å分解能の逆G字型アーキテクチャが明らかになりました(図4B)。
その形状は、最近報告されたSARS-CoV-2 RNPコンフォメーション、およびチクングニアウイルスRNPのinsituコンフォメーションに匹敵します。これもプラス鎖ですが、界面活性剤を使用して放出されるMHVRNPとは異なります。
マップは、頭から尾まで、逆 L 字型の 5 つの密度にセグメント化され、それぞれに N_CTD ([N_NTD]、PDB: 6WKP; C 末端ドメイン [N_CTD]、PDB: 6WJI)によって二量体化された N タンパク質のペアが当てはめられました(図S6AおよびS6C)。
デカマーの表面の静電ポテンシャル分布を分析し、RNA巻きRNPの暫定的な構造モデルを提案しました(図S6BおよびS6D)。
興味深いことに、MHV RNPの初期の観察では、1ターンあたり5つのサブユニットを持つ直径約15nmのヘリックスが示されました。
解像度が限られており、(+)RNAウイルスRNPに関する以前の構造知識がほとんどないため、モデルの解釈には注意が必要です。
RNPのさらなる2D分類により、次の3つのクラスが明らかになりました。(1)エンベローブに対して密に梱包され、(2)六角形および(3)三角形に梱包されたRNP(図4A)。
3Dの改良に続いて、膜近位の「巣の中の卵」型のRNPアセンブリ(「ヘキソン」と呼ばれます)とメンブレンフリーの「ピラミッド」型のRNPアセンブリ(「四面体」と呼ばれます)が現れました(図4CおよびS5E–S5G)。
2つのクラス平均を洗練された座標に投影すると、ヘキソンの大部分が球形のビリオンに由来するのに対し、より多くの四面体が楕円体のビリオンに由来することが明らかになりました(図4D)。
これは統計によって定量化されました。 楕円体のビリオンは、より多くのRNP四面体を詰め込む傾向がありました(図4E)。
さらに、2つの隣接するRNP間の間隔(〜18 nm)は、四面体とヘキソンで同じであり、一部の四面体は、その場の座標に投影するとヘキソンに集合する可能性があります(図S5B)。
このことは、RNPトライアングルが、ウイルス全体の重要かつ基本的なパッキングユニットであることを示唆しています。
さらに、RNPがコロナウイルスの組み立てに関与し、5サイクルの凍結と解凍後も精製されたビリオンが無傷のままであるため、環境的および物理的な課題に対するウイルスの強化に役立つことを提案します(図S7)。
ウイルスアセンブリへのRNPのこのような関与は、RNPがエンベロープの下で格子を形成することを示したNeuman(2006)によっても報告されており、細胞内ビリオンでも見られます。
ただし、RNPの超微細構造が、RNA、MまたはEタンパク質、RNP自体、またはこれらの倍数によって組み立てられているかどうかについては、未解決のままです。
RNPをサブナノメートルの解像度で解決することは、RNPが互いに混雑することによって妨げられました(図S5E〜S5G)。
さらに、おそらく個々のRNPとアセンブリ間の対称性の不一致が原因で、高次アセンブリのRNPの構造的特徴が不鮮明になっています(図4Cおよび4D)。
ルーメン全体で厳密に順序付けられたRNPを持つウイルスは、予測によって検出されませんでした。
ネイティブRNPは非常に不均一で密集していますが、ウイルス内で局所的に順序付けられており、「数珠玉構造」の化学量論でRNAと相互作用している可能性があると結論付けています。
約2,300の無傷のビリオンと300を超える傾斜シリーズから、融合前後のコンフォメーションのスパイクの構造、RNP、およびそれらが本物のウイルスでどのように組み立てられるかについての分子的洞察を提供します。
また、ネイティブスパイクの詳細なグリカン組成を分析しました。
ウイルス マップ (図 1B)、 本物の SARS-CoV-2 の「RBD ダウン」S、「1 RBD アップ」S、脂質エンベロープ、RNP コンポーネントで構成されたウイルス マップ (図 1B)は、アクセッションコードEMD-30430で電子顕微鏡データバンクに寄託されました。これは、完全なウイルス分子動力学シミュレーション、3D プリンティング、教育、または公共メディアのためのモデルを提供します。
以下省略。
この記事が気に入ったらサポートをしてみませんか?
