シールドを超えて:SARS-CoV-2スパイクタンパク質におけるグリカンの役割(2020年9月)
Beyond Shielding: The Roles of Glycans in the SARS-CoV-2 Spike Protein
シールドを超えて:SARS-CoV-2スパイクタンパク質におけるグリカンの役割
元→Beyond Shielding: The Roles of Glycans in the SARS-CoV-2 Spike Protein | ACS Central Science
Abstract
重症急性呼吸器症候群コロナウイルス 2 (SARS-CoV-2) によって引き起こされた進行中の COVID-19 パンデミックにより、これまでに世界中で 2,800 万人以上の感染者と 90 万人の死者が出ています。
抗体開発の取り組みは主に、アンジオテンシン変換酵素 2 (ACE2) に結合して宿主細胞への侵入を仲介する、高度にグリコシル化された SARS-CoV-2 スパイク (S) タンパク質を中心に展開されています。
他の多くのウイルス融合タンパク質と同様に、SARS-CoV-2 スパイクはグリカン シールドを利用して宿主の免疫反応を阻止します。
ここでは、利用可能な構造データと生物学的データを増強し、開いた状態と閉じた状態の両方で、グリコシル化された SARS-CoV-2 S タンパク質の全長モデルを構築しました。
グリカンの役割とタンパク質の構造およびダイナミクスに関する原子論的視点を提供するために、マイクロ秒単位の全原子分子動力学シミュレーションが複数回使用されました。
私たちは、ACE2 認識を担うスパイクの受容体結合ドメイン (RBD) の立体構造ダイナミクスを調節する上で、N165 および N234 部位の N グリカンが重要な構造的役割を果たしていることを明らかにしました。
この発見は、バイオレイヤー干渉法実験によって裏付けられており、N165A および N234A 変異によるこれらのグリカンの削除により、RBD 立体構造が「ダウン」状態へとシフトし、ACE2 への結合が大幅に減少することが示されています。
さらに、エンドツーエンドのアクセシビリティ解析により、SARS-CoV-2 S タンパク質のグリカンシールドの脆弱性の完全な概要が示され、この分子マシンを標的とした治療の取り組みで活用される可能性があります。
全体として、この研究は、SARS-CoV-2 Sタンパク質とそのグリカンコートに関するこれまで知られていなかった機能的および構造的な洞察を提示し、ワクチン開発に活用できるRBDの構造可塑性を制御する戦略を提供します。
Introduction
COVID-19は、2019年末に中国の武漢で始まり、現在では世界的パンデミックとして世界中に広がっている感染性呼吸器疾患です。
コロナウイルスが人間の健康に脅威を与えたのは今回が初めてではありません。
重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)はCOVID-19を引き起こす病原体であり、過去に流行を引き起こしたSARSや中東呼吸器症候群(MERS)関連コロナウイルスと同じコロナウイルス科に属しています。
免疫の欠如により、COVID-19 はすでに世界中で壊滅的な人命損失と重大な経済的損害を引き起こしています。
SARS-CoV-2 を含むコロナウイルスは、脂質エンベロープを持つプラス鎖 RNA ウイルスです。
宿主由来の膜とともに、一連の構造タンパク質がウイルス RNA を包み込み、封じ込める組織的足場を提供します。
その中で最も重要なのはスパイク、つまり S タンパク質です。これはコロナウイルス科全体でさまざまな程度に保存されており、ウイルスが最初に宿主細胞に付着して融合する際に重要な役割を果たします。
S タンパク質はクラス I 融合タンパク質で、1273 アミノ酸の単一のポリペプチド鎖として合成され、三量体として結合します。
各モノマーは、S1 と S2 の 2 つのサブユニットで構成され、頭部、柄部、細胞質尾部 (CT) の 3 つの主要なトポロジカル ドメインに分けることができます (図 1A)。

SARS-CoV-2 Sタンパク質の特に興味深い特徴の1つは、S1とS2(S1/S2)の間に新しいフーリン切断部位を採用していることです。これは、TMPRSS2プロテアーゼによって切断され、スパイクの感染準備を整えると考えられています。
S2′部位での2回目のタンパク質分解切断により融合ペプチド(FP)が放出され、これが宿主細胞膜を貫通して融合の準備を整えます。
最近発表された構造研究の多くは、複数のドメインで構成される SARS-CoV-2 スパイクのヘッド部分について、原子レベルまたは原子レベルに近いレベルでの理解をもたらしました (図 1A および 1B)。

S1 サブユニットには N 末端ドメイン (NTD) と受容体結合ドメイン (RBD) が含まれており、受容体結合モチーフ (RBM) はアンジオテンシン変換酵素 2 (ACE2) 受容体との相互作用を担い、宿主への侵入を促します。
S2 サブユニットは、ウイルスと宿主細胞の膜を統合する上で重要な役割を果たすため、「準安定バネ式融合装置」と適切に表現されています。これには、FP、中央ヘリックス (CH)、および接続ドメイン (CD) が含まれます。
クライオ電子顕微鏡または X 線実験では融合前の状態では解明されていない S2 サブユニット内の追加のドメインには、ヘプタド リピート 2 (HR2) と、ストークおよび CT を形成する膜貫通 (TM) ドメインが含まれます (図 1A および 1B)。
詳細な実験的構造特性が解明されていない S タンパク質のもう 1 つの重要な構造的特徴は、図 1C に示す広範なグリコシル化です。
ウイルスの病原性にはタンパク質のグリコシル化が重要な役割を果たしており、これはウイルス融合タンパク質の特徴的な厚い N グリカンコーティングによって実証されています。
たとえば、HIV-1 エンベロープ スパイク (Env) では、タンパク質がアクセス可能な表面領域はほぼ完全に N-グリカンで覆われています。これらは非常に密集しており、タンパク質の分子量の半分以上を占めています。
ウイルスエンベロープ糖タンパク質の表面に発現する N グリカンの生物学的役割は非常に多様であり、すべてその性質と密接に関連しています。
膜融合によるウイルスの侵入は、細胞表面受容体を含む分子認識イベントを介してエンベロープ糖タンパク質によって開始され、多くの場合、特定の N グリカンエピトープによって媒介されます。
さらに、非免疫原性または免疫原性が弱い複合炭水化物の高密度コーティングは、そうでなければ危険な露出ウイルスタンパク質に、免疫システムを回避するための完璧なカモフラージュ(またはシールド)を構成します。
このため、HIV-1 Env グリカンシールドは、主にオリゴマンノース (Man5–9) N-グリカンで構成されており、ウイルスが免疫系を阻止するのに非常に効果的であることが示されています;また、ウイルスと DC-SIGN C 型レクチンとの相互作用の原因であることもわかっています。
HIV-1 Envとは対照的に、ベータコロナウイルスのSARSおよびMERSのSタンパク質は、それほど効果的に遮蔽されていません。
さらに、SARS-CoV と SARS-CoV-2 のスパイクはどちらも HIV-1 Env とはかなり異なるグリコシル化パターンを示し、オリゴマンノース型に比べて複雑な N グリカンが多く存在します。
より具体的には、SARS-CoV-2 スパイクにはプロトマーあたり 22 の N グリコシル化部位が予測されており、そのうち少なくとも 17 が占有されていることが判明しています。さらに、少なくとも 2 つの O グリコシル化部位が予測されています (図 1C ~ 1E を参照)。

本研究では、ウイルス膜に埋め込まれた全長 SARS-CoV-2 S 糖タンパク質の、グリコミクスデータと一致する完全なグリコシル化プロファイルを持つ、マイクロ秒単位の全原子の明示的に溶媒和された分子動力学 (MD) シミュレーションを複数回提示します。
ここで議論されているシミュレーションは、利用可能な構造および生物学的データを増強および拡張して、SARS-CoV-2 Sタンパク質の全長のグリカンコート、構造、およびダイナミクスに関する原子レベルでの詳細な視点を提供します。
シールドを超えて、私たちの研究は、RBDの立体構造遷移の調整においてN165とN234にリンクされたNグリカンの重要な構造的役割を明らかにしています。
私たちのシミュレーションでこれらのグリカンを削除すると、RBDの「上」立体構造に不安定化効果が引き起こされます。
これらの結果は、ACE2結合の減少を示すバイオレイヤー干渉法実験によって裏付けられており、これがRBDの「下」集団の増加の根底にあります。
さらに、私たちのシミュレーションは、グリカンがSARS-CoV-2 Sタンパク質をカモフラージュして宿主の免疫応答を阻止する方法を強調しています。
S タンパク質のグリカン シールドの詳細な分析により、特に大きな分子に対しては茎に脆弱性がないことが明らかになりましたが、頭部領域はより実行可能なターゲットであるように見えます。
重要なことに、RBM アクセシビリティの詳細な概要は、「上」と「下」の RBD 構造間のグリカン シールドの範囲が著しく異なることを示しています。
全体として、この研究は SARS-CoV-2 S タンパク質に関する原子レベルの視点を提供し、免疫回避のためのシールド デバイスとしてだけでなく、ウイルスの感染性にとって不可欠な構造要素としてもグリカンの重要性を強調しています。
これらの洞察は、RBD 構造の可塑性とウイルスの感染性を調節する可能性のある戦略の基礎を築き、パンデミックの脅威と戦うことを目的とした治療薬の開発に活用できる可能性があります。
Results and Discussion
All-Atom MD Simulations of the Full-Length Model of the SARS-CoV-2 S Protein in Open and Closed States
開いた状態と閉じた状態の SARS-CoV-2 S タンパク質の全長モデルの全原子 MD シミュレーション
※省略します。
N-Glycans at N165 and N234 Modulate the RBD Conformational Dynamics
N165とN234のN-グリカンはRBDの立体配座ダイナミクスを調節する
SARS-CoV と SARS-CoV-2 の S 糖タンパク質は 76% の配列同一性を共有しており、SARS-CoV-2 で見つかった 22 の N グリコシル化部位のうち 18 は SARS-CoV から保存されています。
SARS-CoV-2 S タンパク質の全長モデルのシミュレーションの分析に基づいて、NTD の N165 と N234 にリンクされた 2 つの N グリカンを特定しました。これらの戦略的な位置と構造を考慮すると、RBD のコンフォメーション ダイナミクスで役割を果たす可能性があると仮説を立てました。
シミュレーションでは、Closed では、糖分析データと一致して Man9 としてモデル化された NTD-B 内の N234 の N グリカンが S タンパク質のコアから離れて溶媒に向かっていることが示されています (図 2A);対照的に、鎖 A の RBD が「上」である Open では、NTD-B の N234 にリンクされている同じ Man9 が内側に向けられ、RBD-A の開放によって空いた大きな容積に挿入されます (図 2B および 2C)。

グリカンは、リンクされた GlcNAc (またはキトビオース) 残基の構造的アラインメントによって構築されているため、存在する GlcNAc またはキトビオースの相対的な位置が、N グリカン全体の向きに影響を与える可能性があります。実際、N-グリカンコアは非常に硬い。
ここで説明したシミュレーションに対する力場や初期のクライオ電子顕微鏡構造の影響をさらに評価するために、AMBER ff14SB/GLYCAM06j-1 力場と、N234 にリンクされた GlcNAc がわずかに異なる方向で示されている代替の初期クライオ電子顕微鏡構造 (PDB ID: 6VYB) を使用して、開いた SARS-CoV-2 S タンパク質の頭部の追加シミュレーション セットを実行しました。
SI のセクション 2 で詳しく説明されているこれらのシミュレーションでは、N234 の Man9 グリカンが、反対側にある閉じた RBD ドメインの表面との相互作用を通じて、徐々に挿入されて三量体の頂端コアに到達し (ムービー S2)、図 2B で説明したものと同様の構造配置を採用していることがわかります。
N165 の高度に保存された N グリカンも NTD-B と RBD-A の間に挿入され、RBD-A と広範囲に相互作用するか、開いた後に空いた空間を占有します (図 2C)。

これは、SI のセクション 2 で説明されている追加のシミュレーションでも観察されました。
これらの観察を踏まえて、N165A および N234A 変異を導入して Open から追加の 3 番目のシステム「Mutant」を生成し、それぞれのサイトでグリカンの削除をもたらしました。
次に、Mutant を全原子 MD でシミュレートし、合計で約 4.2 μs の 6 つのレプリカを作成しました (表 S5)。
これら 2 つのグリカンを除去した場合の構造的影響を判断するために、主成分分析 (PCA) を使用して、Open、Mutant、および Closed システムの RBD のコンフォメーション ランドスケープを比較しました。
RBD-A ダイナミクスを最初の 2 つの固有ベクトル (PC1 と PC2)、つまり、軌道の分散が最も大きい 2 つの動き (それぞれ 65% と 15%) に投影した散布図は、Mutant の RBD-A が Open よりも広いコンフォメーション空間を探索し、Open は Closed よりもかなり広いことを明確に示しています (図 2D)。
オープンとクローズの違いは、RBD-A の状態 (「アップ/ダウン」) によってすぐに説明できますが、PCA ランドスケープは、N165 と N234 が存在する場合により安定した RBD の「アップ」コンフォメーションを特定し、それぞれの N 結合グリカンの構造的役割を示唆しています (動画 S3)。
RBD-A によって探索された PCA 空間の詳細な分析 (オープンとミュータントのみの比較、およびすべてのシステムでのレプリカごとの寄与を含む) は、図 S6 と S7 に示されています。
軌道に沿った RMSD の分析により、ミュータントにおける RBD-A の不安定性がさらに強調されています (図 S2)。
この動作は、SI のセクション 2 で説明されているスパイク ヘッドの追加の独立したシミュレーションによって明らかになったように、単一のポイント変異 (N234A) の場合でも確認されています。
RBD-Aとは対照的に、RBD-BとRBD-Cは3つのシステムすべてにおいて「ダウン」構造をしており、ほぼ安定した重なり合うPCA分布を示しています(図S7)。
RBD-A の動的挙動に関するさらなる洞察を得て、PCA によって明らかにされた探索されたコンフォメーション空間の違いを調べるために、RBD-A の変動を表す 2 つの角度 (以下、「横角」および「軸角」と呼ぶ) のダイナミクスに沿った変化を監視しました。
横角は、スパイクの中央ヘリックスを中心とした仮想円に沿った RBD の面内運動を示します (図 3A)。
この角度は、(i) フレーム 0 での RBD-A コア β シートの質量中心 (COM)、(ii - 頂点) CH の上部セクションの COM、および (iii) 軌跡に沿った各フレームでの RBD-A コア β シートの COM に対応する 3 つのポイントによって記述されます。

軸角は、スパイクの CH から離れるか近づく RBD の傾斜運動を識別します (図 3B)。
この角度は、(i) RBD-A コア β シートの COM、(ii - 頂点) CH の COM、(iii) CH の上部セクションの COM に対応する 3 つのポイントによって定義されます。
各システムの軌跡に沿った横方向および軸方向の角度変動の分布は、図 3C および 3D に示されています。

角度の変化は、軌跡のフレーム 0 での初期値に対して計算されました。
角度の定義と計算の詳細は、SI の「材料と方法」セクションに記載されています。
PCA によって明らかになった動的挙動と一致して (図 2D)、RBD-A の横角は、Open ではゼロを中心とした明確な単一の分布を示しますが、Mutant では二峰性の集団を示します (図 3C)。
この分析は、N165 と N234 の NTD-B N-グリカンの存在が、RBD-A の「上」コンフォメーションを安定化し、乱れた動きを防ぐのに重要であることを示しています。
興味深いことに、軸角分析の結果は、Open と Mutant で RBD-A の同様の挙動を示し、両方のシステムでロングテール分布を示しています (図 3D)。
ただし、Mutant は有意な負の傾向を示し、Open は正の傾向を示しています。
これは、N165 と N234 の N-グリカンが、RBD の「上」コンフォメーションを安定化するだけでなく、RBD の開閉を調節する上で重要な役割を果たし、ACE2 への結合に影響を与える可能性があることを示唆しています。
実際、「ダウン」状態の RBD はスパイク三量体の CH 上にあり、ACE2 にアクセスできませんが、「アップ」状態に移行すると中心軸から外れ、溶媒に突き出て結合できるようになります。
シミュレーションによって明らかになった仮説を確認するために、N165 および N234 の欠失が RBD 状態に与える影響を実験的に評価しようとしました。
溶液中の RBD のアクセシビリティを定量化するために、バイオレイヤー干渉法を使用して、N165A または N234A 置換を含む S タンパク質への ACE2 結合を測定しました。
その結果、これらの N 結合部位を除去すると、N165A および N234A 変異体の結合応答がそれぞれ親 S-2P 変異体と同程度に約 90% および約 60% (p = 0.0051 および p = 0.0002、スチューデント t 検定) に低下しました (図 4)。
S タンパク質の S-2P 変異体は、融合前構造を安定化するために導入された S2 サブユニット内に 2 つの連続したプロリン置換を持っていることに注意してください。

重要なのは、S383C/D985C 変異で操作され、ジスルフィド結合を介して 3 つの RBD すべてを閉状態でロックするネガティブ コントロール スパイク (HexaPro) は、ACE2 に結合しないことです。
これらの実験は、N165 および N234 グリカンが RBD の「上」構造を安定化し、ACE2 への結合を促進し、N234 グリカンが N165 よりも大きな役割を果たしているという仮説を裏付けています。
N165 および N234 の N グリカンが RBD を「上」状態で安定化する方法を特徴付けるために、水素結合分析によって S タンパク質との相互作用を調べました。
NTD-B 上の N234 にリンクされた Man9 は、RBD-A の開口によって作成された大きなポケットに深く伸びています (図 5C)。

具体的には、RBD-A の下部 (特に H519) と大部分が相互接続して下から支え、NTD-B の D198 と安定した水素結合を形成し、鎖 B の CH 内にある R983、D985、E988 の深いところで相互作用します (動画 S3)。
これらの水素結合はすべて、オープン システムの包括的な 4.2 μs の軌跡の 40% 以上で安定しており、水素結合の大部分は鎖 B の CH と RBD-A で形成されます (図 5A を参照)。
N165 の N-グリカンは、N234 の Man9 グリカンに比べて溶媒にさらされる割合が高いですが、それでも RBD-A の「上向き」コンフォメーションと広範囲に相互作用し (頻度約 90%)、シミュレーション レプリカ間で水素結合パターンが異なります (図 5B および 5D、図 S8、動画 S3)。
N234およびN165のN-グリカンで観察されたレプリカごとの水素結合ネットワークを図S9に示します。
全体として、PCA と角度分析によって明らかになった不均一な構造ダイナミクスと、バイオレイヤー干渉法実験によって明らかになった ACE2 結合応答の低下は、RBD の構造可塑性が N165 と N234 の N グリカンの欠如によって影響を受けることを示しています。
特に、RBD とその下にある受容体結合 C 末端ドメイン 2 (CTD2) を接続する 2 つの柔軟なリンカー ループは、ACE2 誘導ヒンジ運動などの大規模な構造再編成を RBD が受けるように準備します。
このシナリオでは、RBD の微妙な「アップ/ダウン」平衡は、RBD 内または近傍領域に単一の点変異を導入するか、pH 条件を変えることによって劇的に変化する可能性があります。
私たちの研究により、MERS スパイクの RBD と同様に、SARS-CoV-2 スパイクの RBD の「アップ」状態は、そのコンフォメーション アンサンブル内で準安定であることが明らかになりました。
RBD は、隣接する NTD に位置する 2 つの N グリカン、つまり N165 と N234 を必要とし、「アップ」コンフォメーションを「ロードしてロック」し、ACE2 受容体にうまく結合することがわかりました。
私たちのシミュレーションでは、RBD の完全な「アップからダウン」への遷移は示されていませんが、調査したグリカン削除変異により、「アップ」状態の RBD はより大きなコンフォメーションの自由度を経験し、より広い傾斜運動をします。
その結果、この構造の不安定性により、RBD にある潜在的エピトープが抗体認識にさらされやすくなり、RBD が閉じた状態に向かう構造シフトの結果としてウイルスの感染性が低下する可能性があるため、ウイルスの脆弱性が高まる可能性があります。
ここでは、バイオレイヤー干渉法実験を通じて、ACE2 への結合が N234A 変異体では著しく減少し、N165A 変異体ではわずかに損なわれることを示していますが、3 つの RBD すべてが「ダウン」状態に固定された人工 S タンパク質の場合は完全に失われます。
このように、これらの実験は計算予測を確認し、N234 および N165 グリカンの枯渇の結果として RBD 集団が「ダウン」状態に向かう構造シフトを主張しています。
これは、N234 に結合した Man9 グリカンのダイナミクスと一致しており、スパイクの中心軸の先端部分に向かって這い進み、「上」のコンフォメーションで RBD によって空いたスペースを埋め、同じ RBD「上」と CH の両方と持続的に相互作用しているのが観察されます。
したがって、このグリカンが、溶媒にさらされやすく、相互作用パターンがあまり明確でない N165 のグリカンよりも、RBD「上」を安定化させる上で大きな役割を果たしていることは驚くべきことではありません。
ただし、N165 は着実に RBD と相互作用し、したがって、程度は低いものの、「上」状態のロードとロックにも貢献していることを示しています。
SARS-CoV-2 スパイクは、構造ダイナミクスに影響を与える変異に対して顕著な感受性を示すため、特に RBD の「上/下」平衡の変化を伴う場合には、ワクチン設計の機会となります。
この文脈において、私たちの研究結果は、N165A および N234A 変異を導入することで RBD 構造の可塑性を制御できる可能性を指摘しています。
これらの N 結合部位、特に N234 は、RBD を開いた状態にロックするのに不可欠であり、SARS-CoV-2 ウイルスが ACE2 受容体を認識して宿主細胞に侵入するための準備に寄与している。
これらのグリカンを除去すると、主に「下向き」の RBD を持つ S タンパク質を持つ、感染力の低い、または「弱体化した」ウイルスが生じる可能性がある。
このシナリオでは、RBD が依然として「上向き」の状態に移行できるため、RBM を含む RBD の上部を標的とする体液性免疫応答を防ぐことはできません。 ただし、程度は低くなります。
代わりに、RBD のコア、NTD、または閉じた状態でも関与する可能性のある他の S2 領域にある他の既知のエピトープを認識する抗体の大量生成を刺激する可能性があります。
Glycan Shield of the Full-Length SARS-CoV-2 S Protein
全長SARS-CoV-2 Sタンパク質のグリカンシールド
ワクチン設計の文脈では、ウイルスが宿主の免疫反応を回避するために開発したすべての戦略を考慮することが重要です。
この枠組みの中で、多くのウイルスはグリカンシールドを使用して、中和抗体が標的とする免疫原性エピトープを隠します。
SARS-CoV-2 Sタンパク質の糖化は広範囲に及び、宿主の体液性認識を妨害する能力があります。
スパイクヘッドとRBDに加えて、インフルエンザウイルスの研究でも示されているように、抗体にとって魅力的なターゲットとなる可能性のあるもう1つの部分は柄です。
19 個の N 型糖鎖がスパイクの頭部領域をカモフラージュしているのに対し、茎には 3 つの N 型糖鎖付加部位 (N1158、N1174、N1194) しか存在しません。
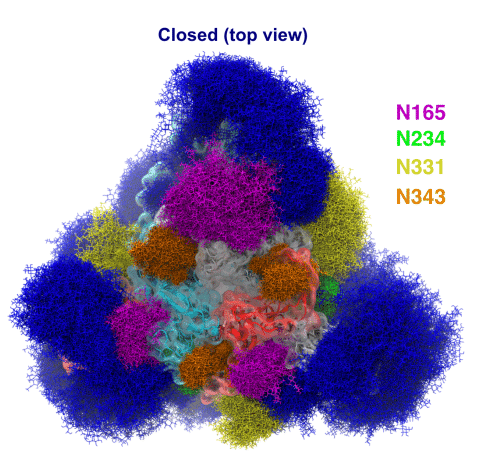
SARS-CoV-2 S 糖タンパク質の全長モデルの全原子シミュレーションから得られた広範なサンプリングを活用して、スパイクの糖鎖シールドのこれまでにないエンドツーエンドの概要を提供します (図 6)。

図 6A では、青いブッシュ状の構造はそれぞれ、1 μs の軌跡に沿ってサンプリングされた単一のグリカンの均等に散在するコンフォメーションの集合を表し、合計 300 個の重ね合わせたポーズがあります。
これらの N グリカンのダイナミクスの時間スケール (ナノ秒) が抗体/スパイク結合プロセス (マイクロ秒からミリ秒) と異なることを考慮すると、この表現は、スパイクを標的とする分子が結合前に遭遇する可能性のある遮蔽の現実的な見方を提供します。
この遮蔽効果を定量化するために、オープン システムのヘッド領域とストーク領域の両方でグリカンで覆われている S タンパク質のアクセス可能な表面積 (ASA) を計算しました。
また、さまざまなサイズの分子に対する遮蔽の有効性を測定するために、1.4 Å から 15 Å までの幅広いプローブ半径をスクリーニングしました。
1.4 Å という値は、通常、水分子の半径に近似するように設定されますが、より大きな半径は、2~5 Å の小さな分子から 10~15 Å のより大きなペプチドおよび抗体サイズの分子まで、より大きな部分を表すために使用できます。
結果は、すべてのプローブ半径において、ヘッドは全体的にストークよりもグリカンによる遮蔽が少ないことを示しています (図 6B および 6C)。
興味深いことに、ストークは抗体などの大きな分子にはほとんどアクセスできず、グリカンの被覆率は 15 Å 半径のプローブのタンパク質アクセス可能領域の 90% に相当します。
逆に、ヘッドは、グリカンのカモフラージュが不十分 (62%) で、その大きな表面積 (「上」コンフォメーションの 1 つの RBD を含む) をカバーするのが容易なターゲットとなります。
より小さなプローブをスクリーニングすると (半径 1.4~3 Å)、ストークとヘッドはそれぞれ平均 26% と 20% で同様に遮蔽され、これは、小分子がどちらの領域にも同じように浸透できることを示唆しています。
オープンでのヘッドとストークの ASA 平均および標準偏差値は、それぞれ表 S6 と S7 に示されています。
全体的に、グリコシル化を考慮すると、ベータコロナウイルスの間で高度に保存されているドメインであるにもかかわらず、茎は頭部よりも潜在的に治療の標的として難しいようです。
その小さな表面は、シアリル化およびフコシル化された大きな四本鎖グリカンによって十分に保護されており、大きな分子をほぼ 100% 遮蔽する効果があることがわかっています。
興味深いことに、N1174 と N1198 のグリカンは常に四本鎖であり、100% フコシル化 (N1174 と N1198)、および 100% シアリル化 (N1198) されていることがわかりました。
しかし、小さな薬剤は、柄の HR2 ドメインに結合することで融合プロセスを妨げる可能性があります。
対照的に、頭部はより脆弱であり、治療薬の開発に活用できます。
Glycan Shield of the Receptor Binding Domain
受容体結合ドメインのグリカンシールド
前のセクションで説明したように、グリカンシールドは、S タンパク質表面を分子認識から隠す上で重要な役割を果たします。
ただし、効果的に機能するには、スパイクが主要な宿主細胞感染経路であるACE2受容体を認識して結合する必要があります。
このため、RBMは完全に露出してアクセス可能になる必要があります。
このシナリオでは、グリカンシールドは、RBD が N グリカンの被覆より上に出現することを可能にする大きな構造変化と連携して機能します。
ここでは、RBD/ACE2 相互作用領域 (残基 400~508) に対応する RBD-A 内の RBM の ASA を、オープン システムとクローズド システムの両方でさまざまなプローブ半径で定量化します (図 7A および 7D、完全なデータは表 S8~S10)。

予想どおり、ASA プロットは、「ダウン」(クローズド) と「アップ」(オープン) RBD 構造の間に大きな違いを示しており、グリカンで覆われた RBM 領域は前者で著しく大きくなっています。
RBD-A が「上」のコンフォメーションにある場合、その RBM は平均 (全半径にわたって) わずか約 9% の表面積しかグリカンで覆われていませんが、クローズド システムでは約 35% です (図 7A および 7D)。
この差は、プローブ半径が 15 Å と大きい場合、さらに大きくなり、オープンとクローズドでそれぞれ最大 11% と 46% になります。
興味深いことに、プローブが小さい場合 (1.4~3 Å)、両方のシステムでシールドが弱くなり、オープンとクローズドでそれぞれ平均 6% と 16% になります。
ACE2 と直接相互作用しない RBD 領域は、「上」と「下」の両方のコンフォメーションでグリカンによって保護されたままであることに注目してください (図 S10)。
この領域は、主に N331 と N343 で RBD 自体に結合した N グリカンと、N165 と N234 で N グリカンに結合した N グリカンの存在により、RBD コンフォメーションに関係なく同様に保護されています (図 S11)。
最終的に、この分析は、RBD が「上」のときは RBM に常にアクセスできるのに対し、RBD が「下」のときは RBM が非常によくカモフラージュされていることを示しています (図 7)。
これは、この重要なドメインのグリカン シールドが「下から上」へのコンフォメーション変化と効果的にペアになっており、RBM がグリカン シールドから一時的に出現して ACE2 受容体に結合できることを示唆しています。
さらに、RBD を標的とする抗体は RBD が「下」の場合には効果がないかもしれないが、小分子はグリカンの覆いをより簡単に回避できる可能性があります。
これは、宿主抗体による RBD 中和の要件として「上」の立体構造を報告する構造データと一致しています。この点では、Sタンパク質を標的とするいくつかのSARS-CoV-2抗体が同定されています(表S11)。
これらの抗体の大部分は RBD 上のエピトープを認識しますが、NTD および CD 内の他の抗原領域に対応することがわかっている抗体はごくわずかです (図 S12)。

RBD 抗体のうち、B38 は RBD/ACE2 インターフェースで RBM と相互作用しますが、S309 および CR3022 は RBD の側面/下部を標的とします。
さらに、4A8 と 1A9 はそれぞれ NTD と CD と結合することがわかっています。
これらのエピトープに有効なグリカンシールドの分析は、SI のセクション 3 に記載されています。
Conclusions
私たちの研究では、ウイルス膜に埋め込まれたグリコシル化 SARS-CoV-2 S タンパク質の全長モデルについて、マイクロ秒単位の全原子の明示的に溶媒和された MD シミュレーションを複数回実施しました。
時間平均グリカンシールドが S タンパク質の表面積の大部分を覆う様子と、タンパク質のコンフォメーション状態に応じて、開いた状態と閉じた状態の間でどのように変化するかを示します。
興味深いことに、シミュレーション モデルでは、N165 と N234 の位置にある N グリカンが、シールドの役割を超えて、RBD コンフォメーション可塑性の調節因子としての役割を担っていることが明らかになっています。
N165A/N234A および S タンパク質の N234A 変異体のシミュレーションは、RBD の「上」の立体構造を安定化させる上でのそれらの重要な構造的役割を強調しています。
シミュレーション結果を確認するために、N165A および N234A 変異体に対して実施したバイオレイヤー干渉法実験では、これらのグリカンが除去されると ACE2 結合が減少し、RBD の立体構造が「下」の状態へとシフトし、N234A の影響がより大きくなることが明らかになりました。
全体として、私たちの研究は、この重要なターゲットの完全な構造に新たな光を当て、小分子とワクチン設計の機会と課題を示しています。
以下省略。
この記事が気に入ったらサポートをしてみませんか?
