糖鎖プロファイルの変化によりオミクロンスパイクタンパク質の表面アクセシビリティが向上(2024年5月)
Enhanced Surface Accessibility of SARS-CoV-2 Omicron Spike Protein Due to an Altered Glycosylation Profile
糖鎖プロファイルの変化により SARS-CoV-2 オミクロンスパイクタンパク質の表面アクセシビリティが向上

Abstract
SARS-CoV-2 スパイク (S) タンパク質は広範囲にグリコシル化され、適切な折り畳みを助け、安定性を高め、宿主の免疫監視を回避します。
この研究では、質量分析を使用して、SARS-CoV-2 オミクロン変異体 (B.1.1.529) 由来の組み換えスパイクタンパク質の N グリコシル化特性とジスルフィド結合を D614G スパイク変異体と比較して解明しました。
さらに、スパイクタンパク質に対してマイクロ秒単位の分子動力学シミュレーションを実施し、異なる N グリカンが 2 つの変異体のスパイク構造サンプリングにどのように影響するかを解明しました。
私たちの調査結果から、オミクロンスパイクタンパク質は、部位特異的なグリカン処理とジスルフィド結合形成の点で、D614G スパイク変異体と全体的に類似していることが明らかになりました。
それにもかかわらず、特定の N グリコシル化部位でグリカンの変化が観察されました。
これらの変化は、オミクロンスパイクタンパク質内の変異と相乗効果を及ぼし、エクトドメイン、受容体結合ドメイン、N末端ドメインを含む高分子の表面アクセシビリティの向上につながります。
さらに、突然変異誘発およびプルダウンアッセイにより、特定のセクオン(N149)のグリコシル化の役割が明らかになりました; さらに、MDシミュレーションとHDX-MSの相関関係により、スパイクタンパク質のいくつかの高動的領域が特定されました。
これらの洞察は、構造と機能の相互作用の理解に貢献し、効果的なワクチン接種および治療戦略の進歩につながります。
TEXT
重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)によって引き起こされるCOVID-19パンデミックは、引き続き重大な世界的健康課題を引き起こしています。
ウイルスは出現以来、複数の遺伝子変化を経て進化しており、多様な特徴を持つ変異体が出現しています。
オミクロンは最も最近流行した変異株であり、その亜種は2023年も依然として世界中で蔓延している優勢な株です。
オミクロン系統は、スパイクタンパク質の異常に高い変異数(図1)により、感染力の上昇、症状の軽度化、感染患者の重症度の低さをもたらし、大きな注目を集めています。
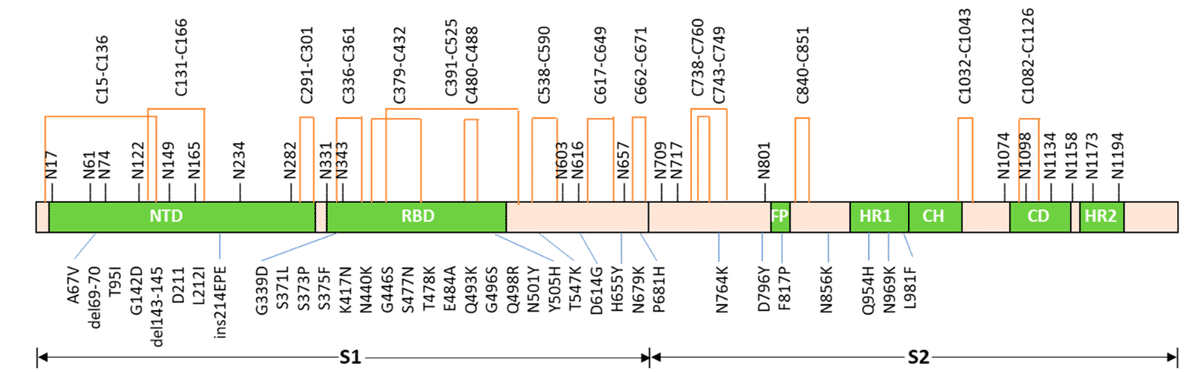
SARS-CoV-2ウイルスのスパイクタンパク質(S)は、ウイルスの侵入と感染において極めて重要な役割を果たします。
この膜固定型 S タンパク質はホモ三量体構造を形成し、それぞれのプロトマーには異なる機能を担う S1 サブユニットと S2 サブユニットが含まれていることが特徴付けられています。
S1 サブユニットは、N 末端ドメイン (NTD) と受容体結合ドメイン (RBD) で構成され、ウイルスが宿主細胞受容体アンジオテンシン変換酵素 2 (ACE2) に付着するのを媒介する重要な構成要素です。一方、融合ペプチドとその他のドメインを含む S2 サブユニットは、ウイルスの膜融合を担っています。
スパイク糖タンパク質は中和抗体の標的であり、したがってワクチン開発と治療介入の主な標的です。
アスパラギン残基への炭水化物部分の付加であるN-グリコシル化は、タンパク質の折り畳み、安定性、および免疫認識に影響を与える重要な翻訳後修飾です。
SARS-CoV-2 ウイルスの S タンパク質は高度にグリコシル化されており、各 S プロトマーには 22 の潜在的な N グリコシル化部位 (sequon、図 1) があります。
これらのグリカンは抗原部位を免疫監視から保護し、ウイルスの中和および免疫回避戦略に影響を与えます。
これまでの研究では、グリカンはSタンパク質を宿主の免疫認識から保護する以外にも役割を果たしていることも実証されている。
COVID-19パンデミックの発生以来、初期の株と進化する変異株におけるSARS-CoV-2 S糖タンパク質のグリコシル化プロファイルは、特に、外部ドメインまたはサブユニット構造の組み換え発現タンパク質、さらにはウイルス由来のSタンパク質を用いたタンデム質量分析法(LC-MS)と組み合わせた高度な液体クロマトグラフィーの使用を通じて、集中的に研究されてきました。
S タンパク質由来の糖ペプチドの分析により、ガンマスパイクタンパク質内の 22 個の保存されたシークオンすべてと 2 個の新規シークオンの N グリカン プロファイルを決定できます。
ジスルフィド結合は、タンパク質の構造的完全性を維持するために不可欠なもう 1 つの重要な修飾です。
システイン残基間のこれらの共有結合は、タンパク質の折り畳み、安定性、および全体的な構造に寄与します。
SARS-CoV-2 Sタンパク質のエクトドメインの一次配列には、30個のシステイン残基があります(図1)。

構造および質量分析により、これらのシステインが15個のジスルフィド結合を形成することが明らかになりました。
S タンパク質のジスルフィド結合は、その構造と機能において重要な役割を果たします。
研究では、チオール系薬剤が S タンパク質と ACE2 の結合を阻害し、人工ジスルフィド結合が S タンパク質を RBD の「ダウン」構造に閉じ込める可能性があることが示されています。
Sタンパク質の融合ペプチドのC840とC851の間のジスルフィド結合も、このペプチドと細胞膜との結合を促進する可能性があります。
最近の研究では、オミクロン S タンパク質の RBD ドメインの変異が 2 つのジスルフィド結合の安定性に影響を及ぼし、この S 変異体の還元に対する脆弱性を高めることが明らかになりました。
本研究の目的は、SARS-CoV-2 オミクロン S タンパク質 (S-オミクロン) の構造特性と変化を、D614G 変異体 (S-D614G) の S タンパク質と比較してより深く理解することでした。
そのために、LC-MS を使用して、それぞれの N グリコシル化プロファイルとジスルフィド結合を分析し、いくつかのセクオンにおけるグリカンの明確な分布を明らかにしました。
LC-MS の結果に基づく S-オミクロンおよび S-D614G 三量体モデルの分子動力学シミュレーションにより、結果としてタンパク質の立体配座サンプリングの変化がさらに解明されました。
要約すると、変異体における構造変化がウイルス構造、免疫認識、および治療戦略に及ぼす潜在的な影響についての洞察を提供します。
Results
・・・中略・・・
Molecular Dynamics Simulations
分子動力学シミュレーション
Sタンパク質の重要な構成要素であるグリカンは、タンパク質構造の安定化や免疫回避の促進など、さまざまな重要な機能を果たします。
CoV-AbDab(コロナウイルス抗体データベース)の最新アップデート(2023年6月13日)によると、合計12,536の抗体エントリのうち、ほぼすべてがSタンパク質を標的としており、抗体ベースの介入における中心的な役割を示しています。
グリコシル化は、Sタンパク質上の重要なエピトープにシールドを作成し、抗体の有効性を低下させることで、ウイルスが免疫システムを回避するのに役立つことがよくあります。
オミクロンとD614Gバリアントのグリコシル化プロファイルの違いがグリカンシールドとグリカンのその他の役割にどのように影響するかを理解するために、グリカンを組み込んだ両方のバリアントのSタンパク質の全原子モデルを構築しました(図S4)。

これらのモデルに統合された特定のグリカンの選択と構築は、上記の質量分析から得られたグリコシル化プロファイルデータによって導かれました。
その後、各システムに対して3つの1.4μs分子動力学シミュレーションを実行しました;また、グリカンが存在しない同じタンパク質モデルもシミュレートしました。
この包括的なアプローチにより、ウイルスの免疫回避戦略におけるグリコシル化の役割を解明し、さまざまな SARS-CoV-2 変異体に対するより効果的な抗体ベースの介入の開発を導くことができます。
Casalino らの研究にヒントを得て、S タンパク質のグリカン シールドの全体像を示します (図 5)。
図 5A と 5B は、シミュレーションの過程でグリカンを重ね合わせたもので、さまざまなコンフォメーションの可能性を示しています。

以前の研究ではより小さな間隔が使用されていましたが、この表現は依然としてグリカン シールドの現実的な描写を提供します。
抗体の結合プロセスはマイクロ秒単位で行われるため、選択された 0.25 μs の間隔は、計算の実現可能性とモデルの精度の間で適切なバランスを保ちます。
グリカン シールド下の S タンパク質のアクセス可能性を包括的に推定し、2 つのバリアントの違いを識別するために、1.4 Å から 8 Å の範囲のプローブ半径を使用してアクセス可能な表面積 (ASA) 計算を実行しました。
これらの異なるプローブ半径により、水などの小さな分子から抗体などの大きな実体まで、さまざまな次元の分子へのアクセス可能性を推定できます。
1.4 Å のプローブ半径は、通常、水のアクセス性を模倣するために使用されます。
抗体のアクセス可能な表面積を評価するために、8 Å に達するより大きなプローブ半径を使用しました。
プローブ半径は、以前の研究で使用された 5 Å ~ 10 Å の範囲と比較して効果的であることが証明されています。
グリカンがなくても、より大きな分子はより小さな分子よりもSタンパク質へのアクセス性が低いことが観察されました(図5C、D)。
さらに、グリカンの存在はこれを悪化させ、より大きな分子がSタンパク質にアクセスすることをますます困難にします。
OmicronとD614GのSタンパク質を比較すると、Omicronバリアントは大きな分子(半径8Å)の遮蔽面積が3%低いことがわかります。
SARS-CoV-2 の S タンパク質に存在するさまざまなエピトープのうち、RBD は最も多くのモノクローナル抗体の標的となっていることが特筆に値します。
実際、CoV-AbDab によると、S タンパク質を標的とする 12,431 個の抗体のうち、8,393 個は RBD に特異的に向けられています。
RBD のエピトープについてさらに詳しく知るために、RBD に存在するグリカン シールドを特に調べました (図 6A-D)。

興味深いことに、観察により、鎖 A の RBD は鎖 B に由来する多数のグリカンで包まれていることが明らかになりました (図 6A、B)。
このグリカン シールドのパターンは、鎖 B と鎖 C の RBD を調べても明らかであり、ウイルスの免疫回避戦略におけるこれらの構造の複雑な相互作用をさらに明らかにしています。
D614G 変異体の場合、鎖 B の N17 の位置にあるグリカンが RBD を包み込みますが、これは Omicron 変異体では観察されません。
N17 の位置にある最も豊富なグリカンは、Omicron では H2H4F1、D614G では N5H4A1 です。
この結果、Omicron では D614G よりも N17 グリカンが短くなります。これが、N17 グリカンが D614G では RBD を包み込みますが、Omicron では包み込まない理由であると考えられます。
オミクロンの RBD 周囲に N17 グリカンが存在しないことにより、この変異体では Ser469 から Val483 までの残基のアクセス可能な領域が広くなります (図 6A、B、S6)。

Delta 変異体の N17 のグリコシル化部位が存在しないことは注目に値しますが、これは T19R 変異によるものと考えられます。
Gamma 変異体の S タンパク質の N17 位置にグリカンが存在しないことも以前に指摘されています。
RBD 内の潜在的なエピトープをさらに特定するために、グリカンの存在を考慮して各残基の ASA を分析しました (図 S7)。
この分析では、Omicron バリアントの Ser469 から Val483 までの残基が再び大きな ASA を示しました。
また注目すべきは、Omicron バリアントの S477N および T478 K 変異により、S タンパク質内の RBD の ASA がさらに増幅されることです。
N17 のグリカンが短いか存在しないことと、タンパク質変異の組み合わせにより、この領域は溶媒にさらされやすくなります。
より広い視点で、オミクロン株の RBD を比較すると、特に大きな分子の場合、RBD 上のグリカン シールドはオミクロン株の方が D614G よりも広範ではありません (図 6C、D)。
具体的には、半径 8 Å の分子の場合、オミクロン株 RBD ではグリカンによって遮蔽された領域の 35% があるのに対し、D614G バリアントでは RBD 領域の 44% が遮蔽されています。

RBD のほかに、もう 1 つの潜在的なエピトープは N 末端ドメイン (NTD) です。
オミクロン株 NTD は、D614G バリアントの NTD よりもグリカンによって遮蔽されていません (図 6)。
特に、半径 8 Å の分子の場合、オミクロン変異体では 45% の領域がグリカンで覆われているのに対し、D614G 変異体では 54% の領域がグリカンで遮蔽されています。

オミクロン株と D614G株では、N149 の位置で最も頻繁に観察されるグリカンはそれぞれ N2H5 と N4H5F1A1 であり、Omicron の N149 グリカンが短くなり、N17 の変化とともに、N 末端ドメイン (NTD) のグリカン遮蔽が減少します。
図 S8 は、NTD のグリカン遮蔽の範囲を示しています。

図 S9 は、N17 と N149 の位置にある全長グリカンを示しています。

オミクロン変異体の二次構造、具体的には残基 140 から 158 にまたがる 2 つの β ストランドとそれらの間のループの安定性が低下していることが観察されました。
この不安定性は、残基 V143、Y144、Y145 の削除、およびこの領域の G142D 変異に起因する可能性があります。
グリカンは免疫回避を促進するだけでなく、S タンパク質構造を安定化させる上で極めて重要な役割を果たします (図 7)。
グリカンのない Omicron S タンパク質の 1.4 μs シミュレーション 3 回のうち 2 回では、ダウン (閉じた) 構造から開始し、「サブダウン」RBD 構造を特定しました (図 7A)。
このサブダウン RBD 構造は、グリカンが存在する状態で実行されたシミュレーションでは現れません (図 7C)。

RBD の立体配座を定量化するために、距離と二面角という 2 つの集合変数を導入しました。これは、以前に使用した定義と一致しています。2 次元 KDE プロットを使用して、これら 2 つの変数の分布を同時に視覚的に表現しました (図 7B、D)。
コンテキストを提供するために、RBD の「上」または開いたコンフォメーションは、距離 70 Å および二面角 0° で定義されます。
グリカンのないシミュレーションでは、2 つのクラスターが識別されました。1 つは「下」または閉じた RBD コンフォメーションを表し、もう 1 つはサブダウン コンフォメーションを表します (図 7B)。
両方のコンフォメーションを図 7A に示します。

対照的に、グリカンが存在する場合の RBD コンフォメーションの分布は、3 つのレプリカすべてで見られるように、閉じた状態のクラスター内にのみ存在します。
さらに、分布の中心の周囲でより強く集中しているように見え、グリカンの存在が RBD コンフォメーションを安定化することを示しています。
さらに、RBD を包むグリカン N165、N234、および N343 を観察しました (図 7C)。これは、以前の観察と一致しています。

これは、RBD を囲むグリカンが RBD を安定化させ、サブダウン構造の出現を防ぐという考えをさらに裏付けています。
ただし、サブダウン RBD 構造は、グリコシル化されていない D164G S タンパク質では観察されません (図 S10)。

分析 (図 7E) により、一貫した傾向が明らかになりました。D614G および オミクロン株の両方のグリコシル化 S タンパク質は、グリコシル化されていない対応するものと比較して、平均二乗偏差 (RMSD) 値が低下しており、グリカンが存在すると構造安定性が向上することを示しています。

・・・中略・・・
Discussion
この包括的な研究では、高度な LC-MS 技術を使用して、D614G S タンパク質を対照として使用しながら、オミクロン変異体由来の組み換えスパイク外部ドメインタンパク質の N グリコシル化およびジスルフィド結合プロファイルの分析を実施しました。
結果の正確性と妥当性を確保するために、同一の実験条件下で発現および精製されたタンパク質を使用し、異なる消化実験からの複数のデータセットが一貫した結果を示した場合にのみ、N グリコシル化データを選択しました。
これらの戦略を通じて、S-オミクロンおよび S-D614G タンパク質の 22 の潜在的な N グリコシル化部位すべてにわたる分布と相対的存在量を決定しました。また、2 つのタンパク質内でジスルフィド結合を検出しました。
データから、Omicron S タンパク質は、ほとんどのシークオンで S-D614G のグリカン処理パターンと密接に一致することが明らかになりました。
一方、特定の部位、すなわち N61、N149、N657、および N1098 では、グリカンの種類に有意な違いが見られました。
さまざまなセクオンでの主なグリカンの存在量に関しては、N17、N149、N331、および N657 を含む複数の部位で違いが明らかでした。
私たちの調査では、ジスルフィド形成ペプチドと遊離硫黄含有ペプチドの強度を比較することにより、2 つのタンパク質間で非常に類似した 15 個のジスルフィド結合のうち 12 個も検出されました。
さらに、主要なグリカンを含む場合と含まない場合の両方のタンパク質バリアントの MD シミュレーションを実行し、N グリコシル化がタンパク質構造に与える影響を明らかにしました。
私たちは、高度な質量分析技術と D614G および Omicron S タンパク質の MD シミュレーション モデルを 2 つの方法で組み合わせました。
まず、両 S タンパク質のボトムアップ グリコプロテオミクス解析の結果を使用して、各シークオンで特定のグリカンを選択し、MD モデリングで生物学的に正確な情報を提供しました。
次に、MD シミュレーションと解析の後、コンピューター ベースの結果を HDX-MS 解析を含む実験室での構造解析と比較しました。
シミュレーションにより、S-Omicron は S-D614G と比較してグリカン シールドが縮小していることが明らかになりました。
この違いは、潜在的なエピトープ領域を含む RBD 領域と NTD 領域内で特に顕著でした。
MD シミュレーションと HDX-MS 解析の結果を比較すると、受容体結合ドメインやフーリン切断部位など、両方の方法で動的 (動いている) と特定された領域は、S タンパク質機能において十分に文書化された役割を持っていることがわかりました。
これらの発見により、グリコシル化、タンパク質構造、免疫回避戦略の複雑な相互作用に対する理解が深まります。
効果的なワクチン接種と治療戦略の追求が世界中で続く中、私たちの研究はこの進行中の取り組みに重要な洞察を提供します。
以下省略。
この記事が気に入ったらサポートをしてみませんか?
