オミクロン株スパイクの構造的多様性(2022年1月)
Structural diversity of the SARS-CoV-2 Omicron spike
SARS-CoV-2オミクロンスパイクの構造的多様性
元→Structural diversity of the SARS-CoV-2 Omicron spike (biorxiv.org)
Abstract
広範なスパイクタンパク質の変異に助けられて、SARS-CoV-2オミクロン株は以前は優勢だったデルタ株を追い越しました。
スパイクコンフォメーションは、受容体結合ドメイン(RBD)の変化と、ウイルスの伝染性と免疫回避に影響を与える中和抗体エピトープの提示を介して、SARS-CoV-2の進化において重要な役割を果たします。
ここでは、オミクロンスパイクとデルタスパイクのクライオEM構造を決定して、それぞれの変異のコンフォメーションへの影響を理解します。
オミクロンスパイク構造は、デルタスパイクでは観察されなかった長距離の影響を伴う、異常に密集したRBD組織を明らかにしました。
結合と結晶学により、オミクロンスパイクの機能的に重要な融合ペプチド部位での柔軟性の向上が明らかになりました。
これらの結果は、高度に進化したオミクロンスパイクアーキテクチャを示しており、その高レベルの免疫回避と伝達性に影響を与える可能性があります。
Introduction
SARS-CoV-2オミクロン(B.1.1.529)バリアントは、南アフリカで2021年11月24日に特定され、11月26日に世界保健機関によって懸念のバリアント(VOC)として宣言されました。そして、世界的に流通しているSARS-CoV 2の主要な形態として、デルタ(B.1.617.2)に急速に取って代わりました。
オミクロンスパイク(S)タンパク質は、30の変異を含み、これまでに同定された中で最も免疫を回避するVOCであり、抗体(Abs)による中和に抵抗する能力においてベータ(B.1.351)を上回っています(補足図1)
構造研究は、VOC Sタンパク質のコンフォメーションの変化を明らかにし、より高い伝達率と免疫回避を促進する原子レベルのメカニズムを理解する上で重要な役割を果たしてきました。
融合前のSARS-CoV-2Sタンパク質は、S1サブユニットとS2サブユニットで構成されており、受容体の結合と宿主細胞膜との融合を促進するために構造が変化します。
S2サブユニットは、受容体が関与する前は立体構造的に安定していますが、モバイルN末端ドメイン(NTD)と受容体結合ドメイン(RBD)を備えたS1サブユニットは本質的に動的です。
RBDは、ACE2受容体の結合部位が閉塞している「閉じた」(「ダウン」)状態と、ACE2結合部位を露出している「開いた」(「アップ」)状態の間で遷移します。
Sタンパク質の受容体結合とタンパク質分解プロセシングに続いて、S2サブユニットは大きなコンフォメーション変化を起こし、その結果、融合ペプチド(FP)が放出され、ウイルスと宿主細胞膜の融合が仲介されます。
RBDダイナミクスは、プロトマー間RBD→RBDおよびRBD→NTDの接触、および他のSタンパク質構造単位の影響を受けます。SD1およびSD2サブドメイン、およびプロトマー内でNTDとRBDを接続する「N2R(NTD→RBD)リンカー」を含みます。
S1ドメインの相互作用がVOCによってどのように調節され、Sタンパク質のRBDの提示が変化するか、およびこれらを免疫原の設計にどのように利用できるかについては、前に説明しました。
ここでは、ネイティブの安定化されていないオミクロンおよびデルタSタンパク質の外部ドメインの構造を決定して、獲得した変異がそれらのコンフォメーション状態をどのように変化させ、受容体結合部位とAbエピトープの提示に影響を与えるかを理解します。
S外部ドメインは、前述のS-GSAS-D614Gプラットフォームで、S2サブユニットのプロリン安定化変異なしで調製されました。これらは融合前のSタンパク質のコンフォメーションランドスケープを変更する可能性があるためです(図1A、図S2)。
オミクロンおよびデルタセクトドメインの3-RBD-down、1-RBD-up、および2-RBD-upの集団の構造を決定しました(図1-3、図S3からS7)。
ミンクに関連するスパイクで以前に説明した外部ドメイン構造と同様に、1つの構造クラスが無秩序なS1サブユニットプロトマーを示すデルタS1サブユニットにかなりのダウンステート変動が見られました。
対照的に、オミクロンSタンパク質は、RBD-RBDインターフェースを安定化する16の免疫回避RBDアミノ酸置換のいくつかでS1変動の減少を示しました。
これらの置換は、デルタスパイクでは観察されない方法で1-RBDアップ状態も安定させました。
オミクロン3-RBDダウン構造でのRBDの密なパッキングにより、RBDの動きが制限され、顕著な単一プロトマーNTD-to-RBD(N2R)リンカーがコンフォメーションを再配置します。
この再配置はデルタ株やその他の亜種で確認されましたが、それらの間では比較的まれでした。
また、Ab DH1058に向けられた融合ペプチド(FP)への結合の強化によるものと同様に、S2コンフォメーションダイナミクスの変化は、2G12および第4級S2グリカンクラスターを標的とする他のFab二量体化グリカン反応性(FDG)抗体へのオミクロンSタンパク質の結合が弱い(G614およびデルタ株 Sタンパク質と比較して)ことによって示されました。
FP-Ab複合体の高解像度の結晶構造は、FPを閉じた状態の位置から解放することで強化を行う必要があることを示しています。
したがって、強化されたFPダイナミクスは、オミクロンの強化された伝達率にリンクされている可能性があります。
まとめると、これらの結果は、免疫回避を超えて、十分に制御された核融合機構と、より容易なFP放出につながる核融合ペプチド(FP)の変化したダイナミクスを備えた、よりコンパクトなアーキテクチャに進化したオミクロンスパイクを示しています。
Results
Conformations of the SARS-CoV-2 Omicron S protein
SARS-CoV-2オミクロンSタンパク質のコンフォメーション
最近の研究では、S2サブユニットにプロリン安定化変異(2PまたはHexaPro変異)を伴うオミクロンSタンパク質の構造が報告されているため、まず、S-GSAS-オミクロンとS-GSAS-2P-オミクロンの受容体ACE2とRBDに向けられた抗体への結合を比較することにより、このような安定化の効果を評価しました(図1、AおよびB)。

不明瞭なRBDエピトープを標的とするAbであるCR3022は、S-GSAS-2P-Omicronへの90%を超える結合を失ったことがわかりました。2P変異が、オミクロンSタンパク質の外部ドメインのコンフォメーションの多様性を制限していることを示唆しています。
2P対非2Pオミクロンスパイクの結合の減少は、DH1047およびS2X259Absの3-RBD-upコンフォメーション結合でも見られます。
したがって、S-GSAS形式のネイティブオミクロンSタンパク質外部ドメインの構造を決定して、変異がそのコンフォメーションに与えた影響を調べました(図1、図S2からS5、表S1)。
クライオEMデータセットでは、Sタンパク質外部ドメインの3-RBD-down、1-RBD-up、および2-RBD-upの集団を特定しました(図1C)。

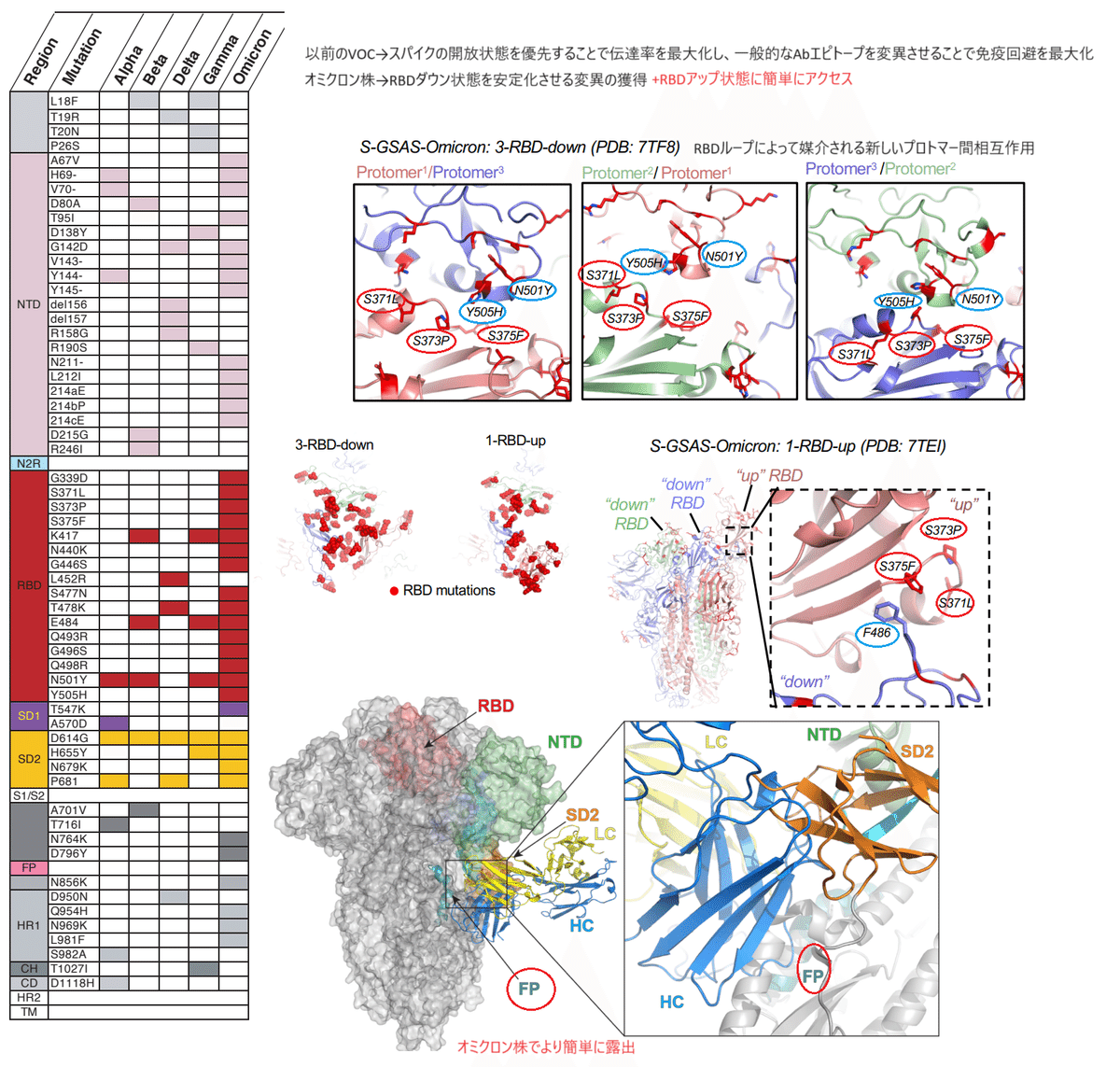
オミクロンスパイクには、RBDに16個のアミノ酸置換があり(図1D)、そのうちのいくつかはACE2認識や免疫回避を仲介することが示されています。

3-RBDダウンの母集団は、2つの非対称再構成に分類され、それぞれが密接なRBD-RBDペアリングを表示します。
O1とO2という名前の2つの3-RBDダウン構造のうち、プロトマー間ドメインの配置は、O1よりもO2の方が対称的であるように見えました。
この非対称性は、構造内のCa原子の各ペアと、2番目の構造内の対応するCa原子のペアとの距離の差を計算することにより、構造のペア間の重ね合わせのない比較を提供する差距離行列(DDM)で視覚化されました(図S5)。
タグProtomer1を、各再構成で最もモバイルRBDが高く、RBDマップ密度が最も低いことを特徴とするプロトマーに割り当てました(図1C)。
「ダウン」RBD間のプロトマー間相互作用は、3つのアミノ酸置換、S371L、S373P、およびS375Fを含むループによって媒介されました。
S373P変異によって引き起こされたループの再配置により、S373PおよびS375Fと隣接するRBDのN501YおよびY505H置換との相互作用を介して、RBD-RBDインターフェースのより緊密なパッキングが容易になりました(図1E)。

1-RBD-up構造では、S375F置換により、隣接するRBD-downプロトマーの残基Phe486とプロトマー間相互作用が生じました(図1F)。

2-RBDアップ状態では、両方のアップRBDが無秩序であり、移動性が向上していることを示しています(図1C)。
全体として、これらの結果は、オミクロンSタンパク質の変異が、RBDのカップリングを誘発し、固有のSタンパク質の閉じた構造状態と開いた構造状態を引き起こしたことを示しています。
以前、RBDのアップ/ダウン遷移には、SD1 / SD2サブドメインの移動、および単一のプロトマーユニット内でNTDとRBDを接続するN2Rリンカー(残基306-334)の移動が伴うことを示しました(図2A)。

RBDの「ダウン」状態では、N2R領域がスタックし、各SD1およびSD2サブドメインにβストランドを提供します(図2B)。

特に、オミクロン 3-RBD-down構造O1(PDB ID:7TF8)(図1Cおよび2B)では、モバイルProtomer1のN2R領域の二次構造が変更されていることがわかりました。R319とNTD残基E298の間に形成された新しいプロトマー内塩橋によって安定化されたSD2関連βストランドの切断を伴います(図2、BおよびC)。
これには、NTD295-303ヘリックスに対する残基F318のパッキングが伴いました。
この再配置により、ここでは「SD2フレックス」と呼ばれるSD2の一般的に無秩序なセグメントとの相互作用が可能になります(残基619-642)。
SD2フレックスの安定化は、残基Ile624(SD2フレックス)とSD2残基V595およびY612(図2C)とのファンデルワールス相互作用、およびNTD残基P295とN2Rリンカー残基R319の間のTrp633パッキングによって促進されました。
NTDヘリックス295-303、SD2ループ、およびSD1ループ554-565の空間配置は、1-RBD-up構造の「アップ」プロトマー(Protomer1)と3-RBD-down構造のモバイルプロトマー(Protomer1)の間と類似していました。モバイルプロトマーが「アップ」の位置に移行する準備ができている可能性があることを示唆しています。
モバイルSD1領域は、3-RBD-downと1-RBD-upの両方のオミクロンSタンパク質で安定化されました。SD1の残基D568およびT572によって媒介されるS2サブユニットとのプロトマー間水素結合を介して、オミクロンSタンパク質融合ペプチドの近くにN856Kアミノ酸置換があります(図2、EおよびF)。

「アップ」プロトマーのN2Rリンカーの残基T315のカルボキシル酸素と、オミクロンSタンパク質のN764K置換の間にも水素結合が形成されました。
したがって、オミクロンSタンパク質に戦略的に配置された残基置換は、S1サブユニットの移動性の高い領域を安定させます。
まとめると、3-RBDダウンオミクロンSタンパク質のRBDの密なパッキングと、「ダウン」プロトマーのN2R再配置は、獲得した変異の広範なネットワークから生じる幅広いコンフォメーションの影響を定義します。
Conformations of the SARS-CoV-2 Delta S protein
SARS-CoV-2デルタSタンパク質のコンフォメーション
次に、Delta Sタンパク質を調べて、オミクロンとの病理生物学の違いの根底にある可能性のある構造特性の違いを理解しました。
S-GSAS-Delta Sタンパク質外部ドメインのクライオEM構造を決定しました(図3、図S6とS7、および表S1)。
デルタバリアントSタンパク質には、NTDでの2つの置換と欠失、2つのRBD置換、フューリン切断部位の近位でのP681R置換、およびS2サブユニットのHR1領域でのD950N置換が含まれます(図3Aおよび図S1)。

オミクロンデータセットの場合と同様に、3-RBD-down(D1)、1-RBD-up(D2)、2-RBD-up(D3)のSタンパク質外部ドメイン集団を特定しました(図3A)。さらにD4を付け加えました。D4は、NTD、RBD、SD1、N2Rリンカー、およびSD2サブドメインの一部を含むS1サブユニット全体がクライオEM再構成で表示されないように、S1サブユニットの1つで非常に高い無秩序を示しました(図3A)。
これは、ミンクに関連するスパイクに見られる状態に似ていました。
7つの異なるサブクラス(D5-D10)は3-RBDダウン集団(図3B)に分類されました。そのうちの1つ(D6)は、オミクロンO1プロトマー1で観察されたN2R構成を示しました(図3C)。

SD2サブドメインとのbストランド配置からのN2R領域の同様の転位は、1-RBDアップサブクラス(D12)の「ダウン」プロトマーでも見つかりました(図3C)。2-RBDアップ状態の中間を表す可能性があります。
一方、ミンクに関連するスパイクで観察された単一のS1プロトマーの無秩序状態の出現(図3、AおよびD)は、NTDを含むモバイルS1領域に起因するSタンパク質の不安定性を示唆しています。

Domain positioning in Omicron, Delta and engineered S proteins
オミクロン、デルタ、および操作されたSタンパク質におけるドメインポジショニング
次に、デルタおよびオミクロン3-RBDダウン構造におけるN2Rリンカーの再配列の原因を調査しました。
S1サブユニットドメインの配置は、隣接する、接触しているRBD、NTD、およびサブドメイン間の通信を通じて、プロトマー間で相互に応答します。
エンジニアリングと以前のバリアントの調査により、この通信がRBDのアップ/ダウン状態の表示に重要な役割を果たすことを実証しました。
オミクロンスパイクのRBD間の密接な相互作用により、導入されたRBDからS2ジスルフィドステープルに対してrS2dと呼ばれる3-RBDダウン状態をロックするように設計された設計が、RBDの移動の制限により、同様のN2Rリンカーの再配置を示すかどうかを尋ねました。
rS2dデザインのS2安定化HexaProバージョン(rS2d-HexaPro)および非安定化rS2d(図S8およびS9、表S1)のクライオEM再構成を取得しました。
両方のデータセットを3次元で分類すると、2つの顕著な構造状態になりました(図4)。
両方のデータセットの1つの再構築は、それぞれ状態1と呼ばれ、デルタD6およびOmicronO1 3-RBD-down構造で観察されたものと同様のSD2再配置を示しました(図4A)。

両方の状態間の構造モーフの調査(ビデオS1)により、リンカー置換プロトマーでSD1がSD2に向かって著しくシフトしていることが確認されました(図4、BおよびC)。
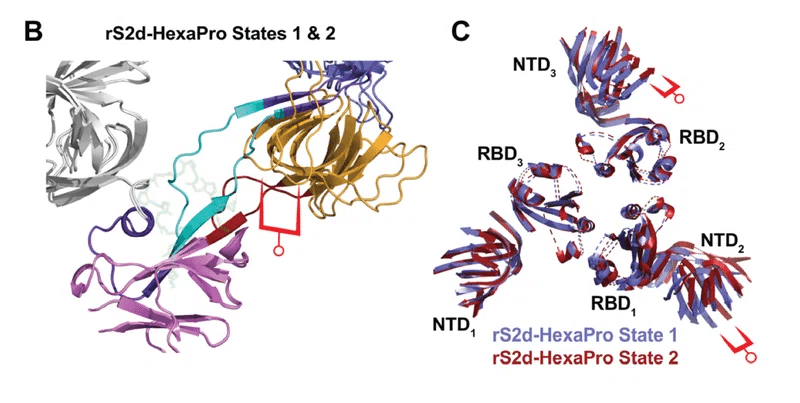
2つのサブドメインが近接しているため、2つをリンクしているbシートの二次構造が壊れており、ペアのN2R bシート構造が壊れています(図4B)。
この二次構造が失われると、リンカーと無秩序なセグメントで観察された再配列が可能になります。
次に、N2Rリンカーの再配置につながるrS2dスパイクドメイン配置の機能が、デルタおよびオミクロン3-RBDダウン状態で発生したかどうかを尋ねました。
N2Rで再配置されたプロトマーのSD2サブドメインの配置は、状態2のプロトマーを除いて、サブドメイン全体とNTDドメインの配置が類似していることを示しています(図4D)。

無秩序なRBD(Protomer1)の位置合わせにより、RBDの位置が三量体間で著しく異なることが明らかになりました(図4D)。
Sタンパク質ドメイン配置のベクターベースの定量化を用いて、スパイクドメイン内の絶対位置は異なっても、その全体構造の傾向は、RBDアップ状態を占める傾向などの重要な構造的特徴と相関していることを以前に発見しています。
SD2が配置されたrS2d状態で観測された最も重要な特徴は、SD1が隣接するSD2に向かって強制されることでした。
各プロトマーのサブドメインとNTDを結ぶ一連のベクターを調べたところ(図5A)、以下のことがわかりました。N2Rの再配列が観察されたSタンパク質三量体のそれぞれについて、角度や距離の値は若干異なるものの、相対的な位置の傾向は同じでした(図4E)。

具体的には、rS2dおよびrS2d-HexaPro状態2に対して、SD1の位置がSD2側にシフトしていることが保持されています。このことは、N2Rの再配列状態を引き起こす上で、これらのサブドメインのシフトが重要な役割を担っていることと一致します。
これらの局所的な効果は、乱れたRBDプロトマー(RBD2からNTD3)の向こう側にあるRBD-NTDペア間の距離の減少を伴っていました。このことは、RBDの位置にばらつきがあるにもかかわらず、この相互作用がN2Rの再配列に関与していることを示唆しています(図4E)。
N2Rの再配列三量体は似ているが、非配列の状態2三量体であるオミクロン三量体とrS2d三量体は顕著に異なっていました。
まとめると、rS2d構造と変異体構造の比較から、S1の局所的および全体的な再配列が再配列状態につながることが示されました。
Vector analysis of intra- and interprotomer domain relationships in the Omicron and Delta S proteins
オミクロンおよびデルタSタンパク質におけるプロトマー内およびプロトマー間のドメイン関係のベクター解析
次に、主成分分析(PCA)を使用して、プロトマー間およびプロトマー内ベクターのセットを利用した以前のバリアント構造とのオミクロンおよびデルタバリアント3-RBDダウン構造のクラスタリングを調べました(図5A)。

オミクロンバリアント構造の目視検査で示唆されているように、オミクロン三量体は、プロトマー間ベクターセットのPCAで別個のクラスターを形成します(図5B)。

デルタ構造は、主成分1に沿って設定されたバリアント構造全体に広がり、クライオEMデータセットのサブ分類によって回復されたかなりの構造変動と一致しています(図3)。
これらの結果は、Sタンパク質がSARS-CoV-2の進化を通じて獲得したかなりの構造的再配列を示しています(図5B)。
遺伝子操作されたSタンパク質とバリアントSタンパク質の間で再配置されたN2R状態が構造的に保存されていることは、これがS1ダイナミクスにおいて特定の役割を果たしていることを示唆しています。
特定のドメイン組織がこの状態の出現を伴うため、株全体に占める割合が低く、地図の密度が目立たないにもかかわらず、以前のバリアント構造がこの状態を示す可能性があるかどうかを尋ねました。
したがって、候補構造を特定するために、PCAによって設定されたプロトマー内ベクターを調べました(図5、AおよびC)。
プロトマー間ベクターと一致して、rS2dコンストラクトのN2R再配列プロトマーと、オミクロンおよびデルタバリアントは、主成分1に沿って異なる位置を占めていました(図5C)。
D614G、ミンク、ベータバリアントのプロトマーは、これらの構造でクラスター化されています。
これらの構造のN2R領域の密度は、rS2d状態1座標をフィッティングすることによって決定された再配置状態と一致していました。この座標では、このN2R再配置状態が特によく解決され、それぞれのクライオEM密度に適合しました(図5D)。

予想どおり、密度はrS2d構造よりも明確ではなく、マルチステートまたは動的な動作を示唆しています。
rS2d、オミクロン、デルタの構造で観察されたように、再配置されたN2Rプロトマーは無秩序なRBDを含み、最も遠いRBD-NTDペアに対応していました。
これらの結果は、N2R再配列状態の安定化を特徴とする、他のバリアントSタンパク質とのオミクロン3-RBDダウン構造の違いを浮き彫りにしています。
Antigenicity of the Omicron and Delta S proteins
オミクロンおよびデルタSタンパク質の抗原性
次に、オミクロンSタンパク質の抗原性と受容体結合特性を調べました。
オミクロン変異体で観察された広範な免疫回避と一致して、そのSタンパク質がいくつかのSARS-CoV-2中和抗体への結合を失ったことがわかりました(図6、AからC、図S10)。

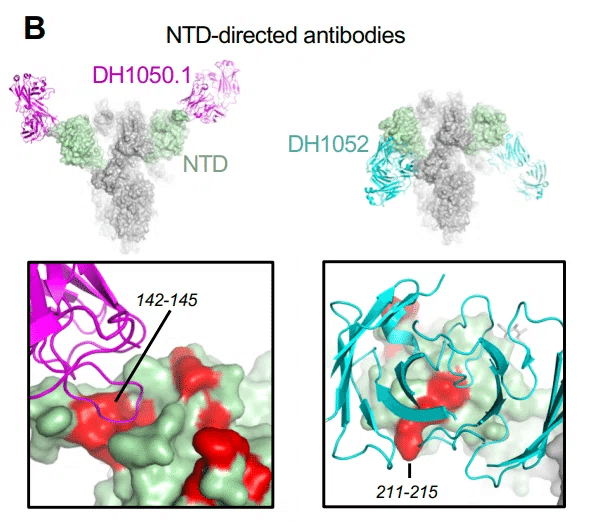

NTDの脆弱性のある部位を標的とするAbDH1050.1は、デルタおよびオミクロンSタンパク質に結合しなくなりました(図6、AおよびB)。
非中和性の保護AbDH1052は、デルタへの結合を保持しましたが、オミクロンSタンパク質への結合は保持しませんでした。その結合は、オミクロンSタンパク質の残基211〜215にまたがる領域の変化によって破壊された可能性があります(図6、AおよびB)。
抗体DH1041およびDH1042に向けられたRBD受容体結合部位は、オミクロンSタンパク質への結合を失いましたが、DH1042もデルタSタンパク質への結合を失いました。
オミクロンとデルタSの両方のタンパク質は、ACE2と同様の結合レベルを保持していました(図6、AおよびC、図S10)。
DH1041およびDH1042は、ACE2結合部位と重複する同様のRBDエピトープに結合します(図6C、図S11およびS12)。エピトープフットプリントの微妙な変化が、Absの残基置換に対する感受性プロファイルを変える可能性があることを示しています。
DH1042の場合、Delta Sタンパク質のL452R置換によって疎水性結合部位内に荷電残基を挿入すると(図S12)、結合が減少し、中和活性が失われます。
実際、DH1042は、S-GSAS-L452RおよびL452R置換を含むS-GSAS-イプシロンSタンパク質外部ドメイン(B.1.429)への結合が大幅に減少していることを示しました。
一方、DH1041バインディングは、L452R置換の影響を受けません(図S12)。
報告されているオミクロン中和の保持と一致して、レベルは低下していますが、操作された治療用Abソトロビマブの親型であるAb S309は、オミクロンスパイクへの実質的な結合を保持していました(図6Aおよび図S10)。
広範なサルベコウイルス中和抗体DH1047は、オミクロンスパイクへの実質的な結合を失い、その結果、オミクロンに対する中和活性が失われました。
S2X259 Abは同様のエピトープを標的としますが、オミクロンに対する結合および中和活性を保持しています。
SARS-CoV-2交差反応性AbCR3022は、スパイク上の不明瞭な変異していないエピトープを標的とし、S1サブユニットのダイナミクスにかなりの違いがあるにもかかわらず、S-GSAS-オミクロンおよびデルタスパイクへの結合を保持します。
ACE2結合をめぐって競合しない2つのRBD指向抗体、DH1044とDH1193は、オミクロンSタンパク質への結合を保持します。
DH1044エピトープは、NSEMによってAbS309のエピトープに隣接する領域にマッピングされました。ただし、残基L452にシフトしているため、デルタスパイクのL452R置換の影響を受けやすくなっています(図6、AおよびC)。
NSEMによるDH1193エピトープのマッピングにより、S309エピトープとDH1044エピトープの間にあるエピトープが明らかになりました(図S13)。
次に、S2ターゲティング抗体への結合を測定することにより、オミクロンS2サブユニットのコンフォメーションを調べました(図6Dおよび7A、図S14からS16)。
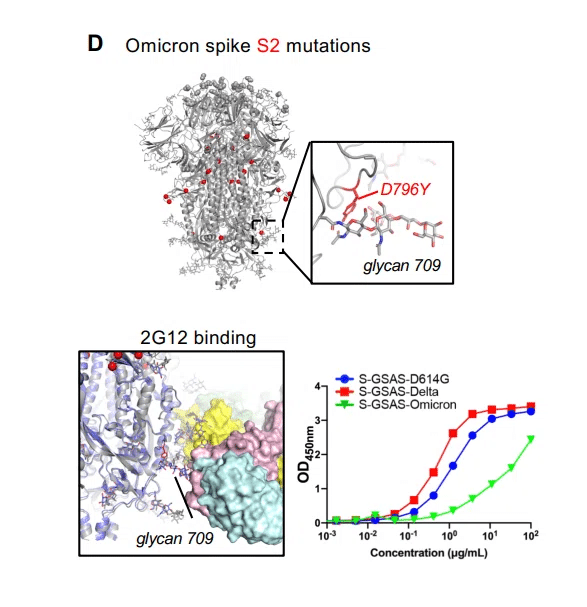
以前、HIV-1中和、Fab二量体化グリカン反応性(FDG)Ab 2G12の、SARS-CoV-2Sタンパク質のS2サブユニットの第4グリカンクラスターへの結合について説明しました。また、2G12の結合は、Sタンパク質のコンフォメーションの変化に敏感であることを示しています(図S14からS16)。
2G12と、同じグリカンクラスターをターゲットとするFDG Abのパネルは、デルタおよびオミクロンSタンパク質へのグリカン依存性結合を示しました(図6Dおよび図S16)。
2G12のオミクロンSタンパク質への結合は、G614およびデルタSタンパク質への結合よりも弱かった。S2コンフォメーションのグローバルな変化、またはD796Y置換によるグリカン709の安定化による局所的な変化のいずれかにより、グリカンクラスターの提示が変化し、グリカンエピトープの提示が変化したことを示唆しています(図6D)。
これらの結果は、かなりのオミクロンSタンパク質の構造変化と一致しており、S2のダイナミクスと柔軟性が後天的な変異の影響を受けることを示唆しています。
Altered fusion peptide dynamics in the Omicron S protein
オミクロンSタンパク質の融合ペプチドダイナミクスの変化
次に、G614、デルタ、オミクロンスパイクのSARS-CoV-2融合ペプチド(FP)に向けられたAb DH1058への結合をELISAでテストしました(図7A)。
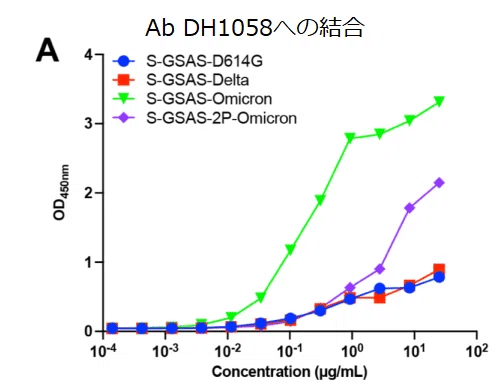
DH1058は、FPを含む残基808〜833にまたがる25残基のペプチドに結合し、G614およびDeltaSタンパク質と比較してSGSAS-オミクロンへの結合が約6倍増加することを示しました。オミクロンSタンパク質のFPへのDH1058のアクセスが多いことを示唆しています(図7A)。
SPRによって測定された結合速度と平衡定数(kon、koff、およびKD)は、テストされたバリアント間に違いがないことを明らかにしました(図7B)。

ELISAアッセイは、SPRアッセイで捕捉されるよりも遅いタイムスケールで結合を測定するため、ELISA実験でAbをスパイクと長時間インキュベートすると、DH1058エピトープのアクセス可能性または提示に時間依存性の変化が生じることが示唆されました。
DH1058の標的となるFP残基は、オミクロンSタンパク質のクライオEM再構成で十分に分解されました(図7C)。 他のバリアントSタンパク質と比較して、クライオEM再構成でより多くの残基が分離されています。

FPの全体的な向きは、D614G、アルファ、ベータ、デルタ、オミクロンSのタンパク質構造の間で保存されていました(図7C)。
フリンで切断されたスパイクと切断されていないスパイクに結合したDH1058のクライオEM構造を取得するためのいくつかの試みは成功しませんでしたが、2.15Åの解像度でFP残基808〜833を含むペプチドに結合したDH1058 Fabの結晶構造を取得しました(P1211空間群)(図7Dおよび表S2)。

DH1058とFPの間の相互作用は、すべての重鎖(HC)相補性決定領域(CDR)によって媒介されます。
残基816と825の間の融合ペプチドの部分は、HCDR2残基Tyr53、Glu54、Arg56、およびAsn57側鎖と水素結合(H結合)を形成するSer816およびAsp820側鎖との相互作用を定義します。
HCDR1 Asp31主鎖カルボニルはFPArg815側鎖とH結合を形成し、HCDR3 Tyr115とTyr116はそれぞれGlu819とLys825と側鎖間H結合を形成しました(図7D)。
重ね合わせのために結晶構造からのFPフラグメントを使用して、三量体の融合前のOmicron Sタンパク質の構造を整列させると、衝突が明らかになりました。SD2サブドメインとバインドされたDH1058HCループ13-17、61-68、84-88、およびS2サブユニットHR1サブドメインとDH1058 HCDR2の間です(図7E)。
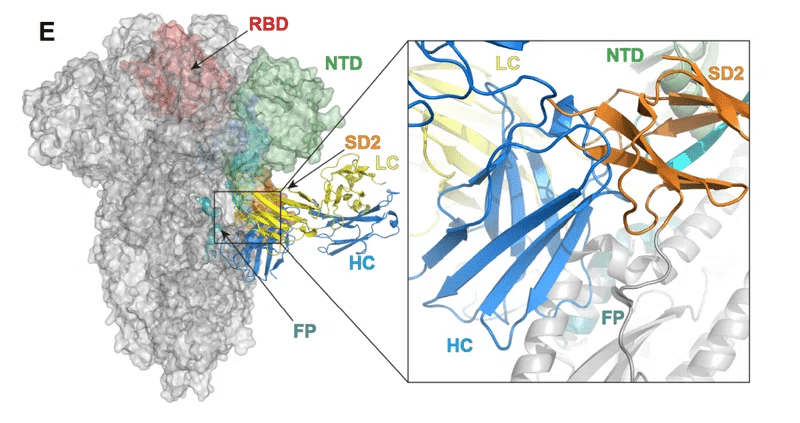
これらのデータは、結晶構造から明らかなように、DH1058のSARS-CoV-2 FPへの結合が、融合前のSARS-CoV-2Sタンパク質の構造と互換性がないことを示しています。
まとめると、これらのデータは、DH1058とFPの弱い初期接触が、SPRアッセイでキャプチャされた融合前のSタンパク質外部ドメインで行われることを示唆しています。その後、スパイクのコンフォメーション変化により、FP曝露が増加し、DH1058Fabが安定して結合します。
DH1058 Fabは、SARS-CoV-2Sタンパク質の融合前のコンフォメーションに安定して結合できません。SARS-CoV-2中和活性の欠如と一致しており、融合前のSタンパク質の隣接領域との衝突を回避する安定した相互作用を行うには、FPのより多くの曝露が必要になります。
ここでのデータは、FP曝露の増加につながるコンフォメーション変化が、オミクロンSタンパク質でより容易に発生することを示唆しています。
まとめると、これらの結果は、他の亜種と比較して、オミクロンスパイクのS2サブユニットの融合ペプチド領域周辺での柔軟性と曝露および放出の容易さの変化を示しています。
Discussion
SARS-CoV-2が進化し続けることで出現したオミクロン株は、前例のない伝染性と免疫回避により、COVID-19パンデミックの進路を変える準備ができています。
これらの特性を定義する中心となるオミクロンスパイクタンパク質は、受容体結合S1サブユニットと融合サブユニットS2の両方に変異があります。
Abと受容体の結合部位の露出は、結合界面での直接置換と重要な部位のコンフォメーションマスキングの両方の影響を受ける可能性があるため、ここでは、一次配列の変化に起因するオミクロンスパイクのコンフォメーション変化を理解しようと努めました。
構造研究は、S2サブユニットに外部の安定化変異を含まない以前に確立されたプラットフォームS-GSASで実行されたため、Sタンパク質のコンフォメーションをよりネイティブな形式で視覚化することを目的としています。
実際、S2サブユニットにプロリン変異を安定化させるコンストラクトを使用したいくつかの研究で明らかになったよりも、オミクロンSタンパク質の構造状態のさまざまなレパートリーを解決することができました。
オミクロンSタンパク質は、他のバリアントと比較して大幅に異なるドメイン構成を示しました(図1)。この違いは、構造内で視覚化し、プロトマー内およびプロトマー間ベクターのセットを使用して定量化することができました(図1、4、5)。
3-RBDダウン状態の密集したRBD-RBDインターフェースが見つかりました。1つのプロトマーにS371L、S373P、S375Fの置換があり、隣接する相互作用するプロトマーにY505Hの置換がある、RBDループによって媒介される新しいプロトマー間相互作用があります。
オミクロンでのダウン状態のRBDの密集は、以前のVOCの進化の仕方とは異なります。
以前のVOCは、スパイクの開放状態を優先することで伝達率を最大化し、一般的なAbエピトープを変異させることで免疫回避を最大化しましたが、RBDダウン状態を安定化させる変異の獲得は、別の方向への大きな変化です。
RBDダウン状態を安定させると、非常に強力な抗体に結合する免疫原性の高い部位が閉塞されるため、免疫回避が強化されます。
さらに、オミクロン株は、これまでに分離されたどの亜種よりも感染性が高くなっています。
オミクロンスパイクは、RBDダウン状態の安定性を強化しながら、どのようにして高い伝染性(受容体と結合して融合するためにスパイクの開いた状態が必要)を達成しますか?
これに対する答えは、構造分析の2つの側面にある可能性があります。
第一に、通常のモバイルRBDをロックダウンすると、プロトマーのNTDとRBDを接続する重要なペプチドN2Rが再配列され、準安定スパイクが発生することを示しました。3-RBDダウンスパイクの1つのプロトマーが、アップ状態に移行するように準備されます。
したがって、3-RBDダウン状態の安定化は、N2Rリンカーの再配置によるアップ状態を採用する傾向の強化によってバランスがとられています。
次に、結合アッセイ、X線結晶学、および低温電子顕微鏡法を組み合わせることにより、デルタを含む他のバリアントと比較して、オミクロンスパイクの融合ペプチドの可塑性が変化している証拠を明らかにしました。
オミクロンスパイクの広範な安定化にもかかわらず、機能的に重要なFPは、Abに向けられた融合ペプチドに結合することによって測定されるように、オミクロンでより簡単に露出されます。
したがって、FPのより準備が整ったリリースの効果と同様に、次の2つの複合効果によってオミクロンスパイクの伝達率の増加が促進される可能性があります。つまり、①ダウン状態のRBD-RBDインターフェースが安定しているにもかかわらず、RBDアップ状態に簡単にアクセスできること、②多数のRBD変異にもかかわらず、ACE2相互作用に対する親和性を保持できること。
オミクロンの病理生物学を深く理解し、さらに進化した変異体の免疫回避の可能性を予測するには、オミクロンテンプレート上の将来の変異体の構造の継続的な進化を綿密に監視する必要があります。
以下省略。
この記事が気に入ったらサポートをしてみませんか?
