JN.1・KP.2・KP.3のスパイク構造、立体配座アンサンブル、結合エネルギーの原子レベルでの予測(2024年7月)
Atomistic Prediction of Structures, Conformational Ensembles and Binding Energetics for the SARS-CoV-2 Spike JN.1, KP.2 and KP.3 Variants Using AlphaFold2 and Molecular Dynamics Simulations: Mutational Profiling and Binding Free Energy Analysis Reveal Epistatic Hotspots of the ACE2 Affinity and Immune Escape
SARS-CoV-2 スパイク JN.1、KP.2、KP.3 変異体の構造、立体配座アンサンブル、結合エネルギーの原子レベルでの予測 (AlphaFold2 と分子動力学シミュレーションを使用):変異プロファイリングと結合自由エネルギー分析により、ACE2 親和性と免疫回避のエピスタシス ホットスポットが明らかになる。

Abstract
BA.2 および BA.2.86 から派生した SARS-CoV-2 オミクロン変異体の最新の波は、機能的ホットスポットの収斂進化により、ウイルスの成長と適応度が向上しました。
これらのホットスポットは連携して機能し、効果的な感染と免疫回避効率の両方の受容体結合を最適化し、それによって全体的なウイルスの適応度を維持します。
最新の FLiRT および FLuQE 変異体と ACE2 受容体および抗体の構造、ダイナミクス、結合エネルギーに関する分子の詳細が不足しているため、この研究ではかなりの課題が検討されています。
我々は、最も優勢なオミクロン変異体 JN.1、KP.1、KP.2、KP.3 について、SARS-CoV-2 スパイク複合体の構造とコンフォメーション アンサンブルの AlphaFold2 ベースの原子予測と宿主受容体 ACE2 を組み合わせ、ACE2 結合と抗体回避のバランスをとる収束進化ホットスポットの役割の根底にあるメカニズムを調べました。
スパイク タンパク質残基のアンサンブル ベースの変異スキャンと結合親和性の計算を使用して、結合エネルギー ホットスポットを特定し、収束変異ホットスポット間のエピスタシス結合の根底にある分子基盤を特徴付けました。
結果は、L455、F456、Q493の位置にある収束変異部位間のエピスタシス相互作用の存在が、ACE2結合親和性を保護および回復しながら有益な免疫回避をもたらすことを示唆しました。
免疫回避メカニズムを調べるために、BA.2.86、JN.1、KP.2、KP.3 に対する中和が損なわれたいくつかのクラスの抗体とスパイクタンパク質の結合の構造ベースの変異プロファイリングを実行しました。
結果は、L455SおよびF456L変異を有するJN.1、KP.2、およびKP.3がクラス1モノクローナル抗体の中和活性を著しく損なう可能性がある一方で、F456Lによって媒介されるエピスタシス効果は、その後のQ493E変化の収束を促進してACE2結合を回復できるという実験データを裏付けました。
構造およびエネルギー分析により、異なる結合エピトープに結合するBD55-5840およびBD55-5514抗体が、検査されたすべての変異体BA.2.86、JN.1、KP.2、およびKP.3に対して中和効果を維持できることを示した実験結果の根拠が示されました。
結果は、オミクロン変異体の進化が、高いACE2親和性と免疫回避のバランスを制御するエピスタシス結合のメディエーターを含む変異の有益な組み合わせを持つ系統の出現を促進する可能性があるという考えを支持しています。
1. Introduction
SARS-CoV-2 のスパイク (S) 糖タンパク質は、ウイルスが宿主細胞に侵入する能力において極めて重要な役割を果たします。
S 糖タンパク質について実施された豊富な構造的および生化学的調査は、ウイルスの伝染と免疫回避を制御するメカニズムに関する重要な洞察を提供しました。
S 糖タンパク質の構造的柔軟性、特に S1 サブユニットとそのさまざまなドメイン (N 末端ドメイン (NTD)、受容体結合ドメイン (RBD)、および構造的に保存された 2 つのサブドメイン (SD1 と SD2) を含む) により、ウイルス侵入プロセスのさまざまな段階に適応できます。
NTDとRBDの構造変化によって引き起こされる閉鎖状態と開放状態の間の遷移により、ウイルスは構造的変動性を通じて免疫監視を回避しながら宿主細胞受容体と効果的に結合することができます。
これらの構造ダイナミクスを理解することは、ウイルスの侵入を阻止し、SARS-CoV-2 による感染を防ぐことを目的とした、S タンパク質を標的とした治療薬やワクチンを設計する上で非常に重要です。
生物物理学的調査により、熱力学的原理と運動学的因子がSタンパク質のメカニズムをどのように制御しているかが明らかにされています。
これらの研究により、Sタンパク質、特にS1サブユニット内の変異は、その安定性と立体構造のダイナミクス、特にウイルスが宿主細胞に付着するために重要なRBDへのアクセス性に影響を与える可能性のある、タンパク質の開状態と閉状態を切り替える能力に影響を与える構造変化を引き起こす可能性があることが明らかになりました。
さらに、動的なS1サブユニット(N末端ドメインやRBDなどのドメインを含む)とより剛性の高いS2サブユニット(膜融合に関与)との間の長距離相互作用は、Sタンパク質三量体の全体的な構造と機能状態を決定する上で重要な役割を果たします。
BQ.1.1やXBB.1のようなSARS-CoV-2変異体の出現と進化は、優れた成長上の利点や潜在的な免疫回避能力など、その独特の特徴により大きな関心を集めています。
XBB.1.5 亜変異体は、BA.2 系統内での組み換えイベントを通じて進化し、BA.2.10.1 および BA.2.75 亜系統の遺伝物質を組み込んでいます。
XBB.1.5 は XBB.1 と同様に免疫回避性がありますが、ACE2 結合性が高いため、成長に有利である可能性があります。 XBB.1.5 亜変異体の F486P は、好ましい疎水性接触のほとんどを回復できるためです。
さらなる調査により、XBB.1.5系統で観察された増殖の促進と伝染性の増加は、中和に対する耐性が維持され、ACE2受容体への結合親和性が向上したことに起因する可能性が高いことが実証されています。
2023年10月までに、F486P置換を特徴とするXBB.1.5やXBB.1.16などのXBB亜系統が世界中で普及しました(出典:https://nextstrain.org/)。
XBB.1.5と比較すると、XBB.1.16はNTDのE180Vと受容体RBDのT478Rの2つの置換を示しています。
これらの新たな変異体は、以前のオミクロン変異体と比較して感染性と伝染性が向上していることが実証されています。
さらに、RBD のいくつかの残基 (R346、K356、K444、V445、G446、N450、L452、N460、F486、F490、R493、および S494) は、少なくとも 5 つの異なる新しいオミクロン系統で変異しています。
EG.5 および EG.5.1 を含む XBB 子孫の系統は、追加の変異 F456L を持ち、現在、優勢な系統の 1 つになっています。
EG.5 はオミクロン XBB.1.9 から進化し、XBB.1.5 と比較して、1 つの追加置換 F456L のみを持ちます。
その直接の子孫である EG.5.1 は、NTD に Q52H、RBD に F456L を備えています。
EG.5 と EG.5.1 は、XBB.1.5 と比較して抗体中和に対して中程度の耐性を示すことが発見され、この耐性はクラス 1 モノクローナル抗体に対して特に顕著です。 主に RBD の単一の F456L 変異が原因です。
EG.5.1 亜変異体の免疫回避に関するさらなる研究により、観察された中和回避の強化は、Q52H 変異ではなく F456L 変異によって主に引き起こされていることが確認されました。
L455FとF456Lの反転置換の組み合わせを持つXBBサブバリアントは、しばしば「FLip」バリアントと呼ばれ、これらのバリアントは、2023年9月初旬に収集された世界中のXBBサンプルの20%以上で特定されました。
FLip 変異体には、JG.3 (XBB.1.9.2.5.1.3.3)、JF.1 (XBB.1.16.6.1)、GK.3 (XBB.1.5.70.3)、JD.1.1 があり、これらはすべて収束的に出現しました。
この収束は、L455F/F456L 二重変異を獲得すると、ヒト集団内で XBB の成長に有利になる可能性があることを強調しています。
BA.2 変異体から派生したオミクロン亜変異体 BA.2.86 は、以前の形態とは遺伝的に大きく異なっています (表 1、図 1)。

生物物理学的調査により、BA.2.86 変異体は、NTD、SD1 のエピトープ、および RBD 内のクラス 1、2、3 エピトープを標的とするモノクローナル抗体による中和に抵抗できることが確認されました。
注目すべきことに、BA.2.86は、XBB.1.5およびEG.5.1変異体で観察された免疫回避と比較して、RBD標的抗体を回避する可能性が高くなっています。
BA.2.86 亜変異体の免疫回避能力は、XBB.1.5 に有効な中和抗体パネルを使用して評価され、BA.2.86 は XBB.1.5 を標的とする抗体を回避できることが明らかになりました。
BA.2.86スパイクタンパク質とACE2および抗体の構造および結合特性を調査した最近の研究では、BA.2.86が獲得した変異は、XBB.1.5と比較して抗体回避に大きな変化をもたらさないことが示されています。
しかし、BA.2.86のRBDは、XBB.1.5と比較してACE2に対する親和性が2.2倍に増加しており、BA.2.86に伝播上の利点を与えています。
BA.2.86三量体Sタンパク質と複合体を形成したACE2の最新のクライオ電子顕微鏡構造は、BA.2.86の結合親和性の向上は、BA.2.86 RBDとACE2間の静電的相補性によってもたらされ、RBDの柔軟性によっても強化され、三量体構造内でACE2結合がよりよく露出される可能性があるという考えを裏付けました。
別の研究でも、BA.2.86 は、オミクロン XBB ファミリー亜種感染のいずれかによって誘発される中和免疫からの免疫回避能力が XBB.1.5 に比べて高くないが、ACE2 への結合親和性が高いためウイルスの適応度が向上することが示唆されています。
JN.1 は BA.2.86 の変異体で、Omicron BA.2 とは独立して出現し、免疫回避の強化に関与する追加の L455S 変異を抱えています。
BA.2.86/JN.1 は、XBB.1.5 および XBB.1.16 と遺伝的に遠いことも示しました (表 1、図 1)。
JN.1 変異体は、XBB.1.5 変異体とは抗原的に異なります。
表面プラズモン共鳴 (SPR) アッセイを使用した比較生化学分析では、JN.1 に対する ACE2 結合親和性が著しく低下していることが示され、免疫回避能力の強化は ACE2 結合の低下を犠牲にしていることが示されました。
JN.1変異体は、その前身であるBA.2.86と比較して、体液性免疫に対する抵抗性を高める1つの追加変異(L455S)があるだけであるにもかかわらず、ヨーロッパで急速に優勢となり、2024年初頭までにそれまで優勢であったXBB系統に打ち勝ちました。
インビトロACE2結合アッセイでは、JN.1 RBDの解離定数値がBA.2.86 RBDよりも有意に高いことが示され、L455S変異により結合親和性が低下する一方で、特にXBB.1.5ワクチンによって誘導される抗体に対して強力な免疫抵抗性を示すことが示されました。
JN.1 は、S2K146 や Omi-18 などの RBD クラス 1 抗体、およびクラス 3 抗体 S309 に対する回避能力が高まっていることが示され、他の関連研究の知見を裏付けています。
L455、F456、R346の収束ホットスポット部位に変異を持つ一連の変異体が出現し、その中にはJN.1変異(L455S)に加えてF456L変異も持つ「SLip」変異体も含まれています。
さらに最近では、SLipのバックボーンにR346T変異をさらに持つFLiRT変異体の出現がみられました。
最近の研究では、JN.1のF456L(SLip)およびR346T(FLiRT)サブバリアントが、JN.1由来の変異体が中和抗体からさらに逃れるのに寄与していることが明らかになりました。
2024年1月以降、JN.1はいくつかの亜系統に多様化し、その多くはR346T(JN.1.18)、F456L(JN.1.16)、T572I(JN.1.7)、またはこれらの変異の組み合わせ(KP.2変異体)を共有しています。
S:R346T、S:F456L、S:Q493E、V1104LなどのSタンパク質置換を収束的に獲得したKP.2(JN.1.11.1.2)およびKP.3(JN.1.11.1.3)を含むJN.1亜種が同時に出現しました。
KP.1.1 を含む他の FLiRT 変異体も米国で流行していることが確認されていますが、KP.2 や KP.3 ほど広まってはいません。
KP.2(R346T、F456L、V1104L)など、これらの反復変異を1つ以上持つJN.1亜種は、成長上の利点があり、2024年4月現在、複数の地域でより優勢になっているようです。
KP.2 変異体のウイルス学的特性を調べたところ、KP.2 の感染性は JN.1 よりも大幅に低く、KP.2 は JN.1 に比べて免疫抵抗能力が高まっていることがわかりました。
JN.1の別の子孫である「FLuQE」変異体(KP.3)は、「FLiRT」を支配し続けており、力強い成長を示しています。KP.3(JN.1.11.1.3)は、L455S、F456L、Q493E、およびV1104Lの変異を特徴としています。
KP.2およびKP.3の擬似ウイルスの感染性は、JN.1の感染性よりも大幅に低かった。
さらに、上記の置換に加えてS31欠失を収束的に獲得したLB.1やKP.2.3などのJN.1亜種が2024年6月現在出現し、拡散しており、免疫回避と相対有効再生産数の増加に寄与しています。
重要なのは、LB.1 と KP.2.3 は KP.2 よりも高い擬似ウイルス感染性とより強力な免疫抵抗性を示したことで、S31del が感染性の増加と免疫回避の強化に重要であることを示したことです。
KP.2 と KP.3 の両方の変異体は、クラス 1 RBD 抗体への結合を阻害することで抗体回避を強化するために重要な F456L 変異を共有しており、抗体回避がウイルス拡散の大きな選択的利点になり得ることを確認しています。
Cao 研究室の最近の調査結果と 6 月 4 日の非公開の通信 (https://twitter.com/yunlong_cao) によると、KP.3 変異体は、KP.2 よりも高い ACE2 結合親和性と、主に RBD 抗体のクラス I に対する免疫回避の強化を可能にする Q493E 変異により、KP.2 に打ち勝ち始めています。
さらに、KP.3 (JN.1+F456L+Q493E) は最も免疫回避性の高い変異体であり、最も急速に成長している JN.1 サブ系統でもあります。
追加の F456L および Q493E 変異により、KP.3 は JN.1 に有効な抗体、特にクラス 1 抗体のかなりの割合を回避できます。
ディープミューテーションスキャニング(DMS)実験と機能研究により、オミクロン変異体の進化の窓は、変異体変異間のエピスタシス相互作用を通じて強化される可能性があることが示唆されました。この相互作用では、1つの変異の影響が他の変異の存在に応じて変化する可能性があり、その結果、特定の機能に対する変異の非加法的影響が生じます。
これらの実験は、免疫逃避変異が個別にACE2結合を低下させる可能性があるが、親和性を高めるQ498RおよびN501Y変異とのエピスタシス結合によって補償されるという補償的エピスタシスの証拠を示しました。
最近の DMS 実験では、XBB.1.5 RBD のすべての変異変化と単一コドン欠失が ACE2 結合親和性と RBD 折り畳み効率に与える影響が調べられました。 R493Q 逆転変異と位置 Y453、L455、および F456 の変異間のエピスタシス相互作用を含む、RBD 残基間のエピスタシス結合の拡張された特性が明らかになりました。
また、BA.2.86までのオミクロン変異体の変異調査を通じて、Q493Eは典型的にはACE2結合に有害であり、結合親和性が最大10倍低下することが多いことが実証されました。
最近の通信で、Starr と同僚は、Q493E 変異を FliRT 変異体の L455S および F456L 変異と組み合わせると、結合親和性の喪失が逆転し、代わりに KP.3 に対する ACE2 結合が増加することを報告しました。
さらに、Starr 氏と同僚は、BA.2.86 バックグラウンドにおける L455S、F456L、Q493E 変異のすべての組み合わせに対するヒト ACE2 結合アッセイの結果を報告し、L455S+F456L バックグラウンドにおける Q493E の有害な影響が逆転したことを明らかにしました。
Cao 氏の研究室からの最新の報告では、KP.3 変異体の受容体 ACE2 結合能力の向上は、F456L 変異と Q493E 変異の大きな相乗効果によって可能になる可能性があることが示されました。
F456L(KD = 12 nM)およびR346T + F456L(KD = 11 nM)はJN.1(KD = 13 nM)のhACE2結合親和性に大きな影響を与えなかったが、KP.3のQ493E変異は受容体結合親和性を大幅に改善し、KD = 6.9 nMを示した。これは、これらのpre-BA.2.86変異体と比較して、Q493EとKP.3の他の変異、特にF456Lとの間に非相加的エピスタシス相互作用があることを示しています。
この洞察に満ちた研究は、エピスタシスによるKP.3の高い親和性により、KP.3 + A475Vに対するACE2結合親和性のわずかな低下(KDは22 nM)が観察されたことを考慮すると、さらなる免疫回避のためにA475V(この変異はJD.1.1、HK.3.14、JN.4、およびKP.2.3.1サブバリアントで収束的に観察される)が組み込まれる可能性があることも示しています。
この研究は、KP.3変異体のACE2結合親和性の高さが、この亜種とその子孫の急速な伝播と蔓延を促進し、免疫回避変異をさらに獲得する可能性を高めている可能性を示唆しました。
最近のいくつかの研究では、JN.1はBA.2.86と比較してACE2への親和性が低く、免疫回避特性が高いことが確認されており、FLiRT変異体を背景にQ493E変異を持つ新たな亜変異体はACE2結合親和性を回復し、強化された抗体回避プロファイルを示し続ける可能性があることが示唆されています。
コンピューターシミュレーションは、SARS-CoV-2 Sタンパク質のダイナミクスと機能を理解する上で重要な原子論的および機構的進歩をもたらしました。
一連の実験的および計算的研究により、SARS-CoV-2 Sタンパク質は、安定したアロステリックホットスポットによって制御され、特定の調節機能と結合機能を調節するアロステリック調節機構として機能できることが明らかになりました。
我々の最近の研究では、G446S、F486V、F486P、F486S、F490Sなどの収束性オミクロン変異は、主要な安定性および結合親和性のホットスポットとのエピスタシス結合を示す可能性があり、観察された広範な抗体耐性を可能にする可能性があることが実証されました。ACE2宿主受容体とのオミクロンSタンパク質複合体におけるコンフォメーションダイナミクス、結合、およびアロステリックコミュニケーションの分析は、結合親和性ホットスポットN501YおよびQ498Rを中心とするエピスタシス結合の領域を特徴付けました。
MDシミュレーションとマルコフ状態モデルは、XBB.1、XBB.1.5オミクロン変異体とその複合体の立体構造ランドスケープを体系的に特徴付け、収束変異部位が柔軟な適応領域における立体構造可塑性の調節を通じて進化アロステリックポケットを制御できることを示しました。
AlphaFold2ベースの構造モデリング手法は、全原子MDシミュレーションおよび結合エネルギーと安定性の変異プロファイリングと組み合わされ、SARS-CoV-2 Omicron BA.2.86スパイク変異体とACE2宿主受容体のダイナミクスと結合を予測しました。
この研究では、結合自由エネルギーの変化を調節するBA.2およびBA.2.86バックグラウンドの役割を定量化し、BA.2.86変異部位R403K、F486P、およびR493Qの重要な変異体固有の寄与を明らかにしました。
MDシミュレーションによる複数のコンフォメーションのAlphaFold予測により、XBB変異体のコンフォメーションランドスケープと結合エネルギーの重要な違いが特定され、L455FとF456L収束変異間のエピスタシス結合におけるQ493ホットスポットの媒介役割が明らかになりました。
XBBスパイク変異体とACE2およびクラス1抗体パネルの変異スキャンおよび結合解析の結果は、物理的に近接した結合ホットスポットY501、R498、Q493、L455F、およびF456L残基のエピスタシス相互作用が強力なACE2結合を決定し、収束変異部位F456LおよびF486Pが広範な抗体耐性を媒介するのに役立つという実験的証拠に定量的な根拠を提供した。
我々は統合計算アプローチを採用し、まず浅い多重配列アラインメント (MSA) アプローチを用いた AlphaFold2 (AF2) 法を使用して、JN.1、KP.2、KP.3 RBD-ACE2 複合体の構造と立体配座の集合を予測しました。
次に、分子力学/一般化ボーン表面積 (MM-GBSA) アプローチを使用して、Omicron RBD-ACE2 複合体の結合親和性を計算しました。
RBD残基の変異プロファイリングとMM-GBSAアプローチを組み合わせて、サブバリアントBA.2.86、JN.1、BA.2.86+F456L、BA.2.86+Q493E、BA.2.86+F456L/Q493E、BA.2.86+L455S/F456L(KP.1)、BA.2.86+L455S/Q493E、KP.2、およびBA.2.86+L455S/F456L/Q493E(KP.3)にわたるRBD-ACE2複合体の結合親和性を計算しました。
S 残基の変異プロファイリングを使用して、結合エネルギー ホットスポットを特定し、収束変異ホットスポット間のエピスタシス結合を定量化します。
我々は、出現した新しい変異体が、有益な表現型をもたらす L455、F456、Q493 位置での収束変異の組み合わせを許容することで、RBD の構造安定性が脆弱な領域での進化を促進できるエピスタシス パターンを誘発する可能性があるという仮説を検証します。
FLiRT および FLuQE 変異体の最新の機能研究と一致して、Q493E 変異は F456L 変異とエピスタシス的に結合し、他のバックグラウンドで見られる Q493E の有害な影響を逆転させる可能性があることがわかりました。
結果は、RBD-ACE2 複合体の AF2 予測コンフォメーション アンサンブルと RBD-ACE2 結合の MM-GBSA 計算を組み合わせることで、結合メカニズムの堅牢な定量分析を生成できることを示しました。
さらに、いくつかの重要なクラス I モノクローナル抗体を使用して S 複合体の変異スキャンを実施し、収束性オミクロン変異が免疫回避に与える影響と、BA.2.86、JN.1、KP.2、および KP.3 バリアントの ACE2 結合と免疫回避のバランスの基盤となるメカニズムを定量化しました。
研究対象となった抗体のパネルには、S2K146、Omi-3、Omi-18、Omi-42 などの RBD 標的抗体のほか、異なる RBD エピトープに結合し、実験的に BA.2.8、JN.1、KP.2、KP.3 バリアントのエスケープ変異を許容することが知られている抗体 BD55-5514 (SA55) および BD55-5840 (SA58) が含まれていました。
クラス 1 抗体であり、類似の RBD 結合ポーズを示す BD-508、BD-236、BD-629、BD-604、および BD-515 の構造解析により、抗体と RBD の間に多様な相互作用が示され、RBD 抗体が示す可能性のある高い多様性が示されました。
収束変異部位のグループである Y453、L455、および F456 は、クラス I 抗体に対する一般的なエスケープ ホットスポットを表していることを示しています。これらの位置のすべての変更(特に L455S と F456L)により、抗体との結合相互作用が劇的に失われるからです。
特定のクラスの抗体であるF2抗体とF3抗体はACE2と競合し、その結合は、BD55-5514(SA55としても知られる)を含むクラス1/4に対応するT376、K378、D405、R408、およびG504の影響を受けます。
ここでは、SA55 および SA58 抗体が収束変異部位を標的とすることを避け、免疫回避を克服できることを実証します。
結果は、収束オミクロン変異が RBD 安定性と立体構造適応性の相互作用を制御し、宿主受容体への結合と堅牢な免疫回避プロファイルの間の最適な適応トレードオフを可能にすることで、ウイルスの高い伝染性と抗原性を促進するメカニズムを示唆しています。
2. Materials and Methods
※省略します。
3. Results
3.1. Evolutionary and Phylogenetic Analysis of Differences between XBB, BA.2.86 and FLiRT Lineages
XBB、BA.2.86、FLiRT系統間の相違点の進化的および系統学的分析
オミクロン変異体間の XBB および BA.2.86 系統の進化的差異と分岐は、進化する病原体集団をリアルタイムで追跡するオープンソース プロジェクトである Nextstrain の対応する系統名を使用した系統解析によって示されます。
Nextstrain は、SARS-CoV-2 の系統樹を動的かつインタラクティブに視覚化して、ユーザーがさまざまな系統と変異体間の進化的関係を探索できるようにします。
このアプローチでは、SARS-CoV-2 変異体が、いつでも世界中で 20% の頻度に達した場合に、その変異体を系統群として割り当てます。
新しい系統群は、その親となる主要な系統群から少なくとも 2 つの変異が離れている必要があります。
Nextstrain の進化解析 (図 2、3) によると、XBB.1.5 (23A 系統) は XBB (22F 系統) の子孫であるため、組み換え変異体です。
XBB.1.5 には、XBB.1.5+F456L (EG.5) および XBB.1.5+L455F/F456L (XBB.1.5.70) でも共有されている追加の S 変異 S:G252V および S:S486P があります。


さまざまなオミクロン系統の進化分析により、BA.2.86 は、XBB.1 ベースの系統を超えてウイルスに世界的な成長の優位性を与えた段階的な進化を表していることが示されました (図 2、3)。
BA.2.86 S は、SARS-CoV-2 スパイク系統樹に新しい枝をもたらした、新しく独特な一連の変異を持っています (図 2、3)。
BA.2 祖先と比較すると、BA.2.86 には BA.2 と比較して 34 の変異 (置換 29、欠失 4、挿入 1) が含まれており、RBD 変異 I332V、D339H、K356T、R403K、V445H、G446S、N450D、L452W、N460K、N481K、delV483、A484K、F486P、R493Q が含まれます (図 1)。

G446S、N460K、F486P、R493Q などのこれらの変異の多くは、以前に他の変異体で観察されています。
BA.2.86 は、BA.2.86、BA.2.86.1、BA.2.86.2、BA.2.86.3、JQ.1、JN.3、JN.2、JN.1 の 8 つの異なる亜系統から構成されています。
JN.1 は BA.2.86 の主要な子孫系統です。JN.1 は Nextstrain 系統分類 24A を受けました (図 2、3)。
2024 年に JN.1 はいくつかの亜系統に多様化し、その多くは反復変異 R346T (JN.1.18)、F456L (JN.1.16)、T572I (JN.1.7)、またはこれらの変異の組み合わせ (KP.2 変異体) を共有しています。
これらの変異は、以前のオミクロン株の遺伝的背景で見つかった場合、強化された抗体回避 (R346T および F456L)、ACE2 結合 (R346T)、および S タンパク質の安定性と動的プロファイル (T572I) に個別に寄与する可能性があります。
進化図は、SARS-CoV-2 オミクロン XBB 系統と BA.2.86/JN.1 系統の明確な進化の軌跡を示しています (図 3)。
Nextstrain 図 (図 3) で示されている XBB 系統と BA.2.6、JN.1 系統間の現在の進化の分岐は、複雑な組み換え、抗原ドリフト、収束進化など、さまざまなメカニズムを通じてオミクロン系統の進化の軌跡が進む可能性があることを示しており、多くの系統が R346T、L455F/S、F456L、T572I などの変異を独立して獲得したため、収束免疫回避変異につながりました。
3.2 AF2-Based Modeling and Prediction of the BA.2.86, JN.1, KP.2 and KP.3 RBD-ACE2 Complexes and Conformational Ensembles
BA.2.86、JN.1、KP.2、KP.3 RBD-ACE2複合体およびコンフォメーションアンサンブルのAF2ベースのモデリングと予測
FLiRT および FLuQE 変異体 (主に JN.1、KP.2、KP.3 RBD と ACE2 受容体および抗体との結合) の構造、ダイナミクス、結合エネルギーに関する分子の詳細が不足しているため、実験データを合理化し、提案された分子メカニズムの原子論的基礎を確立するには、対処する必要がある大きな課題があります。
これらの重要な目的は、AI ベースの構造モデリング手法、MD シミュレーション、および ACE2 受容体とモノクローナル抗体のパネルを使用したJN.1、KP.2、KP.3 変異体のアンサンブルベースの変異プロファイリングを組み合わせて、現在の研究で対処されています。
まず、AF2 法を用いて、BA.2.86、JN.1、KP.2、KP.3 変異体の RBD-ACE2 複合体の構造とコンフォメーション アンサンブルの AF2 構造予測を、MSA 深度を変えて実行しました。
次の形式の 2 つの AF2 パラメータを使用することで、MSA からサブサンプリングされるシーケンスの数を操作できます (max_seqs は行/列アテンション トラックに渡されるシーケンスの数を設定し、extra_seqs はメインの evoformer スタックによって追加で処理されるシーケンスの数を設定します)。
一般的に、値が低いほど、より多様な予測が推奨され、MSA 深度適応によってコンフォメーション サンプリングが容易になる可能性があります。
BA.2.86、JN.1、KP.2、KP.3 バリアントの予測構造アンサンブルでは、すべてのバリアントで RBD のかなりの異質性が明らかになり、KP.2 と KP.3 では RBM 領域の可動性が徐々に増加していることが示されました (補足情報、図 S1)。
興味深いことに、BA.2.86 および JN.1 バリアントの場合、RBD ループ 444-452 の構造的変異は比較的中程度ですが、KP.2 および KP.3 バリアントではより拡散しています (補足情報、図 S1)。

RBD ループ領域 444-452 および 475-487 には、BA.2.86 変異 V445H、G446S、N450D、L452W、delV483、A484K、F486P が含まれています。
これらの領域は、JN.1、KP.2、および KP.3 バリアントで変異変化を受ける L455、F456、および Q493 位置の構造に近接しています。
生成された RBD 構造は、pLDDT メトリックを使用して評価されました。
pLDDT 値の分布は、調査したすべてのバリアントで pLDDT ~70-85 に顕著なピークを示しました (図 4)。

BA.2.86 変異体に対する構造アンサンブルの pLDDT 評価 (図 4A) では、アンサンブルの主要部分 (pLDDT 約 80-85) が実験構造と非常によく一致しているのに対し、アンサンブル サンプルの少数部分は、ネイティブ RBD 構造に関連しますが、RBD フレキシブル ループの可動性が向上した pLDDT 値約 60-80 を示しています (図 4A)。
他の pLDDT 分布は概ね同様でしたが、JN.1 および KP.2 変異体ではより浅いプロファイルが見られました (図 4B、C)。
KP.3 変異体では、さまざまな pLDDT 値でかなりの数の小さなピークが見られ、pLDDT 約 70-75 で主要な分布ピークが見られました (図 4D)。
pLDDT値が約70~90のAF2予測は通常、高い信頼性と関連しているが、pLDDT値が約50~70の領域は信頼性の低下を示している可能性があります。
全体的に、得られた立体構造集合は、JN.1、KP.2、特にKP.3 RBD変異体の不均一性の増加を示唆しており、これらの変異体がより可動性の高いRBD構造を利用して抗体中和を調節および回避できる可能性があります。
BA.2.86 RBD-ACE2複合体のクライオ電子顕微鏡構造に関して計算された構造類似性メトリックRMSDの分布はpLDDTプロファイルを反映し、BA.2.86(図5A)およびJN.1変異体(図5B)で約1〜2.5Åの低いRMSD値で強いピークを示した。

アンサンブルから機能的に関連するコンフォメーションを選択するために、RBD-ACE2 複合体の予測されたトップモデルは、pLDDT > 70 のコンフォメーションを選択することで、信頼度メトリック pLDDT のみに基づいて選択されました (図 6)。
pLDDT 値が高い構造は、RBD 可動性の機能的に関連する代表として考えられると考えられます。

AF2 モデルと、最近公開された祖先 BA.2.86 RBD-ACE2 複合体 (pdb id 8QSQ) のクライオ電子顕微鏡構造との構造アラインメントでは、RMSD < 0.8 - 1.0 Å が得られ、RBD ループに中程度の偏差がほとんど見られ、特に F486P および N481K 変異を伴う RBM 領域で顕著でした (図 6)。
予測されたコンフォメーション アンサンブルの構造アラインメント (図 6) では、柔軟な RBD ループの異質性は中程度である一方、RBD コアはほぼ固定されたままであることが明らかになりました。
さらに、pLDDT 値が高い AF2 予測アンサンブル コンフォメーションの構造解析では、RBD ループ、特に RBM 結合モチーフの可動性の程度は、すべてのバリアントでかなり類似していることがわかりました。
しかし、BA.2.86 変異体の直系の子孫である JN.1 は、RBD ループ 444-452 を含め、BA.2.86 クライオ電子顕微鏡構造 (図 6A) から非常に小さな偏差を示していることに気付きました。
これは、すべての予測されたコンフォメーションにおける変異位置のマッピングからも明らかであり、V445H、G446S、Q493、L455S、F456 サイトは、上位の予測されたコンフォメーションで実質的に同じ位置に留まっていることを示しています (図 6A)。
JN.1 RBD コンフォメーションのアラインメントにより、RBM モチーフの可塑性は中程度であることが明らかになり、これは F486P および N481K 位置のわずかな横方向の変位に反映されています (図 6A)。
KP.2 RBD 構造でも、V445H/G446S 部位に中程度の変動が見られ、RBM 領域の全体的な可動性にもかかわらず、N481K と F486P の予測位置はほぼ同じままという、概ね同様のパターンが観察されました (図 6B)。
興味深いことに、L455SとF456L周辺のRBDバックボーン構造は、側鎖のわずかな変動のみで、主に剛性のままです。
したがって、RBD 構造の機能的異質性はほぼ同様である可能性があり、ウイルスは収束変異部位を利用して ACE2 結合親和性を中程度に調整する一方、これらの変異の主な役割は免疫回避能力を高めることである可能性が高いことを示唆しています。
機能的 RBD 構造のより大きな構造異質性は、KP.3 RBD-ACE2 複合体で観察されました (図 6C)。
この場合、RBD ループ 444-452 および 475-487 が機能的に関連する可塑性を示す可能性があり、RBB 構造と F486P/N481K の位置はトップ構造間でわずかに異なる可能性があることがわかりました (図 6C)。
変異部位の大部分は、アンサンブルの異なる構造で同様の位置に留まりますが、収束変異 R346T、L455S、F456L、Q493E およびそれらの相互作用する ACE2 部位 K31、H34、E35 に関連する中程度の可塑性も見つかりました。
AF2 予測からのこれらの結果は、JN.1、KP.2、および KP.3 バリアントの L455S、F456L、および Q493E が、本来の位置の周囲で機能的に置換される可能性があることを示唆しています。
したがって、ACE2 との結合親和性のそれぞれの変化は、結合界面での協調的な構造再配置によって部分的に影響を受ける可能性があることを示唆しています。
3.3 MD Simulations and Ensemble-Based Mutational Profiling of the Biding Interface Residues in the JN.1, KP.2 and KP.3 RBD-ACE2 Complexes
JN.1、KP.2、KP.3 RBD-ACE2複合体における結合界面残基のMDシミュレーションとアンサンブルベースの変異プロファイリング
BA.2.86、JN.1、KP.2、KP.3 RBD-ACE2 複合体の全原子 MD シミュレーションを比較しました。
残基ベースの距離変動安定性指標の比較分析を使用して MD シミュレーションを分析します (補足情報、図 S2)。

分布から、すべての RBD-ACE2 複合体の極大値は、RBD コア内の構造的に安定で主に疎水性の領域 (残基 400-406、418-421、453-456) と、結合ホットスポット R498 および Y501 を含む主要な結合インターフェース クラスター (残基 495-505) に揃っていることが示されました。
高い距離変動安定性指標に関連する RBD 位置は、F400、I402、Y421、Y453、L455、F456、Y473、A475、および Y489 です (補足情報、図 S2)。
安定性ホットスポット Y449、Y473、および Y489 は、RBD の安定性と ACE2 宿主受容体との結合を維持するための要件によって制約されています。
プロファイルの最小値は、柔軟な RBD 領域 (残基 355-375、381-394、444-452、455-471、475-487) に関連しています。
すべての Omicron RBD バリアントの分布は同様の形状ですが、JN.1 分布ではより大きなピークが見られました (補足情報、図 S2)。
これは、RBD コアと ACE2 結合インターフェースの位置はどちらも JN.1 ではより硬く、KP.2 および特に KP.3 バリアントではやや柔軟になっていることを意味します。
プロファイルは、すべてのバリアントに共通し、低い安定性指数に関連付けられている柔軟な RBD 領域 (残基 355-375、381-394、444-452、455-471、475-487) を示しました。
特に興味深いのは、KP.2 および KP.3 の RBD 領域 440-460 および 470-487 で距離変動安定性指数が低下していることです (補足情報、図 S2)。
これらの観察結果は、KP.2/KP.3 RBD バリアントでは RBD が一般的に柔軟であることを示唆する AF2 予測と一致しています。
BA.2.86 変異部位を分布にマッピングすると、変異した RBD 位置は典型的には中程度の安定性指数で特徴付けられ、オミクロン変異が RBD 内の構造的に適応可能な領域をターゲットにしていることがわかりました。
変異位置 N440K、V445P、G446S、N460K、F486P は、低い距離変動安定性指数を示しており、これらの領域に局所的な可動性が存在することを示している可能性があります。
いくつかの重要な RBD 結合インターフェース センター L455 と F456 は中程度の安定性指数を示しており、これらの位置はすべてのバリアントで全体的に安定しているものの、L455S と F456L の変化を含む変異型では、これらのサイトが中程度の変動を受ける可能性があることを示しています (補足情報、図 S2)。
同時に、R498、Y501、H505 は高い安定性指数を示し、ACE2 との強い相互作用によりこれらの残基がかなり硬直化したことを反映している。
まず、JN.1、KP.2、KP.3 複合体の予測される結合界面を詳細に調べました。
BA.2.86 RBD-ACE2 複合体 (pdb id 8QSQ) のクライオ電子顕微鏡構造における結合界面は、H34 および K31 ACE2 残基、ならびに RBD 残基 Q493、L455、および F456 の位置が、XBB.1.5 RBD-ACE2 複合体のものと非常に類似していることを示しました (図 7A、補足情報、図 S3)。
XBB.1.5 RBD-ACE2 複合体 (pdb id 8WRL) では、K31 側鎖が Q493 と疎水性 L455 および F456 残基の間に配置されており、H34 および K31 の動きがかなり制限されていることを示しています (補足情報、図 S3A)。

XBB.1.5 +L455F変異体についても同様の界面パターンが予測され、Q493の好ましい位置はXBB.1.5と比較してほとんど変化しませんでした(補足情報、図S3B)。
XBB.1.5と比較して、F456L変異はRBD-ACE2界面の相互作用に実質的に影響しませんが、Q493とH34は、Q493とS494の間にH34側鎖を挿入できるコンフォメーションスペースの増加により、いくらかの再配置を受けます(補足情報、図S3C)。
同様の界面パターンは XBB.1.5 +FLip RBD-ACE2 複合体 (pdb id 8WRH) でもさらに強化され、L455 と F456 の反転により、H34 と Q493/S494 RBD 残基間の相互作用が相乗的に再編成されました (補足情報、図 S3D)。

L455 と F456 の位置付近で予測される結合界面の詳細を調べ、L455 と F456 の変異が L455F/F456L 変異によって誘発される構造再パッキングと結合接触の変化に与える影響を明らかにしました。
特に興味深いのは、KP.3 RBD-ACE2 複合体の結合界面の分析です。主に、Q493E 変異が、ACE2 結合親和性の観察されたエピスタシス改善につながる可能性のある潜在的な協調的構造変化を誘発できるかどうかを調べるためです。
Q493E は、すべてのヌクレオチド変異の中で最もまれな C->G を伴い、重要な残基で発生します。
興味深いことに、Q493E は、ほとんどの Omicron バリアントで RBD 界面領域 (438-506) がより塩基性、つまり正に帯電しているという基本的な傾向を逆転させることで、もう 1 つの大きな変化を示しています。
実際、BA.2.86 の V445H、N481K、および A484K 変異により、このバリアントは正に帯電した RBD サイトの最大の増加を示しました。
まったく予想外なことに、KP.3 変異体の Q493E はこの傾向を逆転させ、RBM をより酸性にします。
AF2 および MD シミュレーションから得られたコンフォメーション アンサンブルを活用して、KP.3 RBD-ACE2 複合体における最も好ましい界面相互作用をマッピングしました (図 7B)。
重要な変異位置 R403、L455、Y453、および Q493 を含む RBD の 2 番目の領域は、ACE2 インターフェース残基 (D30、K31、H34、E35、および D38) の α1 ヘリックス インターフェースの中央セグメントと結合します。
構造解析により、H34 および K31 ACE2 残基はどちらも、XBB.1.5 および BA.2.86 複合体と比較して複合体内でより柔軟になることが示されました (図 7B)。
さらに、H34 と K31 は、Q493Q との安定した水素結合相互作用を形成するための余地を増やす、中程度の構造再配置を可能にするいくつかの優先的な側鎖配向をとることができることがわかりました (図 7B)。
さらに、H34 側鎖の配置により、Y453 残基とのより強い接触が可能になることが観察されています。

追加の比較分析により、KP.3 RBD-ACE2複合体の予測される結合界面と、最近決定されたBA.4/5 RBD-ACE2複合体(pdb id 8H06)の構造との重ね合わせが示されました(図7C)。
興味深いことに、BA.4/5複合体のH34の側鎖は、KP.3複合体のH34の優先側鎖コンフォメーションと本質的に同一である2つの代替コンフォメーションを採用しています。
hACE2のH34は、BA.4/5 RBDのY453と水素結合を形成し、Q493はK31と水素結合しています。
同様に、KP.3複合体の予測されたK31側鎖の位置は、実験的に決定されたBA.4/5複合体のK31の向きと非常に似ています(図7C)。
したがって、アンサンブル予測による KP.3 複合体の結合界面の構造解析から、ACE2 界面部位の可塑性が中程度に増加していることが示唆されました。これは、L455S/F456L 変異によって疎水性パッキングのかさばりが軽減され、柔軟性が高まり、Q493E と H34 および K31 ホットスポットとの相互作用が改善される余地が生まれたことによるものです (図 7C)。
さらに、Q493 は RBD 残基 447-457、467-473、484-484、488-497 の構造的近傍にあることがわかりました。
特に、Q493E は残基 346 (FLiRT バリアントの R346T)、448-456、および BA.2.86 に ∆V483 と A484K がある 483-484 領域に近いです。
BA.2.86、JN.1、KP.2、KP.3 変異体の RBD-ACE2 複合体の MD シミュレーション研究から得られたコンフォメーション アンサンブルを使用して、RBD-ACE2 複合体の RBD 残基の系統的な変異スキャンを実行しました。
BA.2.86 RBD-ACE2 複合体のクライオ電子顕微鏡構造 (補足情報、図 S4) における主要な RBD 位置の変異スキャンは、収斂進化の部位に対応し、JN.1 (L455S)、KP.2 (R346T、L455S、F456L)、KP.3 (L455S、F456L、Q93E) に見られる変化を決定する RBD 位置に焦点を当てて分析されました。
変異のほとんどは、大きな不安定化結合自由エネルギー値を生み出しましたが、Y453F は例外で、変化は無視できるものでした (補足情報、図 S4A)。
これは、Y453F が適応変異としての役割を実証した実験研究と完全に一致しており、この変異は、ヒト ACE2 受容体の利用を損なうことなく、ミンク ACE2 受容体とのウイルスの相互作用を増加させました。

L455 での変異プロファイリングでは、すべての変更、特に L455S が ACE2 結合にかなり有害であることが示されました (ΔΔG 約 1.0 kcal/mol) (補足情報、図 S4B)。
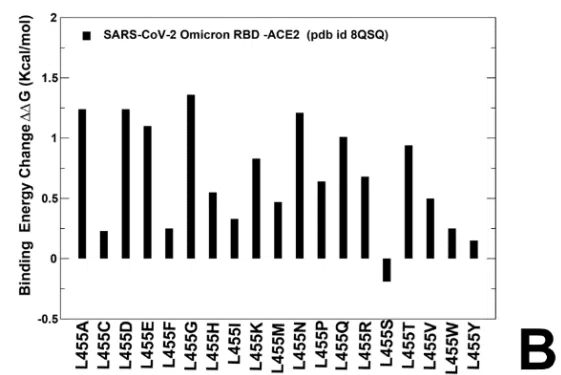
BA.2.86 RBD-ACE2 構造の F456 位置での変異変化は、一般的に非常に不利であり、ΔΔG 約 1.5-2.0 kcal/mol につながります。
ただし、BA.2.86 の遺伝的背景における F456L 変異変化のみはかなり小さく (ΔΔG 約 0.3 kcal/mol)、誤差範囲内であるため、F456L 単独でも ACE2 結合親和性を大幅に低下させることはできないことが示されています (補足情報、図 S4C)。

特に興味深いのは、Q493R と Q493E の比較的中程度の不安定化を示す Q493 位置の変異です (ΔΔG 約 0.6 kcal/mol) (補足情報、図 S4D)。
これらの結果は、XBB.1.5 変異体のバックグラウンドで Q493E を不安定化変異と予測した DMS 研究と一致しています。

私たちの結果は、F456L と Q493E はわずかに有害であり、ACE2 結合に対する効果を逆転させる可能性があることを示唆しています。
次に、AF2 および MD 立体構造アンサンブルを利用して、JN.1、KP.2、および KP.3 で変異している主要な RBD 位置のアンサンブルベースの変異スキャンを実行しました (図 8)。
JN.1 RBD-ACE2 アンサンブルにおける S455 の変異解析により、変異 S455F、S455L、および S455P は有利であり、S455L の逆転は結合親和性のいくらかの改善につながる可能性があることが示されました (図 8A)。
これにより、L455S 変異は ACE2 結合を著しく低下させる可能性があることがさらに確認され、これは実験データと一致しています。
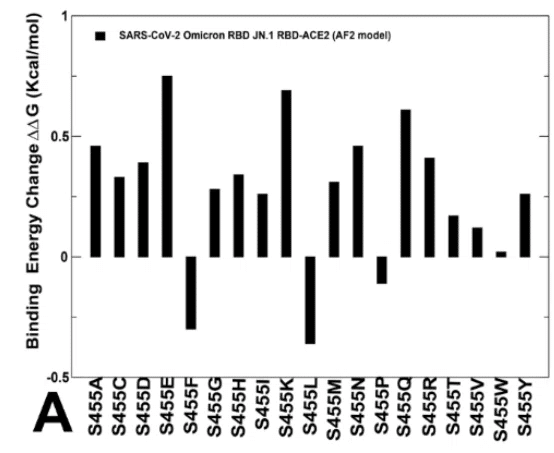
次に、KP.2 (R346T/L455S/F456L) KP.3 変異体の主要部位 (S455、L456、E493) における変異の影響をさらに詳しく調査しました。
結果は、S455F、S455L、S455P が依然として好ましいことを示し、これは L455S 置換が JN.1 と KP.3 の異なる遺伝的背景における ACE2 結合に有害であることを示唆しています (図 8B、D)。


さらに、KP.2 と KP.3 の両方の RBD-ACE2 複合体における L456 の位置は、すべての置換が ΔΔG を約 0.5-2.0 kcal/mol 不安定化させるため、非常に好ましいようです (図 8C、E)。


正の結合自由エネルギー値 ΔΔG は不安定化変化に対応し、負の結合自由エネルギー変化は安定化変化と関連しています。
KP3 RBD-ACE2 複合体における E493 の変異プロファイリングでは、いくつかの変更によって結合親和性が向上するため、より複雑で多様な画像が示されました (図 8F)。これは、E493 が一般に不安定化変化であるという事実と一致しています。
しかし、KP.3 のバックグラウンドでは、逆変異 E493Q はわずかに不安定化しているようでした。つまり、E493 は、KP.3 の L455S の変化、特に F456L と組み合わせると、わずかに有利になります (図 8F)。

以前の研究では、ACE2 の E37、E35、および D38 残基と R493 によって形成される BA.2 RBD の塩橋相互作用は、BA.2.86 RBD-ACE2 複合体では部分的に失われていることが示されました。
BA.2.86 RBD-ACE2複合体では、Q493はD30、K31、N33、H34、E35と安定した界面接触を形成し、これにはACE2のK31側鎖およびE35のカルボキシル基との水素結合相互作用が含まれます。
Q493E は、より柔軟な H34、E35、および K31 側鎖との相互作用を部分的に再配置できることを示しました。
特に、BA.2 の R493 と ACE2 の E35 によって形成される塩橋は、BA.2.75、BF.7、XBB.1、および BA.2.86 では Q493 に置き換えられています。
KP.3 アンサンブルの E493 の変異プロファイリングでは、多くの変化が安定化につながる可能性があることが示されましたが、観察された変化は小さく、多くの場合、小さな疎水性置換基を安定化させるものです (図 8F)。
全体的に、結果は、位置 493 は変更に対して非常に耐性があり、ACE2 結合と免疫回避のバランスをとるために、さまざまなオミクロン株で変異できることを示唆しています。
これらの結果は、S-Q493の小さな疎水性アミノ酸とS-N501の芳香族アミノ酸がACE2結合に非常に豊富であることを示す実験とも一致しており、これらの変化は局所的な疎水性パッキングを増加させると予想されます。
これはDMS実験とも一致しています。
ACE2結合を増加させると予測されるこれらの変異が、循環するSARS-CoV-2変異体では豊富に見られないという事実は、ウイルスの受容体に対する親和性がすでに高い伝染力を発揮するのに十分であることを示唆していると推測されました。
私たちの分析では、最新の KP.3 を含むさまざまなオミクロン株の R493/Q493/E493 が、RBD 上の 456 と 493 の位置にある主要なホットスポット間のエピスタシス結合を通じて進化上の利点を可能にする可能性のある相互作用の収束結合パターンを維持しながら、相互作用ネットワークを部分的に再構築できることも確認されました。
3.4. MM-GBSA Analysis of the Binding Affinities for the XBB RBD-ACE2 Complexes
XBB RBD-ACE2複合体の結合親和性のMM-GBSA分析
正確なSPR測定を用いた最近の実験研究によると、JN.1 RBD-ACE2複合体の結合親和性はKD = 13 nMで、BA.2.86変異体(KD = 1.7 nM)と比較して低下しており、これはJN.1の有害なL455S変化に起因します。
同時に、変異 F456L (KD = 12 nM) および R346T + F456L (KD = 11 nM) は、JN.1 の ACE2 結合親和性に大きな影響を与えることができず、L455S による JN.1 の ACE2 親和性の低下が F456L によって完全に補われなかったことを示しています。
この啓発的な実験研究の中心的な発見は、KP.3 の Q493E 変異により ACE2 結合親和性が著しく向上し、KD = 6.9 nM が明らかになったことです。これは、BA.2.86 変異体の優れた結合親和性よりわずかに低いだけです。
AF2 予測と MD シミュレーションから得られたコンフォメーション平衡アンサンブルを使用して、MM-GBSA 法を使用して RBD-ACE2 複合体の結合自由エネルギーを計算しました。
MM-GBSA 計算におけるノイズを減らし、エラーをキャンセルするために、複合体のみで MD シミュレーションを実行し、単一の軌跡からスナップショットを取得して各自由エネルギー成分を計算しました。
BA.2.86、JN.1 (BA.2.86+L455S)、BA.2.86+F456L、BA.2.86+Q493E、BA.2.86+L455S、BA.2.86+F456L/Q493E、KP.2 (BA.2.86+L455S/F456L/R346T)、および KP.3 (BA.2.86+L455S/F456L/Q493E) の RBD-ACE2 結合親和性を計算し、比較しました (図 9、表 2)。
MM-GBSA 計算の結果は、実験的な結合親和性との堅実な一致を示しました (図 9)。


結合自由エネルギーの総変化は、ΔG = -42.9 kcal/mol で、BA.2.86 RBD-ACE2 の結合親和性の方が良好であることを示しました (図 9A)。
実験と一致して、JN.1 の L455S は BA.2.86 と比較して ACE2 との結合を大幅に減少させることができ、ΔG = -39.3 kcal/mol を示していることがわかりました (図 9A)。

予想どおり、MM-GBSA 結合エネルギーの内訳 (表 2) は、BA.2.86 の好ましいファンデルワールス相互作用 (ΔG = -83.57 kcal/mol) が JN.1 のそれ (ΔG = -79.20 kcal/mol) よりも著しく好ましい重要な役割を担っていることを示しました。
この MM-GBSA 分析により、JN.1 の L455S が相互作用エネルギーを減らし、結合親和性を減らすことができることが確認されました。
BA.2.86+F456L 複合体のエネルギー分析により、F456L 単独では結合親和性にわずかな影響しか及ぼさず、全体的な ΔG = -41.54 kcal/mol (図 9A、表 2) となり、BA.2.86 RBD-ACE2 複合体と同等であることが明らかになりました。
これらの結果は、Starr による未発表の観察結果と一致しています。
MM-GBSA 計算によると、BA.2.86+Q493E、特に BA.2.86+Q493E/F456L 複合体の結合親和性は、結合自由エネルギーが徐々に ΔG = -42.42 kcal/mol および ΔG = - 44.04 kcal/mol に改善されるという、明らかで興味深い傾向を示しました (図 9、表 2)。
したがって、私たちの予測では、Q493E および F456L 変異の結合により、BA.2.86 変異体の結合親和性が完全に回復する可能性があることを示しています。
興味深いことに、エネルギー成分の MM-GBSA 分析では、Q493E 変異を持つ変異体では静電寄与が減少していることが示されましたが、この減少は極性溶媒和ペナルティの減少によって補われ、全体として結合エネルギーがより有利になりました。
包括的な MM-GBSA 分析も実験と一致しており、BA.2.86 L455S/F456L 変異体の結合親和性 (ΔG = -39.61 kcal/mol) は JN.1 (BA.2.86+L455S) の結合親和性と類似していることが示され、したがって F456L 変異だけでは L455S による結合の喪失を補うことができないことが示唆されています。
私たちの分析の重要な発見は、L455S、F456L、および Q493E を持つ KP.3 変異体が、F456L および Q493E 単独の個々の効果と比較して、大幅に改善された結合親和性 (ΔG = -43.78 kcal/mol) を示したことです (図 9A、表 2)。
これらの結果は、Q493EとKP.3の他の変異との間の非加法的エピスタシス相互作用が、KP.3の予想外に強化された親和性の背後にあるエネルギードライバーとなり得ることを示す最新の実験データと一致しています。
研究したすべての変異体における L455、F456、Q493 残基と対応する変異の寄与を評価するために、MM-GBSA 残基ベースの分解も報告しました (図 9B、C、D)。

L455S は、この変異を共有するすべての変異体で一貫して同様の結合相互作用の喪失を引き起こす可能性があることが分かりました (図 9B)。これは、L455S の有害な影響が F456L および Q493E 変異の存在によって影響を受けないことを示しています。
特に興味深いのは、456 の位置での MM-GBSA 分解分析です (図 9)。
F456L は BA.2.86+F456L で結合相互作用の喪失を引き起こす一方で、BA.2.86+F456L/Q493E および KP.3 (BA.2.86+L455S/F456L/Q493E) バリアントでは部分的に傾向を逆転させ、より好ましい寄与を回復できることに気付きました (図 9C)。
全体的に同様の傾向が Q493 位置の MM-GBSA 分解で見られ (図 9D)、KP.3 バリアントでは 493 位置の寄与のわずかな改善が見られました。
しかし、異なる変異体における Q493/E493 の寄与の違いは比較的小さい。
また、L455、F456、Q493 部位との相互作用に関与する個々の ACE2 位置 H34 および K31 の寄与も評価した (補足情報、図 S5)。
F456L/Q493E および L455S/F456L/Q493E を追加した BA.2.86 バリアントは、ファンデルワールス相互作用と静電相互作用の好ましい組み合わせにより、ACE2 上の K31 位置の寄与が強くなることがわかります (補足情報、図 S5A)。
BA.2.86 では Q493 は K31 と水素結合していますが、F456L/Q493E および L455S/F456L/Q493E バリアントでは、E493 と K31 が強力なイオン静電相互作用を形成し、ACE2 の Q493E と K31 の間にはより多くのファンデルワールス接触があります (補足情報、図 S5A)。

また、F456Lを共有する変異体では、H34の寄与がより有利になることもわかりました(補足情報、図S5B)。
さらに、H34の複数の側鎖コンフォメーションは複数の接触に関与し、Y453との好ましい相互作用を形成します。
JN.1、KP.2、KP.3変異体を含むBA.2.86の組み合わせの構造およびエネルギー分析を合わせると、F456LとQ493EがKP.3で協力してエピスタシス結合を誘導し、結合親和性をBA.2.86のレベルに回復させる可能性があることが示唆されました。
Q493EとF456Lの観察されたエピスタシス効果の背後にある正確な分子メカニズムは複雑である可能性が高いが、我々の結果は、KP.3複合体のRBDの柔軟性の向上により、構造アンサンブルで複数の側鎖コンフォメーションを採用しているように見える相互ACE2部位H34とK31の可塑性が高まる可能性があることが示唆されました。
H34 の側鎖は、BA.4/5 複合体でも見られる 2 つの代替コンフォメーションを採用し、Y453 と水素結合を形成することを発見しました。
JN.1、KP.2、KP.3 変異体の RBD-ACE2 複合体の予測アンサンブルの構造検査と MM-GBSA 分析により、L455S/F456L 変異によってもたらされる ACE2 界面部位の可塑性は、Q493E、Y453、F456L の位置によって適切に利用され、結合親和性を高めることができることが示唆されました。
全体として、この分析は、エピスタシス結合の潜在的な調節因子としての F456L の中心的な役割を指摘しましたが、KP.3 変異体における結合界面相互作用の改善をサポートする構造的に近接した L452 および Y453 部位の潜在的な役割も示唆しています。
3.5. Mutational Profiling of Protein Binding Interfaces with Distinct Classes of Antibodies
異なるクラスの抗体によるタンパク質結合界面の変異プロファイリング
我々は、RBDを標的としたさまざまなクラスの抗体と結合するSタンパク質の構造に基づく変異解析に着手し、特にBA.2.86変異が幅広いクラスの抗体に対する潜在的な耐性を媒介し、強力な免疫回避を誘発する役割に焦点を当てました。
我々は特に、BA.2.XBB.1.5およびBA.2.86変異体に対して活性を保持するが、JN.1、KP.2およびKP.3変異体に対しては中和効果が低下すると報告されているモノクローナル抗体のパネルを調べました。
Sタンパク質結合インターフェースの構造に基づく変異スキャンには、クラスI RBD標的抗体のパネルが含まれており、S2K146、Omi-3 Omi-18、Omi-42、および抗体BD55-5514(SA55)とBD55-5840(SA58)が含まれます。 これらは異なるRBDエピトープに結合し、実験的にBA.2.86、JN.1、KP.2、およびKP.3バリアントの回避変異を許容することが知られています。
結果は、広範囲に中和するモノクローナル抗体のパネルからの生化学的測定値と比較されました。
特に、実験では、JN.1のL455SがACE2結合親和性の低下を犠牲にして免疫回避を大幅に強化することが判明しました。
体系的な比較を行うために、S2K146(図10A)、OMI-3(図10B)、OMI-18(図10C)、およびOMI-42抗体(図10D)を含むS複合体のRBDインターフェース残基の変異ヒートマップを作成しました。

驚くべきことに、クラスI抗体を含むすべてのS複合体において、変異ヒートマップは、収束進化ホットスポットに対応する隣接する残基L455とF456のペアが、抗体中和の主要な回避ホットスポットでもあることを明らかに示しました。
これらの結果は、抗体の回避がL455F/SとF456Lの収束進化を駆動し、F456Lによって引き起こされるエピスタシスシフトがその後のL455とQ493の変化の収束を促進してACE2結合を回復することを示す実験データと一致しています。
さらに、変異ヒートマップは、JN.1、KP.2、および KP.3 バリアントに関与する他の重要な位置、特に Q493 部位が、すべてのクラス I 抗体における Q493 の変異による二次エスケープ ホットスポットとして考えられることを示しています (図 10)。
興味深いことに、S2K146 に直接結合する RBD 残基のほとんどは、ACE2 への結合にも関与しています。
実際、S2K146 に対する RBD 結合の変異ヒートマップでは、主要な界面位置のすべての置換が、S2K146 との結合親和性の一貫した大幅な低下を引き起こす可能性があることが特定されました (図 10A)。
これらの部位には、F456、F486、N487、Y489、F490、Q493があり、これらの部位の変異により、S2K146との結合親和性が最も低下しました(図10A)。
これらの知見は、SARS-CoV-2 RBD上のS2K146フットプリントがACE2受容体のフットプリントを模倣していることを示す、完全に一貫した構造研究です。 24個のエピトープ残基のうち18個がACE2結合部位と共有されており、これにはL455、F486、Q493、Q498、N501が含まれます。
変異ヒートマップ解析の重要な発見は、Y421、Y453、L455、F456の位置が、Omi-3、Omi-18、Omi-42クラスI抗体とのS結合における主要なエスケープホットスポットとして現れたことです(図10B-D)。
L455とF456の位置はRBDクラス1抗体のエピトープに位置しており、中和アッセイでは、L455S変異によりJN.1がクラス1抗体を回避できることが実証されました。
変異ヒートマップデータはこれらの実験と一致しており、L455Sを含むL455のすべての変更が抗体結合の大幅な低下を引き起こす可能性があることを示しています。
さらに、結果は、ACE2および抗体との好ましい相互作用に関与するY453の位置の重要性も強調しました。
これらのクラスI抗体のエスケープホットスポットの2番目のグループには、F486、N487、Y489、およびQ493の位置が含まれます(図10B-D)。
F486Pなどのこれらの部位の一部の変異は、BA.2.86変異体の主な免疫エスケープホットスポットとして関与しています。
最後に、ACE2 結合の模倣により、他のエスケープ位置は ACE2 結合親和性ホットスポット Y501 および H505 に対応します (図 10)。
重要なことに、この結果は、このクラス I 抗体の免疫エスケープの主な要因が、JN.1、KP.2、および KP.3 バリアントで変異を受ける L455 および F456 サイトに対応することを確実に示唆しています。 免疫エスケープの強化による進化を可能にします。
次に、BA.286 を遺伝的背景として JN.1、KP.2、および KP.3 変異を調べ、クラス I 抗体との S 結合の結合自由エネルギー変化を定量化しました (図 11)。

S2K146 との複合体における BA.2.86 変異に関連する結合自由エネルギー変化 (図 11A) は、K417N、L455、F456L、F486P、Q493E、および Q498R 変異による結合の顕著な損失を示しました。
興味深いことに、最も大きな不安定化変化は、L455S (ΔΔG = 1.03 kcal/mol)、F456L (ΔΔG = 1.55 kcal/mol)、および Q493E (ΔΔG = 1.59kcal/mol) によって誘発されました。
したがって、JN.1、KP.2、および KP.3 変異体に存在するこれらの重要な変異は、S2K146 からの免疫逃避を徐々に強化する可能性があり、これは実験と一致しています。
L455S および F456L 変異は、Omi-3、Omi-18、および Omi-42 抗体との抗体結合において非常に大きな損失を引き起こすことがわかりました (図 11B-D)。
特に、Omi-3 との結合では、L455S 変異により ΔΔG = 2.3 kcal/mol が発生し、F456L により ΔΔG = 1.76 kcal/mol が発生しました (図 11B)。一方、Omi-42 の場合、これらの損失は L455S で ΔΔG = 1.62 kcal/mol、F456 で ΔΔG = 1.53 kcal/mol でした (図 11D)。
これらの結果は、Omi-3 および Omi-18 抗体による JN.1 変異体に対する回避の増加を示す実験と一致しており、L455 および F456 サイトが主要なホットスポットとして現れ、変異 L455S および F456L が最大の親和性の低下を引き起こしました。
S2K146 および Omi-3 抗体の両方に共通して、F486P 変異による結合の大幅な低下も観察されました (図 11A、B)。
Omi-3 抗体に特有のものは、結合の低下 ΔΔG = 0.83 kcal/mol を引き起こす N460K 変異の不安定化の役割です (図 11B)。
興味深いことに、Q493E は Omi-3 および Omi-18 抗体との結合において 3 番目に重要なエスケープ変異として現れています (図 11B、C)。
BA.2.86 変異の結合エピトープ残基と部位の構造マッピング (図 11E-H) により、これらの抗体の結合モードが類似していることが強調されました。 R403、L455、Y453、および Q493 部位を含む RBD の 2 番目の領域をターゲットにします。
一般的に、結果は、F456L変異を有するKP.2およびKP.3が、単独またはQ493E(KP.3)との組み合わせで、BD-1854、BD57-1302、およびOmi-42などのRBDクラス1モノクローナル抗体の中和活性を著しく損なう可能性があるという実験的発見を裏付けました。
BD55-5514はACE2と競合するクラスF2およびF3抗体に属し、その結合はT376、K378、D405、R408、およびG504の影響を受けます。
BD55-5514 はオミクロン亜種に対して高い効力を示し、最近開発された BD55-5840 (別名 SA58、クラス 3) と BD55-5514 (別名 SA55、クラス 1/4) の非競合抗体カクテルはオミクロン亜種に対して高い効力を示しました。
Cao グループの最近の研究では、BD55-5514 (SA55) 抗体は、JN.1 を含む既知のオミクロン株のほとんどに対して中和効果を維持できることが示されています。
SA55 抗体は、ACE2 結合を犠牲にして他のクラス I 抗体の抗体回避を大幅に増加させることが多い HV.1 (L452R+F456L)、JD.1.1 (L455F/F456L+A475V) などの F456L 変異を共有する他の変異体に対しても、顕著な中和活性を示しました。
BD55-5514 と S タンパク質の相互作用の詳細な変異ヒートマップは、Omi-3、Omi-18、Omi-42 などのクラス I 抗体と比較して非常に異なる図を示しました (図 12A)。
マップは、S373P および S375F 変異が BD55-5514 との相互作用を促進できることを示しました。
T376、D405、および R408 は BD55-5514 との相互作用に関与していますが、これらはすべて BD55-5514 エピトープの周辺に位置しており、これらの位置での変異は結合親和性に中程度の影響を及ぼします (図 12A)。
重要なのは、BD55-5514 結合インターフェースは、L455、F456、Q493 などの BA.2.86、JN.1、KP.2/KP.3 のホットスポット位置を回避しないことです (図 12A)。

BD55-5514 抗体に対する JN.1/KP.3 変異のより詳細なプロファイリングでは、変異 T376A (ΔΔG = 0.81 kcal/mol)、R403K (ΔΔG = 0.65 kcal/mol)、D405N (ΔΔG = 0.79 kcal/mol)、R408S (ΔΔG = 0.34 kcal/mol)、L455S (ΔΔG = 0.7 kcal/mol)、および F456L (ΔΔG = 0.51 kcal/mol) によるわずかな不安定化の変化のみが示されました (図 12B)。
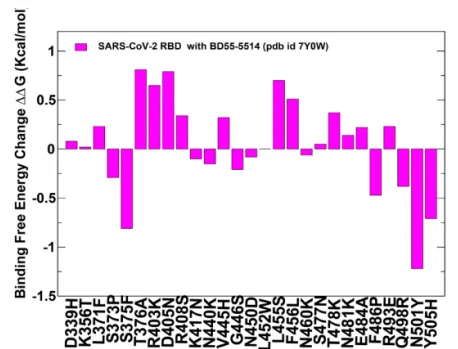
これらの変化は主にRBDの安定性と結合相互作用の軽度の低下を反映しており、これはBD55-5514などのグループF3抗体がBA.2のD405NおよびR408S変異に感受性がなく、この抗体がBA.2.86からKP.2およびKP.3までの最近の変異体の広範囲に対して有効であることを示す機能実験と一致しています。
BD55-5514 および BD55-5840 抗体の結合エピトープと BA.2.86 変異部位の構造マッピング (図 12 C、D) は、BD55-5514 が BD55-5840 とともに干渉なく RBD に結合する様子を示しています。

BD55-5514 と BD55-5840 の相互作用の変異解析により、S373P および S375F 変異がこれらの抗体と S タンパク質の結合親和性を改善できることが確認されました。これは擬似ウイルスデータとも一致しています。
また、BD55-5840 抗体の結合は、BD55-5514 と比較して、変異 T376A、R403K、および D405N による影響がさらに少ないこともわかりました (図 12E)。
重要なのは、L455S および F456L 変異はいずれも、ΔΔG が約 0.5~0.7 kcal/mol で、これらの抗体の結合にわずかに欠けているだけであることです (図 12A、E)。

その結果、これらの抗体は、BA.2.86、JN.1、KP.2、および KP.3 変異の全スペクトルに強力に結合することができました。
我々のデータにより、BD55-5840 と BD55-5514 が相乗的に作用し、RBD の異なる側に結合する複合体におけるこれらの抗体の結合相互作用の定量的なエネルギー分析が可能になりました (図 12C)。
興味深いことに、BD55-5514/BD55-5840 を含む RBD 複合体への BA.2.86 変異部位の構造マッピングにより、抗体の結合モードが変異位置と大幅に重複していないことが明らかになりました (図 12 C)。その結果、この抗体の組み合わせは、免疫逃避ホットスポットをうまく回避することで、並外れた中和活性を発揮できます。
まとめると、構造およびエネルギー分析により、BD55-5840 (SA58) + BD55-5514 (SA55) カクテルが顕著な中和幅と効力を示し、特に SA55 は検査したすべての変異体 BA.2.86、JN.1、KP.2、および KP.3 に対して中和効果を維持できることを示す実験結果の根拠が提供されます。
ごく最近の研究では、CYFN1006-1 および CYFN1006-2 抗体が発表され、試験されたすべての SARS-CoV-2 変異体に対して、SA55 と同等かそれ以上の一貫した中和効果を示しました。
CYFN1006-2は、すべてのSARS-CoV-2変異体に対して高い効力を示したが、KP.2に対する効力はわずかに低下しました。
これらの抗体は、RBDの外表面に位置し、SA55とは異なるRBD領域に結合するLY-CoV1404、REGN10987、S309と重複する結合エピトープを持っています。
したがって、SA55とCYFN1006-1を組み合わせたカクテルは、JN.1、KP.2、KP.3、およびSARS-CoV-2の進化する変異体に対して潜在的に有益である可能性があります。
4. Discussion
この研究の結果は、免疫回避、高いACE2親和性、十分な立体構造適応性の間のトレードオフを最適化する機能的にバランスのとれた置換がウイルス進化の共通戦略であり、新しいオミクロン株(BA.2.86、JN.1、KP.2、KP.3 バリアントを含むがこれらに限定しない)の出現の主な原動力となっている可能性があるという実験的証拠に分子論的根拠と裏付けを提供しました。
出現しつつあるオミクロン変異体、特に進化中の BA.2.86 亜系統に関する最近の研究では、オミクロン RBD 変異体と ACE2 の結合親和性を大幅に改善する進化の道筋は比較的狭く、十分な ACE2 親和性を維持しながら免疫回避を主に最適化するための収束的変異ホットスポットが関与している可能性があることが示唆されました。
収束適応に基づくこれらのメカニズムは、ACE2 結合親和性と安定性を損なうことなく免疫抵抗性を改善する新しい変異をウイルスが適応させるための「進化の機会」の範囲を決定する可能性があります。
私たちの多面的な研究では、BA.2.86、JN.1、KP.2/KP.3 変異体の構造、ダイナミクス、エネルギーの定量的側面を調査し、ACE2 結合およびさまざまなクラスの抗体との相互作用に関する大量の実験データと確実に一致していることを示しました。
調査の結果、L455、F456、Q493 の位置にある収束変異部位間のエピスタシス相互作用の存在が示唆されました。この相互作用により、ACE2 結合親和性を保護および回復しながら、有益な免疫回避を付与できます。
最新の機能研究と一致して、私たちの結果は、Q493E と F456L がエピスタシス結合を介して協力的に作用し、他の遺伝的背景に見られる個々の Q493E 変異の有害な影響を逆転できることを示しました。
Q493E と F456L の観察されたエピスタシス効果の根底にある正確な分子メカニズムは複雑である可能性が高いが、私たちの結果は、これらの部位間のエピスタシス相互作用は、RBD 上の Q493E と ACE2 上の H34 および K31 の相互作用する F456L の側鎖柔軟性の増加により、かなり限定された RBD-ACE2 インターフェース内で発生する可能性があることを示唆しています。
KP.2 および KP.3 複合体における RBD-ACE2 インターフェースの柔軟性が徐々に増加していることは、構造アンサンブルで見られる H34 および K31 の複数の側鎖コンフォメーション、および L455S および F456L 変異によってもたらされる結合インターフェースの強化された可塑性に表れています。
全体として、この分析は、F456Lがエピスタシス結合の潜在的な調節因子として中心的な役割を果たしていることを示唆しているが、KP.3変異体における結合界面相互作用の改善をサポートする構造的に近接したY453部位の潜在的な役割も示唆しています。
私たちの結果は、オミクロン変異体が明確なダイナミクスを誘導し、R346、F486P、Q498、Q493部位間のエピスタシス相互作用を利用して、抗体結合残基の柔軟性を高めながらRBD-ACE2界面を調節できることを示した関連研究と一致してます。
結果はまた、L455、F456、Q493がクラスI抗体に対する耐性の顕著なエスケープホットスポットであることを示した。
これらの部位間のエピスタシス結合は、ACE2結合親和性の回復に役立つだけでなく、個々の変異L455S、F456L、Q493Eによって提供される免疫応答を増幅するメカニズムを表す可能性もあると示唆しました。
これらの議論は、オミクロン変異体の既存の置換と選択実験中に獲得された抗体耐性変異との間に強いエピスタシスがあることを明らかにした進化研究とも一致しており、エピスタシスによって抗体回避の遺伝的障壁も低下する可能性があることを示唆しています。
計算結果と生化学実験の対応に基づいて、BA.2.86、JN.1、KP.2、および KP.3 変異の主な役割は、さまざまなクラスの RBD 抗体に対する広範な耐性を確保することである可能性があり、Q493E などのいくつかの重要な変異は、ACE2 結合親和性の向上をもたらす可能性があると示唆しています。
私たちの研究結果は、最近進化した変異体が、収斂進化を通じてかなり集中した抗体回避部位のグループ(L455、F456、R403、D405)を利用する傾向があるという、ブルームと同僚によって提案されたメカニズムに対する追加の証拠を提供します。これらの位置は中程度の機能的制約を受けており、突然変異はウイルスにほとんどコストをかけません。
免疫回避の効果は、より変異体に依存し、さまざまな適応可能なRBD領域での突然変異部位の採用によって調整される可能性がありますが、現在支配的な変異体は、限られた数の収斂回避ホットスポットで機能します。
同時に、収斂突然変異部位とACE2結合親和性ホットスポット間のエピスタシス相互作用は、最小限のリソースでACE2結合を維持し、免疫回避センターを活用してACE2親和性を保護する実行可能なメカニズムを表している可能性があります。
5. Conclusions
本研究では、最新の優勢なオミクロン変異体 JN.1、KP.1、KP.2、KP.3 について、SARS-CoV-2 スパイク複合体の構造とコンフォメーション アンサンブルの AF2 ベースの原子予測と宿主受容体 ACE2 を組み合わせて、ACE2 結合と抗体回避のバランスをとる収斂進化ホットスポットの役割の根底にあるメカニズムを調べました。
AF2 予測のコンフォメーション アンサンブルは、JN.1、KP.2、特に KP.3 RBD 変異体の不均一性の増加を示唆しており、これらの変異体がより可動性の高い RBD 構造を利用して抗体中和を調整および回避できる可能性があります。
結果は、AF2 アンサンブルの異なるコンフォメーションで変異部位の大部分が同様の位置に留まっているが、収束変異部位 R346T、L455S、F456L、Q493E およびそれらの相互作用する ACE2 部位 K31、H34、E35 は、ある程度の可塑性を示し、結合界面で協調的なコンフォメーション再配置を示す可能性があることを示しました。
BA.2.86、JN.1、KP.2、KP.3 変異体の RBD-ACE2 複合体の MD シミュレーション研究から得られたコンフォメーション アンサンブルを使用して、RBD-ACE2 複合体の RBD 残基の系統的な変異スキャンを実行しました。
結果は、F456L と Q493E はわずかに有害である可能性があり、異なる遺伝的背景では ACE2 結合に対するそれらの影響を逆転させる可能性があることを示唆しました。
KP.3 背景での変異プロファイリングにより、Q493E 変異は、KP.3 の L455S および特に F456L 変異と組み合わせると、より有利になることが示されました。
私たちの研究結果は、KP.2 および KP.3 系統が、L455S、F456L、Q493E、および F486P 変異の補償的エピスタシス効果を介して ACE2 との結合親和性のバランスを取りながら免疫抑制を改善することで、他の Omicron サブバリアントに打ち勝つように進化した可能性があるという最近提案された仮説を裏付けました。
JN.1、KP.2、KP.3 変異体の RBD-ACE2 複合体の立体配座アンサンブルの構造解析と MM-GBSA 計算に基づいて、L455S/F456L 変異によってもたらされる ACE2 界面部位の可塑性は、Q493E、Y453、F456L の位置によって適切に利用され、結合親和性を高めることができると主張します。
全体として、この分析は、F456L がエピスタシス結合の潜在的な調節因子として中心的な役割を果たしていることを指摘しましたが、KP.3 変異体における結合界面相互作用の改善をサポートする構造的に近接した L452 および Y453 部位の潜在的な役割も示唆しています。
さまざまなクラスの RBD 抗体による RBD 結合界面の構造ベースの変異スキャンにより、異なるエピトープ クラスに対する中和に対する広範な抵抗を誘発する特定の変異の役割が明らかになりました。
結果は、L455SS、F456L、Q493E 変異を持つ JN.1、KP.2、KP.3 変異体が RBD クラス 1 モノクローナル抗体の中和活性を著しく損なう可能性があることを示し、また、Y453、L455、F456 がこれらの変異体の主要な回避ホットスポットとして出現したことも明らかにしました。
これらの結果は、抗体回避が L455F/S と F456L の収束進化を促進し、F456L によって媒介されるエピスタシス相互作用が Q493E の好ましい寄与を促進して ACE2 結合を回復できることを示した実験データと一致しています。
この結果は、オミクロン変異体の進化が、ACE2 の高い親和性と免疫回避のバランスを制御するエピスタシス結合のメディエーターを含む変異の有益な組み合わせを持つ系統の出現に有利に働く可能性があるという考えを裏付けています。
私たちの研究は、収束的なオミクロン変異が、宿主受容体への結合と強力な免疫回避プロファイルの相互作用を制御することで、ウイルスの高い伝染性と抗原性を促進できるメカニズムを裏付けました。
これは、中和抗体を回避しながらも ACE2 結合親和性を維持する収束的な変異の組み合わせの探索につながる、オミクロン進化の継続的な共通戦略である可能性があります。
以下省略。
この記事が気に入ったらサポートをしてみませんか?
