BA.2 スパイクのクライオ電子顕微鏡構造(2022年6月)
Cryo-EM structures of SARS-CoV-2 Omicron BA.2 spike
SARS-CoV-2 オミクロン BA.2 スパイクのクライオ電子顕微鏡構造
元→Cryo-EM structures of SARS-CoV-2 Omicron BA.2 spike: Cell Reports
オミクロン BA.2 スパイク (S) タンパク質構造は BA.1 S と異なる
界面ループの突然変異誘発によるリモデリングでBA.2 RBD がより安定
リモデリングされたBA.2 RBD は、3-RBD ダウンS でより良好なインタープロトマーパッキングを示す
BA.2 S タンパク質の融合ペプチドはBA.1 よりも抗体にアクセスしにくい
Summary
重症急性呼吸器症候群コロナウイルス 2 (SARS-CoV-2) オミクロン BA.2 サブ系統は、BA.1 に比べて比例して増加しています。
スパイク (S) タンパク質の変異が病態生物学における違いの根底にある可能性があるため、ここでは BA.2 S エクトドメインのクライオ電子顕微鏡 (cryo-EM) 構造を決定し、これを以前に決定された BA.1 S 構造と比較します。
BA.2 受容体結合ドメイン (RBD) の変異は RBD 構造のリモデリングを誘発し、より密なパッキングと改善された熱安定性をもたらします。
閉じた(または 3-RBD ダウン)BA.2 S ではプロトマー間 RBD 相互作用が強化されていますが、融合ペプチドは BA.1 よりも抗体にアクセスしにくくなっています。
結合および擬似ウイルス中和アッセイでは、BA.1 と BA.2 の両方を中和する 2 つの外側 RBD 面結合抗体 DH1044 および DH1193 のエピトープを定義しながら、広範な免疫回避が明らかになりました。
まとめると、私たちの結果は、プロトマー間 RBD-RBD パッキングによる閉じた状態の安定化が Omicron バリアントの特徴であり、BA.1 および BA.2 S タンパク質の重要な機能領域に違いがあることを示しています。
Introduction
2021年11月に初めて検出された重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)オミクロンB.1.1.529(またはNextstrain 21M)変異株には、BA.1(B.1.1.529.1またはNextstrainクレード21K)、BA.2(B.1.1.529.2またはNextstrainクレード21L)、およびBA.3(B.1.1.529.3またはNextstrainクレード21M)を含むいくつかのサブ系統が含まれています(図1、データS1)。

BA.1は、オミクロンサブ系統の中で世界中に急速に広がった最初のものでした。
その後、報告された BA.2 配列の割合は BA.1 に比べて増加し、BA.2 は BA.1 を追い抜いて米国で支配的なコロナウイルス変異体となりました。
オミクロン変異体は、スパイク (S) タンパク質の変異の数が多いのが特徴です。
BA.1 と BA.2 は、それぞれ 13 と 8 の固有の変異がありますが、共通する S タンパク質変異は 20 個あります (D614G S と比較して)。
これらの違いは、宿主細胞への侵入、ウイルスの伝染、免疫認識などの S タンパク質を介した特性の違いの原因である可能性があります。
BA.1 および BA.2 S タンパク質は、N 末端ドメイン (NTD) が大きく異なり、2 つで共有されているのは G142D 置換のみです (図 1A)。
G142D 置換は、懸念される Delta 変異体 (VOC) のサブ系統でも発生し、免疫回避および高ウイルス量と関連しています。特に、BA.2 S タンパク質 NTD には、BA.1 や Alpha (B.1.1.7)、ミンク関連 (ΔFV) 変異体に存在する H69-V70 欠失 (ΔH69-V70) がありません。
BA.2 NTD には、残基 143~145 の欠失と、位置 214 の 3 つの残基の挿入もありません。
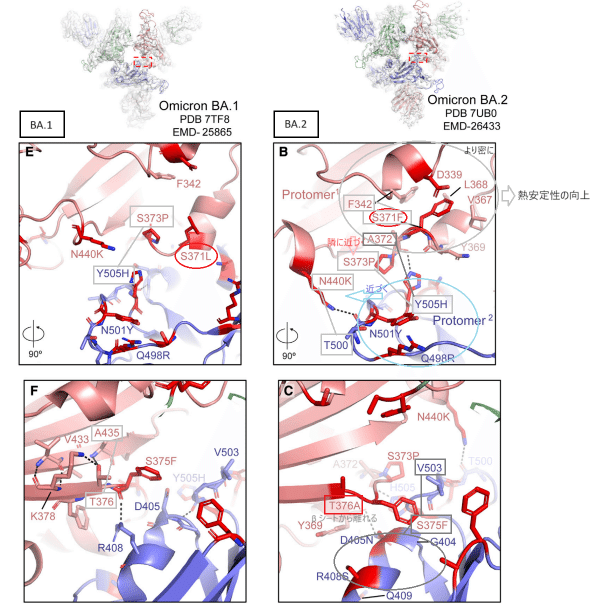
BA.1 と BA.2 の受容体結合ドメイン (RBD) は、12 の共通変異 (2 つ、S373P と S375F) により類似しています。 これらは、3-RBD-down BA.1 S タンパク質における RBD-RBD パッキングの媒介に以前関与していたとされる RBD ループで発生します。
この界面 RBD ループの一部である残基 S371 は、BA.1 ではロイシンに、BA.2 ではフェニルアラニンに変異しています。
Omicron BA.2 S タンパク質には、この界面ループ内に追加のアミノ酸置換 T376A があります。
BA.2 S タンパク質で発生し、BA.1 では発生しない RBD 変異は、T376A、D405N、および R408S です。一方、G446S および G496S は BA.1 で発生しますが、BA.2 S タンパク質では発生しません。
BA.2 S タンパク質には、BA.1 で発生する SD1 T457K および S2 N856K および L981F 置換がありません。
RBD および NTD 領域外の他のすべての変異は、2 つの間で保存されています (図 1A)。
私たちと他の研究者は、オミクロン BA.1 S の構造について説明しました。BA.1 と BA.2 S タンパク質の違いを理解するために、ここでは BA.2 S タンパク質のエクトドメインのクライオ電子顕微鏡 (クライオ EM) 構造を決定します。
BA.2 S クライオ電子顕微鏡データセットは 3-RBD ダウン集団が大部分を占めていましたが、RBD「アップ」集団も解析しました。
3-RBD ダウン状態の優位性は、RBD-RBD パッキングの改善によってもたらされました。
BA.1 S タンパク質で 3-RBD ダウン状態での RBD-RBD パッキングを促進する因子として以前に特定した S373P および S375F 残基置換を含む RBD 界面ループは、BA.2 S タンパク質に追加の残基置換を組み込んでおり、3-RBD ダウン構造の RBD 間の相互作用をさらに密にしました。
これらの追加変異により、各 RBD 内の内部パッキングも強化され、BA.1 RBD に比べてより安定したフォールドが実現しました。
BA.1 と BA.2 の S タンパク質の S2 サブユニットには違いがあり、その中には BA.2 の融合ペプチド (FP) が FP を標的とした抗体にアクセスしにくくなっていることなどが含まれています。
広範囲にわたる免疫回避が観察されましたが、2 つの抗体 (DH1044 と DH1193) が RBD の外面に結合し、擬似ウイルス中和アッセイで BA.1 と BA.2 を中和しました。
まとめると、私たちの結果は、BA.1 と比較した Omicron BA.2 S タンパク質構造の重要な違いを示しており、これが生物学的特性の違いの原因となっている可能性があります。
Results
Structural diversity and ACE2 binding of the SARS-CoV-2 Omicron BA.2 S protein
SARS-CoV-2 オミクロン BA.2 S タンパク質の構造多様性と ACE2 結合
我々は、以前説明した S-GSAS-D614G プラットフォームを使用して、BA.2 S 細胞外ドメインのクライオ電子顕微鏡構造を決定しました (図 1 および S1~S4、データ S2 および S3、表 S1)。
我々は、3 つの RBD が下向きの S 集団 (閉じた、O1BA.2、O2BA.2、および O3BA.2) と、少なくとも 1 つの RBD が上向きの位置にある S 集団 (開いた、O4BA.2、O5BA.2、および O6BA.2) を特定しました。これは、閉じた S と開いた S の比率がおよそ 3:1 で (図 1B)、BA.1 S で観察された約 2:1 の比率よりも高いものでした。

1-RBD-up 集団である O4BA.2 と O5BA.2 は主に up-RBD の位置が異なっていましたが、O6BA.2 は 1 つの RBD が up の位置にあり、2 番目の RBD は部分的に up でした。
3 つの BA.2 3-RBD-down S タンパク質構造のうち、O2BA.2 と O3BA.2 は 3 番目の O1BA.2 よりも対称性が高く、3 つのプロトマー間の RBD 配置に非対称性を示し、O2BA.2 と O3BA.2 は O1BA.2 構造よりも互いに類似していました (データ S3)。
Omicron BA.2 S エクトドメインは、RBD が大きく変異しているにもかかわらず (図 1C)、BA.1 S エクトドメインと同程度、D614G S よりも高いレベルで、ACE2 受容体エクトドメイン構造に強力に結合しており、多数の RBD 変異と 3-RBD ダウン状態の傾向の増加が、ELISA アッセイの条件下で Omicron BA.2 S が ACE2 に結合する能力を損なわないことを示しています (図 1D)。

Thermostability of the SARS-CoV-2 Omicron BA.2 spike and RBD
SARS-CoV-2オミクロンBA.2スパイクとRBDの熱安定性
温度の関数としてタンパク質の固有蛍光の変化を測定する示差走査蛍光測定法 (DSF) アッセイを使用して、D614G、BA.1、および BA.2 S タンパク質エクトドメインと対応するモノマー RBD 構造の熱安定性をテストしました (図 2 および S1、データ S4)。

野生型 (WT) RBD の変曲温度 (Ti) は 約 54.4°C でした (図 2B)。
BA.1 RBD は Ti が約 47.7°C と大幅に安定性が低かった。
WT RBD と比較して BA.1 RBD の安定性が低下したことは、公開されたレポートとほぼ一致しています。
Ti が約 50.4°C の BA.2 RBD の熱安定性は、WT と BA.1 RBD の中間でした。
D614G、BA.1、および BA.2 S タンパク質は、特徴的な DSF プロファイルを示しました。
我々は以前、SARS-CoV-2 SエクトドメインのDSFプロファイル、特に最初の変曲温度(Ti#1)がS安定性に敏感であることを示した。
我々は、BA.1 SエクトドメインのTi#1の大幅な減少を観察したが、BA.2 Sではそうではなかった(図2AおよびS1)。
まとめると、我々の結果は、BA.1と比較して、BA.2の単量体RBD(またはRBDのみ)構造とSエクトドメインの両方が安定化していることを示します。
Intra- and interprotomer RBD packing in the 3-RBD-down Omicron BA.2 S protein
3-RBD-down Omicron BA.2 Sタンパク質におけるプロトマー内およびプロトマー間RBDパッキング
3-RBD-down(またはclosed)SARS-CoV-2 Sタンパク質のCryo-EMマップは、通常、RBD密度にかなりの乱れを示しており、これは高い移動性を示しています。
Omicron BA.2 Sタンパク質の3-RBD-down構造の最も顕著な特徴は、密に詰まっていてよく解像されたRBDです(図1B)。
Omicron BA.1 Sタンパク質の3-RBD-down構造では、プロトマー間のRBDの密なパッキングを観察しましたが、BA.2 SではRBDがより密に詰まっており、これがさらに強化されているように見えました(図1B、1C、3、S2、およびS4)。
BA.1 S では、RBD-RBD 接触は、界面 RBD ループ内で発生した 3 つの獲得アミノ酸置換 S371L/S373P/S375F によって媒介され、S373P 置換によってループが再構築され (D614G S の構造と比較して)、隣接するプロトマーに Y505H 置換を持つ別の RBD 界面ループとの密接な相互作用が促進されました (図 3D~3F および S4)。


3-RBD-down BA.1 S タンパク質の RBD-RBD 界面で観察される S373P、S375F、および Y505H 置換は、BA.2 にも存在します (図 1A、3A~3C、および S4)。
BA.2 は、BA.1 の S371L の代わりに S371F 置換を持ち (図 1A、3B、および S4)、この界面 RBD ループには追加の T376A 置換があります (図 1A、3C、3F、および S4)。
BA.2 S371F 置換により、よりかさばった F371 側鎖と F342 のファンデルワールス相互作用が生じ、RBD 内の 2 つのらせん構造 339–342 と 367–371 がより密に詰め込まれることになります (図 3B)。
BA.2 S371F 置換により、よりかさばった F371 側鎖と F342 のファンデルワールス相互作用が生じ、RBD 内の 2 つのらせん構造 339–342 と 367–371 がより密に詰め込まれることになります (図 3B)。
BA.2 RBD 内の 367-371 らせんターンに対して F371 側鎖がパッキングされているため、残基 371 と 373 の間の領域の位置が反転し、残基 P373 が隣接するプロトマーの RBD に近づき、その H505 側鎖が P373 に対してスタックできるようになります (図 3B)。
さらに、これら 2 つの隣接する RBD 界面ループがより近接しているため、H505 側鎖は残基 A372 の主鎖カルボニルとプロトマー間水素結合を形成できます。
Y505H 置換を持つループには、BA.1 と BA.2 の両方に発生する 2 つの追加の VOC 変異、N501Y と Q498R も組み込まれています。
Y501 と R498 はループ内カチオン-π 相互作用に関与し、この領域に定義された構造を与え、BA.1 と BA.2 間で不変のままです (図 3B および 3E)。
このカチオン-π 安定化ループの内部構造は不変のままですが、残基 H505 を含む BA.2 のより密接なプロトマー間相互作用の結果として、残基 494~507 にまたがるこの領域全体が隣接するプロトマーに引き寄せられます。
2 つの界面 RBD ループのプロトマー間密接なパッキングの結果、K440 の側鎖 (BA.1 と BA.2 の両方で発生する N440K 置換による) は、隣接するプロトマー RBD の残基 T500 の主鎖と水素結合するように配置されます (図 3B および S4)。
この重要な 371-376 界面ループの別の構造変化は、BA.2 RBD の T376A 置換によって調整されます (図 3C および S4)。
BA.1 RBD では、残基 T376 は β ストランドの一部であり、その側鎖は隣接する β ストランドの A435 の主鎖と水素結合相互作用を行います (図 3F)。
BA.2 の T376A 置換による側鎖ヒドロキシルの喪失により、この水素結合が破壊され、A376 は β シートから離れるようになります。その際、S375F 置換の周囲の領域に沿って引っ張られるため、F375 は、隣接するプロトマーの G404-Q409 ヘリックスおよび V503 側鎖に重なるように伸びることができます (図 3C および S4)。
371~376 ループの再構築とともに、新しい相互作用を形成する鍵となる隣接プロトマー RBD にも協調的な変化が起こります。
BA.2 S R408S 置換により、残基 R408 側鎖が残基 375 の主鎖カルボニルと形成するプロトマー間水素結合が破壊されます (図 3C、3F、および S4)。
これにより F375 が解放され、隣接 RBD への移動が容易になります。
R408S 置換によるプロトマー間水素結合の喪失は、Y369 の主鎖カルボニルとのプロトマー間水素結合を媒介する BA.2 S D405N 置換によって補われる可能性があります (図 3C)。
まとめると、私たちの結果は、3-RBD ダウン BA.2 S では BA.1 S と比較してプロトマー間 RBD-RBD パッキングが強化されているという証拠を示しています。これは、界面 RBD ループを再構築して各 RBD 内、および閉じた S 内の RBD 間の密なパッキングを設計する残基置換によって調整されています。
Intra- and interprotomer communication in the Omicron BA.2 3-RBD-down spike
オミクロン BA.2 3-RBD-ダウンスパイクにおけるプロトマー内およびプロトマー間コミュニケーション
我々は以前、プロトマー内の NTD と RBD を連結する残基の配列が RBD のアップおよびダウン遷移の調節因子であることを認識していました (図 4)。
RBD ダウン プロトマーでは、この NTD から RBD (「N2R」) へのリンカーが、各サブドメインに β ストランドを提供することで、SD1 および SD2 サブユニットと相互作用します。
Omicron BA.1 S タンパク質 3-RBD ダウン構造 (O1BA.1 と命名、PDB: 7TF8) は、N2R リンカーの再配置を安定化し、このプロトマーが RBD アップ構成を採用する傾向にあると考えられます (図 4B)。
この N2R 再配置状態は、他のバリアントでも、程度は低いものの発見されており、この N2R 再配置状態は RBD のアップおよびダウン遷移の中間体である可能性が示唆されています。
Omicron BA.1 S クライオ電子顕微鏡データセットでは、この N2R 再配置を示さない別の 3-RBD ダウン集団 (O2BA.1 と命名、PDB: 7TL1) も特定しました (図 4B)。

Omicron BA.2 S 3-RBD ダウン構造の N2R 領域を調べたところ、Omicron BA.1 O1BA.1 構造で観察された N2R 再配置は示されておらず (図 4A および 4B)、各構造内の 3 つのプロトマーは N2R 領域でよく整列していました (図 4A)。

我々は以前、S タンパク質の全体的なドメイン構成を報告する一連のベクターを定義しました (図 4C)。

以前に説明したインタープロトマーベクターの主成分分析 (PCA) を使用して BA.2 S 3-RBD-down 構造を分析したところ、Omicron BA.2 S 3-RBD-down 構造は BA.1 S 3-RBD-down 構造の近くに集まり、この分析に含まれる他のバリアントとは分離されていることが示されました (図 4D)。

3 つの BA.2 3-RBD-down S 構造は、O2BA.1 構造に最も近い PCA 空間の領域に密接に集まっています。
これは、BA.1 および BA.2 S エクトドメイン構造の両方で観察された RBD-RBD パッキングと一致しています。
まとめると、私たちの構造研究は、獲得されたオミクロン BA.2 S 変異が、BA.1 S タンパク質と比較して 3-RBD ダウン状態のさらなる安定化をもたらし、RBD-RBD インターフェースの再構築を通じて、プロトマー間相互作用をさらに安定化させることを示しています。
Antigenicity of the Omicron BA.2 S protein
オミクロンBA.2Sタンパク質の抗原性
Omicron BA.2 S変異の抗原性影響を評価するために、S標的抗体の2つの異なるSタンパク質断片への結合をテストしました。単量体RBDのみの構造と、ここでおよび以前に発表された研究でクライオ電子顕微鏡構造解析に使用されたSタンパク質エクトドメイン(S-GSASプラットフォーム)です(図5、データS5)。
私たちは、2つの代表的なNTD標的抗体の結合をテストしました。NTDの抗原性スーパーサイトを標的とする中和抗体DH1050.1と、異なるNTDエピトープを認識し、in vitroアッセイでは中和しないが、動物モデルではSARS-CoV-2の感染から保護する抗体DH1052です(データS6)。
NTD を標的とする抗体は両方とも、BA.1 および BA.2 S 細胞外ドメインへの結合を失いました。

RBD を標的とする抗体をテストするために、受容体結合モチーフ (RBM) を標的とする抗体 (DH1041 および DH1042)、RBD 内面を標的とする抗体 (DH1047、S2X259、および CR3022)、および RBD 外面を標的とする抗体 (DH1044、DH1193、および S309) の代表的な例を選択しました (図 5A)。RBM を標的とする抗体 DH1041 および DH1042 は D614G S に結合しましたが、Omicron BA.1 および BA.2 S の両方への結合を失いました。これは、ACE2 結合リッジの周囲にエスケープ変異が蓄積していることと一致しています (図 5B)。

試験したRBD外面結合抗体のうち、S309はD614Gコンストラクトと比較してBA.2 Sへの結合が約40%低下し、BA.2に対する中和効果が失われたことと一致しています。ただし、最近の研究では、S309は中和活性を失ったものの、3つのSARS-CoV-2オミクロンサブ系統(BA.1、BA.1.1、およびBA.2)に対する保護効果は保持していると報告されてます。
我々は以前、外面RBDに結合し、Omicron BA.1に対する中和活性を保持する2つの抗体、DH1044とDH1193を報告しました。
両方の抗体は、BA.2 RBD のみおよび S タンパク質エクトドメイン構成体に対する nM 結合親和性を保持し (図 5C)、擬似ウイルス中和アッセイで Omicron BA.1、BA.2、および BA.3 を効果的に中和しました (図 S5)。


DH1044 Fab は、D614G、BA.1、および BA.2 S タンパク質および対応する RBD コンストラクトに、nM レベルで強く結合することが観察されました (図 5C)。
S コンストラクトへの DH1044 の結合プロファイルは、RBD のみのコンストラクトへの結合と同じ傾向を示し、D614G/WT および BA.2 S/RBD は同様のレベルで結合しましたが、BA.1 コンストラクトはより低いレベルの結合を示しました。
対照的に、DH1193 は、WT RBD コンストラクトよりも高いレベルで BA.2 RBD のみのコンストラクトに結合しましたが、BA.2 S エクトドメインは、D614G S に比べて大幅に低いレベルで DH1193 に結合しました (図 5C)。
類似した親和性にもかかわらず結合レベルが異なるのは、S タンパク質のコンテキストで発生し、RBD のみのコンテキストでは存在しないコンフォメーション効果によるものと考えられます。
DH1193 は RBD 上コンフォメーションに結合するため、BA.2 S が RBD 上構成を採用する傾向が低いため、BA.2 S への結合レベルが低下し、利用可能な結合部位が少なくなる可能性があります。
DH1193 は RBD 上コンフォメーションに結合するため、BA.2 S が RBD 上構成を採用する傾向が低いため、BA.2 S への結合レベルが低下し、利用可能な結合部位が少なくなる可能性があります。
Omicron BA.2 変異の最も劇的な抗原的影響の 1 つは、クラス 4 抗体の中和活性の消失です。
注目すべきことに、抗体 S2X259 は Omicron BA.1 に対して活性を保持しますが、BA.2 を中和することはできません。
S371F、T376A、D405N、R408S など、テストされたすべての BA.2 RBD ポイント変異のうち、S371F は擬似ウイルス中和アッセイで中和活性の最も劇的な低下をもたらしました。
BA.2 RBD に結合した S2X259 のモデル (図 6) は、BA.2 S タンパク質の 371–376 RBD ループの再構築により、結合した抗体との衝突が生じることを示しました。


これは、S2X259 の Omicron S タンパク質への結合に反映されており (図 5A)、BA.1 S タンパク質との結合は相当に維持されましたが、BA.2 S とは結合しませんでした。同様の結合傾向は、他のクラス 4 抗体 CR3022 および DH1047 でも観察されました (図 6)。
S2X259 と同様に、DH1047 および CR3022 のエピトープは、371~376 RBD ループの再構築によって破壊される可能性があります。
要約すると、これらの結果は、Omicron BA.2 S タンパク質で獲得された変異が、S のコンテキストでの RBD の上下遷移に関連する構造効果と、RBD 自体の構造変化によって、RBD 指向抗体の結合に影響を与えることを示しています。
Conformational changes in the S2 subunit of the Omicron BA.2 S protein
オミクロンBA.2Sタンパク質のS2サブユニットの構造変化
Omicron BA.1 および BA.2 S タンパク質は、2 つを除くすべての S2 サブユニット変異を共有しています;BA.1 の N865K および L981F 置換は、BA.2 S では発生しません (図 1A および 7A)。

S2 サブユニット内には、Fab 二量体化グリカン反応性 (FDG) 抗体に結合する 4 次グリカン クラスターがあります。
FDG 抗体 2G12 と DH851.3 は、Omicron BA.1 および BA.2 S タンパク質に同程度の結合レベルを示しました (図 6B)。

以前に観察された S2 サブユニット構造変化に対する FDG 抗体結合の感受性を考慮すると、これは、S2 残基位置 856 と 981 の違いが、融合前 BA.2 S S2 サブユニットの全体的な構造に大きな変化を引き起こさないことを示唆しています。
次に、BA.1 と BA.2 で異なる変異の周囲の局所領域を調べました。
BA.1 の N856K 置換により、K856 と T572 の側鎖を含むプロトマー間水素結合が導入されました (図 7C)。
BA.2 にこの変異がないと、BA.1 と比較してこの領域で局所的な不安定化が生じる可能性があります。

BA.1 S タンパク質の L981F 変異は、ヘプタッド リピート 1 (HR1) と中央ヘリックス (CH) の接合部にある残基 K986 と V987 に近い構造的に重要な領域で発生します。この領域では、一般的に使用される「2P」変異である 2 つの連続したプロリンを改変することで、融合前構造から融合後構造への移行がブロックされます。
オミクロン BA.1 構造では、F981 が 3 つのプロトマー間ヘリックス間のポケットに挿入され、その際に残基 E748、L752、L977、I980、A989、I993、および I997 を含む広範なファンデルワールス相互作用ネットワークが構築されます (図 7D)。
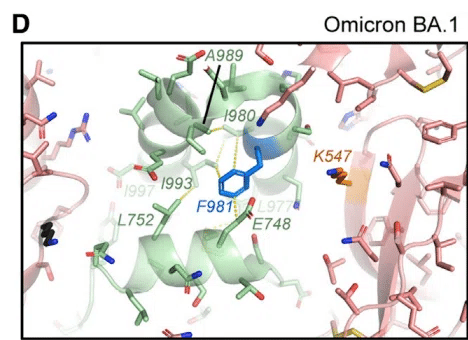
興味深いことに、BA.1 の唯一の T547K SD1 サブドメイン変異 (BA.2 には存在しない) は、残基 F981 を含むヘリックスセグメントの隣にあり、K547 側鎖がこのヘリックスに対して積み重ねられています。
Omicron BA.2 S のかさばらない L981 は、BA.1 S タンパク質の F981 のようにファンデルワールス ネットワークを媒介できません (図 7E)。

この変異の周囲の局所領域に変化があるかどうかを調べたところ、Omicron BA.1 S の CH ヘリックスが、D614G および BA.2 S タンパク質のこれらのヘリックスの位置と比較して、中央の三量体軸に向かって引っ張られていることがわかりました (図 7F)。

このシフトの程度を定量化するために、残基 995 の Cα 間の距離を測定したところ (図 7G)、D614G S ヘリックスが最も離れており、995 の Cα 間の距離は 12.6 Å であることがわかりました。
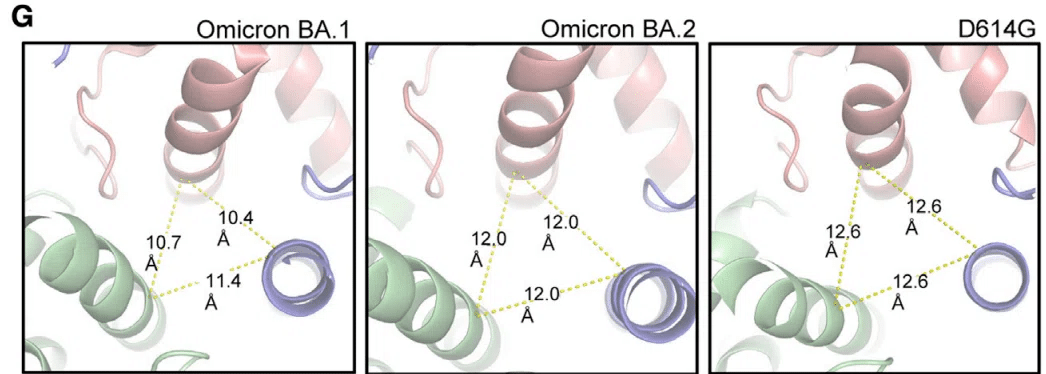
Omicron BA.2 ヘリックスは残基 995 の Cα 原子間の距離が 12 Å とわずかに接近していますが、Omicron BA.1 ヘリックスは平均で約 2 Å 近く、距離は 10.4 Å、10.7 Å、11.4 Å とより非対称性を示しています。
前述のように、これらの局所的な変化は構造全体に浸透せず、FDG抗体によるグリカンパッチの立体構造に敏感な認識は変化しませんでした(図7Aおよび7B)。
次に、FP を標的とする抗体 DH1058 と DH1294 を使用して、FP 領域の立体構造を調べました (図 7H~7K および S6、データ S7)。


両方の抗体は、ELISA によって SARS-CoV-2 FP 領域にまたがる 25 アミノ酸ペプチドにマッピングされました (図 7H)。
DH1058 と同様に、DH1294 はさまざまな CoV S タンパク質に結合しました (図 S6、データ S7)。
これらの多様な S タンパク質の FP 配列を分析すると、FP に結合した DH1058 の結晶構造 (PDB: 7TOW) で DH1058 抗体の接触として特定された残基が著しく保存されていることが明らかになりました (図 7J)。

以前、ELISA による DH1058 結合に対する Omicron BA.1 FP (D614G および Delta バリアント S タンパク質の FP と比較して) のアクセス性の向上を観察しました。
私たちは同様に Omicron BA.1 S の DH1294 への結合性の向上を観察しましたが、DH1058 と DH1294 はどちらも Omicron BA.2 S に非常に低いレベルで結合しました (図 7I)。これは、BA.2 FP が BA.1 FP と比較して FP 指向抗体にアクセスしにくいことを示唆しています。

免疫沈降アッセイでは、FP 指向性抗体を SARS-CoV-2 S 細胞外ドメイン構造物とインキュベートすると、時間依存的に結合が強化されることが示されました。 Omicron BA.1 は最も高い結合レベルを示しました (図 7K、データ S7)。

要約すると、構造分析と結合データから、Omicron BA.1 と BA.2 S タンパク質間の S2 サブユニットの違いが明らかになりました。これには、重要な HR1-CH 領域の構成の違いや FP アクセシビリティの変化が含まれます。
Discussion
SARS-CoV-2 オミクロン変異体の出現は、その高度に変異した S タンパク質のために広範囲にわたる警戒を引き起こしました。
オミクロン変異体の急速な拡散は、当初は BA.1 サブ系統によって支配されていました (データ S1)。
やがて、2 番目のオミクロン サブ系統である BA.2 が拡散し始め、現在ではいくつかの場所で BA.1 を追い越しています。
S タンパク質は変異体の拡散に影響を与えるいくつかの特性において中心的な役割を果たすため、ここでは BA.2 S の構造を決定し、以前に決定したオミクロン BA.1 S タンパク質の構造と比較し、系統学的に関連するこれら 2 つのサブ系統の S タンパク質主導の特性の類似点と相違点を構造に基づいて理解することを目指しました。
オミクロン変異体の統一的な構造的特徴は、受容体結合部位と多くの免疫優勢エピトープにアクセスできない 3-RBD ダウン (または閉じた) S での RBD の密集です。
私たちは以前、3 つの変異 (S371L、S373P、および S375F) を含む BA.1 S タンパク質の界面 RBD ループが、閉じたオミクロン BA.1 S タンパク質でのプロトマー間 RBD-RBD パッキングを媒介することを認識していました。
BA.2 S タンパク質は、この界面 RBD ループに戦略的に配置された追加の変異を組み込んでおり、この領域の再構築につながり、RBD コアの内部パッキングの改善と 3-RBD-down S の RBD-RBD 界面のパッキングの改善の両方を促進します。
BA.3、BA.4、および BA.5 オミクロン変異体サブ系統の S タンパク質も同様に密集した 3-RBD ダウン状態になると思われます。これらはすべて BA.2 の重要な S371F、S373P、および S375F 変異を含んでいるからです。
さらに、BA.4 および BA.5 S タンパク質には、BA.2 S で発生する T376A 置換も含まれています (BA.1 および BA.3 では発生しません)。
閉鎖状態の安定化が進むと、S タンパク質の免疫原性が低下する可能性があり、他の変異体に感染したことのない未接種の個人に対するオミクロン感染からの保護効果が低下する可能性があります。
同じ理由で、オミクロン S に基づくワクチンは、免疫原性 RBD 部位の大部分が閉塞されるため、免疫原性が低下する可能性があります。
この変異誘発による RBD 内および RBD 間構造のリモデリングは、BA.2 で観察される免疫回避の強化、特にクラス 4 RBD 指向抗体の活性低下に寄与しており、RBD 変異によって引き起こされる局所構造効果と S 構造効果の組み合わせが、観察される活性の劇的な低下に役割を果たしている可能性があることを示しました。
変異誘発による BA.2 RBD の内部構造のリモデリングは、RBD および S ベースの免疫原の設計に影響を及ぼし、BA.2 RBD の安定性が高いため、BA.1 の RBD よりも堅牢な免疫原になる可能性があります。
BA.2 S は BA.1 S よりも閉じた状態で優れたプロトマー間 RBD パッキングを示したにもかかわらず、in vitro ELISA アッセイではどちらも ACE2 受容体に同程度のレベルで結合したため、プロトマー間結合が修正されているにもかかわらず、受容体結合能力は 2 つの変異体間で同程度のままであることが示唆されました。
BA.1 S タンパク質と BA.2 S タンパク質では、FP へのアクセス性に違いが見られました。
これらの FP 指向抗体が FP 露出を誘導できることを実証した私たちの結果は、これらの広範囲に反応する抗体の作用メカニズムと、機能的に重要で高度に保存されたこの S2 サブユニット領域のダイナミクスに関する興味深い洞察を提供します。
DH1058 や DH1294 などの FP 指向抗体は非中和性ですが、生体内で保護効果があるかどうかはまだ判明していません。
これらの高度に保存された領域を効果的に標的にできれば、将来の変異体や CoV の発生に対抗するための広範な保護反応を誘発する戦略につながる可能性があります。
BA.1 と BA.2 の S タンパク質の主な違いは、(1) BA.2 の閉じた状態での RBD-RBD のパッキングがより強化されていること、(2) BA.2 RBD の内部パッキングがより優れていること、(3) FP へのアクセスがより困難であることです。
これらすべての要因、および BA.1 3-RBD-down O1BA.1 構造で観察される N2R 再配置状態が存在しないことから、BA.1 S と比較して BA.2 S のアーキテクチャがより安定していることが示唆されます。
SARS-CoV-2 の進化の過程で、特に VOC として出現した変異体では、S タンパク質が融合前の状態の安定性と、より伝染性が高くなる可能性のあるよりオープンな状態を採用する傾向が高いなどの特徴との間でバランスを取っているのが観察されました。
BA.2 S では、バランスが BA.1 と比較して安定性が高くなる方向に傾いていることがわかります。
BA.1 の S タンパク質を BA.2 よりも不安定にする構造的特徴 (RBD がアップ状態に移行する傾向が高く、FP がより容易に放出されるなど) が、オミクロン波の開始時に急速に拡散した原因である可能性があると推測したくなりますが、一方で BA.2 S の安定性が高く、免疫回避特性が向上したことで、BA.1 波の衰退後に現在の勢いを増している可能性があります。
全体として、私たちの研究はオミクロン変異体の特徴を明らかにし、原子レベルの構造決定を通じて主要な変異に目的を割り当てています。
さらに、BA.1 と BA.2 のサブ系統間の機能領域における顕著な違いが、それらの生物学的違いの原因である可能性を浮き彫りにしています。
Limitations of the study
BA.2 S エクトドメインクライオ電子顕微鏡データセットでは 3-RBD-down S タンパク質の割合が高かったにもかかわらず、ELISA では BA.1 および BA.2 S タンパク質の ACE2 結合レベルは同程度でした。
ELISA 形式の ACE2 密度は生理的 ACE2 受容体密度を代表していない可能性があり、生理的条件下では BA.1 および BA.2 S タンパク質の ACE2 結合に違いが観察される可能性があります。
さらに、BA.1 および BA.2 S タンパク質間の 1 つ以上の固有の変異が BA.2 S の ACE2 結合の増加に寄与し、閉じた BA.2 S の割合の増加をある程度相殺する可能性があります。
BA.1 および BA.2 変異が ACE2 結合レベルに与える影響を正確に解析するには、詳細な変異誘発および結合研究が必要になります。
3-RBD-down BA.2 S タンパク質の構造は、変異によって誘発される RBD のリモデリングの原子レベルの詳細を明らかにし、プロトマー内およびプロトマー間のパッキングの改善につながります。
BA.2 S 構造で観察された違いに対する各置換の寄与を明らかにするには、さらなる変異研究と点変異体の慎重な評価が必要になります。
以下省略。
この記事が気に入ったらサポートをしてみませんか?
