
組換えDNAとバイオテクノロジー
年寄りの読書で、いろんな分野の書籍を探したり、眺めたりしているのですが、若い頃から好きでよく読んでいたBLUE BACKS に、アメリカの『大学生物学の教科書』というシリーズがあることを偶然知り、少しずつゆっくり読み進めています。
あくまでも自分の知識の整理のためのまとめですが、その中から興味のあるページを少しずつまとめていきたいと思います。
ということで、今回は「組換えDNAとバイオテクノロジー」というページのまとめに挑戦したいと思います。難しいのは覚悟の上。まずは前半部分から。
1:起源の異なるDNAが組換えDNAを形成する。
組換えDNAは、少なくとも2つの異なる起源を持つDNAを用いて実験室で作成されたDNA分子です。
組換えに用いるDNAは、同じ個体(例えば、異なる染色体に由来する)のものでも、同種の異なる個体、あるいは全く別の生物から得たものでもかまいません。
制限酵素(制限エンドヌクレアーゼ)は2本鎖DNAを切断します。
1960年代後半に、科学者たちはDNA断片の結合を触媒する酵素、DNAリガーゼを発見しました。
DNAリガーゼの機能の1つが、DNA複製時の岡崎フラグメントの結合です。

*岡崎フラグメントとは、DNAが合成される時に作られる小さなDNA断片(フラグメント)のことです。岡崎フラグメントのDNAの塩基数は約100塩基です。
上図のように、リーディング鎖は、DNAの合成方向が複製フォークと同じ、これに対してラギング鎖は、DNAの合成方向が複製フォークと逆になります。
これは、DNAを合成するDNAポリメラーゼが、5’→3’の方向にしか進めないためです。
ラギング鎖が複製フォークと逆方向に合成されるために生じる問題があります。
複製フォークと逆方向にDNAを合成している間にも複製は進行していきます。
リーディング鎖の場合、既存の鎖を伸長すれば新しい鎖を合成できるため問題はありません。
ラギング鎖の場合、複製フォークとは逆方向にDNAを合成するため、既存の鎖を伸長することでは新しい鎖の合成はできません。
ではどうしているのか?というと
ラギング鎖では、5’→3’の方向に複数のRNAプライマーを作成し、断片的な鎖を合成しています。RNAプライマーとは、DNA複製時に一時的に作られるRNAのことです。
DNAを合成する酵素はDNAポリメラーゼですが、DNAポリメラーゼは無から1を生み出すことはできません。材料となるプライマーがなくてはDNAを合成できないのです。
プライマーぜによってRNAプライマーが作成されると、RNAプライマーの3’末端に、DNAポリメラーゼIIIが新しい鎖を合成していきます。この繰り返しで、新たにRNAプライマーが作成され、新生鎖が伸長します。その結果、ラギング鎖では、複製フォークの伸長方向とは逆方向に、断片的に鎖が合成されていきます。
この断片的な鎖のことを岡崎フラグメントと言います。
このままでは連結したDNA鎖になりませんのげ、DNAポリメラーゼI によって、RNAプライマーがDNAに置き換えられます。
そして、置き換えられたDNAと、隣のDNAの間にはすき間があるため、DNAリガーゼですき間部分を連結します。
この連続した過程で形成される1本鎖がラギング鎖です。


制限酵素を手に入れた科学者たちは、DNA分子を切断し、断片を継ぎ合わせて新しい組み合わせを作る、つまりDNA断片を組み換えることができるのではないかと考えました。
1973年には、スタンフォード大学のスタンレー・コーエンとカリフォルニア大学サンフランシスコ校のハーバート・ボイヤーらがついにそれを実現しました。
彼らはまず大腸菌から2つの異なるプラスミド(細菌細胞中で主たる染色体とは別個に複製する小さな環状DNA分子)を分離しました。
この2つのプラスミドは、それぞれ異なる抗生物質耐性遺伝子を含んでいました。
彼らは制限酵素で2つのプラスミドを切断して、その断片を混合し、DNAリガーゼを用いてそれらを再結合させました。
次に、このライゲーション(連結)反応で得た組換えプラスミドを新たな大腸菌細胞へ挿入し、その細胞を両方の抗生物質を含む培地で培養しました。
両方の抗生物質耐性遺伝子を含む組換えプラスミドで形質変換されたごく一部の細菌は培地上でコロニーを作りましたが、組換えプラスミドを欠いた大腸菌は耐性を示しませんでした。
この実験によって、組換えDNA技術が誕生したわけです。

多くの制限酵素はDNAの回文配列(パリンドローム配列:2本鎖をそれぞれ逆向きに読むと同一になる配列)を認識します。
例えば、制限酵素EcoRIの認識塩基配列は、2本鎖上で5’から3’方向に読むと、以下の通りどちらも同じ配列(GAATTC)になります。
5’…GAATTC…3’
3’…CTTAAG…5’
制限酵素の一部は回文配列の中央をまっすぐに切断し、「平滑末端」を持つ断片を生じます。
一方、EcoRIのようなその他の酵素は、二重らせんの片方の鎖のホスホジエステル結合を、他方の鎖の切断点から数塩基離れた場所で切断し、末端が凹凸の断片を生じます。
EcoRIにより相補鎖が別々の位置で切断されると、切断部位の2本鎖状態は4塩基対の間の水素結合のみで保たれることになります。

暖かい温度(室温以上)では、当該部分の水素結合の力は弱すぎて2本鎖状態を保てないため、DNAは断片として解離してしまいます。各断片の切断部位には、1本鎖の「突出部分(オーバーハング)」が生じます。この突出部分は、相補的な別の末端と塩基対合によって結合できる特定の塩基配列を持っているので、粘着末端と呼ばれます。

相補的な2つの粘着末端は、水素結合を形成できます。
もとのDNA分子の粘着末端同士が再結合することも、2つの異なるDNA断片の粘着末端が結合することもあります。
さらに言えば、例えばヒト由来の断片を細菌由来の断片と結合するといった具合に、起源の異なる断片を結合させることもできます。
結合当初は、2つの断片は水素結合の弱い力だけで2本鎖状態を保っていますが、酵素DNAリガーゼの触媒によって断片末端の隣接するヌクレオチド間で共有結合が形成されると、両断端は連結してより大きな1つのDNA分子となります。
制限酵素はEcoRI以外にも何百と存在し、どれも固有の認識配列を持っています。
これらの手段(制限酵素とDNAリガーゼ)によって、人工合成したDNA配列をはじめ、いかなる起源を持つDNA分子であろうと、科学者は切断して再結合することができるようになってきました。
最近になって、組換えDNAの新しい作製方法が開発されました。
ポリメラーゼ連鎖反応(PCR)に基づく複製の手法は、適当な制限酵素部位さえ必要とせずに、どんな2つのDNA分子も結合することができます。
例えば、連結する一方のDNA断片の5’末端と他方の5’末端に相補的な1本鎖を付加するように設計したプライマーを用いたPCR法によって、直接に2つのDNA断片を連結し増幅する方法はフュージョンPCRと呼ばれています。
2:細胞にDNAを挿入する方法は複数ある。
組換えDNAを作製する目的の1つは、特定の遺伝子やDNA配列のクローンの作製、すなわち同一コピーを大量に作ることです。
遺伝子も大腸菌のような細菌細胞に挿入することでクローン化できます。
遺伝子を挿入した細菌を増殖させれば、全て当該遺伝子のコピーを持った何百万もの同一細胞ができます。
クローニングは、配列決定とその後の解析に十分なDNAの入手や大量のタンパク質産物の合成のため、あるいは新しい表現型を持つ生物を創造するための第一段階として行われます。
組換えDNAは、形質転換(トランスフォーメーション)…宿主細胞が動物に由来する場合は形質移入(トランスフェクション)…として知られる操作によって宿主細胞へ挿入することでクローン化されます。
組換えDNAを持つ宿主細胞や生物を形質転換(トランスジェニック)細胞あるいは生物といい、外来DNAは導入遺伝子(トランスジーン)と呼ばれます。
形質転換細胞の作製には様々な方法が用いられます。
しかし一般に、これらの方法だけでは非効率的で、組換えDNAに曝された細胞のうち、実際に導入遺伝子で形質転換されるものはごく一部にすぎません。
形質転換細胞のみを分離し増殖させるために、抗生物質耐性を付与する遺伝子のような遺伝子マーカー(選抜マーカー)がしばしば組換えDNA分子の一部として組み込まれます。
抗生物質耐性遺伝子を選抜マーカーとして利用する場合、形質転換実験で得られた細胞を抗生物質の存在下で生育させると、非形質転換細胞は抗生物質で全て死滅し、形質転換細胞のみが生き残ります。
理論的には、組換えDNAの導入宿主にはどんな細胞でも生物でもなりえます。
とはいえ、多くの研究ではモデル生物が使用されてきました。
細菌は実験室で容易に増殖させたり操作したりできます。
中でも特によく研究されてきた大腸菌(Escherichia coli)のような細菌の分子生物学的機構は、大部分が判明しています。
さらに細菌は、組換えDNAを宿主細胞に導入するよう簡単に操作できるプラスミドを持っています。
しかし、転写、翻訳、翻訳後修飾の過程は原核生物と真核生物とでは進行が異なるから、細菌は真核生物遺伝子の発現宿主として常に適切であるとは限りません。
*ここで真核生物と原核生物の違いについて。(「休み時間の分子生物学」から)
真核生物は細胞の中にタンパク質の集合体からなる発達した構造(細胞内骨格)を有しますが、原核生物にはそのような構造が見当たりません。
大腸菌は原核生物ですが、酵母菌は真核生物です。
つまり酵母菌は私たちの仲間ですが、大腸菌は違います。
原核生物の細胞のことを原核細胞、真核生物の細胞のことを真核細胞と呼びます。
真核細胞には細胞核と呼ばれる構造があります。
これは核ラミナと呼ばれる中間径フィラメント(細胞内骨格の一つ)が核膜を裏打ちするように支持しているために実現しています。
一方、真核細胞が有するような細胞内骨格がない原核細胞には細胞核もありません。
真核細胞には中間径フィラメント以外にも、アクチンフィラメントや微小管などの発達した細胞内骨格が存在します。
また、真核細胞の細胞質中に存在する小胞体やゴルジ体などの膜構造がダイナミックに動くことができるのは、これらの細胞内骨格の存在に起因しています。
DNAについても、真核生物と原核生物では大きな違いがあります。
最大の違いは、真核細胞のDNAは末端がある線状であることに対して、原核細胞のDNAは末端がない環状構造をしている点です。
また、遺伝子情報の記録方式や利用の仕方にも大きな違いがあります。
あるタンパク質の情報を記録している遺伝子領域から、原核細胞では1種類のタンパク質しか作れないのに対し、真核細胞はエキソンと呼ばれる配列を転写時に自在に選択することによって(スプライシング)、多種類のタンパク質を作り出すことが出来ます。スプライシングにはスプライソソームというタンパク質複合体が関わります。
出芽酵母(イースト、Saccharomyces cerevisiae)は、組換えDNA研究のための真核細胞の宿主として汎用されます。
酵母を用いる利点として、細胞分裂が速いこと(2〜4時間で生活環が完了する)、実験室での増殖が容易なこと、ゲノムサイズが比較的小さいことなどが挙げられる。
加えて、酵母細胞は多細胞生物に特徴的な性質を除けば、真核細胞の有するその他の性質のほとんどを共有しています。
植物細胞は、成熟した組織から全能性の幹細胞を作り出すことが出来る優れた宿主です。この未分化細胞を組換えDNAで形質転換して、培養細胞として研究したり、新しい植物体にまで育てたりすることができます。
(幹細胞は、植物にも動物にも存在します。この細胞は絶えず分裂し続けていて、適切な信号が与えられれば分化が可能です。)
細胞培養の段階を経ずに完全な形質転換植物を作る方法もあります。
このような方法を用いて作製した植物は、生殖細胞系列を含む全ての細胞が組換えDNAを持つことになります。
実験室で培養した動物細胞は、ヒトや動物の遺伝子発現の研究、例えば医療的な目的などに用いることができます。
卵細胞に新たなDNAを挿入すれば、完全な形質転換動物さえも作製できます。
挿入されたDNAは通常、宿主の染色体中に組み込まれます。
細胞を化学的に処理することで外膜の透過性を高め、混合したDNAを細胞内へ拡散させることができます。
この他にも電気穿孔法(エレクトロポレーション)と呼ばれる方法があります。
この方法では、短時間の電気ショックを与えて、細胞膜に一時的に穿孔を作り、DNAを送り込めるようにします。
ウイルスに変更を加えて、組換えDNAを細胞内に運ばせることも可能です。
植物の形質転換には、DNAを植物細胞に挿入するある特別な細菌が汎用されています。
形質転換動物は、受精卵の核に組換えDNAを挿入することで作製可能です。
DNAでコーティングした金属の微粒子で宿主細胞を「射撃」する「遺伝子銃」もあります。
細胞に新たなDNAを挿入する上での課題は、宿主細胞に注入するだけでなく、宿主細胞の分裂に伴って注入したDNAも複製されるようにしなくてはならないことにあります。
DNAポリメラーゼはどんな配列にも結合してそのコピーを作るわけではありません。新たなDNAを複製させるためには、それが複製開始点(複製起点)を含むDNA断片の一部となる必要があります。
そのようなDNA分子は複製単位、あるいはレプリコンと呼ばれます。

新たに導入されたDNAを宿主細胞でレプリコンの一部とするためには、代表的な方法が二つあります。
①DNAを宿主染色体に直接連結する。
②運搬体として働くDNA配列(ベクターと呼ばれます)の一部としてDNAを宿主細胞に導入し、その後、宿主染色体に組み込ませるか、ベクター自体に備わった複製起点から複製させる。
宿主細胞へDNAを注入するために、数種類のベクターが利用されています。
・ベクターとしてのプラスミド
プラスミドは多くの原核細胞に存在し、主要なDNAとは別に独自に複製する小さな環状DNA分子です。いくつかの特徴から、プラスミドは有用な形質転換ベクターです。

プラスミドは比較的小さい(大腸菌のプラスミドは通常2000〜6000塩基対に過ぎません)ので、実験室での操作が容易です。
プラスミドは、配列中にそれぞれが1回ずつしか登場しない制限酵素の認識部位を一つ以上持つことが多いです。
このような制限酵素を用いれば、宿主細胞の形質転換に用いる前に、新規DNAを原核生物プラスミドへ容易に挿入できます。
多くのプラスミドが抗生物質耐性を付与する遺伝子を含んでいて、選抜マーカーとして利用できます。
プラスミドは細菌由来の複製地点(ori)を持ち、宿主染色体から独立して複製できます。細菌細胞が数百個もの組換えプラスミドを内包することも珍しくありません。
(宿主細胞中で多数のコピーとして存在するようなプラスミドをリラックス型プラスミドと呼びます。一方、宿主染色体の複製機構に依存し、少数しか存在できないものをストリンジェント型プラスミドと呼びます。)
このため、細菌の形質転換を活用した遺伝子増幅法は並外れて効率的です。
典型的なプラスミドに挿入したヒトβーグロビン遺伝子を内包した1 l の細菌細胞は、典型的な成人1人分のヒト細胞に存在する遺伝子と同程度の数の遺伝子(10の14乗個)を持っています。
実験室でベクターとして用いるプラスミドは大幅に改変され、次のような便利な性質が付与されています。
①多重クローニング部位(マルチクローニングサイト、MCS)
多くの場合、クローニングを目的とした20以上の特異的制限酵素の認識部位を持ちます。(下図。プラスミドpBR322。宿主:大腸菌。Hind III、BamHI、Sal I、Pst I など)

②ある種の宿主細胞で機能する複製地点(上図のori)
③選抜マーカー遺伝子のような1個以上のレポーター遺伝子

・植物用のプラスミドベクター
様々な種類の植物に新たなDNAを導入するためのベクターとして重要なのが、
アグロバクテリウム・ツメファシエンス(Agrobacterium tumefaciens)という細菌が持つプラスミドです。
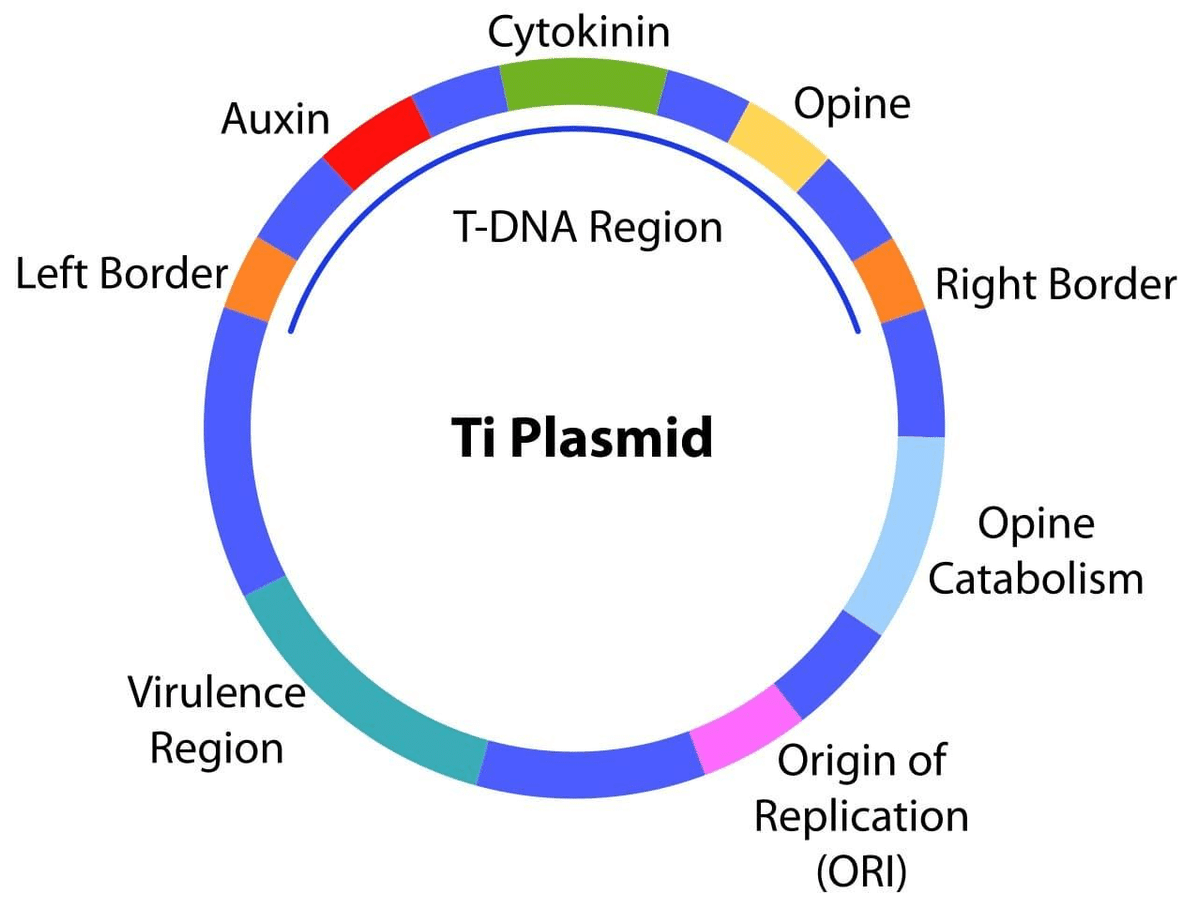
この細菌は土壌中に生息し、植物体に感染して根頭癌腫(クラウンゴール)と呼ばれる病気(異常増殖による腫瘍組織の形成が特徴)を引き起こします。
アグロバクテリウムはTi (tumor-inducing、「腫瘍を誘発する」の意味)と呼ばれるプラスミドを持っています。
アグロバクテリウムが植物に感染すると、Ti プラスミドのT-DNA(transferred DNA)と呼ばれる領域が細胞中に挿入され、植物細胞の染色体の一つに組み込まれます。Ti プラスミドはこのT-DNAの輸送と組み込みに必要な複数の遺伝子を備えています。T-DNAに含まれる遺伝子は宿主細胞で発現し、腫瘍の増殖とアグロバクテリウムが窒素源及びエネルギー源として用いる特殊な糖の合成をもたらします。
(腫瘍の増殖は植物ホルモンであるオーキシンとサイトカイニンの合成遺伝子がもたらします。特殊な糖はオパインと総称されるイミノ酸です。)
科学者は植物ゲノムに外来遺伝子を挿入するため、自然界のこの腕利きの「遺伝子工学技術者」を利用してきました。
Ti プラスミドを植物の形質転換用ベクターとして用いる際には、T-DNA上の腫瘍誘発遺伝子と糖合成遺伝子を取り除き、外来DNAで置き換えます。
組換えTi プラスミドはまず、もともとあったTi プラスミドを取り除いたアグロバクテリウム細胞の形質転換に用いられます。その後、組換えプラスミドを持ったそのアグロバクテリウム細胞を植物に感染させます。
・ベクターとしてのウイルス
プラスミド複製の限界のために、プラスミドに挿入できる外来DNAの大きさは10k(1k=1000)塩基対程度までとなります。
多くの原核細胞遺伝子はこれより小さいですが、イントロン(mRNA前駆体からmRNAとなる過程で不要なものとして切り取られる領域で、それに相当するDNAの領域もイントロンと呼ばれる場合もあります。)と広範な隣接配列を持つ真核生物遺伝子はほとんどが10k塩基対よりもずっと大きいです。こうした遺伝子の導入には、より大きなDNA配列を挿入できるベクターが必要です。
真核生物のDNAベクターとしては、原核生物に感染するウイルスと真核生物に感染するウイルスのどちらも利用できます。
大腸菌に感染するバクテリオファージ・ラムダ(λ)は約48k塩基対のDNAゲノムを持ちます。そのうち約20k塩基対はバクテリオファージが生活史を全うするのに不必要です。この20k塩基対を取り除いて、別の生物のDNAで置き換えることが可能で、置き換えられたDNAはファージDNAとともに複製されます。
ウイルスは特別な操作がなくても自然に細胞に感染しますから、宿主細胞にうまく侵入させるために人工的な手段が必要なプラスミドに比べてはるかに大きな利点を持っています。
・レポーター遺伝子は組換えDNAを含む宿主細胞の選抜と同定に役立つ
宿主細胞の集団に適当なベクターを投与しても、実際にベクターを取り込むのはそのごく一部にすぎません。また、組換えDNAを作製する工程も完璧からは程遠い状態です。ライゲーション反応(DNAリガーゼを用いてDNA分子を連結する反応)の間にDNA分子は様々に組み換わることができるので、その多くが望み通りの組換え分子になりません。
そこで、望んだ組換えが起こる確率を上げるための方法が開発されました。
単純な方法としては、二つの異なる制限酵素を用いて近接した位置でベクターを切断することでした。この操作により、非相補的な二つの粘着末端を両端に持つベクター分子ができて、ライゲーション反応の間にベクターが単にもとの環状に戻る確率がぐっと下がります。挿入したい分子を同じ2種類の酵素で切断すると、理論的にはベクターと挿入断片が連結しさえすれば、機能を持つ環状プラスミド分子ができることになります。その場合でも、ライゲーション産物の一部しか望み通りの組換え配列を持たないことがしばしばです。
どうしたら望ましい配列を含んだ宿主細胞を同定あるいは選抜することができるのでしょうか?初期の組換えDNA実験で用いられた抗生物質耐性遺伝子のような選抜マーカーを用いるのも一つの方法でしょう。
選抜マーカーはレポーター遺伝子の一種と言えます。
レポーター遺伝子とは、その発現が容易に観察できる遺伝子を指します。
数種類のレポーター遺伝子を以下に示します。
①プラスミドその他のベクターに含まれる抗生物質耐性遺伝子は、選抜用の抗生物質の存在下で増殖できる形質転換宿主細胞の検出を可能にします。宿主細胞が通常ある抗生物質に対して感受性ならば、細胞がベクターで形質転換された時にだけ、その抗生物質を含む培地で増殖可能となります。この方法は、原核細胞でも、植物細胞や動物細胞などの真核細胞でも、遺伝子組換え細胞の選抜に用いられています。
②大腸菌lacオペロンのβーガラクトシダーゼ遺伝子(lacZ)は、人工基質のX-Galを明るい青色の産物に変化させることができる酵素をコードしています。

多くのプラスミドが多重クローニング部位(すなわち、標的DNA配列が挿入されうる多くの特異的制限酵素の認識部位が集まった領域)をその配列内に伴うlacZ遺伝子を持っています。
これらのプラスミドは抗生物質耐性遺伝子も併せ持っていますので、プラスミドを保持する細菌コロニーは抗生物質を含んだ固形培地で選抜できます。
この培地にはX-Galも含まれます。lacZ遺伝子に挿入された外来DNAを保有する組換えプラスミドを細菌コロニーが持っていれば、βーガラクトシダーゼが作られずコロニーは白色となります。挿入配列を持たないもとのプラスミドを含むクローンは、lacZ遺伝子を発現して青色のコロニーを作ります。


③オワンクラゲ(Aequorea victoria)が通常生成している緑色蛍光たんぱく質(GFP)は、紫外線(UV)に当たると目に見える緑色の蛍光を発します。
このたんぱく質をコードする遺伝子が分離され、ベクターに挿入されている。
現在、GFPはレポーター遺伝子として汎用されています。
今では、GFPは紫外線に当たると他の色を発するように改変されており、こうした新しい変異型も分子生物学者たちに広く利用されています。
(ホタルやクラゲなどの発光生物の発光メカニズムを研究し、1962年にオワンクラゲからイクオリンとGFPを発見したのは下村脩博士で、2008年にノーベル化学賞を受賞しました。)

ここまでDNAが制限酵素でどのように切断され、ベクターに挿入されて宿主細胞に導入されるかをまとめてきました。また、組換えDNAを取り込んだ宿主細胞を同定する方法も学んできました。
それにしても下村脩博士の発見は画期的でしたね。
組換えDNAとバイオテクノロジーの世界、年寄りには難しすぎる内容が多いですが(当たり前ですが)、頑張ってもう少し勉強してみたいと思いました。
この記事が気に入ったらサポートをしてみませんか?
