
マッカロー博士の芸術作品
https://viroliegy.com/2022/11/30/dr-mcculloughs-works-of-art/
芸術
想像力と技術で作られ、美しいもの、または重要な考えや感情を表現しているもの。
https://www.britannica.com/dictionary/art

最近、ピーター・マッカラ博士の論文で、「SARS-COV-2」の存在を否定し難い証拠が含まれていることを知った。この証拠によって、これまで病人の体液から直接精製・分離され、自然な方法で病原性が証明された「ウイルス」は存在しないとする我々の意見を封じ込めることができるはずであった。このように、私はマッカロー博士が何を用意しているのか、少し興味があった。私たちが求めていた証拠をついに示してくれるのだろうか?科学的手法に則った実際の科学的研究をして、架空の存在の存在を主張するのに必要な立証責任を果たすことができるのだろうか?
さっそく、マッカロー博士がどのような「否定しがたい」証拠を持っているのか、授業で明らかにしていこう。私は、研究者が「SARS-COV-2」の様々な株を扱いながら、低温電子顕微鏡を使って粒子の画像化と研究を行ったと主張するマッカローの論文全文を提供しました。なお、McCullough氏の記事のタイトルは、"SARS-COV-2 "ではなく、"Covid "を間近で見たと主張しています。これは、「SARS-COV-2」が「ウイルス」であるのに対し、「Covid」は「病気」であることから、博士による興味深い誤りであると言えます。幸先の悪いスタートとなった。
COVIDを間近で見ると、その存在を否定するのが難しくなります
SARS-CoV-2の危機とパンデミックへの対応に果てしなく続くフラストレーションから、ウイルスの存在を完全に否定することを後押しする声もある。
ウイルス学の実験方法はよく受け入れられており、細胞への侵入、複製、移動、反復感染を実証するために一連の実験を利用している。 ホールゲノムシークエンスは、変異体および亜変異体の同定に役立ち、次に何が起こるかを予測する上で大いに役立っています。 CDC Nowcastシステムは、ウイルスサンプルのターゲットシークエンシングの優れたアプリケーションです。
それにもかかわらず、SARS-CoV-2をバクテリアのように培養して「分離」できないのであれば、それは存在しないのだと言う者がいる。 私は、実験室でのウイルス学、配列決定、そしてマックス・プランク研究所が行っている複雑な技術システムの力学のようなウイルスの大量生産の原理は、ウイルスの存在に依存する具体的なプロセスであると常に答えてきた[ii]。
医学文献の集積と実際の臨床経験から得た私の理解は、COVID-19は確かにインフルエンザや他のウイルス感染症とは区別される独特の病気であるという結論に一致しています。 COVID-19をインフルエンザや他のウイルス性症候群と区別する特徴として、細菌による重感染や微小・巨大血栓症がないことに、私はいつも感心している。
フランシス・クリック研究所のカルダー(Calder)らは、電子顕微鏡を使ってウイルスを間近で観察し、さらに一歩進んだ研究を行っています[iii]。

写真は千の言葉を語り、最も懐疑的な「ウイルス否定論者」であっても、リスクの高い患者を治療し、馬鹿げた伝染病対策をやめ、我々の世界を正常に戻そうとする合理的なチームに加わることができるはずです。
だから、今度カクテルパーティーで「COVID-19はデマだ、ウイルスは分離されていない」と言う人がいたら、「この芸術作品を見せなさい!」と言うのです。

マッカロー博士によれば、「SARS-COV-2」の存在を否定する人たちが間違っていることを決定的に証明するために必要な「否定しがたい」証拠は、自然な方法で病原性を証明した精製・分離した「ウイルス」粒子ではないとのことである。マッカロー博士の厳重な証拠は、提供された低温電子顕微鏡画像、すなわち博士が言うところの「芸術品」の形で提供される。添付の低温電子顕微鏡画像が存在の必要証拠であると主張しようとするときに使う言葉としては、興味深い選択である。百聞は一見にしかず、ですね。したがって、サンタクロースの写真は赤い服を着た不思議な男の存在を証明する直接的な証拠であると結論づけることができる。ビッグフットは何度も写真に撮られているので、絶滅危惧神話生物リストから外れたということになるのだろう。ネス湖の怪物?ネッシーは好奇心旺盛な野次馬にポーズをとることで有名だからだ。このように、写真を直接の証拠とすることは、合理的な思考プロセスであるようだ。このマッカロー博士の考え方に間違いはないので、私はマッカロー博士のチーム・ラショナルに入ることにしよう。
しかし、あえて言えば、この画像がどのように作成され、どのように入手されたかということが、マッカロの "論理 "に穴をあける絶好のスタート地点になるのかもしれない。明らかに、サンタの防犯カメラ画像は、どこかの子供の親が撮影したもので、サンタが行為に及んだトップ10回を収めた非常に「信頼できる」Youtube動画から取られたものであろう。これは、「否定しがたい」証拠とは到底言えない。物議を醸したビッグフットの画像は、猿の着ぐるみを着た男が撮影したものである可能性が高いことも分かっている。ネス湖の怪獣の写真は、おもちゃの潜水艦にプラスチックの頭を取り付けたものである。このように、画像の出所や作成方法、入手方法は、実際の画像そのものよりも重要なのかもしれない。よくよく考えてみると、私のTeam Rationalへのコミットメントは、ここで少し揺らいでいるのかもしれない。では、マッカローの作品がどのように作られたのか、見てみよう。
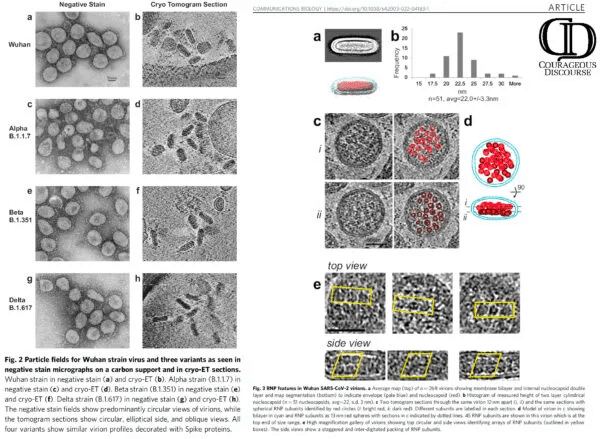
マッカロー博士が魅了された論文のハイライトを見ると、研究者は「SARS-COV-2」の4つの異なる株(武漢、アプラ、ベータ、デルタ)の構造をクライオトモグラフィーで調べようとしたことがわかる。これは電子顕微鏡の一種で、『ネイチャー』によると、極低温に保たれた生体試料を傾けることで電子顕微鏡が一連の二次元画像を記録する手法である。イメージングに使用された「ウイルス株」は、英国ロンドンのフランシス・クリック研究所にある世界インフルエンザセンターの敷地外で、Vero細胞で培養され「増殖」したものである。その後、「ウイルス」は、100 U/mlペニシリン、100 μg/mlストレプトマイシン(Pen-Strep)、10%(v/v)熱不活性化ウシ胎児血清(FCS)を加えたダルベッコ変法イーグル培地(DMEM)Gibco™で維持された。この研究者は、病気の人間の体液から直接精製・分離した「ウイルス」粒子ではなく、「ウイルス」粒子を含むと想定される未精製の細胞培養上清を使用しているので、我々はすでに悪いスタートを切っているのである。
研究者たちは、この研究の結果を掘り下げてみると、観察された粒子は円筒形で、表面からトゲが突き出ていたと主張している。これは、以前の研究で、粒子の形態や形状が球形や楕円形であることを認めていたのとは全く対照的である。このように、マッカローが提示しているスラムダンクの証拠は、奇妙なことに、"SARS-COV-2 "の形状を全く違うものにしているのである。

残りのハイライトでは、フィデューシャルの位置合わせ、動き補正、線量重み付け、位相反転、バックプロット、後処理、参照マッピングなど、培養粒子の3D画像を再構築するために使用した方法とプログラムのいくつかを詳細に説明している。3Dモデルを作成するために、様々な粒子の最良のビューから粗いモデルを生成するために、EMANプログラムを使用して粒子を対称化したと記載されている。このプロセスがどのようなものかを簡単に説明する。
「単粒子クライオ電子顕微鏡画像は、決定すべき3次元構造のランダムビューでの2次元投影であるため、逆問題は、これらの2次元画像から計算機画像処理法を用いて3次元構造を決定することである。現在の画像処理法は、3次元再構成を繰り返しながら改良していく方法である。完全な改良に進む前に、最初の3次元モデルが正しく構築されていることが重要である。」
「シンメトリービュー法 このEMAN法は、5回、3回、2回の対称性の特性が最も良い粒子画像を意図的に探し、これらの粒子を使用して、さらに洗練される最初の粗い3Dモデルを構築します。この方法はEMANプログラムstarticosで利用可能です。」
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4020923/
このような再構築の努力の結果、多くのプロセスとコンピュータプログラムが使用され、実際の画像が作成されたのか、それともコンピュータソフトウェアによって作り出された芸術家の解釈なのかは、読者の判断に委ねたいと思います。
SARS-CoV-2ビリオンの電子線結晶構造解析により、二重層のRNP集合体を持つシリンダー状粒子が発見された
「SARS-CoV-2は脂質を内包したベータコロナウイルスであり、Covid-19のパンデミックの原因である。このウイルスの3次元構造を調べるために、SARS-Cov-2ウイルスと3つの変異型について電子線低温トモグラフィーを行ったところ、規則的な円筒形の形態をした粒子が発見された。ビリオン内部でゲノムをパッケージングしているリボ核タンパク質粒子は、粒子全体の形態に関連した円筒形の密な二層集合体を形成していることがわかった。この組織は、ウイルスの組み立てに重要な構造的相互作用を示唆している。
はじめに
Covid-19の原因ウイルスであるSARS-Cov-2は、高解像度での個々の構成タンパク質の低温電子顕微鏡による構造研究、ウイルスや感染細胞のクライオトモグラフィー研究1,2,3など、精力的に研究が行われてきた4,5。凍結水和ウイルス粒子のクライオトモグラフィーは、粒子の外部と内部を3次元的に可視化できるため、多形脂質を内包したウイルスの3次元構造を理解する上で重要な方法である。これまでの研究では、SARS-CoV-2ウイルスは球形あるいは楕円形であり、また、主にウイルス表面の融合前および融合後のスパイクタンパク質(S)とウイルス内部のリボ核タンパク質集合体を可視化してきた。また、膜タンパク質(M)、エンベロープタンパク質(E)などの他のウイルス構造成分や非構造タンパク質が、ウイルスの構造と集合に重要な役割を果たしていると考えられる。
ウイルスの構造については、ウイルスの構成要素がどのように3次元的に相互作用して粒子を組み立てているのか、大きな正鎖ゲノムはどのようにパッケージされているのかなど、多くの基本的な疑問が残っている。そこで、我々はクライオトモグラフィーにより、SARS-CoV-2のオリジナル株Wuhanとパンデミックの最初の1年間に生じた3種類の変異型のウイルスの構造について研究を行っている。断層写真から、粒子は均一な円筒形をしており、スパイクタンパク質がエンベロープ全体に分布していることがわかった。また、粒子内部ではRNPが組織的に集合していることがわかり、ウイルス集合体としての意味を持つことがわかった。
結果および考察
SARS-CoV-2のオリジナル株(Wuhan)、Alpha(B.1.1.7)、Beta(B.1.351)、Delta(B.1.617)変異体の凍結乾燥ウイルスの傾斜系列と再構成したCTを記録し、このCT画像を用いて、ウイルス粒子の形状を調べた。断層図は、ビリオンが極めて平らな円筒形である単一かつ均一な形態が優勢であることを示している。"
方法
SARS CoV-2ウイルス種苗の作製
「以下の4種類のウイルス株を入手した。
Wuhan (hCoV-19/England/02/2020), (GISAID EpiCov™ accession EPI_ISL_407073), The Respiratory Virus Unit, Public Health England (PHE), UKから入手した。
Alpha variant B.1.1.7 (hCoV-19/England/204690005/2020) Wendy Barclay教授(インペリアルカレッジ、ロンドン、イギリス)を通じてPHEから提供された。
ベータ・バリアント B.1.351 (501Y.V2.HV001) は、アフリカン・ヘルス・インスティチュート、ダーバン、南アフリカ共和国から。
Delta variant B.1.617.2 (GISAID accession EPI_ISL1731019) Wendy Barclay教授より Genotype-to-Phenotype National Virology Consortiumを通じて提供された。
すべてのウイルスは、英国ロンドン大学セントジョージ病院のスティーブ・グッドボーン教授から提供されたVero V1細胞で、バイオセーフティレベル3の条件下、世界インフルエンザセンター、フランシス・クリック研究所で培養し、100 U/mlペニシリン、100 μg/mlストレプトマイシン(ペンストレップ)および10%(v/v)熱不活化ウシ胎児血清(FCS)が含まれるダルベッコ変法イーグル培地(DMEM)Gibco™ 41965039中で維持した」
断層像の生成
「Talosで取得した傾斜系列をIMODパッケージ18でフィデューシャルアライメントし、SIRTで再構成し、5回反復した。得られたトモグラムは4倍ビン化され、Nonlinear Anisotropic Diffusionの10回繰り返しでフィルタリングされた。
Krios が取得したチルト シリーズのムービー フレームは、IMOD パッケージを使用して動きを補正し、線量加重を加え、基準位置合わせを行いました。コントラスト伝達関数は CTFFIND4 で推定され19、断層像は位相反転によって CTF 補正され、novaCTF で再構成され20、重み付き逆投影断層像と SIRT のようなフィルター断層像が生成されました。視覚分析のために、SIRT のようなフィルター処理された断層像を 4 倍にビニングし、Bsoft 21 プログラム bnad をデフォルトのパラメーター (λ = 0.1) で使用して非線形異方性拡散フィルター処理を 20 回繰り返しました。
サブトモグラムの平均化
EMAN 2.91 22を使用して、20 の断層像から 268 の全ビリオンを 4 倍ビニングされた SIRT/NAD フィルター処理された断層像から手動で選択しました。次に、Relion 3.1 を使用して、4 倍のダウンサンプリング (ボックス サイズ 1598 Å、8.88 Å/ピクセル サンプリング) を使用して、ビニングされていない CTF 補正済み WBP トモグラムから粒子を抽出しました23。すべてのサブトモグラムの参照なしの位置合わせにより、初期マップが作成され、これがさらなる 3D 分類および位置合わせの参照として使用されました。ビリオンエンベロープの外面の周りのゆるくて柔らかいマスクは、さらなる位置合わせ、分類、および後処理中に使用されました。2 層ビリオンの平均マップは、Relion 後処理によって FSC = 0.143 カットオフで 44 Å の解像度を持つと推定されました。すべてのサブトモグラムの位置合わせ後、位置合わせなしの 5 つのクラスへの 3D 分類を実行して、ビリオンのさまざまな形態を調べました。
PCA解析のために、ビリオンのサブトモグラムをEMAN 2.9122に再インポートし、Relionマップに対してアライメントを行い、PCAベースの分類を用いて解析した(最初の基底ベクトルは3つで、出力クラスは3つであった)。
スパイク粒子は、IMODを使用して、4回ビン化されたSIRT/NADフィルター断層像から手動で選択された。次に、Relion 3.1でサブトモグラム平均化を行うために、2倍のダウンサンプリング(ボックスサイズ444Å, 4.44Å/pixel sampling)でフルサイズのWBP断層像から18断層像の251ビリオンから4418粒子が抽出された。C1によるリファレンスフリーの初期モデル生成により、明確な3回折りの特徴を持つマップが生成され、これを対称化し、C3対称性を適用し、エクトドメインの周りに緩いマスクを付けて、さらなる精密化のリファレンスとして使用された。精製の収束後、C3対称性を緩和して粒子を再分類したところ、1-RBD-upコンフォメーションのクラスが明らかになった。このクラスは分離され、閉じたコンフォメーション粒子はC3対称で最終的に精製され、1-RBD-up粒子は対称性を持たずに精製された。Relionの後処理により、最終的な分解能はFSC = 0.143 cutoffで30 Å (closed conformation)と28 Å (1-RBD-up conformation)と見積もられた。
サブトモグラム平均化マップは、社内スクリプト(著者から入手可能)を用いて、オリジナルの断層像の参照フレームにバックプロットされた。スパイク粒子は目視で検査し、位置がずれている(すなわち、ウイルスエンベロープの法線に対して90°以上の相対的な傾きがある)場合、または位置合わせ中に同じ位置に収束した重複する粒子を削除した。」
https://www.nature.com/articles/s42003-022-04183-1

この研究における「SARS-COV-2 ビリオン」の画像が、以前の多くの研究で見られたものとは異なる形状と形態を持っていたのはなぜですか? 電子クリオトモグラフィーに関連するさまざまなプロセスと制限を調べると、なぜそうなったのかがわかります。クライオ EM 画像は 3D 再構成であり、見られる画像を組み合わせて作成するには、さまざまなコンピューター プログラムとソフトウェアが必要であることを理解する必要があります。このプロセスでは、サンプルを凍結し、一連の記録を行います。サンプルを異なる軸上でさまざまな角度に傾けて複数の画像を取得し、それらを結合して 3D 再構成を作成します。しかし、これらの「芸術作品」を生み出すためには、最終的な結果を歪める可能性のあるいくつかの問題に定期的に遭遇します。
凍結生体サンプルの場合、放射線による損傷が大きな懸念事項です。サンプルがさまざまな傾斜を介して電子ビームにさらされる時間が長ければ長いほど、サンプルは急速に加熱され、使用できなくなります。より多くの電子が使用されると、高分子構造の本来のシャープなエッジが劣化し、最終的に「気泡」が発生して歪みが生じ、解釈が困難になります。別の問題は、特定の角度で画像を取得する際の制限により、測定されないままの情報のウェッジが欠落していることです。これにより、電子ビームに平行な方向の 3 次元再構成の解像度が、垂直方向の解像度よりも悪くなります。これにより、球状のオブジェクトが楕円体に見える場合があります。これらの問題を修正しようとするさまざまなコンピューター ソフトウェアとアルゴリズムが作成されましたが、それらは依然として残っており、構造の特定を困難にする可能性があります。これはすべて、以下のソースのハイライトで詳しく説明されています。
電子クライオトモグラフィー
はじめに — FtsZ のストーリー
「ECTは無傷の細胞を分子レベルの分解能(約4nm)で3次元に再構成することができるため、バクテリアの細胞の超微細構造について前例のない視点を提供し始めています。
ティルトシリーズの取得と基本的な制限事項
"トモグラフィ "という言葉は、断面や切片によるイメージングを意味します。最も身近なところでは、医療用のCT(Computed Tomography)スキャンで、被写体を透過したX線投影画像を複数の方向から記録し、それを統合して3次元の解剖学的モデルを作成する。同様に、電子断層撮影では、バクテリアの細胞のような1つの物体を1軸、場合によっては2軸に少しずつ傾けて撮影した「チルトシリーズ」の投影画像を記録し、これらの画像を統合して3Dの「再構成」または「断層像」を作成します(図2)。基本的なワークフローは、グリッドをEMに挿入し、ターゲットを選んで電子ビームの中心に置き、投影画像を記録し、サンプルを1~2度回転(傾斜)させ、別の投影画像を記録し、回転と撮影のサイクルを有用な画像が得られる限り(サンプルが非常に厚くなるかグリッドまたはグリッドホルダーがビームをブロックし始めるまで、通常約65°)繰り返すというものである。逆ティルト角(すなわち、0°〜-65°)の画像も同様に記録されるが、代わりに、ティルトシリーズは、ある極端なティルト角(例えば65°)から始まり、その反対の極端な位置(すなわち、-65°)に至るまでのティルト位置を経て進むことも可能である。残念ながら、凍結水和された生体物質では、放射線損傷により、このようなことはできない。イメージング電子が試料を通過する際、電子は散乱しない、弾性的に散乱する、非弾性的に散乱する、あるいは複数の散乱事象に見舞われることがあります。画像のコントラスト(情報量)は、非弾性散乱電子と弾性散乱電子の干渉によって作られるが、非弾性散乱電子は試料を徐々に破壊していく。非弾性散乱イベントは共有結合を破壊し、熱を蓄積し、まれに原子核を打ち砕くことさえある。有用な弾性散乱事象1つに対して、有害な非弾性散乱事象が約3つあるため(Henderson 1995)、画像を構築するために使用する電子の数が増えるにつれて、試料の損傷が蓄積されます。高分子構造の元々シャープなエッジが劣化し、最終的には(おそらく)放射線分解フラグメントの「バブル」が現れ、構造を壊滅的に破壊します(Comolli and Downing 2005; Iancu et al.2006b; Wright et al.2006 )。したがって、ECTにおける最も根本的な制限要因は、試料が破壊される前に画像を記録するために使用できる電子の総数です。"
"最も薄い試料でさえ、約65〜70°より高い傾斜角での有用な画像は通常収集できないので、未測定のままの情報の「くさび」(90°周辺の傾斜角)が存在するのである。その結果、電子ビームに平行な方向の3次元再構成の分解能は、垂直な方向の分解能よりも著しく悪くなる。視覚的にわかりやすく言うと、球状の物体が楕円形に見えたり、フィラメントや膜などの連続した物体が他の方向よりも見えやすくなる。このため、図1Dのような「xz」や「yz」の断層像では、細胞の「上」と「下」のあたりで膜がつながっているように見えないのである。グリッドを90°回転させ、第2の直交傾斜系列(いわゆる「2軸」データセット)を収集することによって、欠落した楔を欠落した「ピラミッド」に減らすことができるが、この手順は2倍以上の時間がかかり、画像ごとに使用できる線量は半分になり、傾斜系列間のアライメント誤差は利益を侵食しうる(Nickellら、2003;Iancuら、2005)。したがって、ECTにおける第3の基本的な限界は、傾斜制限("ミッシングウェッジ")によって引き起こされる異方的な解像度である。"
3D再構成と解釈
「前述したように、ゴニオメータは完全ではないため、試料はチルトシリーズを通してカラム内で横方向と縦方向に移動します。そのため、3D再構成を計算する前に、画像を正確にアライメントする必要があります。さらに、電子光学の物理的性質から、カラム内の高さや焦点が変わると、画像が回転し、微妙に異なる倍率で表示されることも課題となっています。さらに、各画像の傾き角度はおおよそ分かっているが、実際に到達する角度はより正確に決定する必要がある。そのため、傾斜シリーズの各画像の平行移動、回転、倍率、傾斜軸、傾斜角の推定を精密に行う高度なソフトウェアが開発された(Mastronarde 2008)。試料に通常添加されるコロイド金ビーズは、このプロセスを容易にするための正確なフィデューシャルマーカーとなる。
画像の位置合わせが完了したら、さまざまなアルゴリズムで3D再構成を計算することができる。最も直感的なのは「逆投影」で、各画像の密度を投影された方向とは逆の方向に空間を介して「塗抹」することによって再構成を構築する(図2B)(Crowther et al.) この再構成をフーリエ空間で理解するには、投影画像の2次元フーリエ変換は、対象物の3次元フーリエ変換の中心スライスであることが重要な原理となります(「投影定理」)(Crowther et al.1970a)。したがって,サンプルの3次元フーリエ変換を2次元画像の変換で「埋め」,それを通常の(例えばデカルト)座標系に再サンプリングし,逆変換することで実空間の再構成を行うことができる(Lee et al.) これらの計算を行うために、IMOD (Mastronarde 2008)、TOM toolbox (Nickell et al. 2005)、RAPTOR (Amat et al. 2008) など、さまざまなソフトウェアパッケージが作成されています。一度再構成された断層像は、画像コントラストを改善し、解釈可能性を高めるために「ノイズ除去」(Frangakis and Hegerl 2001; Narasimha et al. 2008)および/または特定の特徴を分離または表面として視覚化できるように「セグメント化」(Pruggnaller et al.
"要約すると、ECTは「分子分解能」に近い状態でインタクトな細胞の3D画像を生成しますが、サンプルは薄くなければならず(<0.5μm)、得られた断層像の解釈可能性は放射線損傷によって制限され、分解能は傾斜制限により異方的で、手順は複雑で高価な電子低温顕微鏡を必要とし、断層像の関心構造の識別は困難でありえます。"
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2869529/

この最後の資料を掲載した理由は、生成されたデータからクライオトモグラフィーの画像を得るために何が必要なのか、非常に詳しく説明されているためです。私は、単粒子画像とクライオ電子顕微鏡による断層像の両方を取り上げました。これは、マッカローが楽しんでいるファインアートや、このプロセスを用いて作られた他の「芸術品」の制作に関わる多くのステップを紹介するためです。紙面の都合で割愛させていただきましたが、興味深い情報がたくさんありますので、このテーマをより明確にしたい方は、ぜひ記事全文をお読みください。繰り返しになりますが、コンピュータで再現された映像から何を読み取るかは、読者の判断にお任せします。
この資料では、データの前処理として、スクリーニング、ボクシング、CTF判定といった画像の再構成のための3つのステップに分かれていることが述べられている。複数の単一粒子画像からの「ウイルス」の3次元再構築は、粒子の位置合わせという計算集約的なプロセスの最後のステップである。3Dマップの大半は正20面体対称を仮定しており、非正20面体対称の再構成が理想的なターゲットであるにもかかわらず、再構成に必要なデータがはるかに多いため、これは依然として複雑です。粒子配列と精密化に関して、3D再構成のためのプログラムは数多くあり、最終的にどれを使うかは個人の好みによります。これらのプログラムはアライメントアルゴリズムを使用しており、正確な初期モデルを必要としますが、その必要がないとされているプログラムもあります。モデルを生成するためには、再構成に使用する粒子の数と、低品質の画像には収差があり、再構成の解像度を高める上で益となるよりも害となる可能性があるため、どの粒子が高品質であるかを決定する必要があります。特定の解像度の再構成を達成するために必要な粒子の数を決定するためには、ノイズがない場合とある場合の両方について、データについて仮定する必要があると言われています。こうしたさまざまなステップを経て、CTF補正後のすべての2次元粒子画像をコンピュータプログラムで1つの3次元マップに統合することで、「ウイルス」の3次元モデルを再構築することができるのである。トモグラフィーの場合、画像の位置合わせが重要であり、GUI(グラフィカル・ユーザー・インターフェース)コンピュータープログラムを用いて行われる。ウイルス」の構造決定における最終ステップは、解像度決定、セグメンテーション、モデルフィッティングに関する3次元密度マップの解釈であり、その最適な近似解像度を超えてマップを解釈しないように注意する必要がある。残りの工程は、可視化のためのモデルへのフィッティングである。
1.16 クライオ電子顕微鏡法とウイルス粒子のトモグラフィー
「ウイルスの再構築は通常、単一粒子とトモグラフィーという 2 つの主要な画像診断法のうちの 1 つに依存します。どちらを選択するか は、in vitro または宿主細胞の存在下でのウイルスの構造的不均一性に依存します。2 つの手法の主な違いは、データの記録方法です。単一粒子データ収集では、グリッド上の個々のウイルス粒子が一度だけ画像化され、ウイルスを再構築するために使用されるのは、これらの数千の画像の収集です (図 2 )。クライオ ET では、多くのウイルス粒子を含むグリッドの単一領域が、さまざまな傾斜角度で複数回画像化されます。これらの画像の組み合わせが、断層像とその後の平均密度マップの生成に使用されます (図 3 )。単一粒子データの収集は、サブナノメートル分解能の構造研究によく使用されますが、構造的またはコンフォメーションの不均一性が存在する場合 (例えば、細胞のウイルス感染)、トモグラフィーはより実りある努力を証明します。使用される手法に関係なく、これらの手法のそれぞれに暗黙的に生データを処理する必要があり、その方法は非常に独特ですが、データの整列と平均化の基本原則は変わりません。
この章では、データ収集から処理および解釈まで、ウイルス再構築のためにクライオ EM およびクライオ ET の分野で使用される一般的な手法について概説します。これらの技術の多くは過去に十分に検討されてきましたが、[18]、 [21]、 [19]、 [20] クライオ EM は急速に進化している分野であるため、データ収集手順とデータ処理アルゴリズムの両方の進歩が重要な課題となっています。頻発。この章で説明する方法論は、クライオ EM およびクライオ ET によるウイルスの再構築のために国立高分子イメージング センター (NCMI) で一般的に使用されています。」
1.16.2. 単一粒子の再構成
「ウイルス粒子が生成される立体構造の均一性と、ウイルス粒子の精製が容易な結果として、二十面体ウイルスの構造を解明するために、単一粒子の再構築が長い間使用されてきました。[1]、 [2] 単一粒子の再構成は、数百から数十万のウイルス粒子をイメージングし、各粒子をコンピューターで分離し、個々の粒子を単一の 3 次元 (3-D) 密度マップに結合することに依存しています (図 2 ) 。 . クライオ EM は記録媒体上にウイルスの投影を提供するため、粒子の 3-D 方向の均一な分布の欠如は、データを再構築する際にバイアスを導入する可能性があります。したがって、単一の粒子イメージング セッションでは、一連の 2D 画像を収集する必要があります。これらの画像を組み合わせることで、粒子のさまざまな方向の十分にサンプリングされたビューが得られます。単一粒子再構成のプロセスには、一連の画像から各ウイルス粒子を分離し、その方向と中心を決定し、この情報を使用してデータをつなぎ合わせてウイルスの単一の 3D 表現にすることが含まれます。
最初の単粒子クライオ EM 再構成は約 40 年前に発表されましたが、[1]、 [2]、 [22] 、これらの再構成の解像度をサブナノメートルのしきい値をはるかに超えることを可能にする分野での進歩がまだ行われています。 . [4]、 [6]、 [8]、 [10]、 [5]、 [9] これらの進歩の背後にある原動力は、二次構造要素だけでなく、Cα バックボーンと側鎖も同定する能力です。ウイルスの個々のサブユニット。
1.16.3. トモグラフィー
構造の均一性が高い精製サンプルは単一粒子の再構成に理想的ですが、単一粒子アプローチに適したシステム内で作業できるとは限りません。構造的に多様なサンプルが存在する場合、類似または同一のコンフォメーションのほんの数個の粒子の情報コンテンツを利用できる技術が必要です。トモグラフィーの背後にある理論は、さまざまな傾斜角度でサンプルから一連の画像を収集することにより、2D 投影から 3D 体積密度を再構築できるというものです (図 3 (a))。[23]、 [24] このアプローチにより、トモグラム内の少数の粒子から大量の低解像度の 3D 情報を抽出できます。標準的な断層撮影条件では、一連の 71 枚の画像が -70° から 70° まで 2° のステップ サイズで撮影されます (60° および 80° の傾斜ホルダーも利用可能です)。これらの画像は、画像内に存在する基準マーカーを使用して互いに位置合わせすることができ (図 3 (b))、そこからボリューム (断層像) を抽出できます (図 3 (c))。 このボリュームから、各粒子に対応する個々のサブトモグラムを抽出することが可能です。次に、これらのサブトモグラムを分類、整列、および平均化して、単一の 3D モデルを取得できます。[26]、 [28]、 [27]
トモグラフィーに関する考慮事項の 1 つは、チルト シリーズ イメージング中にサンプルが受ける高線量です。単一粒子のデータ収集では、サンプルは多くの場合、顕微鏡写真ごとに 1 Å2 あたり 20 ~ 25 個の電子にさらされます。[20]、 [29] しかし、トモグラフィーでは、グリッドの単一領域が約 70 回撮像されるため、サンプルははるかに多くの放射線にさらされ、損傷が考慮されます。一般に、断層撮影中、1 画像あたりの線量は、1 断層像 (71 画像) あたりの総線量が Å2 あたり 80 ~ 100 電子となる点に設定されます。27 この線量レベルでも、放射線損傷が問題になり始め、この手法で達成可能な最大解像度が制限されます。[30]、 [31] したがって、単一の断層像で照射される総線量を制限するには、平均して、画像あたりの線量を、単一粒子の顕微鏡写真で一般的に使用される線量のほぼ 1/20 に減らす必要があります。画像ごとの総線量を下げると、データの信号対雑音比 (SNR) が低下し、データから達成可能な最大解像度が効果的に低下しますが、「線量分割」によるゲインは無視されます。32 単一の断層撮影シリーズが記録される方法により、これらの技術でこれまでに達成された最大解像度はナノメートルのしきい値に近づくだけであり、33 これは単一粒子アプローチによって設定された現在の基準をはるかに下回っています。[4]、 [11]、 [5]、 [6]、 [7], [8], [9], [10]”
1.16.6. データの前処理
「再構成ワークフローの次のステップは、顕微鏡から収集した電子顕微鏡写真を前処理することです。単一粒子の再構成の場合、前処理は通常、スクリーニング、ボックス化、および CTF の決定という同じ 3 つの手順に従います。これらの各ステップは再構築プロセスに不可欠であり、データ収集プロセスと同じくらい時間がかかる場合がありますが、それらを自動化するための努力が払われています. [51]、 [52] あるいは、断層像データの前処理は、一連の傾斜画像を互いに位置合わせすることを伴う。断層像データが収集される性質上、データ収集中にデータがスクリーニングされ、傾斜シリーズに異常な画像がある場合、同じ傾斜角度で別の画像を収集して、その角度のデータの損失を防ぐことができます。 .
1.16.7. 単一粒子の再構成
複数の単一粒子画像からのウイルスの 3 次元再構築は、計算集約的な粒子整列プロセスの最後のステップです。1 理論の背後にある一般原則は、一連の単一粒子画像が与えられた場合、それらの正二十面体 (または非対称) 方向、データを組み合わせて 1 つのまとまりのある 3-D モデルを形成できます。このプロセスは少数の粒子に適用できますが、多数の粒子を使用しない限り、優先配向、限られた焦点ぼけ範囲、ノイズなどの多くの機能により、再構成が高解像度に達しません。これらの問題を回避するために、ますます多くの粒子を使用することが一般的に行われてきました。」
1.16.7.1. 粒子の整列と改良
「3 次元再構築は、この分野で多くの研究の源となっており、EMAN、51 EMAN2、52 MPSA、58 Spider、61 XMIPP、62 IMAGIC、63 FREALIGN、64 を含む多くのプログラムの開発につながっています。 AUTO3DEM、65 SPARCX、66、および IMIRS.67 これらのプログラムの中には、他のプログラムよりも効率的なものもありますが、通常、どちらを使用するかは個人の好みの問題です。」
1.16.7.1.2. 非対称アライメント
「文献に掲載されているウイルスの再構築の大部分は、マップに対称性があると想定されている標本のものです。多くのウイルスでは、想定される対称性は正二十面体です。ただし、これらのウイルスが構築されて成熟する性質のために、それらの全体的な構造には対称ではない重要な分子成分があります。したがって、対称性を課すことなくこれらのウイルスの構造を決定することへの関心が高まっています。これは、ウイルスのアセンブリと成熟のプロセスに光を当てるのに役立つからです。[6] , [12] , [16] , [70] , [13] , [14] , [15] 対称性の仮定は、方向決定のプロセスを単純化するため、しばしば行われます。さらに、マップを正二十面体で平均化することにより、データセットの情報量が強化され、対称性を仮定しない再構成よりも約 60 倍少ないデータで高解像度の再構成を実現できます。68 非対称再構築のための方向決定のプロセスは、ウイルス粒子の非二十面体成分を識別できなければならないという点で、従来の対称方向検索とは異なります。6 P-SSP7 などの大きな尾部とポータル構造を持つファージの場合、非正二十面体対称の特徴が生の顕微鏡写真に表示されるため、粒子の非対称方向を決定する方法を概念的に理解するのは簡単です。 (図 15 (a))。しかし、生の顕微鏡写真で容易に識別できる突出した特徴を構造が持たない HSV-1 などのウイルスの場合、粒子の真の非対称方向を見つけることはより困難です (図 15 (b) )))。すべての粒子の非対称方向が決定されると、ウイルスを非対称に再構築するプロセスは、対称性が想定される他の再構築と同じです。
1.16.7.2. 単一粒子データのサブセットの選択
従来、マップの解像度は、より多くの粒子が再構成に追加されるにつれて改善されます。ただし、これは、一部のデータは他のデータよりも品質が高く、収差を含むデータを含めると、再構成の解像度を向上させるのに実際には害を及ぼす可能性があるという観察と比較検討する必要があります。単一粒子の品質は、コンフォメーションの不均一性、正二十面体対称性、およびイメージング条件の指標と考えることができます。56 これら 3 つの要因すべてが優れているほど、粒子が高解像度再構成の生成に寄与する可能性が高くなります。
特定の解像度の再構成を達成するために必要な粒子の数は、ノイズの有無にかかわらず、データに関する仮定によって異なります。[71]、」
「多くの場合、「最高の」粒子は、反復的な位置合わせと改良のプロセスを通じて、他の粒子よりも一貫して高いスコアを獲得することが決定された粒子です。58 どの粒子が最適かをアプリオリ に決定する具体的な方法はありませんが 、再構成に使用するデータの品質が可能な限り最高の品質であることを保証するために実行できる多くの手順があります。まず、サンプルの正確な初期モデルが存在する場合、2 μm を超えるデフォーカスでデータを収集する必要はありません。さらに、フロントエンドでデータを慎重にスクリーニングすることで、品質の低いデータの顕微鏡写真全体を排除することができます。2 番目のレベルのスクリーニングは、ボックス化のレベルで実行できます。このレベルでは、局所帯電の影響を受けている可能性のある粒子を特定して除去することができます。サンプル内のコンフォメーションの不均一性を直接制御する方法はありませんが、生の粒子データに目に見える違いがある場合は、データのサブセットを選択することでこの問題を改善できます (図 9 )。それにもかかわらず、「悪い」粒子がスクリーニングのこれらのステップを通過したとしても、場合によっては、データの改良プロセスによって、既存のデータ プールと一致しない粒子が排除されます。
1.16.7.3. 三次元再構築
データセット内のすべての粒子の方向が決定されると、ウイルスの 3D モデルを再構築することが可能になります。このプロセスは、CTF 補正後にすべての 2D 粒子画像を 1 つの 3D マップにマージすることによって機能します。通常、3 次元再構成プログラムには、これらの 3 次元密度マップを生成する方法があり、このタスクを達成するために、EMAN と EMAN2 はそれぞれプログラム make3d と e2make3dを使用します。これらの 2 つのプログラムは、直接フーリエ逆変換法に基づいて 3D ボリューム データを生成し、ユーザーが対称性やデータの前処理ステップを指定できるようにするさまざまなコマンド ライン パラメーターを備えています。」
1.16.8.2. アライメントと「ミッシングウェッジ」
「単一粒子クライオ EM と同様に、抽出されたサブトモグラム内の粒子の方向と配置を決定するためのさまざまなスキームがあります。[25]、 [26]、 [28]、 [51]、 [76] したがって、十分に解像された断層撮影の平均では、抽出された粒子を含む個々のサブトモグラムが互いに整列している必要があります。単粒子クライオ EM のように簡単です。ほとんどのクライオホルダーは最大傾斜角度が ±70° であるため (一部の 80° ホルダーは利用可能ですが)、断層像の総カバー範囲はせいぜい 140° に制限されます (図 17 (a))。理想的には、トモグラムには 180 度全体で収集されたデータが含まれます。ただし、これは現在利用可能なクライオホルダーでは不可能であるため、この 70° から 90° までの情報の欠如は、フーリエ空間のデータのウェッジの欠落として明らかになります (図 17 (b) および (c))。修正されていないため、最終的な再構成が歪む可能性があります。さらに複雑な要因は、1 つの断層像内の粒子グループの傾斜角が同じであるため、すべての粒子に同じ欠けているウェッジがあることです。ただし、粒子はサンプル内でランダムに方向付けられているため、各粒子にはその方向に関して異なるウェッジ データの欠落があります (図 17 )。(b) および (c))。ウェッジの欠落はデータの FFT でゼロの値として明らかになるため、2 つの粒子間の相互相関を計算すると、フーリエ空間で大量の情報が削除される可能性があります。これは、相互相関がこれらのゼロによる乗算を伴うためです。 (図 17 (c))。フーリエ空間に欠損データが存在すると、サブボリュームの適切な位置合わせが妨げられる可能性があるという事実に対処するために、[27]、 [28] 手順が開発され、異なる方向にある 2 つの粒子について計算された相互相関を正規化することによって、この問題を回避しました。 (図 17 (f))。28 このアプローチは成功を収めていますが、特に高解像度の断層撮影再構成では、ウェッジの欠落の問題が解決されたという保証はありません。
断層撮影データの位置合わせには、計算で抽出された粒子ごとに 3 つのオイラー角 (α、β、γ) を、一連の方向検索によって決定する必要があります。このプロセスは通常、ウイルスの近似モデルとの比較によって開始されます。ただし、適切な参照モデルが利用できない場合は、利用可能なデータから初期モデルを生成する必要があります。生データを使用して、「全対全」比較として知られるプロセスでサブトモグラムを互いに比較することにより、適切な初期モデルを生成できます。この初期モデルは、個々のサブトモグラム が分類され、整列され、単一または複数の 3D モデルに平均化される一連の反復改良によってさらに改善することができます。さらに、単一粒子クライオ EM のように、粒子を非対称の特徴に関して整列させることができるため、マップに対称性を課すことなく単一粒子クライオ ET マップを生成することが可能になります。それにもかかわらず、単粒子クライオ EM の場合と同様に、再構成中の対称性の仮定により、マップの解像度が劇的に向上します。16 クライオ ET データから 3D モデルを生成するには、方向検索が完了し、粒子が整列したら、失われたウェッジを考慮しながら個々のサブトモグラムを平均化することにより、データを 3D モデルに再構築できます。各粒子からの情報。28
1.16.9. データの解釈
ウイルスの構造決定の最終ステップは、得られた 3-D 密度マップの解釈、特に解像度の決定、セグメンテーション、およびモデル フィッティングです。ただし、最適な概算解像度を超えてマップを過度に解釈しないように注意する必要があります。総構造では 20 Å、個々のタンパク質ドメインでは 12 Å、長くて滑らかな α ヘリックスと大きなβ シートでは 9 Å、でこぼこした α ヘリックスでは 4.7 Å です。おそらくβストランド、おそらくCαバックボーントレースとかさばる側鎖の特徴を決定するために4.5Å未満、βストランドとループ接続のあいまいさを解決するために3.6Å未満。[77]、 [79]、 [78]
1.16.9.2. セグメンテーション
データ解釈の重要なコンポーネントの 1 つは、マップのセグメンテーションです。ほとんどの複雑な高分子は、より小さな自己集合タンパク質複合体 (セグメント) に分解できるため、これらの断片を全体から区別することは、ウイルスの構造と集合プロセスを理解することの一部です。ウイルスの場合、再構成で対称性が想定される場合、非対称単位のみを抽出し、それに応じてセグメント化することができます。[29] , [48] しかし、より多くのウイルスが対称性を課されることなく再構築されるにつれて、マップを全体として考慮する必要が生じる. [6]、 [13]、 [15]、 [14] マップは、密度マップの目視検査によって手動でセグメント化できますが、適合する結晶学的同族体を使用したマップの同時視覚化により、プロセスが容易になります。従来、セグメンテーションは手動で行われていました[75]、 [84]、 [85] が、セグメンテーション プロセスの特定の側面を自動化するツールが開発されています。これらのプログラムは、流域86 および主成分分析87 またはマルチスケール セグメンテーション88など、さまざまなアプローチを使用します。 ウイルスの個々のタンパク質成分を特定します。これらのアルゴリズムはセグメンテーション中の時間を大幅に節約しますが、精度は解像度と隣接分子間のオーバーラップ密度に依存します。
使用するソフトウェアに関係なく、正確なセグメンテーションは依然として困難な作業です。たとえば、バクテリオファージの解像度 9 Å のマップでは、キャプシド内に 2 つの別個のコートタンパク質が存在することは明らかになりませんでした。しかし、ε15 の 4.5 Å マップが得られたとき、そのタンパク質の Cα バックボーン トレースが実際には 2 つの異なるタンパク質であることが明らかになった後、2 番目のコートタンパク質が発見されました。4」
1.16.11。今後の展望
「この章で説明したように、ほとんどのウイルスの構造的特徴は、電子顕微鏡から記録されたノイズの多い画像では失われています。幸いなことに、大規模なデータ処理と再構築の後、これらの特徴を解決することが可能になります。場合によっては、原子に近い解像度で。この作業のほとんどは、計算によって画像のコントラストを強調する画像処理技術によって強化されていますが、製造技術の進歩により、顕微鏡学者は電子顕微鏡の光学系を変更することで直接これを行うことができるようになりました。」
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7151817/#!po=21.0983

要約すれば:
McCullough 氏によると、「SARS-CoV-2」がバクテリアのように培養して「分離」できない場合、それは存在しないと言う人もいます。
間違い。これらの粒子の存在は、病気の患者から採取したサンプルから直接精製および分離することにより、まず人間の体内で証明されなければならないと述べています。これが完了するまで、細胞培養は会話の一部であってはなりません。
McCullough 氏は、医学文献と直接の臨床経験から得た彼の理解は、「COVID-19」が実際にインフルエンザや他の「ウイルス」感染と区別できる独特の病気であるという結論と一致していると述べています。
彼は、1枚の写真が千の言葉を語り、最も懐疑的な「ウイルス否定論者」でさえ合理的なチームに参加するのに役立つはずだと信じています。
McCullough は読者に、次回カクテル パーティーで誰かが「COVID-19」はでっち上げだと言ったり、「ウイルス」は分離されていないと言ったりした場合は、「これらの芸術作品をいくつか見せてください」とアドバイスします。

研究者らは、「SARS-Cov-2 ビリオン」と 3 つのバリアントに対して電子低温断層撮影法 (cryo-ET) を実行し、規則的な円筒形態の粒子を明らかにしました。
CDC によると、「SARS-COV-2」は球形であるため、これはかなり奇妙です。
研究者らは、以前の研究では「SARS-CoV-2 ビリオン」を球状または楕円体として説明していると述べています。
彼らのトモグラムは、粒子がエンベロープ全体にスパイクタンパク質が分布する均一な円筒形であることを示していると再び述べています。
彼らは、凍結水和した「SARS-CoV-2ビリオン」の傾斜シリーズと再構築されたクリオトモグラムを記録しました
トモグラムは、「ビリオン」が主に (つまり、すべてではありません)非常に平らな円筒形である単一で均一な形態の
研究者は、英国ロンドンのフランシス・クリック研究所の世界インフルエンザセンターでバイオセーフティーレベル3の条件下でVero V1細胞で「増殖」させた4つの菌株(武漢、アルファ、ベータ、デルタ)を使用しました。 (サル腎細胞)
細胞培養物は、100 U/ml ペニシリン、100 μg/ml ストレプトマイシン (Pen-Strep)、および 10% (v/v) 熱不活化ウシ胎児血清 (FCS )
断層像の生成では、Talos が取得した傾斜シリーズをIMOD パッケージ に合わせて基準位置合わせし、 SIRT で 5 回繰り返して再構築しました。
Krios が取得したチルト シリーズのムービー フレームは、IMOD パッケージを使用してモーション補正、線量加重、および基準位置合わせが行われました。
コントラスト伝達関数はCTFFIND4で推定され、断層像は位相反転によってCTF補正され、novaCTFで再構築され、重み付き逆投影断層像とSIRTのようなフィルター断層像が生成されました
EMAN 2.9122 を使用して、20 個の断層像からの268 個の全「ビリオン」が、4 倍ビニングされた SIRT/NAD フィルター処理された断層像から手動で選択されました。(つまり、彼らは複数の画像から再構成した最良の粒子を選びました)
すべてのサブトモグラムの参照なしの位置合わせにより、初期マップが作成され、これをさらに 3D 分類および位置合わせの参照として使用しました
「ビリオン」エンベロープの外面の周りの緩いソフトマスクは、さらなるアライメント、分類、および後処理中に使用されました
すべてのサブトモグラムの整列後、「ビリオン」のさまざまな形態を調べるために、整列なしで 5 つのクラスへの 3D 分類が実行されました。
PCA 分析では、「ビリオン」サブトモグラムが EMAN 2.91 に再インポートされ、Relion マップに対して整列され、PCA ベースの分類を使用して分析されました。
スパイク粒子は、IMOD を使用して 4 倍ビニングされた SIRT/NAD フィルター処理された断層像から手動で選択されました。
次に、18 のトモグラムの 251 の「ビリオン」から4418 の粒子が、Relion 3.1 でのサブトモグラムの平均化のために 2 倍のダウンサンプリングを使用して、フルサイズの WBP トモグラムから抽出されました。
C1でのリファレンスフリーの初期モデル生成により、明確な3倍の特徴を持つマップが作成されました。これは対称化され、C3対称性が適用され、外部ドメインの周りに緩いマスクが適用されたさらなる改良のためのリファレンスとして使用されました
サブトモグラム平均化マップは、社内スクリプトを使用して元のトモグラムの参照フレームにバックプロットされました
スパイク粒子は視覚的に検査され、位置がずれている場合(つまり、「ウイルス」エンベロープの法線に対して 90° を超える相対傾斜がある場合) 、または位置合わせ中に同じ位置に収束した粒子が重複している場合は除去されました。

ECT は無傷の細胞の3 次元 (3-D)再構成を行うと言われています
電子断層撮影法では、細菌細胞のような単一の物体が 1 つ、場合によっては 2 つの軸の周りで徐々に傾斜する投影画像の「傾斜シリーズ」が記録され、これらの画像が結合されて 3D の「再構成」が生成されます。 」または「トモグラム」
基本的なワークフローは、グリッドが EM に挿入され、ターゲットが選択され、電子ビームの下で中央に配置され、投影画像が記録され、サンプルが 1 ~ 2 度回転 (傾斜) され、別の投影画像が記録され、回転とイメージングのサイクルは、有用な画像が得られる限り繰り返されます(サンプルが非常に厚くなるか、グリッドまたはグリッドホルダーがビームをブロックし始めるまで、通常は〜65°)。
残念ながら、凍結水和生物材料の場合、放射線損傷が問題になります
イメージング電子がサンプルを通過すると、散乱されないままになるか、弾性的に散乱するか、非弾性的に散乱するか、または複数の散乱イベントが発生する可能性があります。
非弾性散乱イベントは、共有結合を破壊し、熱を蓄積し、まれに原子核を所定の位置にノックすることさえあります
有用な弾性散乱イベントごとに、画像を構築するために使用される電子が増えるにつれて、約 3 つの有害な非弾性散乱イベントが発生するため、サンプルの損傷が蓄積されます。
高分子構造の元々鋭いエッジが劣化し、最終的に (おそらく) 放射線分解フラグメントの「気泡」が現れ、壊滅的に構造を破壊します。
ECT における最も根本的な制限要因は、サンプルが破壊される前に画像を記録するために使用できる電子の総数です。
最も薄いサンプルでも、約 65 ~ 70° を超える傾斜角での有用な画像は通常収集できず、測定されないままの情報 (90° を囲む傾斜角) の「くさび」があります。
その結果、電子ビームに平行な方向の 3-D 再構成の解像度は、垂直方向の解像度よりも大幅に悪くなり、これにより、球状のオブジェクトが多少楕円体に見えます(ビームの方向に不鮮明になります)。
グリッドを 90° 回転させて 2 番目の直交傾斜系列 (いわゆる「2 軸」データ セット) を収集することにより、欠落したウェッジを欠落した「ピラミッド」に縮小することができますが、この手順は 2 倍以上の時間を要します。画像ごとに使用できる線量が半分になり、一連のチルト間の位置合わせエラーが利点を損なう可能性があります
したがって、ECT の 3 番目の基本的な制限は、傾斜の制限 (「ウェッジの欠落」) によって引き起こされる異方性解像度です。
ゴニオメーターは完璧ではないため、標本は傾斜シリーズ全体を通してカラム内で横方向および垂直方向に移動するため、3D 再構成を計算する前に画像を正確に位置合わせする必要があります。
そのため、洗練されたソフトウェアは、傾斜シリーズの各画像の並進、回転、倍率、傾斜軸、および傾斜角度の推定値を改善するために作成されています。
画像の位置合わせが完了すると、さまざまなアルゴリズムを使用して 3 次元再構成を計算できます。
最も直観的なのは「逆投影」です。これは、各画像の密度を、投影されたのとは反対の方向に空間を介して「塗りつぶす」ことによって再構築されます。
これらの計算を実行するために、IMOD (Mastronarde 2008)、TOM ツールボックス、RAPTOR などのさまざまなソフトウェア パッケージが作成されており、一度再構成すると、断層像を「ノイズ除去」して画像のコントラストを改善し、解釈可能性を高めることができます。
ECT の制限事項は次のとおりです。
サンプルは薄くする必要があります (< 0.5 µm)
得られた断層像の解釈可能性は、放射線損傷によって制限されます
傾きの制限により、解像度は異方性です
手順は複雑で、高価な電子クライオ顕微鏡が必要です
トモグラムで関心のある構造を特定することは困難な場合があります

クライオ ET では、多くの「ウイルス」粒子を含むグリッドの 1 つの領域が、さまざまな傾斜角度で複数回撮像されます。これらの画像の組み合わせを使用して断層像が生成され、続いて平均密度マップが生成されます。
クライオ EM およびクライオ ET の分野では、データ収集から処理および解釈まで、「ウイルス」再構築のために使用される一般的な手法があります。
単一粒子の再構成は、数百から数十万の「ウイルス」粒子をイメージングし、各粒子をコンピューターで分離し、個々の粒子を単一の 3 次元 (3-D) 密度マップに結合することに依存しています。
クライオ EM は記録媒体上に「ウイルス」を投影するため、粒子の 3D 方向が均一に分布していないと、データを再構築する際にバイアスが生じる可能性があります。
単一粒子再構成のプロセスには、一連の画像から各「ウイルス」粒子を分離し、その向きと中心を決定し、この情報を使用してデータをつなぎ合わせて「ウイルス」の単一の 3D 表現にすることが含まれます。
構造的に多様なサンプルが存在する場合、類似または同一のコンフォメーションのわずか数個の粒子の情報コンテンツを利用できる技術が必要です。
トモグラフィーの背後にある理論は、さまざまな傾斜角度でサンプルから一連の画像を収集することにより、2D 投影から 3D 体積密度を再構築できるというものです。
標準的な断層撮影条件の下で、一連の 71 枚の画像が-70° から 70° まで 2° のステップ サイズで撮影されます (60° および 80° の傾斜ホルダーも利用可能です)。
単一粒子の再構成の場合、前処理は通常、スクリーニング、ボックス化、および CTF の決定という同じ 3 つの手順に従います。
あるいは、断層像データの前処理では、一連の傾斜画像を互いに位置合わせする必要があります
複数の単一粒子画像からの「ウイルス」の 3 次元再構成は、計算集約的な粒子整列プロセスの最後のステップです。
この理論の背後にある一般原則は、一連の単一粒子画像が与えられた場合、それらの 20 面体 (または非対称) の方向を決定したら、データを組み合わせて 1 つのまとまりのある 3D モデルを形成できるということです。
文献で公開されている「ウイルス」の再構築の大部分は、 対称性が想定されている標本のものです。(互いに向かい合っている、または軸の周りにあるまったく同じ部品で構成されているという性質)地図へ
対称性の仮定は、方向決定のプロセスを簡素化するため、しばしば行われます。
HSV-1 などの「ウイルス」の場合、生の顕微鏡写真で容易に識別できるような突出した特徴が構造にない場合、粒子の真の非対称方向を見つけることはより困難です。
すべての粒子の非対称方向が決定されると、「ウイルス」を非対称に再構築するプロセスは、対称性が想定される他の再構築と同じです。
マップの解像度は、再構成に追加される粒子が増えるにつれて改善されますが、これは、一部のデータが他のデータよりも品質が優れているという観察と比較検討する必要があり、異常を含むデータを含めると、実際には、再構成の解像度を高める
特定の解像度の再構成を達成するために必要な粒子の数は、ノイズの有無にかかわらず、データに関する仮定に依存します。
「最良の」粒子とは、反復的な位置合わせと改良のプロセスを通じて、他の粒子よりも一貫して高いスコアを示すと判断さ れた粒子ですが、どの粒子が最良であるかをアプリオリに判断する具体的な方法はありません 。
コンフォメーションの不均一性を直接制御する方法はありませんが、(性格または内容が多様であるという性質または状態)サンプル内で、生の粒子データに目に見える違いがある場合、データのサブセットを選択することでこの問題を改善することができます
「悪い」粒子がスクリーニングのこれらのステップを通過したとしても、場合によっては、データの改良プロセスによって、既存のデータ プールと一致しない粒子が除去されます。
3-D モデルを作成するプロセスは、CTF 補正後に、通常これらの 3-D 密度を生成する方法を備えた3-D 再構成プログラムを使用して、すべての 2-D 粒子画像を単一の 3-D マップにマージすることによって機能します。マップ
十分に解像された断層撮影の平均では、抽出された粒子を含む個々のサブトモグラムを相互に整列させる必要があります。このプロセスは、単一粒子クライオ EM ほど簡単ではありません。
理想的には、トモグラムには 180 度全体で収集されたデータが含まれます。ただし、これは現在利用可能なクライオホルダーでは不可能なため、70° から 90° までのこの情報の欠如は、フーリエ空間でのデータのウェッジの欠落として現れます。再建
追加の複雑な要因は、単一の断層像の粒子グループの傾斜角が同じであるため、すべての粒子に同じ欠けているくさびがあることです。
フーリエ空間に欠損データが存在すると、サブボリュームの適切な位置合わせが妨げられる可能性があるという事実に対処するために、異なる方向にある 2 つの粒子について計算された相互相関を正規化することにより、この問題を回避する手順が開発されました。
このアプローチは成功していますが、特に高解像度の断層撮影再構成の場合、ウェッジの欠落の問題が解決されたという保証はありません。
断層撮影データの位置合わせには、計算で抽出されたすべての粒子の 3 つのオイラー角 (α、β、γ) が、一連の方向検索によって決定される必要があります。
このプロセスは通常、「ウイルス」の近似モデルとの比較によって初期化されますが、適切な参照モデルが利用できない場合は、利用可能なデータから初期モデルを生成する必要があります
この初期モデルは、個々のサブトモグラムを分類、整列、および単一または複数の 3-D モデルに平均化する一連の反復改良によってさらに改善できます。
クライオ ET データから 3D モデルを生成するには、方向検索が完了し、粒子が整列されたら、欠落しているウェッジ情報を考慮しながら個々のサブトモグラムを平均化することにより、データを 3D モデルに再構築できます。各粒子から
「ウイルス」の構造決定の最終ステップは、得られた 3D 密度マップの解釈、具体的には解像度の決定、セグメンテーション、およびモデル フィッティングです。
ただし、最適な近似解像度を超えてマップを解釈しすぎないように注意する必要があります。
データ解釈の重要な要素の 1 つは、マップのセグメンテーションです。
「ウイルス」の場合、再構成で対称性が想定される場合、非対称単位のみを抽出して、それに応じてセグメント化することができます。ただし、より多くの「ウイルス」が対称性を課されることなく再構築されるにつれて、マップ全体を考慮する必要が生じます
「ウイルス」内の個々のタンパク質成分を特定するために、分水界および主成分分析やマルチスケールセグメンテーションなど、さまざまなアプローチを使用する多くのプログラムがあります。
これらのアルゴリズムはセグメンテーションの時間を大幅に節約しますが、精度は解像度と隣接分子間のオーバーラップ密度に依存します。
使用するソフトウェアに関係なく、正確なセグメンテーションは依然として困難な作業です
これらの画像は、画像に存在する基準マーカーを使用して互いに位置合わせすることができ、そこからボリューム (断層像) を抽出できます。
このボリュームから、各粒子に対応する個々のサブトモグラムを抽出することができ、これらのサブトモグラムを分類、整列、平均化して、単一の 3D モデルを取得できます。
ただし、トモグラフィーでは、グリッドの単一領域が約 70 回画像化されるため、サンプルははるかに多くの放射線にさらされ、損傷が考慮されます。
ほとんどの「ウイルス」の構造的特徴は、電子顕微鏡から記録されたノイズの多い画像では失われています
大規模なデータ処理と再構築の後、これらの特徴を解決することが可能になったと言われています – 場合によっては、原子に近い解像度で
ただし、この作業のほとんどは、画像のコントラストを計算によって強化する画像処理技術によって強化されています。
製造技術の進歩により、顕微鏡学者は電子顕微鏡の光学系を変更することで直接これを行うことができるようになりました

ピーター・マッカラ博士は、低温電子顕微鏡で撮影された3D画像から再構成された「SARS-COV-2」と名乗るきれいな写真を見た。そして、この画期的な証拠によって、否定的な人々を説得し、「チーム・ラショナル」に参加させるべく、急いで記事を書いたのである。というのも、これまでにも「ウイルス」と称する多くのTEM画像を丹念に調べ、納得できないでいた人たちが、このコンピュータで再現した画像は、否定派を納得させる視覚的証拠になると、マッカロー博士は確信していたのである。「百聞は一見に如かず」とマッカローは嬉々として歌った。"芸術作品を見せてやれ "と。しかし、マッカロー博士が無謀にも忘れていたのは、絵の背後にある千の言葉が重要だということだ。そして、この "芸術作品 "の場合、映像そのものよりも、その背後にある言葉の方が、はるかに大きな意味を持つのである。
マッカロー博士の芸術作品の制作方法を分解してみると、この粒子もまた、未精製の細胞培養上清が直接の原因であることがわかる。問題の「ウイルス」は、その場でVero細胞で「増殖」させ、ダルベッコ変法イーグル培地(DMEM)Gibco™に、100 U/mlペニシリン、100 μg/mlストレプトマイシン(Pen-Strep)、10%(v/v)熱不活性化ウシ胎児血清(FCS)入れて維持したものである。したがって、撮影された粒子が病気の宿主の体液中にあったという証拠はなく、この粒子が何らかの形で病原性を持つという証拠もない。あるのは、シャーレの中でアフリカミドリザル腎臓細胞に多数の毒物や異物を混ぜて何日も培養すると、細胞がバラバラになって死に、その跡にさまざまな粒子が残るという証拠である。ウイルス粒子と称する画像は、細胞の残骸か、画像処理の過程で生じたアーチファクトである可能性があります。再構成された粒子が複製能力のある "ウイルス "であるという証拠は全くない。
しかし、それでもこれらの画像の「否定しがたい」力を疑うには十分でないとしたら、これらの芸術作品を再構築するためだけに行わなければならない様々なプロセスを考慮に入れてみてください。細胞培養の過程で手を加えた試料を瞬間冷凍し、適当な大きさに薄め、電子ビームの高熱で叩いて画像を生成する。試料を軸方向に傾けながら、何度も記録を取る。一方、この高熱にさらされる時間が長いほど、試料は傷つき、劣化し、画像が歪む。記録後、さまざまな2次元画像をコンピュータープログラムと異なるアルゴリズムで整列・統合し、欠落しているデータを補う。コンピューターソフトウェアは、その結果を、粒子がどのようなものであるかを決定した3D表現にマッピングする。この過程で、多くの仮定、推定、解釈がなされ、目的の再現がなされるのです。
そう考えると、マッカローの言う「芸術品」という表現が最もしっくりくる。人間の技と想像力から生まれた映像。ある種のアイデア、感情、感覚を表現するために使われる。再現は創造的なプロセスから生まれ、見る人の主観的な解釈のために展示されます。クライオ電子顕微鏡画像は、どこから見ても「芸術品」なのです。しかし、これらは「SARS-COV-2」やその他の「ウイルス」の存在を決定的に証明するものではありません。それらは、見る人の目にとってのみ意味を持つランダムな粒子の画像である。だから、マッカローの説明はこれらの画像にはぴったりかもしれないが、彼が考えているようなメッセージを伝えているかどうかはわからない。もし、マッカロー博士がこの低温電子顕微鏡画像を「SARS-COV-2」の存在を証明するものとして真剣に受け止めてもらいたいのなら、彼が喜びの声明の中で考えていなかったかもしれない一つのことに、我々は同意できるだろうと思う。

________________________________________________
この記事が気に入ったらサポートをしてみませんか?
