
【Pythonで統計モデル】SEM8:二次因子分析・階層的因子分析
はじめに
初めまして、株式会社GA technologiesのAI Strategy Center部署に所属している宋です。
現在所属しているデータサイエンスチームでは、構造方程式モデリングの勉強会を行っています。構造方程式モデリングをPythonのsemopyパッケージでどこまでlavaanの分析結果を再現できるかを調べることを目的にしています。
今まで出していた「Pythonで統計モデル」のSEMシリーズは以下で:
【Pythonで統計モデル】SEM1:速習・構造方程式モデリング
【Pythonで統計モデル】SEM2:母数に関する制約
【Pythonで統計モデル】SEM3:パス解析(逐次モデル)
【Pythonで統計モデル】SEM4:パス解析(非逐次モデル)
【Pythonで統計モデル】SEM5:多変量回帰分析
【Pythonで統計モデル】SEM6:MIMICモデル
【Pythonで統計モデル】SEM7:PLSモデル
このシリーズに興味のある方は、ぜひAISC Magazineのフォローをお願いします!
学生時代にSEMの基礎について学んだ経験はありますが、その応用に関してはまだ不足していると感じています。もしご指摘や補足、感想などあれば教えていただけると幸いです!
また、この記事では東京図書から出版されている『共分散構造分析[R編]―構造方程式モデリング―』で取り上げられていた事例を、Pythonを使って再現していきます。分析用データは東京図書のホームページからダウンロードできますので、興味のある方はぜひ試してみてください。
ちなみに、GA technologiesにはエンジニアのスキルアップをサポートする「テックチャージ」という制度が導入されています。これにより、上司の許可を得れば、書籍の購入費やカンファレンスへの参加費を無制限で会社が負担してくれます。今回の書籍の購入もその制度を利用して、会社にサポートしてもらいました。
環境
本記事の分析は、OS: MacOS 13.4、Python 3.9.7の環境下で行っています。
今回使用しているsemopyはPythonで構造方程式モデリング(SEM)を構築するためのパッケージで、詳しいドキュメントは以下のリンクで確認できます。
二次因子分析モデル・階層因子分析モデル
今回はsemopyを用いて二次因子分析モデルと階層因子分析モデルを行います。
2次因子モデルは、「因子間に強い相関が観察されたときに、因子に共通して影響を与える上位の因子を想定する」モデルです。観測変数に直接影響を与えるのは一次因子、上位因子が二次因子と呼ばれています。
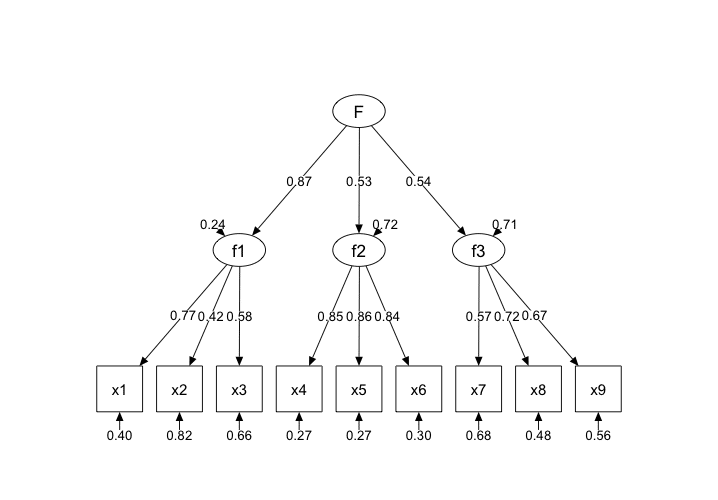
階層因子分析は、「全観測変数を説明する一般因子と一部の観測変数を説明するグループ因子を想定する」モデルです。『共分散構造分析[R編]―構造方程式モデリング―』のRコードで階層因子モデルのパス図を描画したら、下のような意味がわからないエラーメッセージが出ました。検索したがそのエラーが起きた原因はまだわからないです。
#パス図の描画 :図3.9
semPaths(fit5_H,layout="spring",
whatLabels="stand",sizeMan=5,
shapeLat="ellipse",sizeLat=8,sizeLat2=5,
exoCov=F,residuals=F,nCharNodes=0,
edge.label.cex=0.9,edge.color="black")Error in data.frame(label = pars$label, lhs = ifelse(pars$op == "~" | :
arguments imply differing number of rows: 37, 1, 38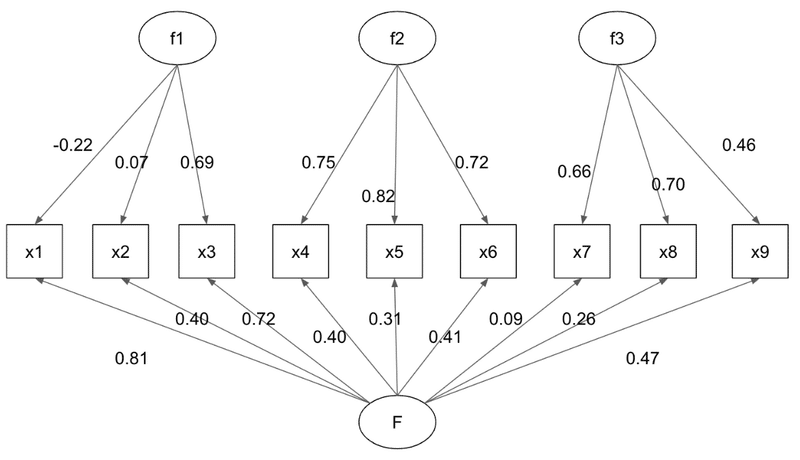
パッケージとデータの読み込み
import pandas as pd
import numpy as np
import semopydata5 = pd.read_csv("dat/intelligence.csv")今回使用するのは中学生を対象とした「知能検査データ」で、項目として
x1:視覚
x2:立方体
x3:ひし形
x4:段落の理解
x5:文章完成
x6:言葉の意味
x7:足し算
x8:点の数え上げ
x9:真っすぐな文字と歪んだ文字の区別
が含まれています。
x1, x2, x3は「視覚」因子、x4, x5, x6は「言語」因子、x7, x8, x9は「速度」因子から影響を受けています。
print(data5.head())# 出力結果
x1 x2 x3 x4 x5 x6 x7 x8 x9
0 3.333333 7.75 0.375 2.333333 5.75 1.285714 3.391304 5.75 6.361111
1 5.333333 5.25 2.125 1.666667 3.00 1.285714 3.782609 6.25 7.916667
2 4.500000 5.25 1.875 1.000000 1.75 0.428571 3.260870 3.90 4.416667
3 5.333333 7.75 3.000 2.666667 4.50 2.428571 3.000000 5.30 4.861111
4 4.833333 4.75 0.875 2.666667 4.00 2.571429 3.695652 6.30 5.916667
... ... ... ... ... ... ... ... ... ...
296 4.000000 7.00 1.375 2.666667 4.25 1.000000 5.086957 5.60 5.250000
297 3.000000 6.00 1.625 2.333333 4.00 1.000000 4.608696 6.05 6.083333
298 4.666667 5.50 1.875 3.666667 5.75 4.285714 4.000000 6.00 7.611111
299 4.333333 6.75 0.500 3.666667 4.50 2.000000 5.086957 6.20 4.388889
300 4.333333 6.00 3.375 3.666667 5.75 3.142857 4.086957 6.95 5.166667
301 rows × 9 columns二次因子モデル分析の実行
今回は「視覚」、「言語」、「速度」という3つの因子は「知能」という共通因子から影響を受けていることを想定し、以下のようなモデルを作成します。
model5_S = """
f1 =~ x1 + x2 + x3
f2 =~ x4 + x5 + x6
f3 =~ x7 + x8 + x9
intelligence =~ f1 + f2 + f3
x1 ~~ x1
x2 ~~ x2
x3 ~~ x3
x4 ~~ x4
x5 ~~ x5
x6 ~~ x6
x7 ~~ x7
x8 ~~ x8
x9 ~~ x9
"""semopyとRのlavaanパッケージとの間にはいくつか違いがあります。一つは外生潜在変数の分散(f1 ~~ f1)は自動で計算されますが、外生変数の分散(x1 ~~ x1, x2 ~~ x2)を明記しないとsemopyでは推定が行われない点です。
model5_S = semopy.Model(model5_S)
result = model5_S.fit(data5)
inspect = model5_S.inspect(std_est=True)
print(inspect)分析結果を表示すると小数点第2位の精度でテキストと一致していることが確認できます。
# 出力結果
lval op rval Estimate Est. Std Std. Err z-value
0 f1 ~ intelligence 1.000000 0.873819 - - \
1 f2 ~ intelligence 0.660532 0.525070 0.172682 3.825137
2 f3 ~ intelligence 0.424222 0.538340 0.117817 3.600677
3 x1 ~ f1 1.000000 0.772503 - -
4 x2 ~ f1 0.552572 0.423294 0.099537 5.551426
5 x3 ~ f1 0.728117 0.580666 0.108941 6.68359
6 x4 ~ f2 1.000000 0.851622 - -
7 x5 ~ f2 1.112935 0.855061 0.065407 17.015574
8 x6 ~ f2 0.926060 0.837995 0.055441 16.703654
9 x7 ~ f3 1.000000 0.569884 - -
10 x8 ~ f3 1.179383 0.723218 0.164781 7.157292
11 x9 ~ f3 1.080643 0.665024 0.150931 7.159852
12 f1 ~~ f1 0.191701 0.236440 0.170952 1.121372
13 f2 ~~ f2 0.709608 0.724302 0.107095 6.625983
14 f3 ~~ f3 0.273020 0.710190 0.068968 3.958632
15 intelligence ~~ intelligence 0.619078 1.000000 0.183513 3.373478
16 x1 ~~ x1 0.547855 0.403240 0.113711 4.817949
17 x2 ~~ x2 1.134082 0.820822 0.101714 11.149692
18 x3 ~~ x3 0.844991 0.662827 0.090601 9.326527
19 x4 ~~ x4 0.371130 0.274740 0.047719 7.777425
20 x5 ~~ x5 0.446261 0.268871 0.058388 7.642954
21 x6 ~~ x6 0.356259 0.297764 0.043037 8.277986
22 x7 ~~ x7 0.799282 0.675233 0.081397 9.819596
23 x8 ~~ x8 0.487604 0.476955 0.074193 6.572102
24 x9 ~~ x9 0.566164 0.557743 0.070715 8.006242
p-value
0 -
1 0.000131
2 0.000317
3 -
4 0.0
5 0.0
6 -
7 0.0
8 0.0
9 -
10 0.0
11 0.0
12 0.26213
13 0.0
14 0.000075
15 0.000742
16 0.000001
17 0.0
18 0.0
19 0.0
20 0.0
21 0.0
22 0.0
23 0.0
24 0.0 続いてモデルの適合度指標を求めると以下のようになりました。
stats = semopy.calc_stats(model5_S)
print(stats.T)# 出力結果
Value
DoF 2.400000e+01
DoF Baseline 3.600000e+01
chi2 8.530578e+01
chi2 p-value 8.501720e-09
chi2 Baseline 9.188516e+02
CFI 9.305594e-01
GFI 9.071604e-01
AGFI 8.607407e-01
NFI 9.071604e-01
TLI 8.958390e-01
RMSEA 9.227509e-02
AIC 4.143318e+01
BIC 1.192825e+02
LogLik 2.834079e-01Rでも出力した結果の一部を抜粋したものが以下になります。
lavaan 0.6.15 ended normally after 1 iteration
Estimator ML
Optimization method NLMINB
Number of model parameters 21
Model Test User Model:
Test statistic 85.306
Degrees of freedom 24
P-value (Chi-square) 0.000
User Model versus Baseline Model:
Comparative Fit Index (CFI) 0.931
Tucker-Lewis Index (TLI) 0.896
Root Mean Square Error of Approximation:
RMSEA 0.092自由度などの基本情報と、CFIやTFIなどの適合度指標が小数点第2位まで一致しているのを確認できました。
なお、SRMRについてはsemopyでは出力するためのメソッドは準備されていないため、別途自分で計算する必要があります。詳しくはPythonで統計モデル(SEM編)第1回の記事をご覧ください。
階層因子モデル分析の実行
「視覚」、「言語」、「速度」という3つの因子とは無関係な一般因子「勝負強さ」が全ての観測変数に影響を与えていることを想定し、以下のようなモデルを作成します。
model5_H = """
f1 =~ x1 + x2 + x3
f2 =~ x4 + x5 + x6
f3 =~ x7 + x8 + x9
F =~ x1 + x2 + x3 + x4 + x5 + x6 + x7 + x8 + x9
F ~~ 0*f1 + 0*f2 + 0*f3
F ~~ 1*F
f1 ~~ 1*f1
f2 ~~ 1*f2
f3 ~~ 1*f3
x1 ~~ x1
x2 ~~ x2
x3 ~~ x3
x4 ~~ x4
x5 ~~ x5
x6 ~~ x6
x7 ~~ x7
x8 ~~ x8
x9 ~~ x9
"""ここの「視覚」、「言語」、「速度」因子と「勝負強さ」一般因子は外生変数であるため、分散を1に固定する必要があります。本では、Rでの実行だとx3が不適解になるため、制約を書きましたが、semopyだと
x3 ~~ a*x3
CONSTRAINT(a > 0)
## もしくは
x3 ~~ a*x3
BOUND(0, inf) aという書き方ができます。しかしsemopyで制限を書かなくても不適解にはならないので、制限を書かないようにモデルを構築しました。
model5_H = semopy.Model(model5_H)
result = model5_H.fit(data5)
inspect = model5_H.inspect(std_est=True)
print(inspect)分析結果を表示するとテキストとまったく一致していないことが確認できます。
# 出力結果
lval op rval Estimate Est. Std Std. Err z-value p-value
0 x1 ~ f1 1.000000e+00 7.071068e-01 - - -
1 x1 ~ F 1.000000e+00 7.071068e-01 - - -
2 x2 ~ f1 -6.250664e-02 -5.215699e-02 0.09248 -0.675891 0.49911
3 x2 ~ F 6.522873e-01 5.442836e-01 0.087195 7.480774 0.0
4 x3 ~ f1 8.200249e-02 6.965272e-02 0.094423 0.86846 0.385143
5 x3 ~ F 7.624492e-01 6.476225e-01 0.079635 9.574275 0.0
6 x4 ~ f2 1.000000e+00 8.165128e-01 - - -
7 x4 ~ F 3.735659e-01 3.050213e-01 0.079177 4.718129 0.000002
8 x5 ~ f2 1.114462e+00 8.316804e-01 0.058127 19.172958 0.0
9 x5 ~ F 3.525764e-01 2.631144e-01 0.088462 3.985638 0.000067
10 x6 ~ f2 8.791352e-01 7.689241e-01 0.049236 17.855556 0.0
11 x6 ~ F 4.112433e-01 3.596885e-01 0.073388 5.603653 0.0
12 x7 ~ f3 1.000000e+00 8.680642e-01 - - -
13 x7 ~ F 4.152716e-03 3.604824e-03 0.089944 0.04617 0.963175
14 x8 ~ f3 6.258610e-01 6.013732e-01 0.069081 9.059792 0.0
15 x8 ~ F 3.246296e-01 3.119280e-01 0.07423 4.373271 0.000012
16 x9 ~ f3 4.581160e-01 4.372228e-01 0.066276 6.91225 0.0
17 x9 ~ F 5.759311e-01 5.496647e-01 0.07146 8.059513 0.0
18 F ~~ f1 0.000000e+00 0.000000e+00 - - -
19 F ~~ f2 0.000000e+00 0.000000e+00 - - -
20 F ~~ f3 0.000000e+00 0.000000e+00 - - -
21 F ~~ F 1.000000e+00 1.000000e+00 - - -
22 f1 ~~ f2 3.319790e-01 3.319790e-01 0.077763 4.269104 0.00002
23 f1 ~~ f3 1.515739e-01 1.515739e-01 0.089117 1.70085 0.088971
24 f1 ~~ f1 1.000000e+00 1.000000e+00 - - -
25 f2 ~~ f3 2.141704e-01 2.141704e-01 0.069514 3.080977 0.002063
26 f2 ~~ f2 1.000000e+00 1.000000e+00 - - -
27 f3 ~~ f3 1.000000e+00 1.000000e+00 - - -
28 x1 ~~ x1 4.880801e-17 2.440400e-17 0.13909 0.0 1.0
29 x2 ~~ x2 1.006855e+00 7.010350e-01 0.119197 8.447 0.0
30 x3 ~~ x3 7.979936e-01 5.757335e-01 0.100713 7.923421 0.0
31 x4 ~~ x4 3.603889e-01 2.402688e-01 0.04665 7.725411 0.0
32 x5 ~~ x5 4.292972e-01 2.390786e-01 0.059523 7.212282 0.0
33 x6 ~~ x6 3.652075e-01 2.793800e-01 0.041952 8.705287 0.0
34 x7 ~~ x7 3.270605e-01 2.464516e-01 0.093136 3.511656 0.000445
35 x8 ~~ x8 5.860113e-01 5.410512e-01 0.062278 9.409614 0.0
36 x9 ~~ x9 5.562890e-01 5.067049e-01 0.061992 8.973594 0.0続いてモデルの適合度指標を求めると以下のようになりました。
stats = semopy.calc_stats(model5_H)
print(stats.T)# 出力結果
Value
DoF 19.000000
DoF Baseline 36.000000
chi2 56.155675
chi2 p-value 0.000015
chi2 Baseline 918.851637
CFI 0.957914
GFI 0.938885
AGFI 0.884203
NFI 0.938885
TLI 0.920258
RMSEA 0.080737
AIC 51.626873
BIC 148.011739
LogLik 0.186564Rでも出力した結果の一部を抜粋したものが以下になります。
lavaan 0.6.15 ended normally after 1 iteration
Estimator ML
Optimization method NLMINB
Number of model parameters 30
Number of inequality constraints 1
Model Test User Model:
Test statistic 26.016
Degrees of freedom 15
P-value (Chi-square) 0.038
User Model versus Baseline Model:
Comparative Fit Index (CFI) 0.988
Tucker-Lewis Index (TLI) 0.970
Root Mean Square Error of Approximation:
RMSEA 0.049外生変数の分散を1に制限したため自由度などの基本情報と、CFIやTFIなどの適合度指標も一致できなかったです。
パス図の描画
続いてsemplotを使ってパス図を書いていきます。
g = semopy.semplot(model5_S, "model5_S.png", plot_covs=True,std_ests=True)
g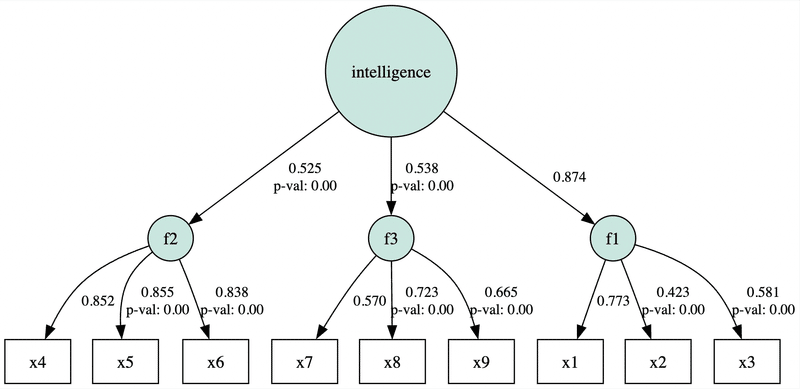
g = semopy.semplot(model5_H, "model5_H.png", plot_covs=True,std_ests=True)
g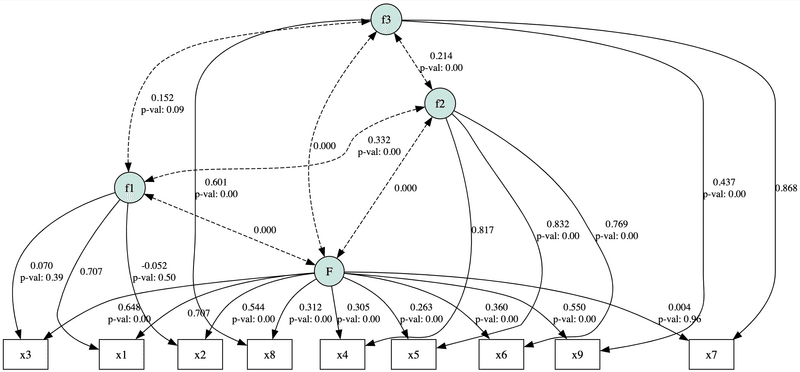
パラメータ「std_ests」は「標準化するかどうか」を、「plot_covs」は「共変関係のパスを表示するかどうか」を指定しています。
lavaan結果との比較
今回の分析を実行する中で、semopyでの階層因子分析がlavaanとの違いは以下だったのではないかと思います。
・外生変数の分散をすでに1に固定したのに、semopyがまた観測変数に刺すパスのうちの任意の1つを1に固定しないといけません。そしてこの固定制限を解除することもできません。(x1 ~ F、x1 ~ f1、x4 ~ f2、x7 ~ f3)
・そもそもデータと階層因子分析モデルの適合が良くありません。Rのlavaanで実行した際にx3の分散が不適解になってしまいました。
最後に
このシリーズは構造方程式モデリング(SEM:Structural Equation Modeling)をPythonで実行する方法について解説していきます。二次因子分析モデルと階層因子分析モデルはここで解説済みなので、「さまざまなパス図の描画」はここで終わりです。続きはSEMのもっと上級者向けの内容です。
1因子分析モデルによる信頼性係数の推定
多特性多方法行列のための加法モデル
構成概念間の因果モデル
平均共分散構造分析
多母集団同時分析
潜在成長曲線モデル
ランダム効果
ご覧いただき、ありがとうございました。Pythonを使用した統計モデルの学習がお役に立ったことを願っています。
この記事が気に入ったらサポートをしてみませんか?
