超簡単Seuratによるシングルセル遺伝子解析(scRNA-seq)を試してみる!
非エンジニアのための機械学習:
— Toshi@MD,PhD 🇺🇸 (@tokitky) October 1, 2019
seuratによる単細胞遺伝子解析(scRNA-seq解析)にだいぶ慣れてきた!!
治療前(D000)、治療後(D034)で白血病患者サンプルの中の正常(normal)および腫瘍細胞(malignant)細胞がどのように変化するかわかる! pic.twitter.com/2pG7RUrWH4
最近シングルセル遺伝子解析(scRNA-seq)のデータが研究に多用されるようになってきており、解析方法をすこし学んでみたので、ちょっと紹介してみたい!
簡単なのはSUTIJA LabのSeuratというRパッケージを利用する方法。scRNA-seqはアラインメントしてあるデータがデポジットされていることが多いのでそれを使って色々試すことができるのも面白い!
まずはパッケージのインストール!
Rでやるのだと
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("edgeR")
BiocManager::install("made4")
BiocManager::install("fgsea")
BiocManager::install("genefilter")
install.packages("pheatmap")
install.packages("RColorBrewer")
install.packages("ggplot2")
install.packages("gplots")
install.packages("calibrate")
install.packages("maptools")
install.packages("FactoMineR")
install.packages("rgl")
install.packages("caret")
install.packages("BBmisc")
install.packages("survival")
install.packages("survminer")
install.packages("dplyr")
install.packages("data.table")
install.packages("reshape2")
install.packages("beeswarm")
install.packages("devtools")
devtools::install_github("vqv/ggbiplot")
install.packages('Seurat')
install.packages('cowplot')
devtools::install_github('satijalab/seurat-data')このくらいを入れておくと大体困らないのではないだろうか?
ライブラリの読み込みはこんな感じになる
library(edgeR)
library(made4)
library(fgsea)
library(genefilter)
library(caret)
library(pheatmap)
library(RColorBrewer)
library(ggplot2)
library(gplots)
library(calibrate)
library(maptools)
library(FactoMineR)
library(rgl)
library(BBmisc)
library(survival)
library(survminer)
library(dplyr)
library(data.table)
library(reshape2)
library(beeswarm)
library(Seurat)
library(cowplot)
library(SeuratData)さて肝心の解析は以下の白血病データのうち
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE116256
GSM3587946のデータを用いて行います。このデータは治療前Day0と治療後Day34のscRNA-seqからなっており、データファイルとアノテーションファイルがからなります。それぞれ読み込んで
D000.data <- read.table("GSM3587946_AML371-D0.dem.txt" , header=T, row.names=1, check.names=T,sep="\t")
D034.data <- read.table("GSM3587948_AML371-D34.dem.txt" , header=T, row.names=1, sep="\t")
D000.anno <- read.table("GSM3587947_AML371-D0.anno.txt" , header=T, row.names=1, sep="\t")
D034.anno <- read.table("GSM3587949_AML371-D34.anno.txt", header=T, row.names=1, sep="\t")次にSeuratオブジェクトを作成し、タイムポイントとアノテーション(正常細胞か白血病細胞かです。このアノテーションはデータの作成者が機械学習を用いて予測したものです)を挿入します。
#create Seurat Object
D000 <- CreateSeuratObject(D000.data, project = "AML371", min.cells = 5)
D034 <- CreateSeuratObject(D034.data, project = "AML371", min.cells = 5)
D000@meta.data$stim <- "D000"
D034@meta.data$stim <- "D034"
D000@meta.data$mal <- D000.anno$PredictionRF2
D034@meta.data$mal <- D034.anno$PredictionRF2
D000@meta.data$mal
D034@meta.data$mal死細胞などを除くQCを行いNormalizationを行います
#QC check
D000[["percent.mt"]] <- PercentageFeatureSet(D000, pattern = "^MT-")
D034[["percent.mt"]] <- PercentageFeatureSet(D034, pattern = "^MT-")
v1 <- VlnPlot(D000, features = c("nFeature_RNA", "nCount_RNA", "percent.mt"), ncol = 3)
v2 <- VlnPlot(D034, features = c("nFeature_RNA", "nCount_RNA", "percent.mt"), ncol = 3)
plot_grid(v1, v2)
D000 <- subset(D000, subset = nFeature_RNA > 200 & nFeature_RNA < 2000 & percent.mt < 5)
D034 <- subset(D034, subset = nFeature_RNA > 200 & nFeature_RNA < 3000 & percent.mt < 5)
#normalize
D000 <- NormalizeData(D000, scale.factor = 10000)
D034 <- NormalizeData(D034, scale.factor = 10000)特徴となる遺伝子を抽出し、共通の遺伝子(ハウスキーピングジーン?)を用いて二つのデータを統合します。
#feature selection
D000 <- FindVariableFeatures(D000, selection.method = "vst", nfeatures = 3000)
D034 <- FindVariableFeatures(D034, selection.method = "vst", nfeatures = 3000)
#integration of Data
AML371_all.list <- NULL
AML371_all.list <- list(D000, D034)
names(AML371_all.list) <- c("D000","D034")
AML371_all.list
AML371_all.anchors <- FindIntegrationAnchors(object.list = AML371_all.list, dims = 1:30)
AML371_all.integrated <- IntegrateData(anchorset = AML371_all.anchors, dims = 1:30)
DefaultAssay(AML371_all.integrated) <- "integrated"クラスタリングを行い、結果を可視化します。
# Run the standard workflow for visualization and clustering
AML371_all.integrated <- ScaleData(AML371_all.integrated, verbose = FALSE)
AML371_all.integrated <- RunPCA(AML371_all.integrated , npcs = 30, verbose = FALSE)
AML371_all.integrated <- RunUMAP(AML371_all.integrated , reduction = "pca", dims = 1:5)
AML371_all.integrated <- FindNeighbors(AML371_all.integrated , reduction = "pca", dims = 1:5)
AML371_all.integrated <- FindClusters(AML371_all.integrated , resolution = 0.5)
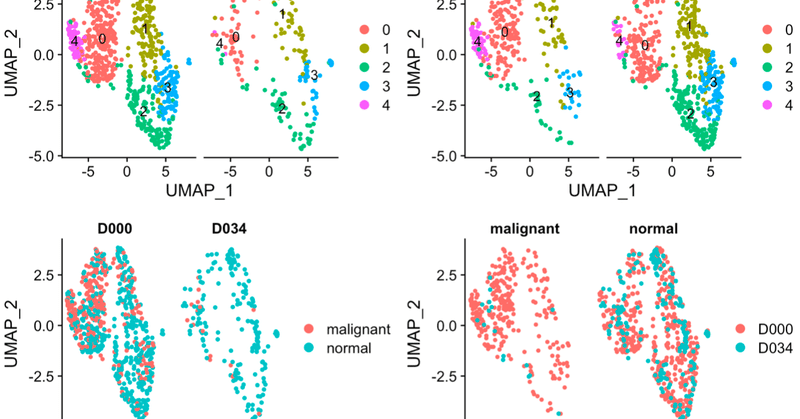
p1 <- DimPlot(AML371_all.integrated , reduction = "umap", group.by = "stim")
p2 <- DimPlot(AML371_all.integrated , reduction = "umap", group.by = "mal")
p3 <- DimPlot(AML371_all.integrated , reduction = "umap",label = TRUE,
repel = TRUE) + NoLegend()
plot_grid(p1, p2, p3)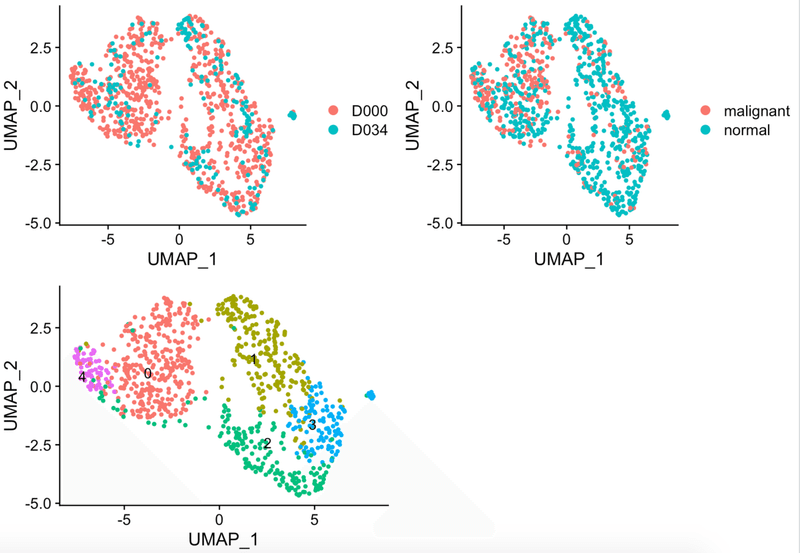
どのような細胞集団がいるのか?またタイムポイントや正常か白血病かでどう違うのかがよくわかると思います。
さらにはこんな可視化も面白いかもしれません。
v1 <- DimPlot(AML371_all.integrated , reduction = "umap", label = TRUE, split.by="stim")
v2 <- DimPlot(AML371_all.integrated , reduction = "umap", label = TRUE, split.by="mal")
v3 <- DimPlot(AML371_all.integrated , reduction = "umap", group.by="stim", split.by="mal")
v4 <- DimPlot(AML371_all.integrated , reduction = "umap", group.by="mal", split.by="stim")
plot_grid(v1, v2, v4, v3)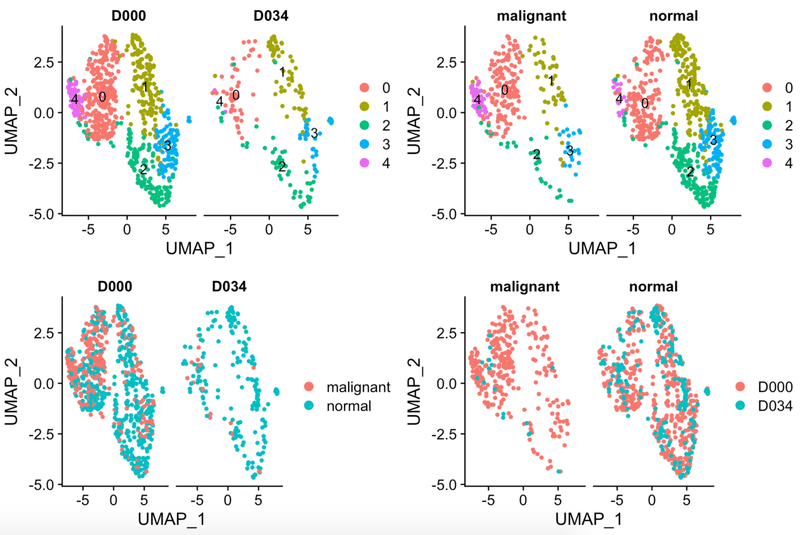
次にこれだけだと面白くないので、治療前後の正常または白血病細胞でどのように遺伝子が変化したのか取り出してみることにする。メタ情報として$malに正常か白血病か$stimにタイムポイントが入っているので、それらを統合して$mal.stimというメタ情報として格納する。
AML371_all.integrated$mal.stim <- paste(AML371_all.integrated$mal, AML371_all.integrated$stim, sep = "_")
AML371_all.integrated$mal.stim 出力はこんな感じ

このメタ情報をidentifier として正常もしくは白血病それぞれで治療前後で優位な遺伝子(特性)を探してみる。
#Before and after in leukemia
mal.chemo.response <- FindMarkers(AML371.integrated, ident.1 = "malignant_D034", ident.2 = "malignant_D000", verbose = FALSE)
#Before and after in Normal
normal.chemo.response <- FindMarkers(AML371.integrated, ident.1 = "normal_D034", ident.2 = "normal_D000", verbose = FALSE)試しに出力させてみると、白血病の方では
head(mal.chemo.response, n = 50)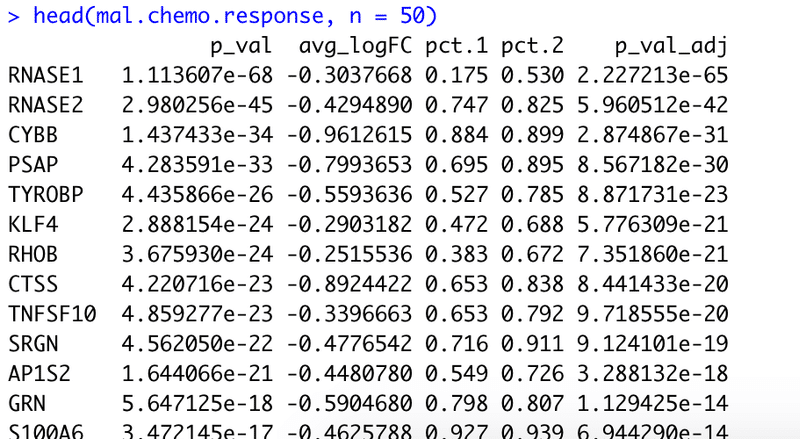
正常の方では
head(normal.chemo.response, n = 50)
といった感じでした。この後は色々と個々人で調べるということでしょうか?
ファイルに落として、一先ず解析おしまいです(さらに0−4のクラスタ毎で調べても面白いと思います)。
write.table(mal.chemo.response,"AML371_AML_chemo.txt",sep="\t")
write.table(normal.chemo.response,"AML371_normal_chemo.txt",sep="\t")といった形で割とバカチョンでささっと解析できてしまうのが、目から鱗でした。最近のテクノロジーは凄いです!
この記事が気に入ったらサポートをしてみませんか?
