Rを使った安定同位体比データ解析(Part 3)
はじめに
安定同位体比は、生体内の代謝や、食物網における食う–食われるの関係、生息地の移動など、わたしたちの身の回りで起きている様々な「物の流れ」を調べるツールとして用いられてきました。
近年、分析共同利用拠点や依頼分析サービスが増え、ますます多くの研究者に安定同位体比分析が使われていることと思います。
また、消費者の餌資源推定や栄養ニッチの算出など、安定同位体比データを用いた解析の幅が広がるとともに、
同位体研究において、Rを活用する機運が高まっていると感じています。
この記事は、Rの使い方を学びながら、
安定同位体比データをどうやって図示するか、どのように統計解析をおこなうか、Rのパッケージをどう動かすかを伝えることが目的です。
このページはRを使った安定同位体比データの解析シリーズの Part 3 です
Part 1 >>> データファイルの作り方 / 散布図の作り方
Part 2 >>> 箱ひげ図の作り方
Part 3 で学べること
Rで平均値や標準偏差を計算する方法
計算結果をcsvファイルで出力する方法
集計する
安定同位体関連の論文を見ていると、グループごとに平均±SDを集計した表や図が出てくるケースが非常に多いです。
Excel で計算しても良いですが、Rを使うことでかなり労力が抑えることができます。
Rを用いた平均値やSDの計算では、主に次の2つのコマンドが使われるのではないかと思います。
aggregate() を使う
dplyr パッケージを使う
この記事では、両方を紹介しつつ、用途に応じて適した方法を検討します。
なお、データセットはPart 1,2 と同じものを使います。
dt <- read.csv("Isotope.csv", header = T)
colnames(dt)[4] <- c("delta15N")
colnames(dt)[5] <- c("delta13C")
dt$delta15N <- round(dt$delta15N, digits = 1)
dt$delta13C <- round(dt$delta13C, digits = 1)
fish <- dplyr::filter(dt, Type == "Fish")aggregate関数で計算する
aggregateは、Rのデフォルトで用意されている関数です。
さっそく、安定同位体比の平均値とSDを魚種ごとに計算してみましょう。
aggregate(delta15N ~ Species, data = fish, FUN = mean)
aggregate(delta15N ~ Species, data = fish, FUN = sd)
解説
#1,2 aggregate() で平均値やSDを計算します。
関数のルールは、aggregate(数値型データが入った列 ~ 文字型データが入った列, data = データセット, FUN = 計算方法) です。
aggregate(delta15N ~ Species, data = fish, FUN = sd) はつまり「データセットfishに対して、delta15NのSDをSepciesごとに計算しなさい」というコマンドです。
FUN = では mean, sd のほかにも sum, summary などを指定できます。
また、計算の対象とする列を複数指定することも可能です。
aggregate(cbind(delta13C, delta15N) ~ Species, data = fish, FUN = mean)
aggregate(cbind(delta13C, delta15N) ~ Species, data = fish, FUN = sd)
解説
#3,4 aggregate(cbind(delta13C, delta15N) ~ Species, data = dt, FUN = ..)
コード#1,2とほぼ同じですが、"数値型が入った列"の箇所を cbind(delta13C, delta15N) としました。
これによって、delta13C と delta15N を一括でデータ操作することができます。
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
~Rの基礎を理解するコーナー~
#B1 cbind() は、2つ以上のデータ列(一次元)またはデータフレームを連結する関数で、「新しい列」としてデータを追加していきます。
以下のコードを実行して、cbind() で何が起きているかを確かめてみましょう。
> d1 <- c("Tomato","Tomato","Potato")
> d2 <- c("Beer","Beer","Chips")
> cbind(d1,d2)
d1 d2
[1,] "Tomato" "Beer"
[2,] "Tomato" "Beer"
[3,] "Potato" "Chips"データ列d1とd2はそれぞれ3つのデータを含みます。
cbind()によって、独立したデータ列d1とd2が連結し、3行2列のデータフレームが作られました。
#B2 ちなみに、rbind() という関数もあります。これは、2つ以上のデータ列(一次元)またはデータフレームを連結する関数で、「新しい行」としてデータを追加していきます。
rbind(d1,d2)
[,1] [,2] [,3]
d1 "Tomato" "Tomato" "Potato"
d2 "Beer" "Beer" "Chips" rbind()によって、独立したデータ列d1とd2が連結し、2行3列のデータフレームが作られました。
つまり、cbindは "column[列]にデータをbindする"、rbindは "row[行]にデータをbindする" ということです。
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
dplyrで計算する
ここからは、dplyrパッケージを活用してデータを集計する方法に移ります。
さっそく、以下のコードを実行してみましょう。
library(dplyr)
fish %>%
group_by(Species) %>%
summarise(mean(delta15N))
fish %>%
group_by(Species) %>%
summarise(sd(delta15N))
解説
#5 library(dplyr) でライブラリー"dplyr"を呼び出します。
以前のパートで dplyr を使った列または行抽出方法を紹介しましたが、データの集計でも大活躍します。
まだインストールが済んでいない場合は、install.packages("dplyr")でダウンロードしましょう。
#6,7 fish %>% group_by() %>% summarise() でグループ分けと計算を一括で実行します。
たとえば fish %>% group_by(Species) %>% summarise(mean(delta15N)) は、
「データセット fish を Species 列に格納されているIDにしたがってグループ分けし、 delta15N 列のデータの mean を計算しなさい」ということです。
summarise () で自由に計算方法を指定できます。
また、複数のデータ列を指定し、複数タイプの計算を一括で実行することも可能です。
たとえば、以下のようにfunctionを増やします。
fish %>%
group_by(Species) %>%
summarise(mean(delta13C), sd(delta13C), mean(delta15N), sd(delta15N))
解説
#8 解説するまでもないかもしれませんが、 summarise() でデータ列や計算方法(function)を追加すれば OKです。
aggregate() と dplyr どちらを使うべきか?
結論から言うと、dplyrの使用を推奨します。
まず、計算速度が aggregate() よりも dplyr で速いことが挙げられます。
また、お気づきの方もいるかもしれませんが、
aggregate では 複数のfunctionを一括で実行できません。
mean と sd を計算する際、コードを分けて実行する必要があり、少し面倒です。
計算結果をcsvファイルで保存する
最終的に論文で結果を示すことを想定して、計算結果を .csv 形式で保存する方法を紹介します。
その前に、dplyrでの計算結果をデータフレームに保管しましょう。
また、計算結果を確認すると、あることに気が付きます。
困ったことに、安定同位体比データの小数がかなり遠いところまで続いています。
Part 1 で説明したように、安定同位体比分析結果は、小数第1位までしか使えません。
小数第2位以降を切り捨てる作業を忘れずにおこないましょう。
fish_m_s <- fish %>%
group_by(Species) %>%
summarise(mean(delta13C), sd(delta13C), mean(delta15N), sd(delta15N))
colnames(fish_m_s) <- c("Species","Mean_d13C","SD_d13C","Mean_d15N","SD_d15N")
fish_m_s$Mean_d13C <- round(fish_m_s$Mean_d13C, digits = 1)
fish_m_s$SD_d13C <- round(fish_m_s$SD_d13C, digits = 1)
fish_m_s$Mean_d15N <- round(fish_m_s$Mean_d15N, digits = 1)
fish_m_s$SD_d15N <- round(fish_m_s$SD_d15N, digits = 1)
write.csv(fish_m_s, "SIA_mean_sd.csv", row.names = F)
解説
#9 コード#8と同じですが、計算結果を"fish_m_s"というオブジェクトで保管しておきます。
#10 colnames() で、データフレームの列名を変更します。
これは必須の作業ではありませんが、fish %>% group_by() %>% summarise() の集計表そのままだと
列名が 「Species `mean(delta13C)` `sd(delta13C)` `mean(delta15N)` `sd(delta15N)`」となり、特に``の部分が扱いづらいです。
このため、列名を扱いやすい(理解しやすい)文字列に置き換えます。
関数のルールは、colnames(データセット) <- c("新しい列名", "新しい列名", ,,) です。
#11-14 round(データ, digits = 1) で、データを小数第1位で揃えます。
※round() 関数の詳細は Part 1 参照
#15 write.csv() で、任意のデータフレームを.csv形式で保存します。
関数のルールは、write.csv(データセット, "ファイルの名前.csv", row.names = F) です。
row.names = F はあってもなくても良いです。
何も指定しないと、出力されたcsvファイルの1列目に数字(行番)が入ります。
今回のような用途では不要だと思うので、行番が入らないように row.names = F としました。
#11-14のコードは、以下のようにfor文を使って行数を省略してもOKです。
for (i in 2:5) {
fish_m_s[,i] <- round(fish_m_s[,i], digits = 1)
}基本は以上です。
応用編
ここからは応用編です。余力のある方はぜひ読んでみてください。
"平均±SD" 表記で出力する
これまでに紹介した方法では、平均とSDの集計結果がそれぞれ別の列に入っていました。
一方、論文等で見かける結果の表は "mean±SD" 表記になっていることが多いと思います。
Excelで "mean±SD" 表記に編集しても良いのですが、地味に面倒な作業だと思います。
これもRで編集してしまいましょう。
作業プロセスを要約すると、
①魚種ごとにδ13Cとδ15Nの平均とSDを計算し、計算結果をデータフレームで保存
②δ13Cの平均とSDを小数部を整えたあと、"平均", "±", "SD" を連結して一つのカラムに保存
③δ15Nの平均とSDを小数部を整えたあと、"平均", "±", "SD" を連結して一つのデータ列に保存
④②と③のデータ列を連結して、一つのデータフレームとして保存
⑤④のデータフレームを.csv形式で保存
となります。それでは、コードで表してみましょう。
#fish_m_s <- fish %>%
# group_by(Species) %>%
# summarise(mean(delta13C), sd(delta13C), mean(delta15N), sd(delta15N))
d13C <- c()
d15N <- c()
for (i in 1:length(fish_m_s$Species)) {
tem_d13C <- paste(round(fish_m_s[i,2], digits = 1),
round(fish_m_s[i,3], digits = 1),
sep = "±")
d13C <- c(d13C, tem_d13C)
tem_d15N <- paste(round(fish_m_s[i,4], digits = 1),
round(fish_m_s[i,5], digits = 1),
sep = "±")
d15N <- c(d15N, tem_d15N)
}
fish_table <- data.frame(Species = fish_m_s$Species,
delta13C = d13C,
delta15N = d15N)
write.csv(fish_m_s, "SIA_mean±sd.csv", row.names = F)
解説
#16,17 d13C <- c(), d15N <- c() で、空のデータ列を作成します。
δ13Cやδ15Nの出力結果 "平均", "±", "SD" を保存するためのデータ列をあらかじめ宣言しておきます。
#18 for 文の設定
1:length(fish_m_s$Species) はつまり1:8を表しており、魚種の数だけ試行を繰り返します。
#19 paste(round(fish_m_s[i,2], digits = 1), round(fish_m_s[i,3], digits = 1), sep = "±")
まず、fish_m_s[i,2]は、データフレームfish_m_sのi行2列にアクセスしなさいというコマンドです。
つまり、i番目の魚種の"mean(delta13C)" 列にアクセスします。
同様に、fish_m_s[i,3]では、i番目の魚種の"sd(delta13C)" 列にアクセスします。
次に、round(データ, digits = 1)で、データを小数第1位で揃えます。(コード#11-14と同じ)
最後にpaste()関数について説明します。
paste()は、()の中の要素を(文字型として)連結します。
以下に例を示します。
paste("apple","pen")
[1] "apple pen"
paste("pineapple","pen","apple","pen")
[1] "pineapple pen apple pen"また、paste()では、sep =で要素と要素の間をどのように連結するかを指定できます。
以下に例を示します。
paste("Tackey","Tsubasa", sep = "&")
[1] "Tackey&Tsubasa"
paste("CX", 5, sep = "-")
[1] "CX-5"今回のコードの場合、paste(round(fish_m_s[i,2], digits = 1), round(fish_m_s[i,3], digits = 1), sep = "±")
δ13Cの出力結果 "平均" と "SD" を"±" で連結することになります。
この文字列("平均 ± SD")をtem_d13Cというデータ列で保管しておきます。
#20 d13C <- c(d13C, tem_d13C)
コード#19でi番目の魚種の"平均 ± SD"を計算し、"tem_d13C"にデータを格納しました。
このままでは、データが「上書き」され、i+2番目の試行が終わった時点で"tem_d13C"にあった "i番目の魚種の平均 ± SD"は消えてしまいます。
そこで、出力結果を都度、データ列(コード#16)に並べていきます。
これによって、i~i+n番目のデータをすべて保存できます。
#21,22
δ15Nでも同様の作業を行います(コード#19,20)
詳細な説明は省略しますが、fish_m_s[i,4]ではi番目の魚種の"mean(delta15N)" 列に、
fish_m_s[i,5]ではi番目の魚種の"sd(delta15N)" 列にアクセスします。
#23 data.frame(Species = fish_m_s$Species, delta13C = d13C, delta15N = d15N)
δ13Cの"平均 ± SD"列とδ15Nの"平均 ± SD"列を一つのデータフレームとして保存します。
data.frame() 関数のルールは、data.frame(列名1 = データ列1, 列名2 = データ列2,,,) です。列名を改めて指定できます。
データフレームを fish_table で保管します。
#24 write.csv(fish_m_s, "SIA_mean±sd.csv", row.names = F) で、出力結果をcsv形式で保存します(コード#15と同じ)
平均±SDの散布図を作成する
最後に、平均とSDデータで散布図を作成する方法を紹介します。
#fish_m_s <- fish %>%
# group_by(Species) %>%
# summarise(mean(delta13C), sd(delta13C), mean(delta15N), sd(delta15N))
library(ggplot2)
ggplot(fish_m_s, aes(x = Mean_d13C, y = Mean_d15N, colour = Species)) +
geom_point(size = 3) +
geom_errorbar(aes(ymin = Mean_d15N - SD_d15N,
ymax = Mean_d15N + SD_d15N),
data = fish_m_s,
width = 0.05,
colour = "black",
alpha = 0.5) +
geom_errorbarh(aes(xmin = Mean_d13C - SD_d13C,
xmax = Mean_d13C + SD_d13C),
data = fish_m_s,
height = 0.05,
colour = "black",
alpha = 0.5)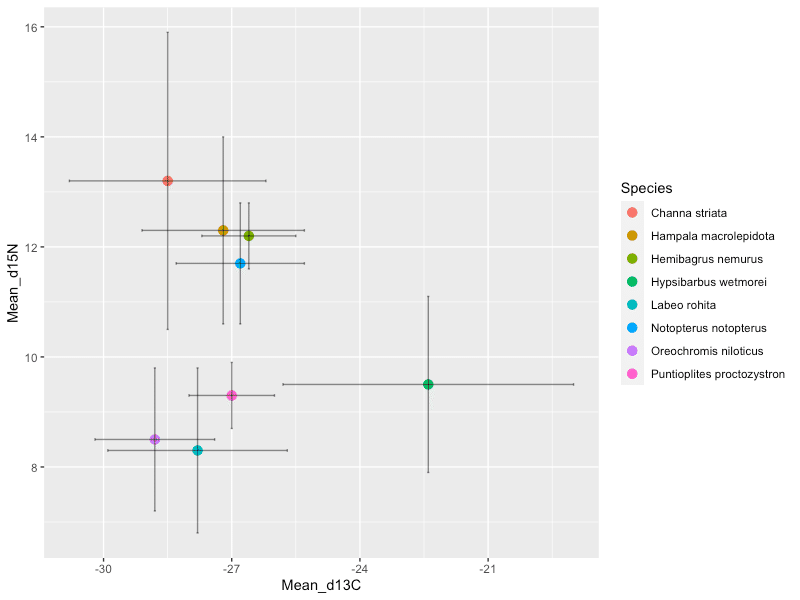
解説
#25 library(ggplot2) で、ggplot2ライブラリーを呼び出します。
#26 ggplot(fish_m_s, aes(x = Mean_d13C, y = Mean_d15N, colour = Species)) + geom_point(size = 3)
で散布図を作ります。
データセットは、コード#9 で作成したデータフレーム fish_m_s を使用します。
ここまででまず、各魚種の平均値のプロットを設定できました。
ここから、 + でコマンドを追加していきます。
+ geom_errorbar() で、Y軸方向のエラーバーを設定します。
ここで、各魚種におけるδ15Nの平均値とSDの集計結果を使います。
関数のルールは、ggplot()とだいたい同じです。
aes(ymin = , ymax = ) でバーの範囲を指定します。
また、width = でエラーバーの幅を、colour = でエラーバーの色を、alpha = でエラーバーの透明度を指定します。
+ geom_errorbarh() で、X軸方向のエラーバーを設定します。
ここで、各魚種におけるδ13Cの平均値とSDの集計結果を使います。
関数のルールは+ geom_errorbar() と同じです。
散布図の説明は以上です。
Part 1 の散布図では、全データをプロットしましたが、データ数が多いと少し見づらいかもしれません。
そのような場合は、平均とSDで図示することを検討しても良いと思います。
***************************************************
ご不明な点があれば、お気軽にご連絡ください。
***************************************************
>> Part 4 統計解析
おまけ
平均±SDの散布図を作成する / 食性ごとに色分けする
dplyrでδ13Cとδ15Nの平均値およびSDを集計した時点で、データフレーム (fish_m_s ) のカテゴリカルな情報は"Species"しか残されていませんでした。
この状態では、散布図の作成で"Species"の情報をもとにした色分けしかできません。
そこで、大元のデータセット fish から必要な情報を抽出してデータフレーム (fish_m_s ) に結合し、散布図の色分けパターンを増やしてみます。
今回は、食性(Feeding.habits列)を抽出し、データフレームに結合します。
id <- unique(fish_m_s$Species)
feeding <- c()
for (i in 1:8) {
id_name <- id[i]
da <- fish %>% filter(Species == id_name)
feeding_temp <- da %>% select(Feeding.habits)
feeding_temp <- feeding_temp[1,]
feeding <- c(feeding,feeding_temp)
}
fish_m_s_ <- cbind(fish_m_s,feeding)
cols1 <- c("#fb6d5c","#57ae26","#29a8bd","#8890fc")
ggplot(fish_m_s_, aes(x = Mean_d13C, y = Mean_d15N, colour = feeding)) +
geom_point(size = 3) +
geom_errorbar(aes(ymin = Mean_d15N - SD_d15N,
ymax = Mean_d15N + SD_d15N),
data = fish_m_s_,
width = 0.05,
colour = "black",
alpha = 0.5) +
geom_errorbarh(aes(xmin = Mean_d13C - SD_d13C,
xmax = Mean_d13C + SD_d13C),
data = fish_m_s_,
height = 0.05,
colour = "black",
alpha = 0.5) +
scale_color_manual(values=cols1)+
theme_light() +
theme(panel.grid=element_blank()) +
xlab(expression({delta}^13*C~'\u2030')) +
ylab(expression({delta}^15*N~'\u2030'))
(コードの解説が必要な場合はお声掛けください)
この記事が気に入ったらサポートをしてみませんか?
