Rを使った安定同位体比データ解析(Part 2)
はじめに
安定同位体比は、生体内の代謝や、食物網における食う–食われるの関係、生息地の移動など、わたしたちの身の回りで起きている様々な「物の流れ」を調べるツールとして用いられてきました。
近年、分析共同利用拠点や依頼分析サービスが増え、ますます多くの研究者に安定同位体比分析が使われていることと思います。
また、消費者の餌資源推定や栄養ニッチの算出など、安定同位体比データを用いた解析の幅が広がるとともに、
同位体研究において、Rを活用する機運が高まっていると感じています。
この記事は、Rの使い方を学びながら、
安定同位体比データをどうやって図示するか、どのように統計解析をおこなうか、Rのパッケージをどう動かすかを伝えることが目的です。
このページはRを使った安定同位体比データの解析シリーズの Part 2 です
Part 1 >>> データファイルの作り方 / 散布図の作り方
Part 2 で学べること
Rで安定同位体比データから箱ひげ図を作る方法
dplyrを使ったデータ操作
for文の活用方法
箱ひげ図の作成
グループ(種、年、地点)間で安定同位体比に差があるかを調べる際、箱ひげ図がよく使われると思います。
ここでは、δ13Cとδ15Nを魚種ごとに箱ひげ図で図示する方法を解説します。
なお、"複数グループの箱ひげ図" のコードはやや応用的ですので、読み飛ばしても構いません。
基本プロットの作成
まずは、基本操作を覚えましょう。
ためしに、データセットから魚類のデータのみを抽出し、δ13Cとδ15Nを箱ひげ図で出力します。
なお、データセットはPart 1と同じものを使います。
##### データセット #####
#dt <- read.csv("Isotope.csv", header=T)
#colnames(dt)[4] <- c("delta15N")
#colnames(dt)[5] <- c("delta13C")
#dt$delta15N <- round(dt$delta15N, digits = 1)
#dt$delta13C <- round(dt$delta13C, digits = 1)
##### ここから #####
library(dplyr)
fish <- dplyr::filter(dt, Type == "Fish")
boxplot(fish$delta15N)
解説
#1 library(dplyr) でライブラリー"dplyr"を呼び出します。
dplyrは、データ操作や集計で大活躍するRのパッケージです。
まだインストールが済んでいない場合は、install.packages("dplyr")でダウンロードしましょう。
#2 fish <- dplyr::filter(dt, Type == "Fish")
dplyr::filter() で、任意の列から条件が一致する行を抽出します。
関数のルールは、dplyr::filter(データセット, 列名 == 変数) です。
データセットdt のType 列のなかに、グループ(Fish, Aquatic Insect, Periphytonなど)の情報が入っています。
dplyr::filter(dt, Type == "Fish") とすることで、Fish が入っている行のみを抽出します。
ここで抽出したデータを fish という箱で保管しておきます。
#3 boxplot(fish$delta15N)
boxplot(データ列1, データ列2,,, )で、箱ひげ図を出力します。
ここでは、fish$delta15Nに含まれるデータをすべて使って箱ひげ図を作りました。
ちなみに、boxplot()では、複数のデータ列を指定できます。
たとえば、boxplot(fish$delta15N, fish$delta13C) を実行すると、箱ひげ図が2つ出力されます。
2グループの箱ひげ図
さて、δ13Cとδ15Nを魚種ごとに箱ひげ図を作るためには、
①魚種ごとにデータセットを分割し、さらに
②δ13Cのみが格納されたデータ列とδ15Nのみが格納されたデータ列に分割し
③boxplot(データ列1, データ列2,,, )で箱ひげ図を出力
というプロセスをこなす必要があります。
まずは、①~③のプロセスを理解するために次の作業をこなしてみましょう。
シナリオ
データセット dt には、2種の植物食性魚類 [herbivore] が含まれている。植物食性魚類のδ13Cとδ15Nを箱ひげ図で図示し、データの範囲を比較する。
herbivore1 <- dplyr::filter(fish, Species == "Oreochromis niloticus")
herbivore2 <- dplyr::filter(fish, Species == "Hypsibarbus wetmorei")
boxplot(herbivore1$delta15N,herbivore2$delta15N)
boxplot(herbivore1$delta13C,herbivore2$delta13C)

解説
#4,5
dplyr::filter() で、任意の列から条件が一致する行を抽出します。(コード#2と同じ)
データセットdt のSpecies 列のなかに、魚種の情報が入っています。"Oreochromis niloticus"と"Hypsibarbus wetmorei"が植物食性魚類です。
dplyr::filter(fish, Species == "Oreochromis niloticus") または dplyr::filter(fish, Species == "Hypsibarbus wetmorei") とすることで、各魚種のデータのみを抽出します。
ここで抽出したデータをそれぞれ、herbivore1とherbivore2 という箱で保管しておきます。
#6,7
boxplot(データ列1, データ列2,,, )で、箱ひげ図を出力します。(コード#3と同じ)
boxplot(herbivore1$delta15N,herbivore2$delta15N) では、herbivore1とherbivore2 の δ15Nを使って箱ひげ図を
boxplot(herbivore1$delta13C,herbivore2$delta13C)では、herbivore1とherbivore2 の δ13Cを使って箱ひげ図を作成します。
①~③のプロセスをイメージできたでしょうか?
複数グループの箱ひげ図
ここからは、全魚種のデータセットを使って箱ひげ図を作っていきましょう。
データセット dt には、8魚種のデータが含まれています。
コード#4,5のように、dplyr::filter() で各魚種のデータを抽出できることがわかりました。
しかし、今回は8魚種あるので、dplyr::filter() を8回も実行するのはなかなか面倒に感じます。
そこで、少し応用的な内容にはなりますが、for文を活用しましょう。
species <- unique(fish$Species)
list_c <- list()
list_n <- list()
for (i in 1:length(species)) {
spe <- species[i]
da <- dplyr::filter(fish, Species == spe)
list_n[[i]] <- da$delta15N
list_c[[i]] <- da$delta13C
}
names(list_n) <- species
names(list_c) <- species
boxplot(list_n, las = 2)
boxplot(list_c, las = 2)

解説
#8 species <- unique(fish$Species)
unique() で、任意の列から重複無しでIDを抽出します。
unique(fish$Species)
[1] "Puntioplites proctozystron" "Notopterus notopterus"
[3] "Channa striata" "Hampala macrolepidota"
[5] "Hemibagrus nemurus" "Oreochromis niloticus"
[7] "Hypsibarbus wetmorei" "Labeo rohita" fish$Species のデータ列には魚種名の情報が入っており、
全部で8種類、計157個のデータが格納されています。
イメージができない場合は、コンソールで fish$Species を実行してみてください。以下が出力結果(一部抜粋です)
fish$Species
[1] "Puntioplites proctozystron" "Puntioplites proctozystron"
[3] "Puntioplites proctozystron" "Puntioplites proctozystron"
[5] "Puntioplites proctozystron" "Notopterus notopterus"
[7] "Notopterus notopterus" "Notopterus notopterus"
[9] "Notopterus notopterus" "Notopterus notopterus"
...
[157] "Puntioplites proctozystron"今回は、「どんなタイプの魚種がいるのか?」がわかれば良く、157個すべての情報を保管しておく必要はないため、
unique(fish$Species) を使いました。
unique(fish$Species) の情報を species に保管しておきます。
#9,10
list() で、空のリストを作成します。
先ほど、boxplot()関数は boxplot(データ列1, データ列2,,, ) のように使うと解説しましたが、実は boxplot(リスト形式データ) としても使えます。
ということで、各魚種におけるδ13Cとδ15Nのデータをリスト形式のデータに保管しておき、
あとでまとめてboxplot(list)として箱ひげ図を出力しようという算段です。
#11
ここからは、for文の記述です。ここでの目標をとても簡単に説明すると、
「魚種の数だけdata <- dplyr::filter() を実行する」
ということです。
今回、魚種数は8種類あります。unique(fish$Species) の出力結果を目視でカウントし、for (i in 1:8) としてもOKですが、見落としがあると怖いので、
列数をカウントしてくれるコード length() を使いました。
文章だけでは理解しづらいかもしれないので、次のコードを実行してみてください。
length(species)
[1] 8for (i in 1:length(species)) はつまり for (i in 1:8) を表しています。
#12 spe <- species[i] で、speciesからi番目のデータを抽出します。
for文のカウンターが i =1 のとき、species[1] が実行されます。
そして、speciesの1番目に格納されているデータは"Puntioplites proctozystron"ですので、
spe には "Puntioplites proctozystron" が入ることになります。
#13
da <- dplyr::filter(fish, Species == spe) で、 データセットfishから、コード#12で抽出した種名に一致する行のみを抽出します。
(つまり、 i =1 のとき、"Puntioplites proctozystron" のデータが抽出される)
作業内容そのものは、コード#2,3,4と同じです。
#14,15
list_n[[i]] <- da$delta15N
list_c[[i]] <- da$delta13C で、各魚種の δ15Nとδ13C のデータをリストに格納します。
例として、list_cの中身を確認してみます。
抽出したδ13C のデータが、試行回ごと分割して納められています。

#16,17
names(list_n) <- species
names(list_c) <- species で、リストの名前を置換します。
各リストの名前は1~8の数値で与えられていましたが、これだと魚種との対応が分かりづらいです。
names(list_n) <- species とすることで、species列(魚種のIDが入ったデータ列)の情報に置き換えてくれます。

#18,19
boxplot(list_n, las = 2)
boxplot(list_c, las = 2) で、箱ひげ図を作成します。
las = 2 とすることで、横軸のラベルの向きを変えてくれます。
これで、全魚種のデータセットを使って箱ひげ図を出力することができるようになりました。
正直、ちょっと大変だなあと思われるかもしれません。
次の項目で紹介する ggplot2を活用したコードでは、もっと簡単に複数グループの箱ひげ図を出力できます。
ggplot2 を活用する
ここからは、ggplot2を使った作図方法に移ります。
Part 1 で説明したように、ggplot2は Rでもっとも使われるデータ可視化ツールです。
これまでに作成した図を ggplot2版で作ってみましょう。
library(ggplot2)
ggplot(fish, aes(x = Species, y = delta15N)) +
geom_boxplot()
ggplot(fish, aes(x = Species, y = delta13C)) +
geom_boxplot()

解説
#20 library(ggplot2) でライブラリー"ggplot2"を呼び出します。
※コード#2で作成した fish データセットを使います
#21,22
ggplotで箱ひげ図を作成します。関数のルールは、
ggplot(データセット, aes(x = 文字型データ, y = 数値型データ)) + geom_boxplot() です。
ggplot(fish, aes(x = Species, y = delta15N)) + geom_boxplot() とすることで、データセットfishに対して、種ごとにδ15Nの箱ひげ図を描画してくれます。
たった1行のコードで、魚種ごとのデータ分布を示す箱ひげ図を出力できました。
応用編
この先は、ggplotで作成した箱ひげ図の体裁を整える方法を紹介します。
ラベルの向きと順番を変える
ggplot(fish, aes(x = Species, y = delta15N)) +
geom_boxplot() +
theme(axis.text.x = element_text(angle = 45, hjust = 1, size = 12)) +
scale_x_discrete(limits = c("Channa striata",
"Notopterus notopterus",
"Hemibagrus nemurus",
"Hampala macrolepidota",
"Puntioplites proctozystron",
"Oreochromis niloticus",
"Hypsibarbus wetmorei",
"Labeo rohita")) 
解説
#23 コード#21,22 と同じです。これ以降、+ でコマンドを追加していきます。
#24 theme(axis.text.x = element_text(angle = 45, hjust = 1, size = 12)) で、X軸のラベルの向きとフォントサイズを調整します。theme() にはたくさんの機能が備わっているのですが、ここでは説明を省きます。
angle = で文字列の向きを調節します。これだけだと、文字がプロットと重なってしまうため、hjust = を設定して文字列の位置を下方向にずらしました。
#25 scale_x_discrete(limits = c("文字型データ", "文字型データ", ,, "文字型データ") で、箱ひげ図を並べる順番を指定します。
注意点は、ggplot(fish, aes(x = Species, y = delta15N)) の x = で指定した列(ここではSpecies)に含まれるIDを、scale_x_discrete() で一つも残さずに記述することです。そうでないとエラーが出ます。
色を変える
cols1 <- c("#fb6d5c","#57ae26","#29a8bd","#8890fc")
ggplot(fish, aes(x = Species, y = delta15N, fill = Feeding.habits)) +
geom_boxplot() +
theme(axis.text.x = element_text(angle = 45, hjust = 1, size = 12)) +
scale_x_discrete(limits = c("Channa striata",
"Notopterus notopterus",
"Hemibagrus nemurus",
"Hampala macrolepidota",
"Puntioplites proctozystron",
"Oreochromis niloticus",
"Hypsibarbus wetmorei",
"Labeo rohita")) +
scale_fill_manual(values=cols1)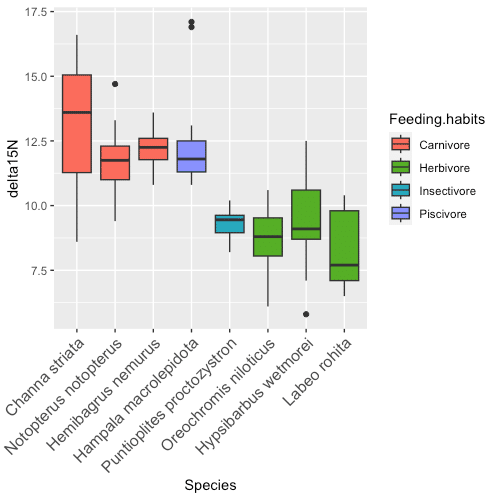
解説
#26 カラーパレットを作成します。
#27 コード#21,22 とほぼ同じですが、 fill = Feeding.habits を追加しました。これによって、Feeding.habits列のIDにしたがって箱ひげ図の色をわけてくれます。もし、種ごとに色分けをしたい場合は、fill = Species とします。
#28,29 コード#24,25と同じ
#30 scale_fill_manual(values=cols1) で、カラーパレットを指定します。
ちなみに、箱ひげ図の塗りつぶしの色ではなく、枠線の色を変えたい場合は、
ggplot(fish, aes(x = Species, y = delta15N, color = Feeding.habits)) + scale_color_manual(values=cols1)
を実行します。つまり、fill ではなく color を使います。
カラーパレットの作成や、ggplotでのカラーパレットの指定については、Part 1で詳しく説明しました。
概念がよくわからなかったという人は、Part 1 の内容をご確認いただければと思います。
また、軸ラベル(delta15Nなど)の変更や高画質で画像を保存する方法についても、Part 1 をご参照いただけると幸いです。
***************************************************
ご不明な点があれば、お気軽にご連絡ください。
***************************************************
この記事が気に入ったらサポートをしてみませんか?
