Rを使った安定同位体比データ解析(Part 1)
はじめに
安定同位体比は、生体内の代謝や、食物網における食う–食われるの関係、生息地の移動など、わたしたちの身の回りで起きている様々な「物の流れ」を調べるツールとして用いられてきました。
近年、分析共同利用拠点や依頼分析サービスが増え、ますます多くの研究者に安定同位体比分析が使われていることと思います。
また、消費者の餌資源推定や栄養ニッチの算出など、安定同位体比データを用いた解析の幅が広がるとともに、
同位体研究において、Rを活用する機運が高まっていると感じています。
この記事は、Rの使い方を学びながら、
安定同位体比データをどうやって図示するか、どのように統計解析をおこなうか、Rのパッケージをどう動かすかを伝えることが目的です。
※このページはRを使った安定同位体比データ解析シリーズの Part 1 です※
Part 1 で学べること
Rで操作するためのデータファイルの作り方
RとRstudioの使い方と、Rで頻出のコード
安定同位体比データで散布図を作る方法
それでは、はじめていきましょう
準備する
データファイルの作成
まずは、データファイルの準備をしましょう。
Excel アプリケーションで以下のようなデータセットを作成してください。

最低限必要なデータは炭素安定同位体比(δ13C)と窒素安定同位体比(δ15N)ですが、
ほかにも個体IDや、種名、採取年などがデータファイルに含まれていても、まったく問題ありません。
データのファイル形式は、 ".csv" が好ましいです。
なお、この記事で用いるサンプルデータセットは、以下のリンクから入手できます。
データセットの説明
ある湖で採取したジャイアントなナマズと、その湖の食物網を構成する生物[魚類9種、水生昆虫、付着藻類、POMなど] の炭素・窒素安定同位体比のデータが含まれる。
ナマズ以外の魚類には、食性の情報が付与されている。
Datasets_ver.2.1.xlsx の "Stable Isotope ratios" タブからデータをコピーし、別ウィンドウにデータを貼り付けて、"Isotope.csv" と名前を付けて保存します。
4列目"Corr. δ15N (‰)"が窒素安定同位体比、
5列目"Corr. δ13C (‰)"が炭素安定同位体比のデータです。
Rのインストール
ここからは、Rの操作に移ります。
もしRのインストールが済んでいない場合は、以下のサイトからダウンロードしてください。
Rstudio での操作が便利なので、ぜひこちらも使ってみてください。
RとRstudioのインストール方法は、こちらのサイトに丁寧な説明があります。
インストール方法に自信がない方は要チェックです。
RStudioの操作
簡単にRstudioの操作方法を説明しておくと・・・

左上のボックスはただのメモです。ここにコードを書いても、何も起きません。
左下のボックスがコンソールで、コードの入力欄(&出力欄)です。
コンソールに書かれたコード(命令)にしたがってRが動き、そしてRがここにお返事を書いてくれます。
コンソールに書いてあるコードや出力結果は、コードを実行している間にどんどん流れていってしまうので、
必要なコードはメモ欄に書きためておくと良いでしょう。
また、いちいちコードをメモ欄からコンソールにコピペする必要はありません。
メモ欄で、任意のコードの行にカーソルをあわせておき、
Macの場合は "command + Enter"、Windowsの場合は "control + Enter" とすれば、
メモ欄のコードがコンソールに入力されます。
データセットを読み込む
それでは、Rでcsvファイルを読み込んでみましょう。
以下のコードを実行してください。
dt <- read.csv("Isotope.csv", header=T)(↑のコードをメモ欄にコピペし、この行にカーソルをあわせて、"command + Enter"です!)
解説
#1 read.csv() で、csvファイルをRに読み込みます。
今回のデータセットでは、一行目に"列名"が入っています。

列名の情報は、R上でデータファイルを操作する際には必要ですが、データ解析の際には不要な情報です。
そこで、"header = T" を指定します。これによって、列名の情報を維持したまま、2行目以降のデータのみを解析に用いることができます。
そして、読み込んだデータセットを"dt"という箱に入れて保管しておきます。
read.csv("Isotope.csv", header=T) を実行するだけだと、
csvデータを読み込んだログがR上に残るものの、後からcsvデータを「思い出させる」ことが難しいです。
dt <- read.csv("Isotope.csv", header=T) としておけば、その後は "dt" と命令するだけで、Rさんはデータセットのことを思い出してくれます。
また、データセットをいったん何かしらの箱に入れておくことで、
データをさらに別の箱に仕分ける作業が楽になるというメリットもあります。
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
~Rの基礎を理解するコーナー~
#B1 "<-" で、データを代入します。
たとえば以下のように使います。
age <- 19
age
[1] 19
apple <- "pen"
apple
[1] "pen"つまり dt <- read.csv("Isotope.csv", header=T) は、「Isotope.csvのデータ」を「dtというオブジェクト(箱)」に代入する、というコマンドです。
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
安定同位体比データを整える
ここからは、Rでデータセットを整形します。
具体的には、安定同位体比データの桁数を整えます。
測定機器(EA-IRMS)のメーカーや研究機関が保証する安定同位体比測定の精度は、
だいたい小数第1位まで(δ13Cで±0.3‰…?)だと思います。
特にワーキングスタンダードで補正したあとの安定同位体比データを見ると、
小数点8位くらいまでの情報を持っているように見えます。
しかし、実際には、小数第2位より小さい部分は測定精度が保証されていないため、研究成果としては使えない情報です。
したがって、データ解析に進む前に、小数点の繰り上げ作業をおこないます。
Excel アプリケーション上でこの処理をおこなっても良いのですが、Rのコマンドでも簡単にできます。
colnames(dt)[4] <- c("delta15N")
colnames(dt)[5] <- c("delta13C")
dt$delta15N <- round(dt$delta15N, digits = 1)
dt$delta13C <- round(dt$delta13C, digits = 1)解説
#2,3 colnames() で、列名を変更します。
関数のルールは、colnames(データセット) <- c("新しい列名") です。
colnames(dt)では、dtのすべての列名を取得します。colnames(dt)[4]とすることで、dtの4列目だけにアクセスします。
この作業をおこなう理由は、
4列目の名前にはカッコや‰などRで表示できない文字が含まれており、列名指定の際に扱いにくいからです。
列名変更前の状態で4,5 列目を読み込むと、dt$Corr..δ15N....またはdt$Corr..δ13C....と表示されます。
ということで、データセットを作成する際には、文字化け対策のためにも
アルファベットのみを使って列名を付与すると良いでしょう。
#4,5 round() で、任意の数値データを指定の桁で四捨五入します。
関数のルールは、round(データ, digits = 桁の位置) です。
ここでは安定同位体比データを少数第1位で表したいので、round(dt$delta15N, digits = 1) とします。
※サンプルデータセットでは、もともと安定同位体比データが小数第1位の数値で格納されています。
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
~Rの基礎を理解するコーナー~
#B2 dt$delta15N は、データセット"dt"の列"delta15N"にアクセスしなさい、というコマンドです。
dt$delta15N <- round(dt$delta15N, digits = 1) とすることで、「データセット"dt"の列"delta15N"」に「小数第1位で四捨五入したデータセット"dt"の列"delta15N"データ」を代入(置換)できます。
#B3 c() は、指定した情報をベクトルかリストの形式で結合してくれる関数です。Rの操作で非常によく使われます。
c("pen","pineapple","apple","pen")
[1] "pen" "pineapple" "apple" "pen" このように、任意のカテゴリカルデータや数値データを結合して、ひとつのデータ列を作ります。
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
データセットの準備はここまで。
この先は、安定同位体比データを使って作図する方法の紹介です。
散布図の作成
それでは、論文でもっともよく使われているであろう
δ13C–δ15Nの二次元プロット(散布図)を作ってみましょう。
基本プロットの作成
まずは、もっともシンプルな散布図を作ります。
plot(dt$delta13C, dt$delta15N)
解説
#6 plot() で、二次元のプロットを作成します。
関数のルールは、plot(X軸データ,Y軸データ) です。
plot(5,3) や、plot(c(-23,-25,-25,-30),c(5,8,9,3)) のように、データを手入力してもOKです。
plot(dt$delta13C, dt$delta15N) とすれば、
データセット dtのdelta13C列と dtのdt$delta15N列におさまっている
データを一行ずつあたりながらプロットしてくれます。
とりあえず基本的なプロットができました。
ここから、図の体裁を整えていきます。
プロットの色や形を変更する
データセットには魚や昆虫など様々な生物のデータが含まれています。
このグループごとに安定同位体比データの傾向をつかむため、
プロットの色や形を変えてみましょう。
library(RColorBrewer)
type <- as.numeric(as.factor(dt$Type))
cols <- brewer.pal(9, "Set3")
shapes <- c(1:9)
plot(dt$delta13C, dt$delta15N,
col = cols[type],
pch = shapes[type])
解説
#7 library(RColorBrewer) で、RColorBrewerライブラリーを呼び出します。
まだインストールが済んでいない場合は、install.packages("RColorBrewer") を実行してください。
RColorBrewerはカラーパレットの作成で役立ちます。
RColorBrewerライブラリーの詳細は、以下のサイトをご参照ください。
#8 type <- as.numeric(as.factor(dt$Type))
グループIDの情報は、 dt データセットの Type 列に入っています。
ただし、Type 列のデータはすべて文字型です。
これだと後のプロット作成において不都合なので、データ型を変更します。最終的な目標は、数値型に変更することです。
Rでは、文字型を直接数値型に変換することができないため、文字→ファクター→数値の順番で、データ型を変換します。
数値型になった Type 列の情報を、"type" という箱に保管しておきます。
※
種ごとに色分けしたい場合はdt$Species、採取年ごとに色分けしたい場合はdt$Year とします
#9 cols <- brewer.pal(9, "Set3") で、カラーパレットを作成します。
関数のルールは、brewer.pal(色の数, "パレット名") です。
RColorBrewerライブラリーでは、様々なタイプのカラーパレットが用意されています。
今回、"Set3"というカラーパレットから、9色を取得します。
なお、パレット名と配色を知りたい場合は、次のコードを実行してみてください。
display.brewer.all()
RColorBrewerを使わずに、自由にカラーパレットを作ることも可能です。
cols1 <- c("色名", "色名", "色名", "色名", ,, "色名") で作成します。
色名は、"red" や "blue" でも良いし、"#CD5C5C", "#8B0000" のようにカラーコードで指定してもOKです。
ちなみに私は、huey というサイトを活用して配色を決めています。
#10 shapes <- c(1:9) で、プロットの形を指定します。
たとえば、0 = □, 1 = ○, 15 = ■, 16 = ● のように、形に対して数字でIDが割当られます。
なお数字と形の対応表は、以下のサイトをご参照ください。
#11 plot(dt$delta13C, dt$delta15N, col = cols[type], pch = shapes[type])
コード#6に、col = と pch = を追加しました。
col = でプロットの色を、pch = でプロットの形を指定します。
たとえば、col = "red" とすれば、すべてのプロットは赤色になります。
pch = 0 とすれば、すべてのプロットは白抜き四角(□)になります。
今回は、Type ごとにプロットの色や形を変えたいので、col = cols[type], pch = shapes[type] としました。
colsには、コード#9 で作成したカラーパレットの情報が入っています。そして type には、「数値型になった Type 列の情報」が入っているのでした。
cols[type] とすることで、「数値型になった Type 列の情報」を一行目から読み込んでいき、
各ID (魚、昆虫など)に対して、カラーパレット cols 中の色1,2, .., 9を順番に割り当ててくれます。
同様に、shapes[type] とすることで、「数値型になった Type 列の情報」を一行目から読み込んでいき、
各ID (魚、昆虫など)に対して、shapes 中の形1,2, .., 9を順番に割り当ててくれます。
プロットの凡例は、以下のコードで出力します。
plot(dt$delta13C, dt$delta15N,type = "n")
legend("bottomleft",
legend=c(unique(dt$Type)),
col=cols[(unique(type))],
pch=shapes[unique(type)])
ラベルを変更する
それでは最後に、軸ラベルや向きを変えてみましょう。
plot(dt$delta13C, dt$delta15N,
col = cols[type],
pch = shapes[type],
xlab = expression({delta}^13C~'\u2030'),
ylab = expression({delta}^15N~'\u2030'),
las=1)
解説
#12 コード#11に、xlab = , ylab = , labs = を追加しました。
まずxlab = で、X軸のラベルを指定します。基本的な使い方は、xlab = "文字列"です。ちなみに、何も表示したくない場合はxlab = ""とします。
同様に、ylab = で、Y軸のラベルを指定します。
labs = で、軸の向きを指定します。1を指定すると、X軸とY軸の数値が水平方向を向きます。
labs = では1~3までパターンが用意されています。気になる方は数字を変えてプロットしてみてください。
最後に、高画質で図を保存する方法を紹介します。
Rstudioでは、右下のボックスに図が表示されます。そして、"Export" タブから図を保存することができます。
しかし、この方法では手間がかかる上に、解像度が低いといった問題があります。
よって、コードでの画像保存を推奨します。
png("biplot.png", width = 1200, height = 1200, res = 300)
plot(dt$delta13C, dt$delta15N,
col = cols[type],
pch = shapes[type],
xlab = expression({delta}^13C~'\u2030'),
ylab = expression({delta}^15N~'\u2030'),
las=3)
dev.off()ルールは、
①png("保存する図のファイル名"、width = 幅, height = 高さ, res = 解像度)
②任意の plot()
③dev.off()
の順でコードを実行することです。
ggplot2 を活用する
ここからは、ggplot2を使った作図方法に移ります。
ggplot2は、Rでもっとも使われるデータ可視化ツールです。
Rのデフォルト関数で作成した図よりも可読性が高く、
またプロットの色や形の変更操作をより直感的に動かすことができます。
これまでに作成した図をggplot2版で作ってみましょう。
library(ggplot2)
ggplot(dt, aes(x = delta13C, y = delta15N)) +
geom_point(size = 3)
#13 library(ggplot2) で、ライブラリーggplot2を呼び出します。
まだインストールが済んでいない場合は、install.packages("ggplot2") を実行してください。
#14 ggplotで散布図を作ります。
基本的なルールは、ggplot(データセット, aes(x = X軸データ, y = Y軸データ)) + geom_point()
です。
geom_point(size= )で、プロットの大きさを操作します。
次に、図の体裁を整えます。
ggplot2では、上記の基本コードに "+" でコマンドを足していきます。
#cols <- brewer.pal(9, "Set3")
#shapes <- c(1:9)
ggplot(dt, aes(x = delta13C, y = delta15N, color = Type, shape = Type)) +
geom_point(size = 3) +
scale_color_manual(values=cols) +
scale_shape_manual(values=shapes) +
theme_light() +
theme(panel.grid=element_blank()) +
xlab(expression({delta}^13*C~'\u2030')) +
ylab(expression({delta}^15*N~'\u2030'))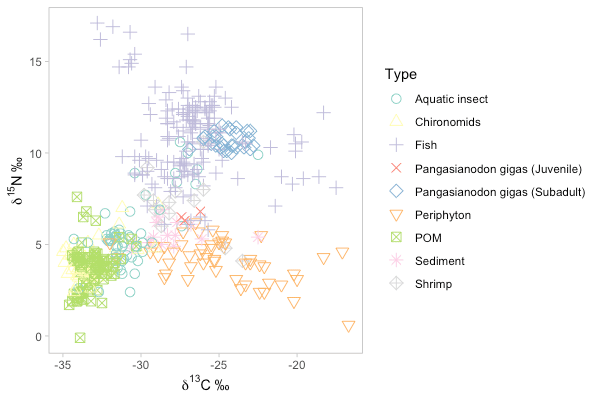
#15 ggplot(dt, aes(x = delta13C, y = delta15N, color = Type, shape = Type)) + geom_point(size = 3)
コード#14 に color = Type と shape = Type を追加しました。
これは、Type 列のIDに応じてプロットの色と形を変えてください、ということです。
#16 + scale_color_manual(values=cols)
scale_color_manual(values= )でカラーパレットを指定します。
ここでは、コード#9で作成したカラーパレット"cols"を使いました。
#17 + scale_shape_manual(values=shapes)
scale_shape_manual(values= )でプロットの形を指定します。
ここでは、コード#10で作成したデータセット"shapes"を使いました。
#18,19 + theme_light() + theme(panel.grid=element_blank()) で、
図のテーマ(配色、軸の種類など)を指定できます。
ggplot2では、いくつかのテーマが用意されていますので、
ほかのテーマにも興味がある方は、こちらのサイトを参照ください。
#20 + xlab(expression({delta}^13*C~'\u2030'))
X軸のラベルを指定します。基本的な使い方は、+ xlab("文字列") です。
#21 + ylab(expression({delta}^15*N~'\u2030'))
同様に、Y軸のラベルを指定します。
最後に、ggplotの図を高画質で保存する方法を紹介します。
ggplot(dt, aes(x = delta13C, y = delta15N, color = Type, shape = Type)) +
geom_point(size = 3) +
scale_color_manual(values=cols) +
scale_shape_manual(values=shapes) +
theme_light() +
theme(panel.grid=element_blank()) +
xlab(expression({delta}^13C~'\u2030')) +
ylab(expression({delta}^15N~'\u2030'))
ggsave("biplot_gg.png", width = 8, height = 8, dpi = 300)ルールは、
①任意の ggplot()
②ggsave("保存する図のファイル名"、width = 幅, height = 高さ, res = 解像度)
です。直前に出力したggplot() に対して、保存作業がおこなわれます。
***************************************************
ご不明な点があれば、お気軽にご連絡ください。
***************************************************
この記事が気に入ったらサポートをしてみませんか?
