プログラマブルタンパク質(Programmable proteins):標的特異性、プログラミング性、将来の方向性
山形方人 Masahito Yamagata
2022年10月に、私が編集委員をやっている合成生物学の国際誌SynBioに発表した総説「Programmable proteins: target specificity, programmability and future directions」の日本語訳です。DeepLを用いたのち、手動で修正しましたので、不完全な箇所が沢山あります。
なお、この総説は、「Programmable proteins in synthetic biology」という特集の1つとして掲載されています。この特集では、論文の投稿をお待ちしています。
投稿する前に、私に連絡すると、全く無料でオープンアクセス発表することができます(15万円ほどのAPCが完全免除となります)。
Programmable proteins: target specificity, programmability and future directions Masahito Yamagata
SynBio 2022, 1(1), 65-76
© 2022 by the author. Licensee MDPI, Basel, Switzerland. This article is an open access article distributed under the terms and conditions of the Creative Commons Attribution (CC BY) license (https://creativecommons.org/licenses/by/4.0/).
要約
プログラミング可能なタンパク質(プログラマブルタンパク質Programmable protein)は、in vitro および in vivo で選択的にタンパク質を検出、可視化、調節、除去することで、認識されるターゲットとその後の生物学を研究するために不可欠である。プログラマブルタンパク質の特異性は、その配列や構造を設計することによって容易に変更することができる。このようなタンパク質の柔軟性とモジュール性は、現在、合成生物学や様々な医療への応用に極めて重要である。プログラマブル・ヌクレアーゼ、シングルドメイン抗体、その他のタンパク質足場など、個々のプログラマブルタンパク質の概念と応用については、多くの総説が存在する。本稿では、このようなプログラマブルタンパク質を、そのプログラマブルな原理と生体分子(核酸、タンパク質、糖鎖)への標的特異性に基づいて拡張した概念フレームワークを提案し、その利点、限界、将来の方向性について概説している。
キーワード: programmable protein; nuclease; CRISPR; TALEN; Cas9; Argonaute; nanobody; DARPin; glycan-binding protein; lectin
1. プログラマブルタンパク質とは何か?
「プログラマブル」という言葉は、一般的に、一連の指令やルールを受け入れて、意図したとおりにさまざまな作業を行うことができるコンピュータや機械を指す言葉として使われている。この指令やルールは通常、特定のプログラミング言語で記述されている。生体においては、生体分子、分子間や細胞間の回路を人工的に改変し、特異性や機能を変換することを「プログラマブル」と呼んでいる。核酸やタンパク質の機能は、その配列を変えることでプログラム可能である。DNA分子の優れたプログラム性を利用すれば、核酸にユニークな化学的性質を付与することが可能である[1]。アプタマーは、抗体に匹敵する特異性と親和性で様々な分子を認識することができる設計可能なオリゴヌクレオチド配列である[2,3,4]。リボスイッチ、リボザイム、デオキシリボザイムは、塩基配列を変えることで設計できる可能性のある機能性の核酸である[5,6]。しかし、蛍光性、リガンド結合性、触媒活性などの機能は、SELEX(systematic evolution of ligands by exponential enrichment)によるリガンドの系統的進化を利用しない限り、まだ完全に予測できるわけではない[7]。
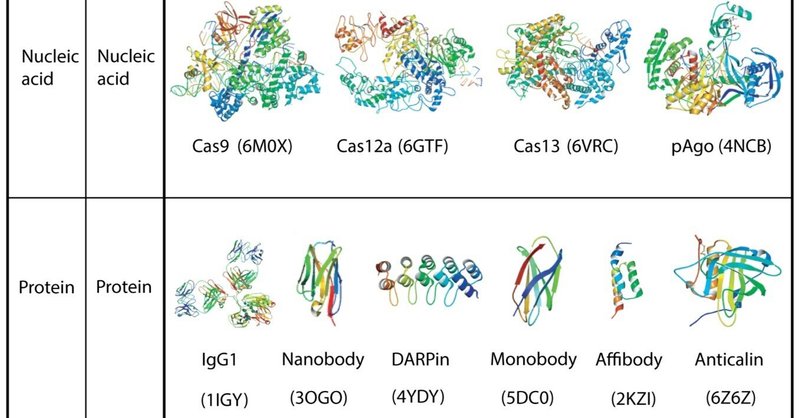
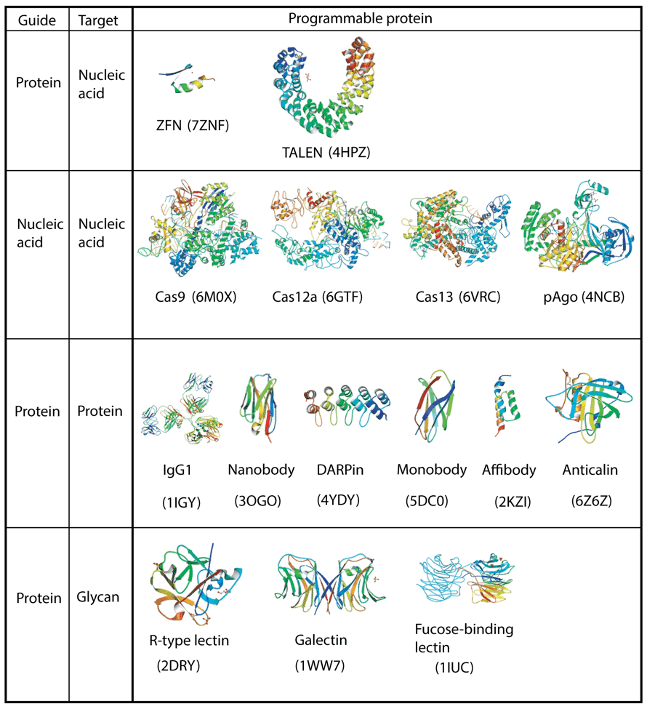
タンパク質は、多様な化学的性質と生物学的な役割を持つ重要な生体分子です。タンパク質と生体分子の相互作用は生物学において極めて重要な役割を果たしており、これらの相互作用を阻害したり変化させたりするタンパク質を設計するアプローチは非常に有用であると考えられる。AlphaFold2を含む最近の深層学習アプローチは、タンパク質の構造が計算機上で予測可能であることを示した[8,9]。場合によっては、結合に関する事前の知識を必要とせず、標的の構造に関する情報のみで新しい結合タンパク質を設計することができる[10,11,12]。このことは、近い将来、多様なタンパク質が機能性タンパク質として人為的にプログラム可能になる可能性を示している。しかし、現状は、プログラマブルタンパク質に用いられるテンプレートは限られている。図1にまとめたように、現在のプログラマブルタンパク質は、核酸ガイド型とタンパク質ガイド型の2種類に大別される。また、相互作用する標的によって、核酸、タンパク質、糖鎖を含む他の分子に分類される。
このようなプログラマブルタンパク質は、生物学、合成生物学、医学の分野で広く利用されている。CRISPR/Cas9 (clustered regularly interspaced short palindromic repeats, CRISPR-associated protein 9)システムの数多くの応用はその好例である[13,14,15,16].また、プログラム可能なヌクレアーゼやシングルドメイン抗体など、個々のプログラム可能なタンパク質についても、すでに多くの総説が存在する。さらに、この分野は急速に発展している。したがって、このトピックに関するいかなる総説記事もすぐに時代遅れになってしまうだろう。その代わりに、この総説では、プログラマブルタンパク質の考え方を他のユニークな結合分子に拡張することを提案し、その利点、限界、将来の方向性について概説する。
2.プログラマブル・ヌクレアーゼ、修飾因子、核酸結合蛋白質
プログラム可能なヌクレアーゼは、天然および合成由来で数種類が報告されている[17,18,19,20]。このような酵素は、タンパク質モジュール-DNA相互作用を利用して特定の配列を認識するタンパク質ガイド型ヌクレアーゼ(例:TALEN:セクション2.1)と、付属の短い相補的DNAまたはRNAを介して特定の配列を認識する核酸ガイド型ヌクレアーゼ(例:Cas9:セクション2.2~2.6)の2つのグループがゲノム工学に使用されている。
2.1 ZFNs and TALENs
最初の概念実証に成功したプログラマブル・ヌクレアーゼは、DNA結合ホメオドメイン(ショウジョウバエのUltrabithorax)とFok1の非特異的DNA切断ドメインを連結して作られた人工ハイブリッド型デオキシリボヌクレアーゼであった [21] 。Fok1はFlavobacterium okeanokoitesから発見された制限酵素で、N末端のDNA結合ドメインとC末端のDNA切断ドメイン(約200アミノ酸)からなるIIS型制限酵素である。しかし、メガヌクレアーゼのようなプログラム可能なヌクレアーゼを作製する初期のアプローチは、その技術的限界(例えば、ホーミング酵素やFEN1 (Flap structure-specific endonuclease-1) の改変など)から普及しなかった [19,20,22].その後、タンパク質誘導型プログラマブルヌクレアーゼとして、エンジニアリングされたジンクフィンガーヌクレアーゼ(ZFNs)と転写活性化因子様エフェクターヌクレアーゼ(TALENs)が発明された。
ZFNは、3bp程度のDNAを認識するプログラム可能なDNA結合タンパク質を用いている。ZFNは、エンドヌクレアーゼFokIドメインと、特定の標的DNA配列を認識するようにプログラムされたジンクフィンガー配列とが融合したモノマーから二量体を形成する[23]。ZFNのDNA結合ドメインは、通常3-4個のジンクフィンガーアレイで構成されている。新しいZFNを作成するために用いられる方法を開発することで、多くの技術的な課題が解決された。しかし、ZFNの欠点は残されたままである。
TALENは、DNA結合ドメインとFokI DNA切断ドメインから構成され、ZFNと同様の構造をしている[24]。TALENは、天然に存在する植物細菌であるXanthomonas属の病原体に由来し、TALEと呼ばれるDNA結合タンパク質を含んでいる[25]。TALEは34アミノ酸ほどの長さで、DNAの3塩基対を認識するZFNとは異なり、1塩基対を認識するため、TALENはZFNよりも高い柔軟性を有しています。TALEはタンデムに連結され、特定のDNA配列を標的とすることができる鎖を作る。しかし、TALENアレイを構築するには、ほぼ同一のリピート配列を複数個組み立てる必要があり、技術的に困難であった。この問題を解決するために、いくつかの研究室での優れた方法が開発されてきた。核酸ガイド型CRISPRは広く使われているプログラム可能なヌクレアーゼだが、タンパク質ガイド型TALENはオフターゲットが少なく、CRISPRのガイドRNAが取り込みにくいミトコンドリアDNAを標的とすることができる[26]。
2.2 Cas9 (Type II CRISPR-Cas)
ZFNやTALENは、エンドヌクレアーゼドメインをブロックガイド型のDNA結合モジュールに連結し、標的DNAの二本鎖切断(DSB)を誘導する戦略をとっている。一方、Cas9は標的DNAとの塩基対形成による核酸ガイド型ヌクレアーゼとなり、特異的かつ効率的に設計しやすく、多様な細胞種や生物のハイスループットかつマルチプレックスな遺伝子編集に適したシステムである。CRISPR-Cas9戦略は、細菌や古細菌がウイルスやプラスミドに対する適応免疫に用いる天然由来のゲノム編集システムから生まれた[27,28]。
CRISPR/Cas9システムは、様々な細菌種由来のCas9単量体ヌクレアーゼ(例えば、.Streptococcus pyogenes Cas9 (SpCas9), Staphylococcus aureus Cas9 (SaCas9), Streptococcus thermophilus Cas9 (St1Cas9 and St3Cas9), Campylobacter jejuni Cas9 (CjCas9), Neisseria meningitides (NmCas9)) 、特異性決定CRISPR RNA (crRNA) および補助トランス活性化RNA (tracrRNA) が使われる。crRNAとtracrRNAは、in vitroまたはin vivoで合成されるデュアルRNAまたはシングルガイドRNA(sgRNA)として使用される[13,14,15,16]。SpCas9が最も広く用いられているが、Cas9、SaCas9、CjCas9、NmCas9はSpCas9より小型で、アデノ随伴ウイルスベクターにパッケージングすることが可能である。Cas9は、ガイドRNAと相補的なDNA鎖(標的鎖)を切断するHNHヌクレアーゼドメインと、非相補鎖(非標的鎖)を切断してDSBを生じるのに必要なRuvCヌクレアーゼドメインを有している。最も広く特徴づけられているSpCas9-RNA複合体は、プロトスペーサー隣接モチーフ(PAM)であるNGGを含む鎖を認識する。PAMの上流にある標的20bpsのうち、8から12bpsが認識に重要である。ブラントエンド切断部位はPAMの上流から3番目のbpsにある。DSBはその後、非相同末端接合(NHEJ)または相同性修復(HDR)により修復される[29]。crRNA-Cas9タンパク質複合体をエレクトロポレーションすることで、GFPレポーターをHDRによってニワトリゲノムの特定の遺伝子に体細胞で組み入れるすることができ、このアプローチが様々な種で可能であることを示している[30]。
ニッキング酵素、塩基エディター、プライムエディター、CRISPR活性化(CRISPRa)、干渉(CRISPRi)など、さまざまなCRISPRを基盤としたゲノムおよびエピゲノム編集ツールは、ヌクレアーゼ活性を排除し他のエフェクターと融合することで正確な認識機構を用いている[16,31,32,33]。
2.3 Cas12 (Type V CRISPR-Cas)
CRISPR-Cas12a (Cpf1) は、ゲノム編集ツールボックスを多様化させたもう一つのCRISPR/Casシステムである[34,35]。CRISPR/Cas9と比較して,CRISPR/Cas12aシステムは,サイズが小さく,tracrRNAを必要とせず,T-rich PAMを使用し,切断部位に粘着性末端を形成する。また、デオキシリボヌクレアーゼとリボヌクレアーゼの両方の機能を持ち、1つの転写産物から複数の機能的なcrRNAを処理することができる。このように、CRISPR/Cas9がRNAガイドを用いてDNAを認識・切断する(R-D nuclease)のに対し、CRISPR/Cas12aはRNAガイドを用いてDNAとRNAの両方を認識・切断する(R-D/R nuclease)。最も一般的に用いられているCas12aは、Francisella novicida(FnCas12a)、Acidaminococcus sp.(AsCas12a)、Lachnospiraceae bacterium(LbCas12a)由来である。
Cas12b(C2c1とも呼ばれる)タンパク質は、Cas9およびCas12aよりも小さい。Cas9と同様に、Cas12bはDNAターゲティングのために、sgRNAとして結合可能なcrRNAとtracrRNAの両方を必要とする。最近開発されたAlicyclobacillus acidiphilus(AaCas12b)およびBacillus hisashii(BhCas12b)からのメソフィリックなCas12bは、哺乳類のゲノム編集に適合させることができる[36,37]。
2.4 Cas13 (Type VI CRISPR-Cas)
Cas13(C2c2とも呼ばれる)は、crRNAを用いて標的の一本鎖RNAを識別し(シス認識・切断)、トランス開裂リボヌクレアーゼ活性(トランス開裂・コラテラル効果)を示すRNAガイド型リボヌクレアーゼ(R-R nuclease)で、ゲノム配列を変化させずにRNAiやCRISPRi並みにRNAサイレンシングするものとして期待されている[38,39,40]。Cas13ファミリーには、Cas13a、Cas13b、Cas13c、Cas13dの4種類のサブタイプが確認されている。全てのCas13ファミリーのメンバーは、Cas9よりも小さく、標的特異性を確保するためにcrRNAを必要とする。Cas12aと同様に、この核酸ガイド型ヌクレアーゼは、しばしば診断のためのRNAまたはDNA標的の配列特異的検出に使用されてきた[41]。
2.5 OMEGA (TnpB, IscB)
IscBとTnpBは、最近研究されたRNAガイド型デオキシリボヌクレアーゼ(R-D nuclease)で、それぞれCas9とCas12の祖先型と思われる。IscBは原核生物のIS200/IS605トランスポゾンの別ファミリーに見いだされた[42].TnpBは,好極性細菌Deinococcusの同じトランスポゾンに由来するものであった[42,43].IscBとTnpBタンパク質 IscBとTnpBは、トランスポゾンがコードする非コードの短いωRNAと右端RNA(reRNA)によってガイドされる。トランスポゾンがコードするRNAをガイドとするデオキシリボヌクレアーゼは、いずれもヒト細胞内でdsDNAを切断し、小型でプログラム可能な非Casヌクレアーゼという新しいグループを提供して、ゲノム編集のツールボックスを拡張している。Feng Zhangのグループは、これらの広範なヌクレアーゼをOMEGA (obligate mobile element-guided activity)と呼ぶことを提案した[43]。
2.6 Argonaute
Ago (Argonaute) タンパク質は、核酸ガイド型プログラマブルタンパク質の2番目のタイプです[20,44]。細菌のCRISPRと同様に、Agoは真核生物や原核生物において、侵入してきた核酸から宿主細胞を守る遺伝子免疫系において極めて重要な役割を担っている。真核生物のAgoタンパク質(eAgos)は、RNA干渉(RNAi)に関与し、RNA標的の認識にガイドRNAを利用する(R-R nuclease)。一方、原核生物のAgo(pAgo)ヌクレアーゼは、DNAガイドとDNAターゲットに天然の特異性を持ち(D-Dヌクレアーゼ)、CRISPRに関連する少数のpAgoは、DNAガイドまたはRNAガイドを用いてDNAターゲットを切断するようにプログラム可能である(D/R-Dヌクレアーゼ)。
しかし、Agoを用いた哺乳類細胞でのプログラミング可能なゲノム編集の成功は報告されていない。これは、当初、好熱性原核生物で同定されたpAgosは、高温(>65 °C)で最も効果的であるが、37 °Cでは効果がないためである(例えば、Thermus thermophilus (TtAgo), Methanocaldococcus jannaschii (MjAgo) and Pyrococcus furiosus (PfAgo) のpAgosなど)。しかしながら、様々な原核生物におけるpAgoタンパク質の多様な構造と機能は、Casヌクレアーゼと並ぶ次世代のゲノム編集ツールになることを示唆している。いくつかのpAgoタンパク質は,実際に37℃でDNAガイド依存的にDNA配列を切断することが実証されている(例えば,Clostridium perfringens由来のpAgos(CpAgo)およびIntestinibacter bartlettii(IbAgo))[45].この試みが成功すれば、ガイドとターゲットのミスマッチの許容度、RNAの二次構造の可能性、PAMの必要性などから、その適用が制限されがちなCRISPR-Cas9ツールの幅を広げることができるだろう。
3. プログラマブルなタンパク質結合タンパク質
核酸を標的とするだけでなく、多様なクラスのプログラマブルタンパク質は、生物学で本質的な役割を持つタンパク質の相互作用を検出、破壊、または調節することができる[46,47,48,49]。抗体は、様々な用途を持つ古くからの免疫学的ツールである。特に、ハイブリドーマ技術や組み換えDNA技術の出現により、モノクローナル抗体や一本鎖可変フラグメント(scFv)を含む組み換え抗体の応用が成功し、多様なタイプの免疫学的試薬や治療薬の開発を触発した[50]。ファージディスプレイ、酵母ディスプレイ、mRNAディスプレイ、リボソームディスプレイ、有向進化、アフィニティ成熟などのin vitro選択法の発明は、さらなる抗体工学を可能にしただけでなく、新規結合タンパク質の開発を促進した[51,52,53,54,55]。しかし、これらのタンパク質は、核酸誘導型プログラマブルタンパク質とは異なり、その正確な特異性を容易に予測することはできない。現在までのところ、効率的なプログラム可能性は、様々なディスプレイ手法とin vitroでの指向性進化法に依存している。しかし、計算機による新たなアプローチにより、プログラム可能なタンパク質が出現する可能性が出てきた[10,11,12,56]。
このようなプログラム可能な結合タンパク質の最初のクラスは、VHHまたはナノボディと呼ばれる珍しい抗体からの一本鎖断片である(セクション3.1)。第二のクラスは、免疫グロブリンの特徴である、新規結合部位を設計するための構造適応性を提供する可変セグメントと折り畳み安定性を提供する定常領域という二つの構造的特徴を持つタンパク質足場をベースにしている。このような2つの構成要素は、すでに可変ループモチーフを持つドメイン構造から出発した場合に実現可能である。そこで、アンキリンリピートやフィブロネクチンタイプIII(FN3)リピートなどの反復ドメインは、一般的な結合タンパク質の強固な足場として魅力的に映ったのである。その結果、DARPinやmonobodyのような一連のタンパク質誘導型プログラマブルタンパク質が作られた(セクション3.2および3.3)。第3のクラスは、熱やタンパク質分解に対して高い安定性を持つ、リガンド結合型非繰り返しタンパク質に基づくものである。アフィボディとアンチカリンと名付けられたプログラム可能なタンパク質は、それぞれStaphylococcus aureus protein Aと輸送タンパク質の一種であるリポカリンから作られた(セクション3.4と3.5)。この第3のクラスは、通常、単一タンパク質として使用されるが、第1および第2のクラスは、単一タンパク質だけでなく、組み換え融合タンパク質として頻繁に適用される。
プログラマブルタンパク質を構築する例は多岐にわたる(例.HEAT様反復配列を含むαRepタンパク質[57]、ロイシンリッチリピートに基づくレペボディ[58]、FynのSH3モチーフに基づくFynomers [59]、シスタチンに基づくaffimer/adhirons [60] 、ユビキチン/ガンマBクリスタリンに基づくaffilin [61] 、種々の膜受容体に基づくavimer [62] および Kunitzドメインペプチド [47] など) を構築した例がある。これらの足場は、プログラマブルタンパク質の骨格のレパートリーを拡大し、その多様な化学的性質、薬物動態、およびユニークな組織への浸透に関して重要な付加価値を提供してきた[46,47,48,49]。ここでは、5つのタンパク質ガイド型プログラマブルタンパク質が、実用的に有用であることが実証されているので、模範となる例として簡単に取り上げる。
3.1 シングルドメイン抗体, ナノボディ, VHH
シングルドメイン抗体は、ナノボディとも呼ばれ、分子量約15 kDa、サイズ約2-4 nmの小さな抗原結合ポリペプチドである。1990年代にラクダ科動物(ラクダとラマ)の血清中に初めて報告された重鎖のみの抗体(VHH)の可変ドメインで構成されている[63]。同様の一本鎖抗体は、軟骨魚類(VNAR、サメ由来)にも存在する[64,65]。
このように、ナノボディは、活性な免疫グロブリンに由来する最小のインタクトな抗原結合タンパク質断片である[66,67]。ナノボディは、サイズが小さく、溶解性が高く、様々な用途で安定であるため、従来の抗体の代替品として有利である。さらに、ファージディスプレイ、リボソームディスプレイ、mRNAディスプレイなどの方法を用いて、in vitroで結合分子を効率的に生成し、最適化することができる。ナノボディは、遺伝子コード化、タグ付けされ、細胞内の組換えイントラボディまたはレポーター融合体として発現し、in vivoでの標的タンパク質の局在や機能研究を行うことができる[68,69,70]。現在、いくつかのナノボディが臨床試験中であり、1つは2019年に後天性血栓性血小板減少性紫斑病に対してFDAによって承認された[71]。
3.2 DARPin
デザインされたアンキリンリピートタンパク質(DARPin)は、遺伝子工学的に設計された抗体模倣タンパク質であり、一般的に高い特異性と高親和性標的タンパク質結合を示す[72,73]。ほとんどの天然アンキリンリピート(AR)タンパク質は、4-6個のARが互いに積み重なっている。DARPinは、N末端とC末端のキャッピングモジュールに挟まれた2-3個の内部ARを含んでいる。各内部ARモジュールは27個の定義されたフレームワーク残基と6個の潜在的なタンパク質結合残基からなり、βターンに続いて2個の逆平行らせんと次のARのβターンに接続するループを形成する [74]。DARPinはサイズが小さく(14-18kDa、内部ARの数に依存)、耐熱性があり、プロテアーゼや化学変性剤に耐性があり、バクテリアで発現させることができる。DARPinは凹型の結合界面を持ち、その結合界面はナノボディの凸型界面とは異なるため、合成リガンドのランドスケープを広げることで価値を高めている [75]。
3.3 モノボディ, Adnectin®, FingR
モノボディは当初、10番目のFN3ドメインに基づいて設計された。このドメインは、7本の鎖が6つのループで接続された免疫グロブリンβサンドイッチフォールドを持つが、ジスルフィド結合はない[76,77]。オリジナルの10番目のFN3は94個のアミノ酸から成り、分子量は約10kDaで、インテグリンと結合する接着性のRGD配列を含んでいる。FN3の片側にある6つの柔軟なループのうち3つは表面に露出しており、目的のリガンドを結合するための合成界面であることが示されている。その後、市販の同等品であるAdnectin®やいくつかのモノボディ様足場が開発され[77,78]、プログラム可能なタンパク質を作り出すためのFN3アプローチの堅牢性が再確認された。様々なディスプレイディープLは、ナノモルからピコモルの範囲の結合親和力を持つリガンド結合タンパク質を選択している。ナノボディベースのイントラボディと同様に、モノボディ由来のタンパク質も細胞内で使用することができる(例えば、神経シナプスで内在性PSD95と共局在化したGFP融合FingR(=モノボディ)がある[79])。注:Adnectin ®はBristol-Myers Squibb R&D CompanyであるAdnexusの登録商標である。
3.4 アフィボディ
アフィボディは、黄色ブドウ球菌のプロテインAの免疫グロブリン結合領域のZドメイン [80,81] をベースに設計されており、3つのヘリックス足場を採用し、ジスルフィド結合を含んでいない。プログラム可能なリガンド結合面は、2つのらせんの間に散在する13個のアミノ酸残基で構成されている。そのコンパクトさ(58アミノ酸、大きさ約7kDa)により、バクテリアで簡単に発現させたり、化学合成で生産することができる。
3.5 アンチカリン ®
アンチカリン®は、ビタミン、ステロイド、臭気物質、様々な代謝産物などの小さな化合物を輸送または貯蔵する分泌タンパク質の大きなグループであるリポカリンをベースに設計されました[82,83]。アンチカリンは、8本鎖の反平行βバレルを採用し、一端が溶媒に開かれており、160-180個のアミノ酸を含む(大きさは〜20 kDa)。アンチカリンはグリコシル化されておらず、ジスルフィド結合も有していない。リガンド結合タンパク質をスクリーニングするためのアンチカリンライブラリーは,各ループに16-24個のランダムなアミノ酸を含んでいる[82].リガンド特異的なアンチカリンは、ファージやバクテリアの表面ディスプレイによって設計され、バクテリアまたは酵母で発現される[82]。 注:Anticalin®はPieris Pharmaceuticalsの登録商標。
4 糖鎖結合タンパク質(GBPs)
糖鎖は、オリゴ糖と多糖を含む炭水化物の総称であり、核酸とタンパク質に続く第3の生体高分子である[84]。糖鎖は遊離糖として存在するが、より一般的には、糖タンパク質、プロテオグリカン、糖脂質などの複合糖質として見出される。糖鎖の幅広い重要性から、新しい糖鎖結合分子の開発への関心が高まっている[85,86,87]。
糖鎖結合タンパク質(GBP)には、抗体、レクチン、擬似酵素、糖鎖結合モジュールが含まれる。糖鎖に対する抗体は自然界にも存在し、多くのモノクローナル抗体は、糖鎖の特異的構造を認識するための試薬として著しく有用である[88]。しかし、一般に糖鎖は抗原性が低い。一方、レクチンは、少なくとも1つの非触媒ドメインを含む非免疫グロブリン蛋白質であり、多くの場合、糖鎖に特異的に結合する。例えば、糖鎖を認識する植物レクチンは数多く存在し、糖鎖を検出するツールとして利用されている[88,89,90]。また、GBPにはレクチンに類似した糖鎖結合モジュールもあるが、これはグリコシダーゼや糖転移酵素などの糖鎖活性酵素に典型的に見られる小さな結合ドメインである[91]。糖質活性酵素の中には、酵素活性を失い、糖鎖結合能を保持する擬似酵素に進化したものもあり、GBPの足場となる可能性を秘めている[92]。
レクチン工学の先駆的な研究では、真核生物および原核生物のレクチンの結合特異性を変換させている。例えば、ランダム変異導入とリボソームディスプレイによる深層Lsを用いて、ミミズ由来のガラクトース結合性R型レクチン(ガレクチン)から新規なシアル酸結合性タンパク質が作製された[93]。また、2種類のキノコ(Agrocybe cylindraceaとAleuria aurantia)レクチンの糖結合スペクトルも変異導入により変化させた[94,95]。これらの先行研究は、天然に存在するマメ科レクチン[98]と同様に、新しいGBPを生成できることを示し、レクチンがプログラム可能であるという考えを支持するものである。さらに、セクション3で述べたタンパク質ガイド型プログラマブルタンパク質など、他の一般的なタンパク質足場も、新しい糖鎖結合特異性を獲得するために利用することができる。最後に、複合糖質研究における最先端の進歩[84]と共に、多数の新しい計算およびデータ科学のアプローチが、さらに複雑な糖鎖を区別するためのツールボックスを拡張するために使用できるだろう[90,92,99]。
5 展望
in vitro および in vivo において、選択した生体分子と結合、標識、阻害、除去するプログラマブルなタンパク質は、認識された生体分子の基本的な役割を理解するために重要である。プログラマブルタンパク質の特異性は、in vitroで配列を変えることにより柔軟に設計することが可能である。特に、DNAの優れた合成技術により、特定の核酸配列を認識する核酸ガイド型タンパク質のプログラム性は、オフターゲットや非効率性などの多くの重要な問題が未解決であるが、実質的に完成している。一方、タンパク質ガイド型のプログラマブルタンパク質は、ビルディングブロックガイド型のモジュール型ヌクレアーゼを除き、まだ開発途上にある。それにもかかわらず、AlphaFold2を含む新興の深層学習アプローチは、多くのタンパク質構造がin silicoで正確に予測できることを実証している[8]。このように、限られたケースではあるが、標的の構造に関する情報のみを用いて結合タンパク質が設計され[10,11,12]、タンパク質や糖鎖の特定の配列や構造を認識するために有用なものとして、多様なタンパク質が設計可能になる可能性が出てきた。このような計算アルゴリズムやオープンソースの巨大データベースの開発は、合成生物学における最先端の「武器倉庫」を実現する鍵となるだろう。
この総説では、核酸、タンパク質、糖鎖という重要な3つの生体高分子を認識するプログラマブルタンパク質に焦点を当ててきた。しかし、プログラミング性には様々なタイプがある。例えば、バクテリア、菌類、植物に広く存在するポリケチド、非リボソームペプチド(NRP)、リボソーム合成翻訳後修飾ペプチド(RiPP)などの生物活性天然物の生合成に関与する酵素は、プログラマブルタンパク質のユニークなグループである [100,101,102].これらの生合成酵素は、組立式パイプラインに似たモジュール式の合成スキームで多様な天然物を生産する。このようなメガ酵素のモジュール性は、合成生物学的な観点から有用である。モジュールの置換や再配置によって新しい天然物を生み出すことができ、生理活性低分子の生合成がプログラム可能であることが示唆されている[101,102,103]。このように、プログラマブルタンパク質が持つ柔軟性とモジュール性は、診断薬や治療薬などの産業・医療分野への応用に極めて重要である。
文献
1. Micura, R.; Höbartner, C. Fundamental studies of functional nucleic acids: aptamers, riboswitches, ribozymes and DNAzymes. Chem. Soc. Rev. 2020, 49, 7331-7353. doi: 10.1039/d0cs00617c.
2. Sun, W.; Du, L.; Li, M. Aptamer-based carbohydrate recognition. Curr Pharm Des. 2010, 16, 2269-2278. doi: 10.2174/138161210791792877.
3. Nimjee, S.M.; White, R.R.; Becker, R.C.; Sullenger, B.A. Aptamers as Therapeutics. Annu Rev Pharmacol Toxicol. 2017, 57, 61-79. doi: 10.1146/annurev-pharmtox-010716-104558.
4. Zhou, J.; Rossi, J. Aptamers as targeted therapeutics: current potential and challenges. Nat Rev Drug Discov. 2017, 16, 181-202. doi: 10.1038/nrd.2016.199.
5. Ma, L.; Liu, J. Catalytic Nucleic Acids: Biochemistry, Chemical Biology, Biosensors, and Nanotechnology. iScience. 2020, 23, 100815. doi: 10.1016/j.isci.2019.100815.
6. Breaker, R.R. The Biochemical Landscape of Riboswitch Ligands. Biochemistry. 2022, 61, 137-149. doi: 10.1021/acs.biochem.1c00765.
7. Lee, G.; Jang, G.H.; Kang, H.Y.; Song, G. Predicting aptamer sequences that interact with target proteins using an aptamer-protein interaction classifier and a Monte Carlo tree search approach. PLoS One. 2021,16, e0253760. doi: 10.1371/journal.pone.0253760.
8. Jumper, J.; Evans, R.; Pritzel, A.; Green, T.; Figurnov, M.; Ronneberger, O.; Tunyasuvunakool, K.; Bates, R.; Židek, A.; Potapenko, A.; Bridgland, A.; Meyer, C.; Kohl, S.A.A.; Ballard, AJ.; Cowie, A.; Romera-Paredes, B.; Nikolov, S.; Jain, R.; Adler, J.; Back, T.; Petersen, S.; Reiman, D.; Clancy, E.; Zielinski, M.; Steinegger, M.; Pacholska, M.; Berghammer, T.; Bodenstein, S.; Silver, D.; Vinyals, O.; Senior, A.W.; Kavukcuoglu, K.; Kohli ,P.; Hassabis, D. Highly accurate protein structure prediction with AlphaFold. Nature. 2021, 596, 583-589. doi: 10.1038/s41586-021-03819-2.
9. Baek, M.; DiMaio, F.; Anishchenko, I.; Dauparas, J.; Ovchinnikov, S.; Lee, G.R.; Wang, J.; Cong, Q.; Kinch, L.N.; Schaeffer, R.D.; Millán, C.; Park, H.; Adams, C.; Glassman, C.R.; DeGiovanni, A.; Pereira, J.H.; Rodrigues, A.V.; van Dijk, A.A.; Ebrecht, A.C.; Opperman, D.J.; Sagmeister, T.; Buhlheller, C.; Pavkov-Keller, T.; Rathinaswamy, M.K.; Dalwadi, U.; Yip, C.K.; Burke, J.E.; Garcia, K.C.; Grishin, N.V.; Adams, P.D.; Read, R.J.; Baker, D. Accurate prediction of protein structures and interactions using a three-track neural network. Science. 2021, 373, 871-876. doi: 10.1126/science.abj8754.
10. Cao, L.; Coventry, B.; Goreshnik, I.; Huang, B.; Sheffler, W.; Park, J.S.; Jude, K.M.; Marković, I.; Kadam, R.U.; Verschueren, K.H.G.; Verstraete, K.; Walsh, S.T.R.; Bennett, N.; Phal, A.; Yang, A.; Kozodoy, L.; DeWitt, M.; Picton, L.; Miller, L.; Strauch, E.M.; DeBouver, N.D.; Pires, A.; Bera, A.K.; Halabiya, S.; Hammerson, B.; Yang, W.; Bernard, S.; Stewart, L.; Wilson, I.A.; Ruohola-Baker, H.; Schlessinger, J.; Lee, S.; Savvides, S.N.; Garcia, K.C.; Baker, D. Design of protein-binding proteins from the target structure alone. Nature. 2022, 605, 551-560. doi: 10.1038/s41586-022-04654-9.
11. Wang, J.; Lisanza, S.; Juergens, D.; Tischer, D.; Watson, JL.; Castro, K.M.; Ragotte, R.; Saragovi, A.; Milles, LF.; Baek, M.; Anishchenko, I.; Yang, W.; Hicks, D.R.; Expòsit, M.; Schlichthaerle, T.; Chun, J.H.; Dauparas, J.; Bennett, N.; Wicky, B.I.M.; Muenks, A.; DiMaio, F.; Correia, B.; Ovchinnikov, S.; Baker, D. Scaffolding protein functional sites using deep learning. Science. 2022, 377, 387-394. doi: 10.1126/science.abn2100.
12. Pan, X.; Kortemme, T. Recent advances in de novo protein design: Principles, methods, and applications. J Biol Chem. 2021, 296, 100558. doi: 10.1016/j.jbc.2021.100558.
13. Doudna, J.A.; Charpentier, E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9. Science. 2014, 346, 1258096. doi: 10.1126/science.1258096.
14. Hsu, P.D.; Lander, E.S.; Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 2014, 157, 1262-1278. doi: 10.1016/j.cell.2014.05.010.
15. Adli, M. The CRISPR tool kit for genome editing and beyond. Nat Commun. 2018, 9, 1911. doi: 10.1038/s41467-018-04252-2.
16. Anzalone, A.V.; Koblan, L.W.; Liu, D.R. Genome editing with CRISPR-Cas nucleases, base editors, transposases and prime editors. Nat Biotechnol. 2020, 38, 824-844. doi: 10.1038/s41587-020-0561-9. Epub 2020 Jun 22. PMID: 32572269.
17. Gaj, T.; Gersbach, C.A.; Barbas, C.F. 3rd. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends Biotechnol. 2013, 31, 397-405. doi: 10.1016/j.tibtech.2013.04.004.
18. Kim, H.; Kim, J.S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 2014, 15, 321-334. doi: 10.1038/nrg3686.
19. Chandrasegaran, S.; Carroll, D. Origins of Programmable Nucleases for Genome Engineering. J Mol Biol. 2016, 428, 963-989. doi: 10.1016/j.jmb.2015.10.014.
20. Kropocheva, E.V.; Lisitskaya, L.A,; Agapov, A.A.; Musabirov, A.A.; Kulbachinskiy, A.V.; Esyunina, D.M. Prokaryotic Argonaute Proteins as a Tool for Biotechnology. Mol Biol. 2022, 1-20. doi: 10.1134/S0026893322060103.
21. Kim, Y.G.; Chandrasegaran, S. Chimeric restriction endonuclease. Proc Natl Acad Sci U S A. 1994, 91, 883-887. doi: 10.1073/pnas.91.3.883.
22. Hafez, M.; Hausner, G. Homing endonucleases: DNA scissors on a mission. Genome. 2012, 55, 553-569. doi: 10.1139/g2012-049. Epub 2012 Aug 14. PMID: 22891613.
23. Urnov, F.D., Rebar, E.J.; Holmes, M.C.; Zhang, H.S.; Gregory, P.D. Genome editing with engineered zinc finger nucleases. Nat Rev Genet. 2010, 11, 636-646. doi: 10.1038/nrg2842.
24. Joung, J.K.; Sander, J.D. TALENs: a widely applicable technology for targeted genome editing. Nat Rev Mol Cell Biol. 2013, 14, 49-55. doi: 10.1038/nrm3486.
25. Boch, J.; Scholze, H.; Schornack, S.; Landgraf, A.; Hahn, S.; Kay, S.; Lahaye, T.; Nickstadt, A.; Bonas, U. Breaking the code of DNA binding specificity of TAL-type III effectors. Science. 2009, 326, 1509-1512. doi: 10.1126/science.1178811.
26. Bhardwaj, A.; Nain, V. TALENs-an indispensable tool in the era of CRISPR: a mini review. J Genet Eng Biotechnol. 2021, 19, 125. doi: 10.1186/s43141-021-00225-z.
27. Nussenzweig, P.M.; Marraffini, L.A. Molecular Mechanisms of CRISPR-Cas Immunity in Bacteria. Annu Rev Genet. 2020, 54, 93-120. doi: 10.1146/annurev-genet-022120-112523.
28. Koonin, E.V.; Makarova, K.S. Evolutionary plasticity and functional versatility of CRISPR systems. PLoS Biol. 2022, 20, e3001481. doi: 10.1371/journal.pbio.3001481.
29. Chiruvella, K.K.; Liang, Z.; Wilson, T.E. Repair of double-strand breaks by end joining. Cold Spring Harb Perspect Biol. 2013, 5, a012757. doi: 10.1101/cshperspect.a012757.
30. Yamagata, M.; Sanes, J.R. CRISPR-mediated Labeling of Cells in Chick Embryos Based on Selectively Expressed Genes. Bio Protoc. 2021, 11, e4105. doi: 10.21769/BioProtoc.4105.
31. Kampmann, M. CRISPRi and CRISPRa Screens in Mammalian Cells for Precision Biology and Medicine. ACS Chem Biol. 2018, 13, 406-416. doi: 10.1021/acschembio.7b00657.
32. Knott, G.J.; Doudna, J.A. CRISPR-Cas guides the future of genetic engineering. Science. 2018, 361, 866-869. doi: 10.1126/science.aat5011.
33. Nakamura, M.; Gao, Y.; Dominguez, A.A.; Qi, L.S. CRISPR technologies for precise epigenome editing. Nat Cell Biol. 2021, 23, 11-22. doi: 10.1038/s41556-020-00620-7.
34. Zetsche, B.; Gootenberg, J.S.; Abudayyeh, O.O.; Slaymaker, I.M.; Makarova, K.S.; Essletzbichler, P.; Volz, S.E.; Joung, J.; van der Oost, J.; Regev, A.; Koonin, E.V.; Zhang, F. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 2015, 163, 759-771. doi: 10.1016/j.cell.2015.09.038.
35. Safari, F.; Zare, K.; Negahdaripour, M.; Barekati-Mowahed, M.; Ghasemi, Y. CRISPR Cpf1 proteins: structure, function and implications for genome editing. Cell Biosci. 2019, 9, 36. doi: 10.1186/s13578-019-0298-7.
36. Teng, F.; Cui, T.; Feng, G.; Guo, L.; Xu, K.; Gao, Q.; Li, T.; Li, J.; Zhou, Q.; Li, W. Repurposing CRISPR-Cas12b for mammalian genome engineering. Cell Discov. 2018, 4, 63. doi: 10.1038/s41421-018-0069-3.
37. Strecker, J.; Jones, S.; Koopal, B.; Schmid-Burgk, J.; Zetsche, B.; Gao, L.; Makarova, K.S.; Koonin, E.V.; Zhang, F. Engineering of CRISPR-Cas12b for human genome editing. Nat Commun. 2019, 10, 212. doi: 10.1038/s41467-018-08224-4.
38. Abudayyeh, O.O.; Gootenberg, J.S.; Konermann, S.; Joung, J.; Slaymaker, I.M.; Cox, D.B.; Shmakov, S.; Makarova, K.S.; Semenova, E.; Minakhin, L.; Severinov, K.; Regev, A.; Lander, E.S.; Koonin, EV.; Zhang, F. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 2016, 353, aaf5573. doi: 10.1126/science.aaf5573.
39. O'Connell, M.R. Molecular Mechanisms of RNA Targeting by Cas13-containing Type VI CRISPR-Cas Systems. J Mol Biol. 2019, 431, 66-87. doi: 10.1016/j.jmb.2018.06.029.
40. Tang, T.; Han, Y.; Wang, Y.; Huang, H.; Qian, P. Programmable System of Cas13-Mediated RNA Modification and Its Biological and Biomedical Applications. Front Cell Dev Biol. 2021, 9, 677587. doi: 10.3389/fcell.2021.677587.
41. Kaminski, M.M.; Abudayyeh, O.O.; Gootenberg, J.S.; Zhang, F.; Collins, J.J. CRISPR-based diagnostics. Nat Biomed Eng. 2021, 5, 643-656. doi: 10.1038/s41551-021-00760-7.
42. Karvelis, T.; Druteika, G.; Bigelyte, G.; Budre, K.; Zedaveinyte, R.; Silanskas, A.; Kazlauskas, D.; Venclovas, Č.; Siksnys, V. Transposon-associated TnpB is a programmable RNA-guided DNA endonuclease. Nature. 2021, 599, 692-696. doi: 10.1038/s41586-021-04058-1.
43. Altae-Tran, H.; Kannan, S.; Demircioglu, F.E.; Oshiro, R.; Nety, S.P.; McKay, L.J.; Dlakić, M.; Inskeep, W.P.; Makarova, K.S.; Macrae, R.K.; Koonin, E.V.; Zhang, F. The widespread IS200/IS605 transposon family encodes diverse programmable RNA-guided endonucleases. Science. 2021, 374, 57-65. doi: 10.1126/science.abj6856.
44. Wu, J.; Yang, J.; Cho, W.C.; Zheng, Y. Argonaute proteins: Structural features, functions and emerging roles. J Adv Res. 2020, 24, 317-324. doi: 10.1016/j.jare.2020.04.017.
45. Cao, Y.; Sun, W.; Wang, J.; Sheng, G.; Xiang, G.; Zhang, T.; Shi, W.; Li, C.; Wang, Y.; Zhao, F.; Wang, H. Argonaute proteins from human gastrointestinal bacteria catalyze DNA-guided cleavage of single- and double-stranded DNA at 37 °C. Cell Discov. 2019, 5:38. doi: 10.1038/s41421-019-0105-y.
46. Škrlec, K.; Štrukelj, B.; Berlec, A. Non-immunoglobulin scaffolds: a focus on their targets. Trends Biotechnol. 2015, 33, 408-418. doi: 10.1016/j.tibtech.2015.03.012.
47. Yu, X.; Yang, Y.P.; Dikici, E.; Deo, S.K.; Daunert, S. Beyond Antibodies as Binding Partners: The Role of Antibody Mimetics in Bioanalysis. Annu Rev Anal Chem (Palo Alto Calif). 2017, 10, 293-320. doi: 10.1146/annurev-anchem-061516-045205.
48. Richards, D.A. Exploring alternative antibody scaffolds: Antibody fragments and antibody mimics for targeted drug delivery. Drug Discov Today Technol. 2018, 30, 35-46. doi: 10.1016/j.ddtec.2018.10.005.
49. Gebauer, M.; Skerra, A. Engineered Protein Scaffolds as Next-Generation Therapeutics. Annu. Rev. Pharmacol. Toxicol. 2020, 60:391-415. doi: 10.1146/annurev-pharmtox-010818-021118.
50. Lu, R.M.; Hwang, Y.C.; Liu, I.J.; Lee, C.C.; Tsai, H.Z.; Li, H.J.; Wu, H.C. Development of therapeutic antibodies for the treatment of diseases. J Biomed Sci. 2020, 27, 1. doi: 10.1186/s12929-019-0592-z.
51. Lipovsek, D.; Plückthun, A. In-vitro protein evolution by ribosome display and mRNA display. J Immunol Methods. 2004, 290, 51-67. doi: 10.1016/j.jim.2004.04.008.
52. Cherf, G.M.; Cochran, J.R. Applications of Yeast Surface Display for Protein Engineering. Methods Mol Biol. 2015, 1319, 155-175. doi: 10.1007/978-1-4939-2748-7_8.
53. Packer, M.S.; Liu, D.R. Methods for the directed evolution of proteins. Nat Rev Genet. 2015, 16, 379-394. doi: 10.1038/nrg3927.
54. Almagro, J.C.; Pedraza-Escalona, M.; Arrieta, H.I.; Pérez-Tapia, S.M. Phage Display Libraries for Antibody Therapeutic Discovery and Development. Antibodies (Basel). 2019, 8, 44. doi: 10.3390/antib8030044.
55. Alfaleh, M.A.; Alsaab, H.O.; Mahmoud, A.B.; Alkayyal, A.A.; Jones, M.L.; Mahler, S.M.; Hashem, A.M. Phage Display Derived Monoclonal Antibodies: From Bench to Bedside. Front Immunol. 2020, 11, 1986. doi: 10.3389/fimmu.2020.01986.
56. Chauhan, V.M.; Pantazes, R.J. MutDock: A computational docking approach for fixed-backbone protein scaffold design. Front Mol Biosci. 2022, 9, 933400. doi: 10.3389/fmolb.2022.933400.
57. Urvoas, A.; Guellouz, A.; Valerio-Lepiniec, M.; Graille, M.; Durand, D.; Desravines, D.C.; van Tilbeurgh, H.; Desmadril, M.; Minard, P. Design, production and molecular structure of a new family of artificial alpha-helicoidal repeat proteins (αRep) based on thermostable HEAT-like repeats. J Mol Biol. 2010,404, 307-327. doi: 10.1016/j.jmb.2010.09.048.
58. Lee, S.C.; Park, K.; Han, J.; Lee, J.J.; Kim, H.J.; Hong, S.; Heu, W.; Kim, Y.J.; Ha, J.S.; Lee, S.G.; Cheong, H.K.; Jeon, Y.H.; Kim, D.; Kim, H.S. Design of a binding scaffold based on variable lymphocyte receptors of jawless vertebrates by module engineering. Proc Natl Acad Sci U S A. 2012, 109, 3299-3304. doi: 10.1073/pnas.1113193109.
59. Grabulovski, D.; Kaspar, M.; Neri, D. A novel, non-immunogenic Fyn SH3-derived binding protein with tumor vascular targeting properties. J Biol Chem. 2007, 282, 3196-3204. doi: 10.1074/jbc.M609211200.
60. Tiede, C.; Bedford, R.; Heseltine, SJ.; Smith, G.; Wijetunga, I.; Ross, R.; AlQallaf, D.; Roberts, A.P.; Balls, A.; Curd, A.; Hughes, R.E.; Martin, H.; Needham, S.R.; Zanetti-Domingues, L.C.; Sadigh, Y.; Peacock, T.P.; Tang, A.A.; Gibson, N.; Kyle, H.; Platt, G.W.; Ingram, N.; Taylor, T.; Coletta, L.P.; Manfield, I.; Knowles, M.; Bell, S.; Esteves, F.; Maqbool, A.; Prasad, R.K.; Drinkhill, M.; Bon, R.S.; Patel, V.; Goodchild, S.A.; Martin-Fernandez, M.; Owens, RJ.; Nettleship, J.E.; Webb, M.E.; Harrison, M.; Lippiat, J.D.; Ponnambalam, S.; Peckham, M.; Smith, A.; Ferrigno, P.K.; Johnson, M.; McPherson, M.J.; Tomlinson, D.C. Affimer proteins are versatile and renewable affinity reagents. eLife. 2017, 6, e24903. doi: 10.7554/eLife.24903.
61. Lorey, S.; Fiedler, E.; Kunert, A.; Nerkamp, J.; Lange, C.; Fiedler, M.; Bosse-Doenecke, E.; Meysing, M.; Gloser, M.; Rundfeldt, C.; Rauchhaus, U.; Hänssgen, I.; Göttler, T.; Steuernagel, A.; Fiedler, U.; Haupts, U. Novel ubiquitin-derived high affinity binding proteins with tumor targeting properties. J Biol Chem. 2014, 289, 8493-8507. doi: 10.1074/jbc.M113.519884.
62. Silverman, J.; Liu, Q.; Bakker, A.; To, W.; Duguay, A.; Alba, B.M.; Smith, R.; Rivas, A.; Li, P.; Le, H.; Whitehorn, E.; Moore, K.W.; Swimmer, C.; Perlroth, V.; Vogt, M.; Kolkman, J.; Stemmer W.P. Multivalent avimer proteins evolved by exon shuffling of a family of human receptor domains. Nat Biotechnol. 2005, 23, 1556-1561. doi: 10.1038/nbt1166.
63. Hamers-Casterman, C.; Atarhouch T.; Muyldermans S.; Robinson G.; Hamers C.; Songa EB.; Bendahman N.; Hamers R. Naturally occurring antibodies devoid of light chains. Nature. 1993, 363, 446-448. doi: 10.1038/363446a0.
64. Greenberg, A.S.; Hughes, A.L.; Guo, J.; Avila, D.; McKinney, E.C.; Flajnik, M.F. A novel "chimeric" antibody class in cartilaginous fish: IgM may not be the primordial immunoglobulin. Eur J Immunol. 1996, 26, 1123-1129. doi: 10.1002/eji.1830260525.
65. Matz H.; Munir D.; Logue J.; Dooley H. The immunoglobulins of cartilaginous fishes. Dev Comp Immunol. 2021, 115, 103873. doi: 10.1016/j.dci.2020.103873.
66. Muyldermans, S. Applications of Nanobodies. Annu Rev Anim Biosci. 2021a, 9, 401-421. doi: 10.1146/annurev-animal-021419-083831.
67. Muyldermans, S. A guide to: generation and design of nanobodies. FEBS J. 2021b, 288, 2084-2102. doi: 10.1111/febs.15515.
68. Ingram, J.R.; Schmidt, F.I.; Ploegh, H.L. Exploiting Nanobodies' Singular Traits. Annu Rev Immunol. 2018, 36, 695-715. doi: 10.1146/annurev-immunol-042617-053327.
69. Yamagata, M.; Sanes, J.R. Reporter-nanobody fusions (RANbodies) as versatile, small, sensitive immunohistochemical reagents. Proc Natl Acad Sci U S A. 2018, 115, 2126-2131. doi: 10.1073/pnas.1722491115.
70. Cheloha, R.W.; Harmand, T.J.; Wijne, C.; Schwartz, T.U.; Ploegh, H.L. Exploring cellular biochemistry with nanobodies. J Biol Chem. 2020, 295, 15307-15327. doi: 10.1074/jbc.REV120.012960.
71. Arbabi-Ghahroudi, M. Camelid Single-Domain Antibodies: Promises and Challenges as Lifesaving Treatments. Int J Mol Sci. 2022, 23, 5009. doi: 10.3390/ijms23095009.
72. Plückthun, A. Designed ankyrin repeat proteins (DARPins): binding proteins for research, diagnostics, and therapy. Annu Rev Pharmacol Toxicol. 2015, 55, 489-511. doi: 10.1146/annurev-pharmtox-010611-134654.
73. Schilling, J.; Jost, C.; Ilie, I.M.; Schnabl, J.; Buechi, O.; Eapen, R.S.; Truffer, R.; Caflisch, A.; Forrer, P. Thermostable designed ankyrin repeat proteins (DARPins) as building blocks for innovative drugs. J. Biol. Chem. 2022, 298, 101403. doi: 10.1016/j.jbc.2021.101403.
74. Binz, H.K.; Stumpp, M.T.; Forrer, P.; Amstutz, P.; Plückthun, A. Designing repeat proteins: well-expressed, soluble and stable proteins from combinatorial libraries of consensus ankyrin repeat proteins. J Mol Biol. 2003, 332, 489-503. doi: 10.1016/s0022-2836(03)00896-9.
75. Veesler, D.; Dreier, B.; Blangy, S.; Lichière, J.; Tremblay, D.; Moineau, S.; Spinelli, S.; Tegoni, M.; Plückthun, A.; Campanacci V.; Cambillau C. Crystal structure and function of a DARPin neutralizing inhibitor of lactococcal phage TP901-1: comparison of DARPin and camelid VHH binding mode. J. Biol. Chem. 2009, 284, 30718-30726. doi: 10.1074/jbc.M109.037812.
76. Sha, F.; Salzman, G.; Gupta, A.; Koide, S. Monobodies and other synthetic binding proteins for expanding protein science. Protein Sci. 2017, 26, 910-924. doi: 10.1002/pro.3148.
77. Akkapeddi, P.; Teng, K.W.; Koide, S. Monobodies as tool biologics for accelerating target validation and druggable site discovery. RSC Med Chem. 2021, 12, 1839-1853. doi: 10.1039/d1md00188d.
78. Lipovsek, D. Adnectins: engineered target-binding protein therapeutics. Protein Eng Des Sel. 2011, 24, 3-9. doi: 10.1093/protein/gzq097.
79. Gross, G.G.; Junge, J.A.; Mora, R.J.; Kwon, H.B.; Olson, C.A.; Takahashi, T.T.; Liman, E.R.; Ellis-Davies, G.C.; McGee, A.W.; Sabatini, B.L.; Roberts, R.W.; Arnold, D.B. Recombinant probes for visualizing endogenous synaptic proteins in living neurons. Neuron. 2013, 78, 971-985. doi: 10.1016/j.neuron.2013.04.017.
80. Nygren, P.A. Alternative binding proteins: affibody binding proteins developed from a small three-helix bundle scaffold. FEBS J. 2008, 275, 2668-2676. doi: 10.1111/j.1742-4658.2008.06438.x.
81. Ståhl, S.; Gräslund, T.; Eriksson Karlström, A.; Frejd, F.Y.; Nygren, P. Å.; Löfblom, J. Affibody Molecules in Biotechnological and Medical Applications. Trends Biotechnol. 2017, 35, 691-712. doi: 10.1016/j.tibtech.2017.04.007.
82. Gebauer, M.; Skerra, A. Anticalins small engineered binding proteins based on the lipocalin scaffold. Methods Enzymol. 2012, 503, 157-188. doi: 10.1016/B978-0-12-396962-0.00007-0.
83. Rothe, C.; Skerra, A. Anticalin® Proteins as Therapeutic Agents in Human Diseases. BioDrugs. 2018, 32, 233-243. doi: 10.1007/s40259-018-0278-1.
84. Griffin, M.E.; Hsieh-Wilson, L.C. Tools for mammalian glycoscience research. Cell. 2022, 185, 2657-2677. doi: 10.1016/j.cell.2022.06.016.
85. Arnaud, J.; Audfray, A.; Imberty, A. Binding sugars: from natural lectins to synthetic receptors and engineered neolectins. Chem Soc Rev. 2013, 42, 4798-813. doi: 10.1039/c2cs35435g.
86. Tommasone, S .; Allabush, F .; Tagger, Y.K .; Norman, J .; Köpf, M .; Tucker, J.H.R .; Mendes, P.M . The challenges of glycan recognition with natural and artificial receptors. Chem Soc Rev. 2019, 48, 5488-5505. doi: 10.1039/c8cs00768c.
87. Ward, E.M.; Kizer, M.E.; Imperiali, B. Strategies and Tactics for the Development of Selective Glycan-Binding Proteins. ACS Chem Biol. 2021, 16, 1795-1813. doi: 10.1021/acschembio.0c00880.
88. Cummings, R.D.; Etzler, M.E. Antibodies and Lectins in Glycan Analysis. In: Varki, A.; Cummings, R.D.; Esko, J.D, et al., editors. Essentials of Glycobiology. 2nd edition. Cold Spring Harbor (NY): Cold Spring Harbor Laboratory Press; 2009. Chapter 45. Available from: https://www.ncbi.nlm.nih.gov/books/NBK1919/
89. Tsaneva, M.; Van Damme, E.J.M. 130 years of Plant Lectin Research. Glycoconj J. 2020, 37, 533-551. doi: 10.1007/s10719-020-09942-y.
90. Lundstr√∏m, J.; Korhonen, E.; Lisacek, F.; Bojar, D. LectinOracle: A Generalizable Deep Learning Model for Lectin-Glycan Binding Prediction. Adv Sci (Weinh). 2022, 9, e2103807. doi: 10.1002/advs.202103807.
91. Armenta, S.; Moreno-Mendieta, S.; Sánchez-Cuapio, Z.; Sánchez, S.; Rodríguez-Sanoja, R. Advances in molecular engineering of carbohydrate-binding modules. Proteins. 2017, 85, 1602-1617. doi: 10.1002/prot.25327.
92. Warkentin, R.; Kwan, D.H. Resources and Methods for Engineering "Designer" Glycan-Binding Proteins. Molecules. 2021, 26, 380. doi: 10.3390/molecules26020380.
93. Yabe, R.; Itakura, Y.; Nakamura-Tsuruta, S.; Iwaki, J.; Kuno, A.; Hirabayashi, J. Engineering a versatile tandem repeat-type alpha2-6sialic acid-binding lectin. Biochem Biophys Res Commun. 2009, 384, 204-209. doi: 10.1016/j.bbrc.2009.04.090.
94. Hu, D.; Tateno, H.; Sato, T.; Narimatsu, H.; Hirabayashi, J. Tailoring GalNAcα1-3Galβ-specific lectins from a multi-specific fungal galectin: dramatic change of carbohydrate specificity by a single amino-acid substitution. Biochem J. 2013, 453, 261-70. doi: 10.1042/BJ20121901.
95. Norton, P.; Comunale, M.A.; Herrera, H.; Wang, M.; Houser, J.; Wimmerova M.; Romano PR.; Mehta A. Development and application of a novel recombinant Aleuria aurantia lectin with enhanced core fucose binding for identification of glycoprotein biomarkers of hepatocellular carcinoma. Proteomics. 2016, 16, 3126-3136. doi: 10.1002/pmic.201600064.
96. Hirabayashi, J.; Arai, R. Lectin engineering: the possible and the actual. Interface Focus. 2019, 9, 20180068. doi: 10.1098/rsfs.2018.0068.
97. Notova, S.; Bonnardel, F.; Lisacek, F.; Varrot, A.; Imberty, A. Structure and engineering of tandem repeat lectins. Curr Opin Struct Biol. 2020, 62, 39-47. doi: 10.1016/j.sbi.2019.11.006.
98. Katoch, R.; Tripathi, A. Research advances and prospects of legume lectins. J Biosci. 2021, 46, 104. doi: 10.1007/s12038-021-00225-8.
99. Mattox, D.E.; Bailey-Kellogg, C. Comprehensive analysis of lectin-glycan interactions reveals determinants of lectin specificity. PLoS Comput Biol. 2021, 17, e1009470. doi: 10.1371/journal.pcbi.1009470.
100. Montalbán-López, M.; Scott, T.A.; Ramesh, S.; Rahman, I.R.; van Heel, A.J.; Viel, J.H.; Bandarian, V.; Dittmann, E.; Genilloud, O.; Goto, Y.; Grande Burgos, M.J.; Hill, C.; Kim, S.; Koehnke, J.; Latham, J.A.; Link, A.J.; Martínez, B.; Nair, S.K.; Nicolet, Y.; Rebuffat, S.; Sahl, H.G.; Sareen, D.; Schmidt, E.W.; Schmitt, L.; Severinov, K.; Süssmuth, R.D.; Truman, A.W.; Wang, H.; Weng, J.K.; van Wezel, G.P.; Zhang, Q.; Zhong, J.; Piel, J.; Mitchell, D.A.; Kuipers, O.P.; van der Donk, W.A. New developments in RiPP discovery, enzymology and engineering. Nat Prod Rep. 2021, 38, 130-239. doi: 10.1039/d0np00027b.
101. Hwang, S.; Lee, N.; Cho, S.; Palsson, B.; Cho, B.K. Repurposing Modular Polyketide Synthases and Non-ribosomal Peptide Synthetases for Novel Chemical Biosynthesis. Front Mol Biosci. 2020, 7, 87. doi: 10.3389/fmolb.2020.00087.
102. Wu, C.; van der Donk, W.A. Engineering of new-to-nature ribosomally synthesized and post-translationally modified peptide natural products. Curr Opin Biotechnol. 2021, 69, 221-231. doi: 10.1016/j.copbio.2020.12.022.
103. Yi, D.; Bayer, T.; Badenhorst, C.P.S.; Wu, S.; Doerr, M.; Höhne, M.; Bornscheuer, U.T. Recent trends in biocatalysis. Chem Soc Rev. 2021, 50, 8003-8049. doi: 10.1039/d0cs01575j.
この記事が気に入ったらサポートをしてみませんか?
