
Photo by
hoho8888
Rを使った主成分分析(2021年8月改訂)
最近、以前書いていた方法ではうまくできなかったので、2021年8月に改定しました。
2021年8月時点で、私のRのバージョンは R version 4.0.3 (2020-10-10) です。Bioconductorも下記サイトで再インストールしました。Bioconductor 3.13です。
https://bioconductor.org
> if (!requireNamespace("BiocManager", quietly = TRUE))
+ install.packages("BiocManager")
> BiocManager::install()ーーー
メタゲノム、トランスクリプトーム、メタボロームなどのオミックスデータのサンプル間の比較でよく用いられる主成分分析の方法。
まずは、準備として、Rのメニューバーの”パッケージとデータ”>"パッケージインストーラ"で"ggplot2" と "rgl" パッケージをインストール。
”meshr" もインストールしておきましょう。
下記のコマンドでもインストールできます。
> BiocManager::install("meshr")例えば、図のようなデータが与えられたとき、
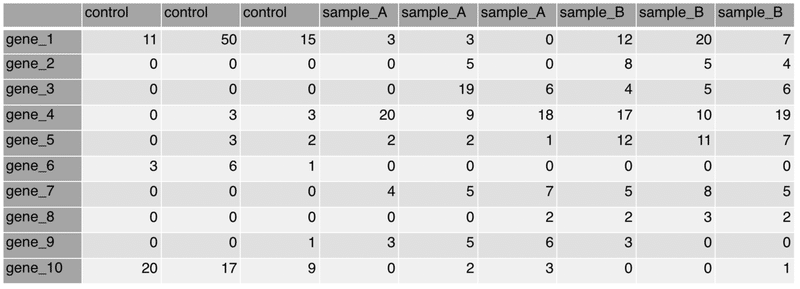
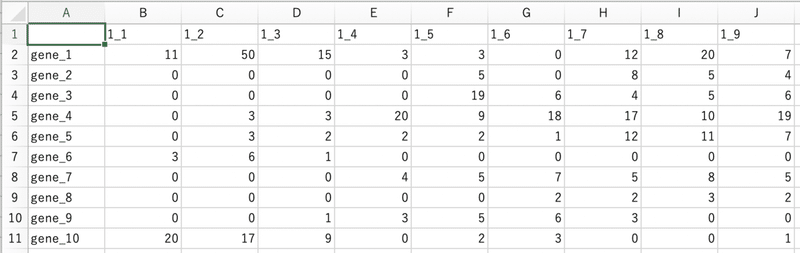
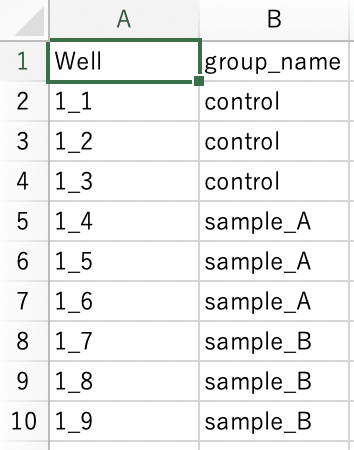
DataのファイルとGroupのファイルを以下のように作成し、csvファイルとして保存します。
Dataファイル;table.csv
Groupファイル;group.csv
ファイルを置いてある作業ディレクトリ、ここではデスクトップへ移動。
> setwd("~/Desktop")
DataとGroupの2つのファイルを読み込み。
> group<-read.csv("group.csv", header=T)
> data<-read.csv("table.csv", row.name=1, header=T)確認
> group
Well group_name
1 1_1 control
2 1_2 control
3 1_3 control
4 1_4 sample_A
5 1_5 sample_A
6 1_6 sample_A
7 1_7 sample_B
8 1_8 sample_B
9 1_9 sample_B
> data
X1_1 X1_2 X1_3 X1_4 X1_5 X1_6 X1_7 X1_8 X1_9
gene_1 11 50 15 3 3 0 12 20 7
gene_2 0 0 0 0 5 0 8 5 4
gene_3 0 0 0 0 19 6 4 5 6
gene_4 0 3 3 20 9 18 17 10 19
gene_5 0 3 2 2 2 1 12 11 7
gene_6 3 6 1 0 0 0 0 0 0
gene_7 0 0 0 4 5 7 5 8 5
gene_8 0 0 0 0 0 2 2 3 2
gene_9 0 0 1 3 5 6 3 0 0
gene_10 20 17 9 0 2 3 0 0 1
データファイルの行列変換
> data2<-t(data)
> data2
gene_1 gene_2 gene_3 gene_4 gene_5 gene_6 gene_7 gene_8 gene_9 gene_10
X1_1 11 0 0 0 0 3 0 0 0 20
X1_2 50 0 0 3 3 6 0 0 0 17
X1_3 15 0 0 3 2 1 0 0 1 9
X1_4 3 0 0 20 2 0 4 0 3 0
X1_5 3 5 19 9 2 0 5 0 5 2
X1_6 0 0 6 18 1 0 7 2 6 3
X1_7 12 8 4 17 12 0 5 2 3 0
X1_8 20 5 5 10 11 0 8 3 0 0
X1_9 7 4 6 19 7 0 5 2 0 1Data2のファイルにWell列を足して、groupファイルのWell列を記入。
> data3 <- data.frame(data2, Well=sub("X", "", rownames(data2)))
> colnames(group)[1]<-"Well"
> data3
gene_1 gene_2 gene_3 gene_4 gene_5 gene_6 gene_7 gene_8 gene_9 gene_10 Well
X1_1 11 0 0 0 0 3 0 0 0 20 1_1
X1_2 50 0 0 3 3 6 0 0 0 17 1_2
X1_3 15 0 0 3 2 1 0 0 1 9 1_3
X1_4 3 0 0 20 2 0 4 0 3 0 1_4
X1_5 3 5 19 9 2 0 5 0 5 2 1_5
X1_6 0 0 6 18 1 0 7 2 6 3 1_6
X1_7 12 8 4 17 12 0 5 2 3 0 1_7
X1_8 20 5 5 10 11 0 8 3 0 0 1_8
X1_9 7 4 6 19 7 0 5 2 0 1 1_9Groupとdata3のファイルを"Well”でマージ。
> data4 <- merge(group, data3, by ="Well")
> data4
Well group_name gene_1 gene_2 gene_3 gene_4 gene_5 gene_6 gene_7 gene_8 gene_9 gene_10
1 1_1 control 11 0 0 0 0 3 0 0 0 20
2 1_2 control 50 0 0 3 3 6 0 0 0 17
3 1_3 control 15 0 0 3 2 1 0 0 1 9
4 1_4 sample_A 3 0 0 20 2 0 4 0 3 0
5 1_5 sample_A 3 5 19 9 2 0 5 0 5 2
6 1_6 sample_A 0 0 6 18 1 0 7 2 6 3
7 1_7 sample_B 12 8 4 17 12 0 5 2 3 0
8 1_8 sample_B 20 5 5 10 11 0 8 3 0 0
9 1_9 sample_B 7 4 6 19 7 0 5 2 0 1Label用のファイルを作成
> Label <- data.frame(group_name=data4$group_name, Color=NA, Name=NA)
> Label$Color[which(Label$group_name=="sample_A")]<- rgb(1, 0, 0, 0.5)
> Label$Color[which(Label$group_name=="sample_B")]<- rgb(0, 1, 0, 0.5)
> Label$Color[which(Label$group_name=="control")]<- rgb(0, 0, 0, 0.5)
> Label$Name[which(Label$group_name=="sample_A")]<- "A"
> Label$Name[which(Label$group_name=="sample_B")]<- "B"
> Label$Name[which(Label$group_name=="control")]<- "control"
> Label
group_name Color Name
1 control #00000080 control
2 control #00000080 control
3 control #00000080 control
4 sample_A #FF000080 A
5 sample_A #FF000080 A
6 sample_A #FF000080 A
7 sample_B #00FF0080 B
8 sample_B #00FF0080 B
9 sample_B #00FF0080 BData4から必要な部分だけ取り出して、行列変換。
> data5<-t(data4[,3:ncol(data4)])
> data5
[,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9]
gene_1 11 50 15 3 3 0 12 20 7
gene_2 0 0 0 0 5 0 8 5 4
gene_3 0 0 0 0 19 6 4 5 6
gene_4 0 3 3 20 9 18 17 10 19
gene_5 0 3 2 2 2 1 12 11 7
gene_6 3 6 1 0 0 0 0 0 0
gene_7 0 0 0 4 5 7 5 8 5
gene_8 0 0 0 0 0 2 2 3 2
gene_9 0 0 1 3 5 6 3 0 0
gene_10 20 17 9 0 2 3 0 0 1PCA計算
> pca <- prcomp(data5, scale = TRUE)
> summary(pca)
Importance of components:
PC1 PC2 PC3 PC4 PC5 PC6 PC7 PC8 PC9
Standard deviation 1.9584 1.7679 0.9906 0.89327 0.39786 0.24857 0.18769 0.06639 0.01529
Proportion of Variance 0.4262 0.3473 0.1090 0.08866 0.01759 0.00687 0.00391 0.00049 0.00003
Cumulative Proportion 0.4262 0.7734 0.8825 0.97112 0.98871 0.99557 0.99948 0.99997 1.000003d グラフを描いて回してみる。
> library("rgl")
> plot3d(pca$rotation[, 1:3], xlab="PC1", ylab="PC2", zlab="PC3", col=Label$Color)2Dで論文用に図を描画。
> library(maptools)
> plot(pca$rotation[,1],pca$rotation[,2], col=Label$Color, pch=19, cex =3)
> pointLabel(pca$rotation[,1],pca$rotation[,2], labels=Label$Name)保存
ラベルなし
> tiff(filename="test_PCA.tiff")
> plot(pca$rotation[,1],pca$rotation[,2], col=Label$Color, pch=19, cex =3)
> dev.off()
quartz
2 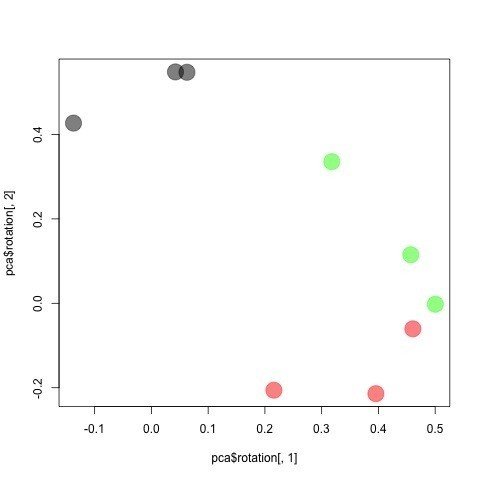
ラベルあり
> tiff(filename="test_PCA.tiff")
> plot(pca$rotation[,1],pca$rotation[,2], col=Label$Color, pch=19, cex =3)
> pointLabel(pca$rotation[,1],pca$rotation[,2], labels=Label$Name)
> dev.off()
quartz
2 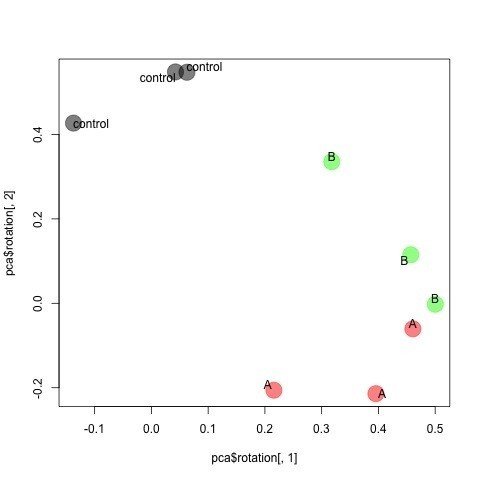
この記事が気に入ったらサポートをしてみませんか?
