
biglasso: Extend Lasso Model Fitting to Big Data in R
先日,貧弱な計算環境で R を使用して分析している時に,そこそこ大きなテーブルデータ(50GB 程)を使って Lasso 回帰を行う必要がでてきてしまいました.無理矢理計算を走らせたところ,普通に計算機が固まり帰宅したくなりました.
が・・・,そこは一応社会人ですので何か解決策はないか調べていたところ,biglasso という下記のリポジトリに行きつきました.
下記論文によると,16 GB しかメモリのないラップトップ上で,31 GB のデータを使った Lasso 回帰が可能 らしいです.
この package を既に試している方(いつもお世話になっております)がいたので,そちらも参考にさせていただきました.
ところで,業務等で未知の package を初めて使う時は,皆さんはどうされているでしょうか?
私の場合は,簡単なデータを使って,信頼性が高く実績のあるモデルとの整合性を検証してから使用しています.既に使用実績がたくさんある package であれば特に疑いもなく使用しても良いと思いますが,そうでない場合は検証してから使用する方が良いですよね.
ということで,今回は biglasso の計算結果の妥当性を検証したいと思います.
具体的には,Friedman・Hastie・Tibshirani といった機械学習・統計学界の著名人らによって開発され,多くの人に使用され検証されてきているであろう glmnet (https://glmnet.stanford.edu/articles/glmnet.html) を使った結果と相違ないことを検証したいと思います.
glmnet は elastic net を行うことができる package で,ハイパーパラメータ $${\alpha}$$ を指定することで lasso や ridge 回帰も行うことができます.
The elastic net penalty is controlled by α, and bridges the gap between lasso regression (α=1, the default) and ridge regression (α=0). The tuning parameter λ controls the overall strength of the penalty.
それでは,検証していきましょう.
1. 準備
最初に,必要なライブラリ群を install + load してしまいましょう.
とても big data とは呼べない too small data ではありますが,今回は MASS パッケージの Boston データセットを使用します.
install.packages("biglasso")
library(MASS)
library(cvTools)
library(glmnet)
library(biglasso)
library(doParallel)
library(Metrics)
data("Boston")次に,Lasso の場合は,交差検証でハイパーパラメータを決定する必要がありますので,validation scheme (cross validation の枠組み) を作成します.
ここで 1 つ注意点があります.
glmnet::cv.glmnet の option である nfolds の値と biglasso::cv.biglasso の option の nfolds の値を同じ値にさせても,内部での fold の切り方が異なるせいか結果が一致しません.
そこで,train data に関しては,明示的に fold の番号を glmnet::cv.glmnet 及び biglasso::cv.biglasso の各 option で与えることにします.そのため,下のコードの最終行で train 内の各データ点に対する fold 割当番号を cvTools::cvFolds で作成しています.
今回は,train : hold out = 4 : 1,train data 内の cv fold 数は 10 としました.
hold.out.idx <- sample(1:nrow(Boston), size = round(nrow(Boston) / 5))
train.x <- Boston[-hold.out.idx, -ncol(Boston)]
train.y <- Boston[-hold.out.idx, ncol(Boston)]
hold.out.x <- Boston[hold.out.idx, -ncol(Boston)]
hold.out.y <- Boston[hold.out.idx, ncol(Boston)]
f <- cvFolds(n = nrow(train.x), K = 10, type = "random")2. glmnet の実装と結果
glmenet のコードと結果は以下のようになります.
前述の通り,foldid option にて明示的に各データ点に対する fold を指定しています.
# - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - -
# Lasso with glmnet
# - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - -
set.seed(2020)
lasso.cv <- cv.glmnet(
x = as.matrix(train.x), y = as.matrix(train.y),
type.measure = "mse",
nfolds = 10,
foldid = f$which,
family = 'gaussian',
alpha = 1 # Lasso
)
plot(lasso.cv)
cat(lasso.cv$lambda.min, lasso.cv$lambda.1se)
# 0.005923991 0.3235848
rmse(hold.out.y,
predict(lasso.cv, as.matrix(hold.out.x), s = "lambda.min"))
# 4.504093
coef(lasso.cv, s = "lambda.min")
14 x 1 sparse Matrix of class "dgCMatrix"
1
(Intercept) 39.06057098
crim -0.10587859
zn 0.04295487
indus 0.02221518
chas 2.17875382
nox -19.32012390
rm 3.54571013
age 0.01537566
dis -1.42856477
rad 0.32492394
tax -0.01195581
ptratio -0.99041597
black 0.00900786
lstat -0.58933656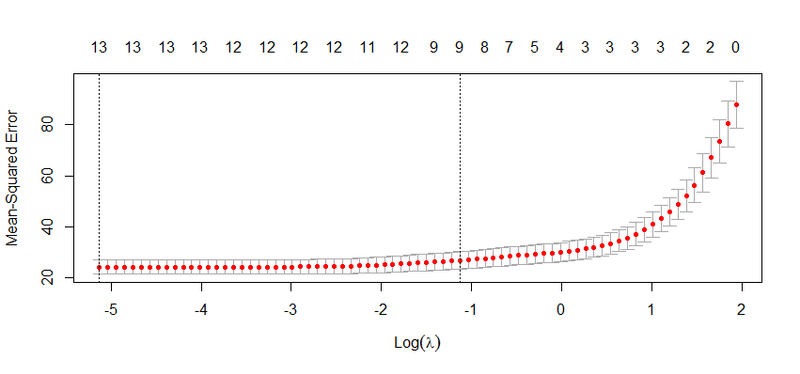
交差検証の結果,fold 平均の mse (family = 'gaussian' なので尤度と等価) を最小にする lasso.cv$lambda.min は ~0.006 となりました(上図で左側の垂直破線の位置に対応)
そして,rmse は約 4.504 となっています(コード参照).
3. biglasso の実装と結果
今度は biglasso で実装をしてみましょう.詳しい使い方は,開発者の reference manual を参照してください.
コードは以下になります.
glmnet 同様に,cv.biglasso では cv.ind option にて各データ点に対する fold を指定しています.
# - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - -
# Lasso with biglasso
# - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - -
train.x.big <- as.big.matrix(train.x)
hold.out.x.big <- as.big.matrix(hold.out.x)
biglasso.cv <- cv.biglasso(
X = train.x.big, y = train.y,
family = 'gaussian',
seed = 2020,
nfolds = 10,
cv.ind = f$which,
ncores = detectCores(),
alpha = 1 # Lasso
)
plot(biglasso.cv)
cat(biglasso.cv$lambda.min)
# 0.006971424
rmse(hold.out.y, predict(biglasso.cv, hold.out.x.big)[, 1])
# 4.502594
coef(biglasso.cv)
14 x 1 sparse Matrix of class "dgCMatrix"
0.007
(Intercept) 38.964676086
crim -0.105399557
zn 0.042754211
indus 0.021102471
chas 2.179749968
nox -19.238829392
rm 3.549205596
age 0.015195943
dis -1.426514650
rad 0.322297298
tax -0.011839676
ptratio -0.989156023
black 0.008999109
lstat -0.589049818
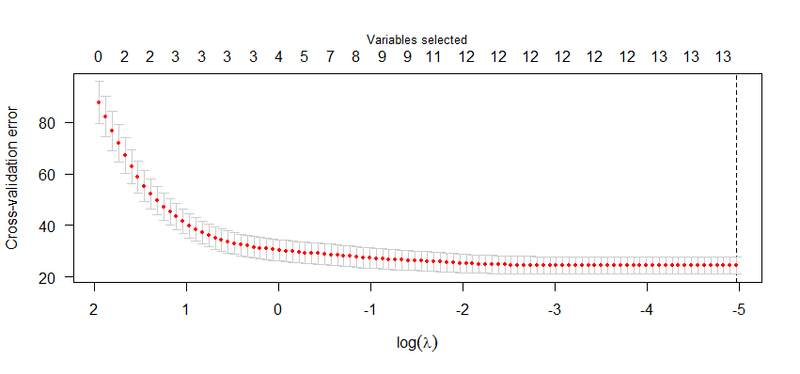
(横軸の log(λ) の向きが glmnet の plot と比較して左右反転している点に注意)
計算の結果,biglasso の lambda.min は 約 0.007 となりました.
glmnet の場合は 約 0.006 でしたので,lambda の値はそもそもぶれやすいことを鑑みると,ほぼ同じといって良さそうです.
また,biglasso における rmse は約 4.503 と,glmnet の 約 4.504 と,こちらもほぼ同じ値になりました.
4. まとめ
今回の検証結果に限っては biglasso の計算結果は信頼して使っても良いといえそうです.
別のデータセットや設定で似たような検証をされた方は是非コメントいただけると嬉しく思います.
この記事が気に入ったらサポートをしてみませんか?
