超・バイオインフォマティクス入門!
こんにちは。翡翠です。
前回の記事は、実はかなり削った部分があります。(まだ読んでいない人は先に読んでください!)
理系院卒の怒り #8:『PCR』の凄さを嘗めるな!
https://note.com/kawasemi_no_hina/n/n5361fa19cc53
途中まで書いた部分があったので、紹介しようと思います。
前回の途中で、WHOの作成した検出マニュアルに記載されている、リアルタイムRT-PCR検査用のリバースプライマーの配列「CTCCCTTTGTTGTGTTGT」について、ヒトゲノムの中にこれと完全に一致する配列が存在することを指摘した記事を紹介しました。
これを根拠に、記事の筆者(FAUXLEX)は「これは、検査結果に大きな影響を及ぼすはずだ!」と言っています。
アデニン(A)、チミン(T)、グアニン(G)、シトシン(C)の4種類からなる、この18塩基のプライマーと全く同じ配列が現れる確率は、約700億分の1ですから(20塩基にすると約1兆分の1ですからそれよりは高いですが、)、この件については、私自身、正直驚きました。
BOMBSHELL: WHO Coronavirus PCR Test Primer Sequence is Found in All Human DNA
https://pieceofmindful.com/2020/04/06/bombshell-who-coronavirus-pcr-test-primer-sequence-is-found-in-all-human-dna/
繰り返しになりますが、リバースプライマーが一致しているだけでは、PCRで増幅されませんので、全く意味がありません。PCRの原理が理解できていない、頓珍漢な指摘です。(ただ、原理の理解が伴っていない部分が残念ではありますが、何でも疑いを持って調べようとする姿勢は素晴らしいと思います。)
FAUXLEX氏は、どのようにしてヒトゲノムの約30億塩基もの中から、検出用プライマーと一致する配列を見つけたのでしょうか?

「ある遺伝子の配列が、動植物や、細菌、ウイルスのゲノムに含まれているかどうか知りたい!」
そういう時に研究者たちが使用するのが、アメリカの国立生物工学情報センター(NCBI)の『BLAST(Basic Local Alignment Search Tool)』です。
バイオインフォマティクス分野で、最もよく使われているプログラムの一つです。
難しいプログラムの知識は一切必要なく、ウェブブラウザ上で使えるプログラムですので、これを使って一緒に配列を探してみましょう!
まずは、こちらのURLをクリックしてください。下の画面が出てきます。
https://blast.ncbi.nlm.nih.gov/Blast.cgi
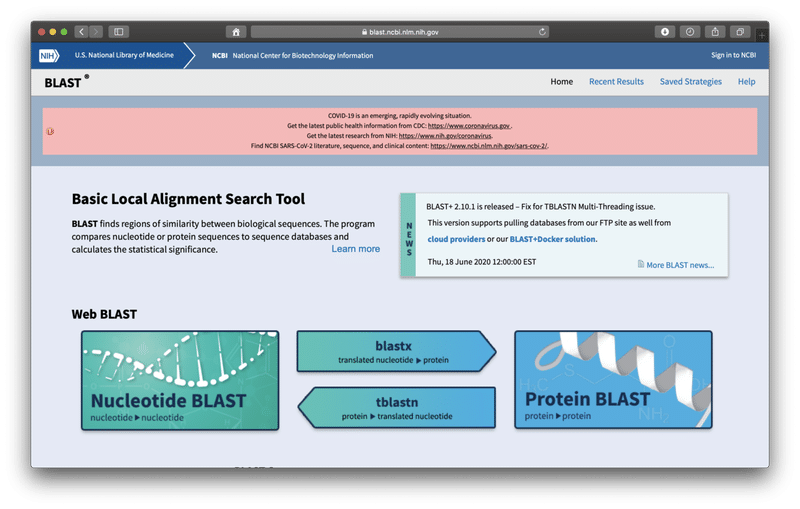
塩基配列を調べたいので、左下の「Nucleotide BLAST」をクリックします。
すると、下の画面に移ります。
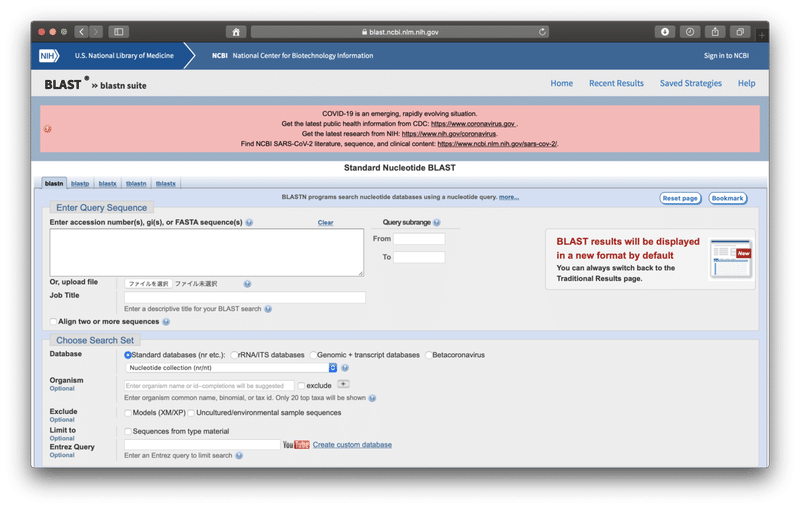
調べたい配列、WHOのリバースプライマー「CTCCCTTTGTTGTGTTGT」を黄色の部分に入力します。
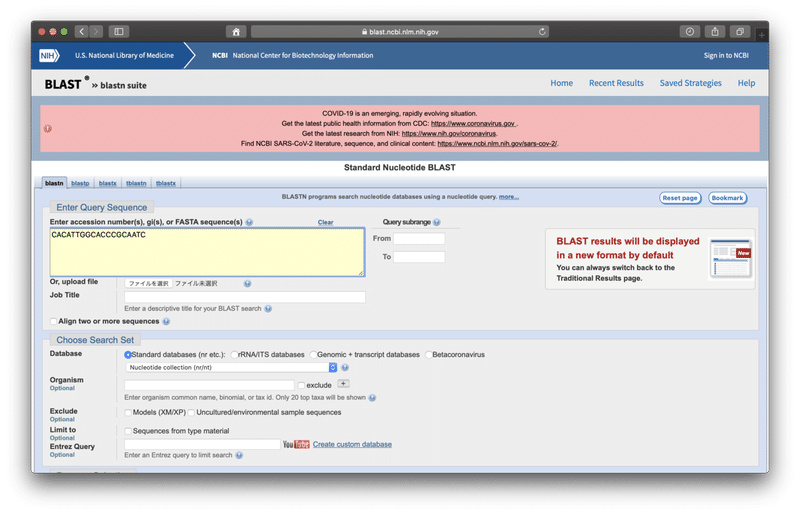
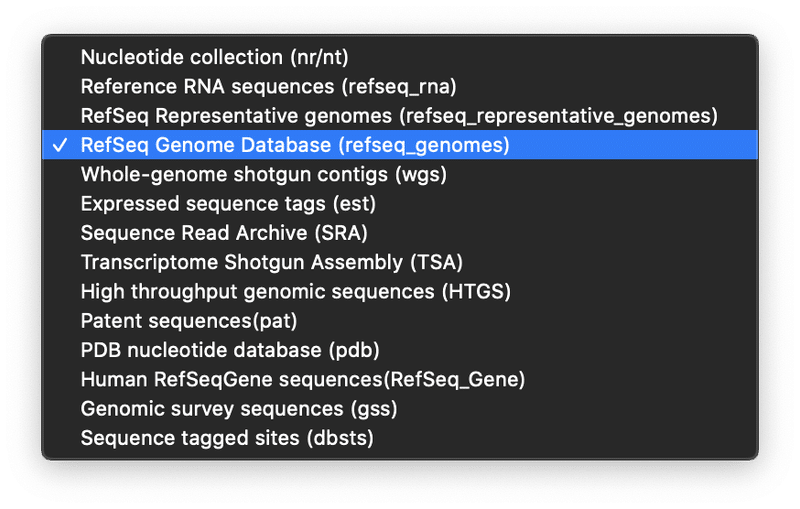
次に、Databaseの中から、「RefSeq Genome Database (refseq_genomes)」を選択します。
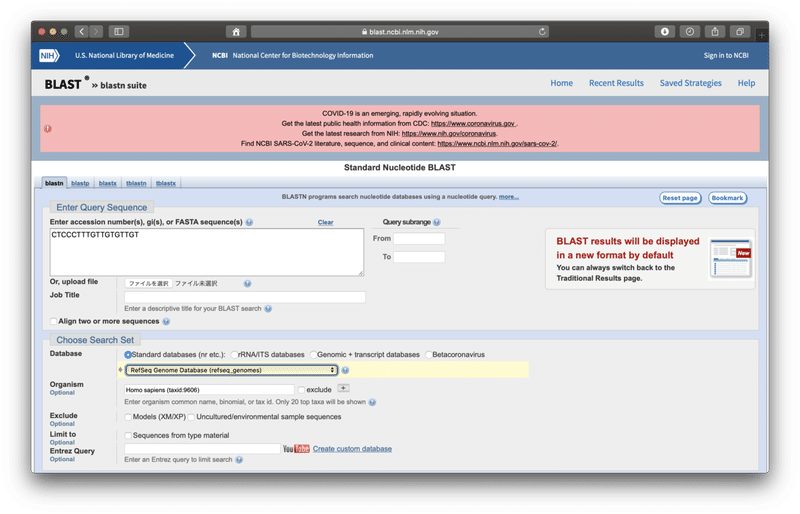
今回は、ヒトゲノムの中にこの配列があるか調べたいので、その下のOrganism(生物種)に「Homo sapiens (taxid:9606)」と入力します。(途中まで入力すれば、選択できるようになります。)
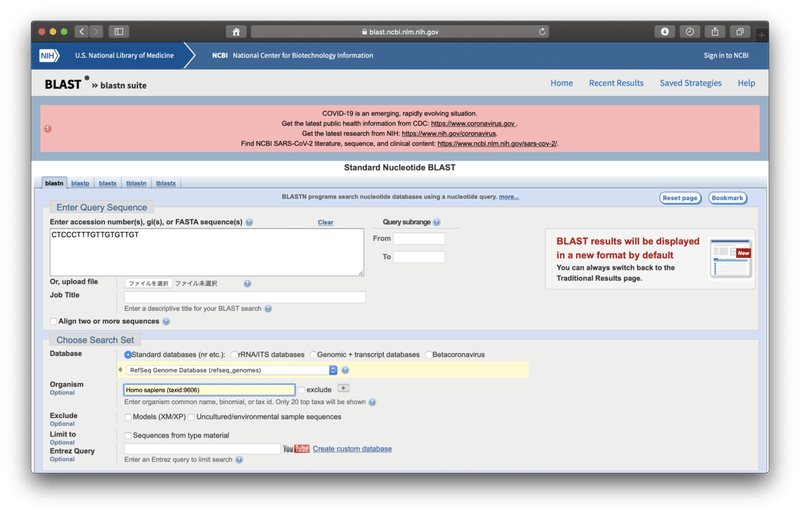
これだけでOKです!
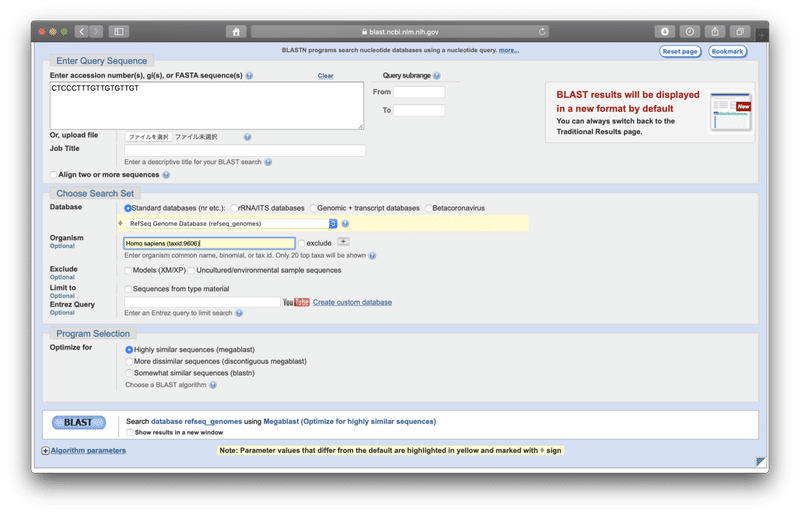
左下の青色の「BLAST」ボタンをクリックして、検索開始!!
しばらくすると、結果の画面に切り替わります(〜3分)。
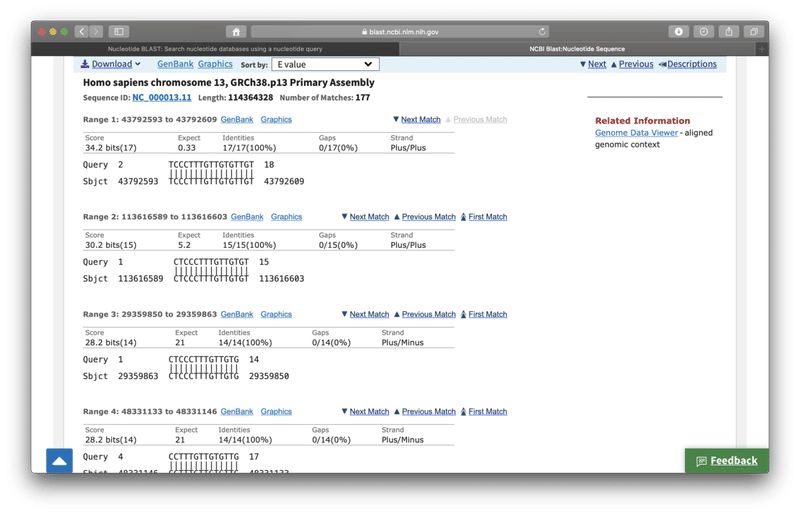
そして、下にスクロールすると、一致する配列(全一致、部分一致全て)が表示されます。上の方から、一致率が高い順になっています。
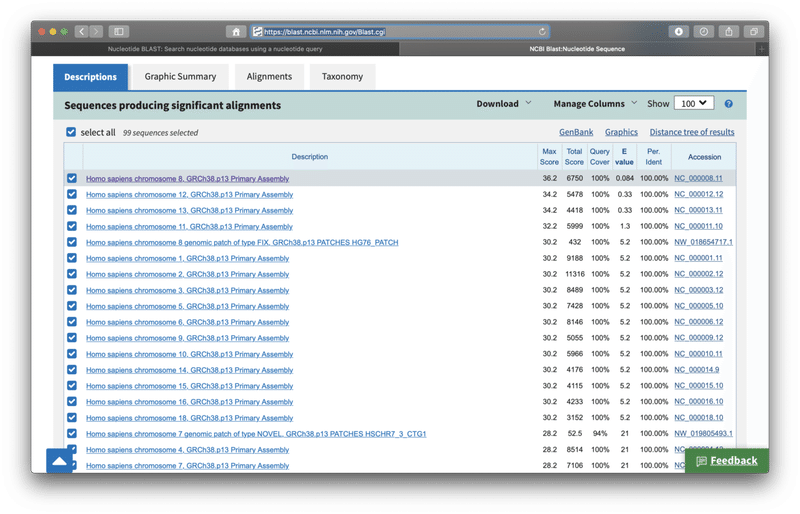
そして、「Alignments」のタブをクリックすると、WHOのリバースプライマーの配列「CTCCCTTTGTTGTGTTGT」とヒトゲノムの中の配列が、どの程度一致しているかが分かります。
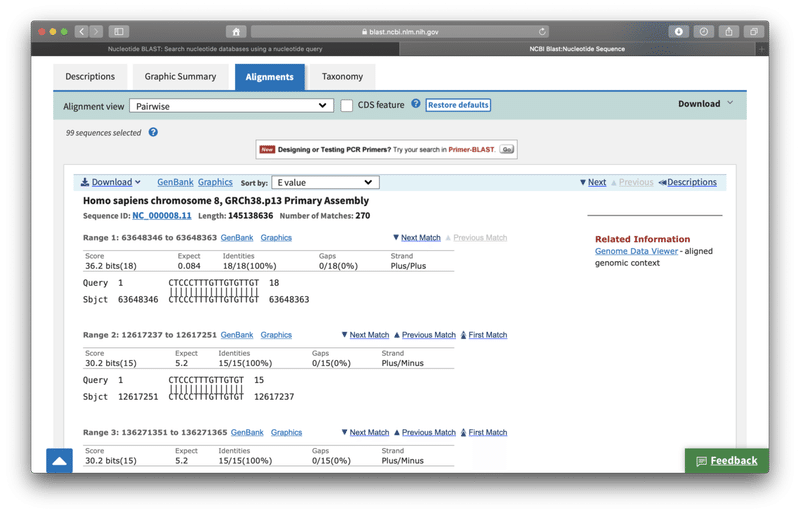
確かに記事の通り、8番染色体の中に18塩基のリバースプライマーと完全に一致する配列が見つかりました!
ちなみに、12番染色体、13番染色体の中にも、プライマーと17塩基一致する配列が見つかりました。
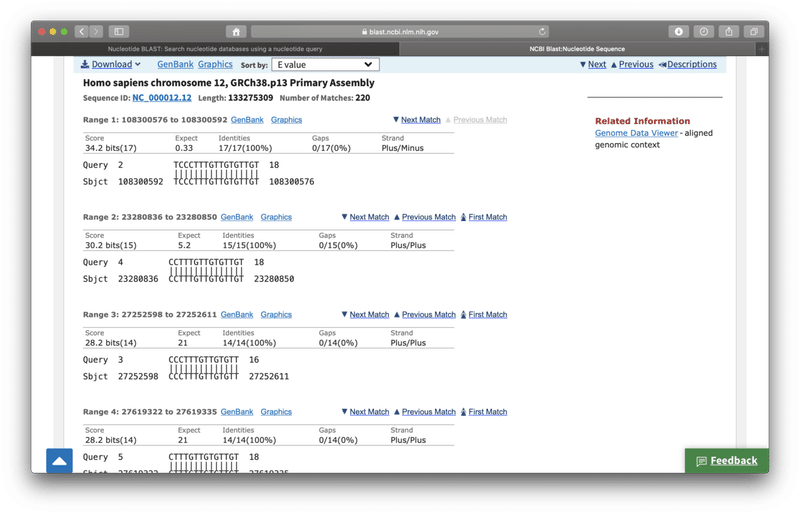
簡単ですね!
このようにして、FAUXLEX氏も記事に掲載された情報を検索したと思います。

世界的にはNCBIのBLASTが使われていると思いますが、日本にはもっと簡単に一致する配列を検索する方法があります!
それが『超絶高速ゲノム配列検索 GGGenome』です。(名前。笑)
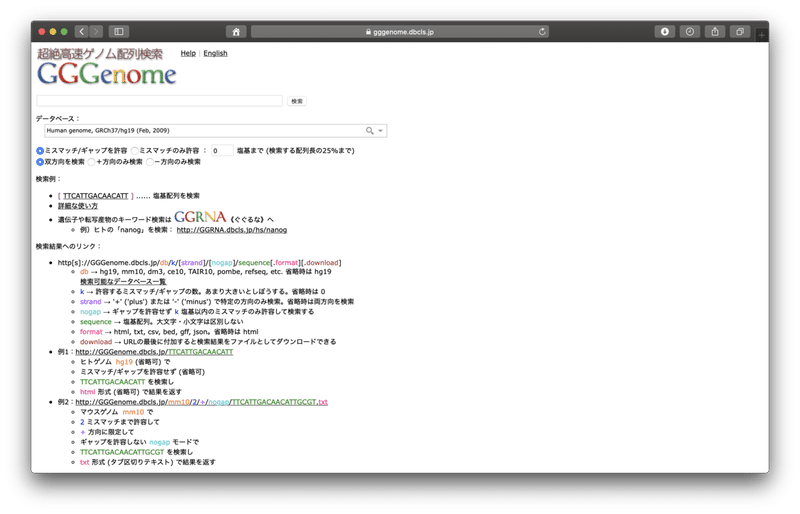
超絶高速かつ超絶簡単(笑)です。
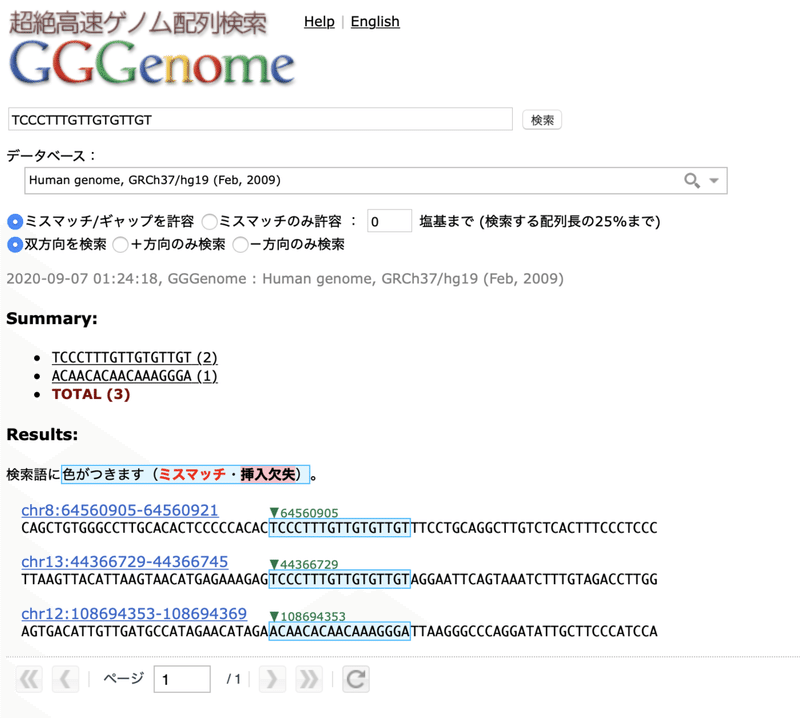
調べたい配列「CTCCCTTTGTTGTGTTGT」を検索欄に入力するだけです。

NCBIのBLASTと同様、8番染色体(chr8)に一致する配列が見つかりました!
早い!

プライマーの先頭のCを削って「TCCCTTTGTTGTGTTGT」の17塩基を検索欄に入力すれば、12番染色体、13番染色体の一致する配列も出てきます。
ただし!
これまで紹介したのは、『ヒトゲノム』と一致する配列を見つける方法です!
ご存知、新型コロナウイルス(SARS-CoV-2)は、RNAウイルスです。
検体(咽頭ぬぐい液や唾液)から精製したRNAサンプル中にはゲノムDNAは含まれません。
DNA分解酵素(DNase)を添加することにより、サンプルからDNAを完全に除去しています。
RNA 抽出を行う前の DNase 処理について
QIAamp Viral RNA Mini Kit を用いて RNA 抽出を行う際、喀痰成分は Buffer
AVL を加える事で溶解する。しかし、Buffer AVL 溶解液中の喀痰由来のゲノム 等の夾雑物により、RNA 抽出効率が著しく低下する事があるため、10%DTT in PBS 溶解液を RNA 抽出に供する場合は溶解液を事前に DNase 処理する必要が ある。
https://www.niid.go.jp/niid/images/lab-manual/2019-nCoV20200319.pdf
したがって、BLAST(GGGenome)で、ヒトゲノム(染色体)を検索して、プライマーと一致する・結合する配列を見つけることは、全く意味のないことです!!
大橋氏は、相変わらずですね。(苦笑
ガチで何も分かっていないのか、それともPCR検査を批判し、信者たちにこれに対する疑念を持たせるためなら、どんなデタラメな情報でも構わないのか...

騒動の原因は、PCRが人の遺伝子に反応するためという説(2020/09/09)
https://youtu.be/0UB8vXDvr6U
『RNAの中に一致する配列があるかどうか。』
これが重要です。
セントラルドグマという言葉をご存知でしょうか?
細胞内では、まず、DNAの塩基配列が、RNAの塩基配列に写し取られます。この過程を転写といいます。次に、RNAの塩基配列がタンパク質のアミノ酸配列に置き換えられます。この過程を翻訳といいます。
この一連の『DNA→(転写)→RNA→(翻訳)→タンパク質』という過程は、全ての生物に共通するもので、これを分子生物学の中心原理『セントラルドグマ』と言います。
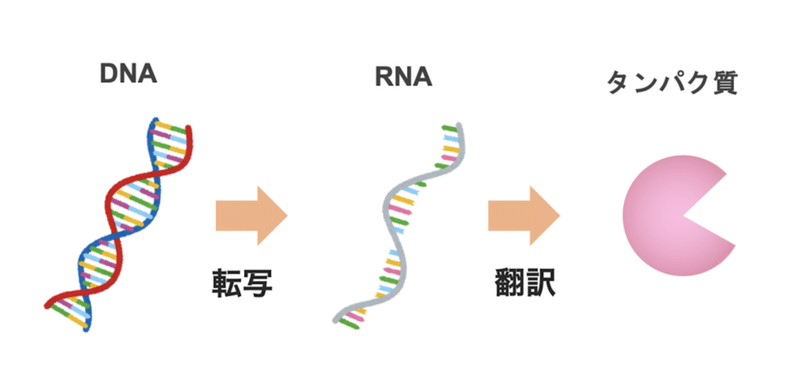
ただし、DNAの遺伝情報がRNAに写し取られただけでは次の翻訳のステップに進みません。
写し取られたばかりのRNAは『mRNA前駆体』と呼ばれますが、それが翻訳に進むためには、RNAプロセシング(成熟化)と呼ばれるステップが必要になります。
この時、mRNA前駆体に対して行われるのが、1. キャップ構造の付加、2. RNAスプライシング、3. ポリA配列の付加です。
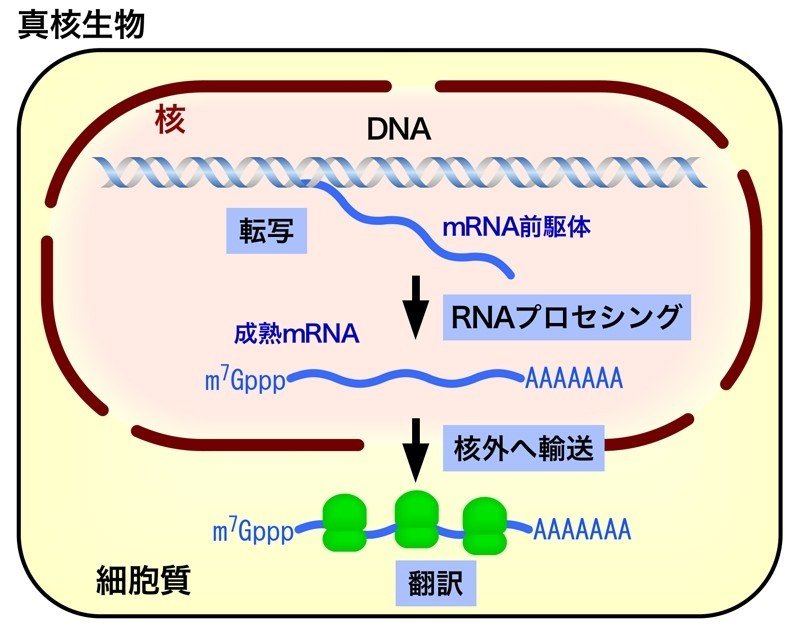
http://nsgene-lab.jp/expression/rna_processing/
今回のキーワードは『RNAスプライシング』です。
ゲノムDNAと、そこから転写されたばかりのmRNA前駆体には、『イントロン』と呼ばれる、タンパク質(アミノ酸配列)には翻訳されない配列が存在します。
RNAスプライシングとは、このイントロンを除き、アミノ酸配列に翻訳される『エクソン(エキソンとも呼ぶ)』と呼ばれる配列を繋げ、完全なmRNA(成熟mRNA)を作ることを言います。
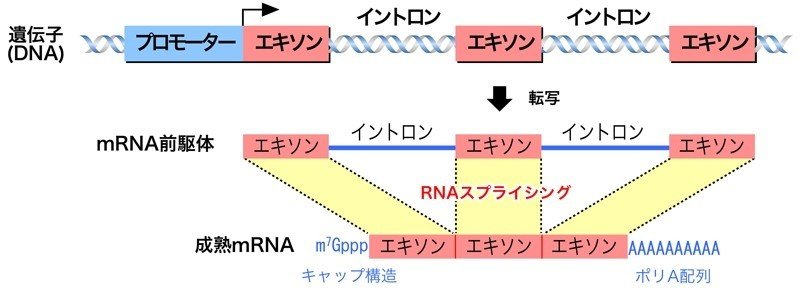
http://nsgene-lab.jp/expression/rna_processing/
補足)どうして、こんなややこしいシステムを持っているのでしょうか?
例えば、1つの遺伝子に5つのエクソン(図中、Exson 1〜5)があったとします。転写されたばかりのmRNA前駆体(図中、RNA)の、どれをエクソンとするか(どのイントロンを切り出すか)を”選択”することにより、1つの遺伝子から塩基配列の異なる複数の成熟mRNAを作り出すことができます。
これを、選択的スプライシング(Alternative Splicing)と呼びます。
それぞれの成熟mRNAからは異なる構造を持つタンパク質(Protein A〜C)が作られるため、生物の複雑性をこの選択的スプライシングが担っていると考えられています。
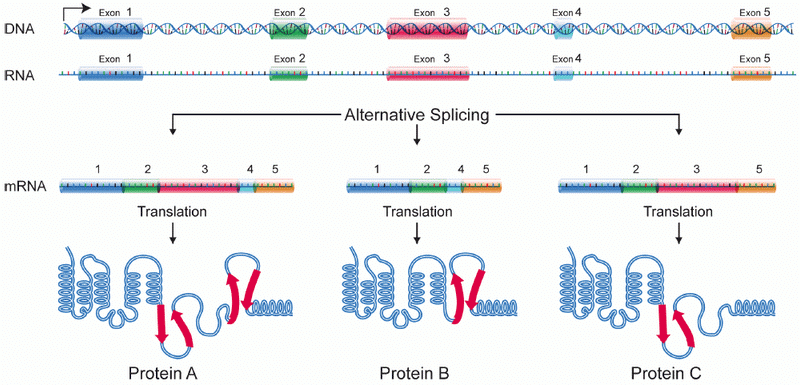
このRNAスプライシングによりイントロンが除かれ、エキソン同士が繋がることで、ゲノムDNAにはなかった新しい配列が生まれます。
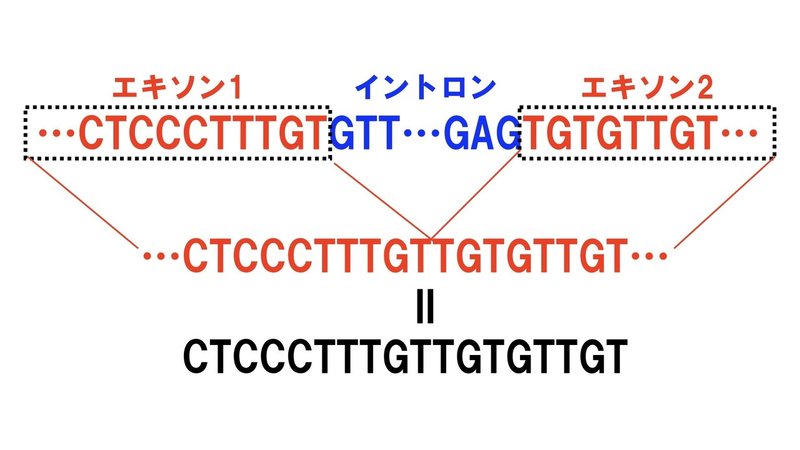
したがって、「ゲノムDNA(染色体)の配列にプライマーと一致する配列が存在する!」と騒ぐのがそもそもバカ丸出しで、本当に調べるべきは、RNAサンプル中のRNAの配列にプライマーと一致する配列があるかどうかです!

では、RNAサンプル中に存在する、WHOのリバースプライマーと一致する配列を見つけるにはどうしたらいいでしょうか?
実際に、『超絶高速ゲノム配列検索 GGGenome』を使って調べてみましょう。(名前は『ゲノム配列検索』ですが、RNAと一致する配列も調べることができます。)
データベースの欄に、今度は「RefSeq human RNA release 200 (May, 2020)」と入力します。
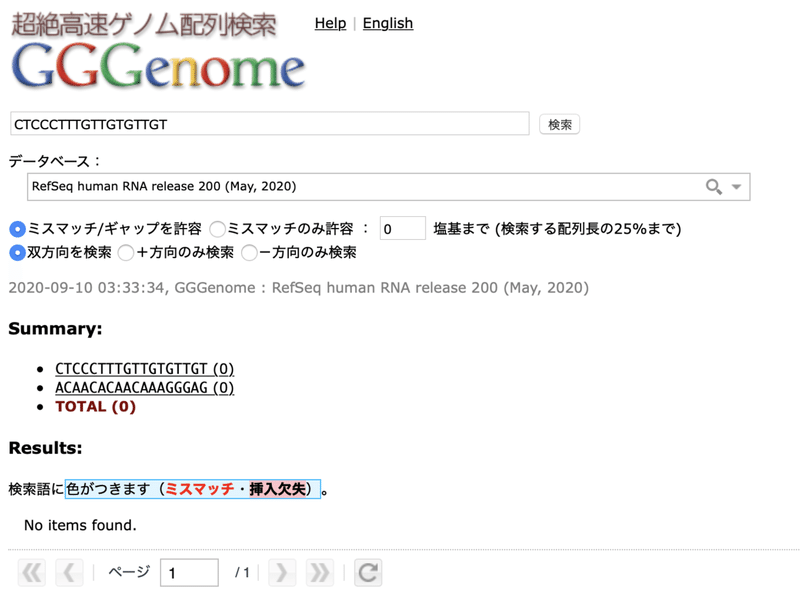
そして、調べたい配列「CTCCCTTTGTTGTGTTGT」を検索欄に入力します。
検索すると「No items found.」と表示されました。
つまり、RNAサンプル中には、WHOのリバースプライマー18塩基と完全に一致する配列は見つかりませんでした。(8番染色体の配列は、成熟mRNAに残らない配列だった訳ですね。)
プライマー内部の配列のうち、数塩基の違いであれば、完全に一致していなくとも結合する可能性があります。ただし、長さが違ってしまうとダメです。
したがって、次は「ミスマッチのみ許容」を選択し、0を「2」に変えて、2つの塩基の違いまで許容して検索してみましょう。
すると、今度はたくさんの配列が出てきました。
これらのRNAにはWHOのリバースプライマーが結合する可能性があります。
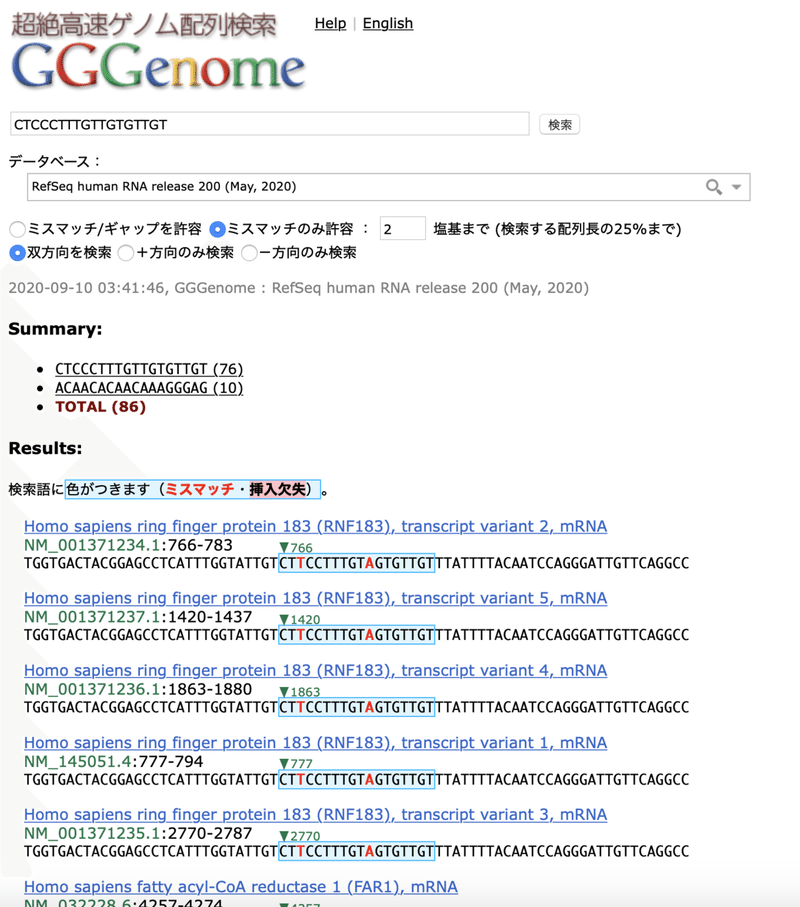
注)同じ名前の遺伝子が並んでいますが、「transcript variant」は、先に説明した選択的スプライシングにより生じた『異なる成熟mRNA』を意味します。
その遺伝子名(Homo sapiens ring finger protein 183)をクリックすると、NCBIのデータベースに飛びます。
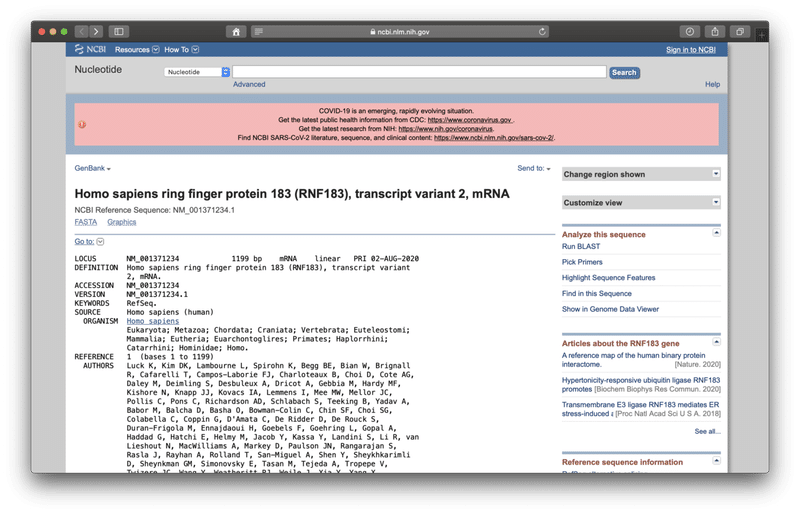
この遺伝子に関する様々な情報が載っていますが、遺伝子の名前の下にある「FASTA」をクリックすると遺伝子の配列だけが表示されます。
水色で示したのが、GGGenomeで見つかったリバースプライマーが結合する可能性のある配列です。
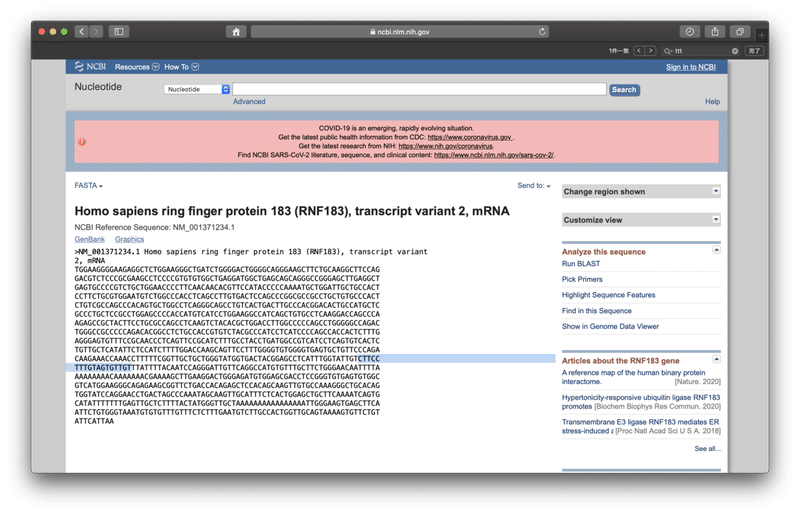
もし、この1199塩基の長さのmRNAに、WHOのリバースプライマーとセットになったフォワードプライマー「ATGAGCTTAGTCCTGTTG」と、プローブ「AGATGTCTTGTGCTGCCGGTA」が結合すれば、リアルタイムRT-PCRで検出されます。
つまり、新型コロナウイルスが感染していなくても『陽性』となる訳です!
大橋眞信者は大喜びですね!笑
日本におけるPCR検査では、以下のプライマーとプローブのセットが使われていますから、検索してみてください。
Nセット
フォワードプライマー:CACATTGGCACCCGCAATC
リバースプライマー:GAGGAACGAGAAGAGGCTTG
プローブ:ACTTCCTCAAGGAACAACATTGCCA
N2セット
フォワードプライマー:AAATTTTGGGGACCAGGAAC
リバースプライマー:TGGCAGCTGTGTAGGTCAAC
プローブ:ATGTCGCGCATTGGCATGGA
https://www.niid.go.jp/niid/images/lab-manual/2019-nCoV20200319.pdf
ただし、仮に同じ遺伝子上に、この3つが結合する配列が見つかったとしても、その『並び』にも気を付けなければなりません。
DNAには『向き』があります。
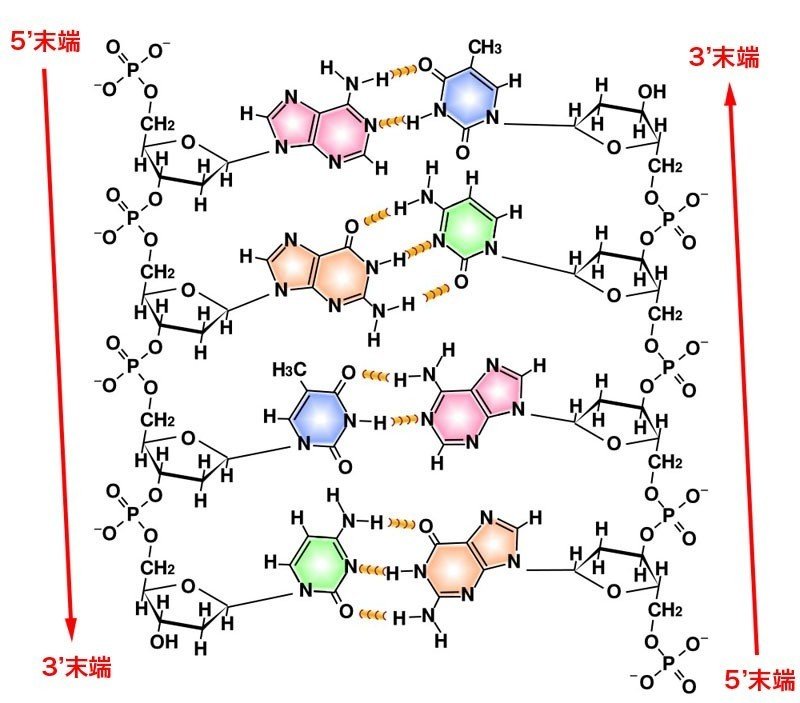
DNAを構成する2本の鎖は逆平行になっています。
5’末端→3’末端という方向性があり、一方の鎖の5’末端側には他方の鎖の3’末端側が並び、3’末端側には他方の鎖の5’末端側が並びます。
この2本の鎖を、それぞれセンス鎖(5’–––––3’)とアンチセンス鎖(3’–––––5’)と呼びます。
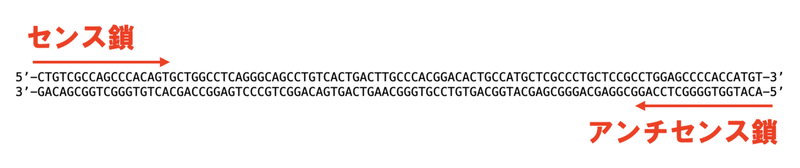
PCRでは、1つのプライマー(赤色)が鋳型DNAのアンチセンス鎖に、もう1つのプライマー(青色)が鋳型DNAのセンス鎖に結合することで、この区間の配列が増幅されます。
この位置関係が大事です!
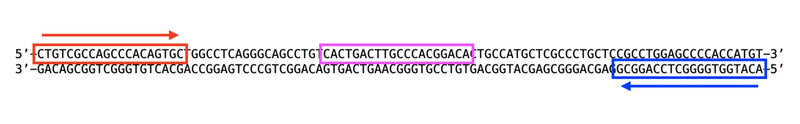
さらに、リアルタイムPCRであれば、TaqManプローブ(ピンク色)がその間に結合しなければ検出できません。
これをRNAのデータベースの中から発見したら大したものですね。
堂々と「新型コロナウイルスが感染していなくても『陽性』になる!」、「PCR検査はインチキ!」と主張できますよ。
バズること間違いなしです!大橋眞氏もYouTubeで取り上げてくれるでしょう。
歴史に名を残しませんか?
私にTwitterで絡んでくる人は基本的に自分で調べようとしない人が多いです。

たまには自分で調べましょう。
私が嘘をついているかもしれませんよ?笑
自分でデータベースを検索して「あぁ、ないんだ」と理解することが”深い理解”に繋がります。
「面倒だ...」と思うか、「面白そう!」と思うかは自分次第!
今回も、最後までご覧いただきありがとうございました。
ではまた。
追記)WHOのプライマーはゴミクズです。プライマー設計には、その特異性を高めるために、いくつかの守るべき『ルール』があります。そのうちの一つが『3'末端がTになる配列は避ける。』ですが、WHOのリバースプライマーは「5’-CTCCCTTTGTTGTGTTGT-3’」ですから守れていません。
おい!WHO!こんなプライマーをよく恥ずかしげもなく全世界に公開できるな!しっかりしろよ!!(全く期待してないけど。笑

リアルタイムPCRのプライマー設計について
https://catalog.takara-bio.co.jp/product/basic_info.php?unitid=U100004242
この記事が気に入ったらサポートをしてみませんか?
