CourseraWeek8で紛らわしかったところ(K-mean, PCA)
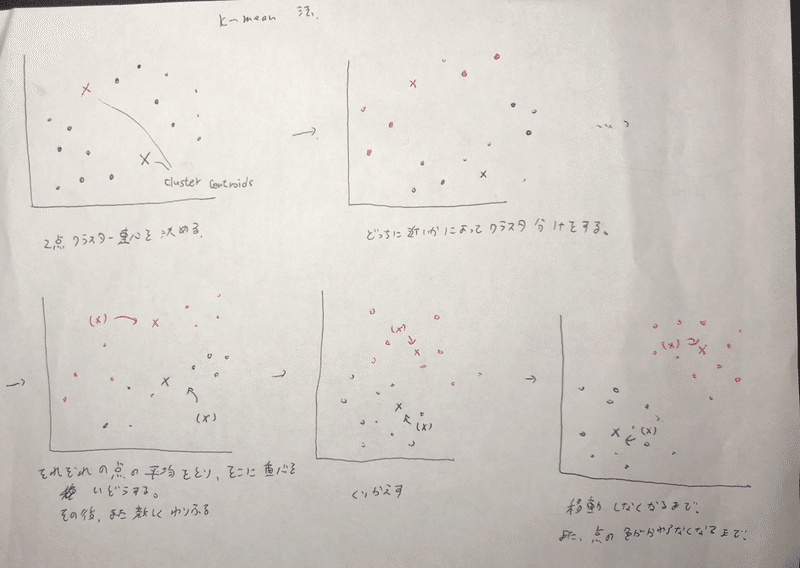
K-mean法
教師なし学習で、クラスタリングの一つ。
1.クラスター重心(centroid)を決める。(上記は2点)
2.どっちのcentroidに近いかで、クラスタ分けをする。
3.それぞれのクラスタの値の平均をとり、その平均の場所にcentroidを移動させる。
1~3を繰り返し、移動しなくなったらおわり。
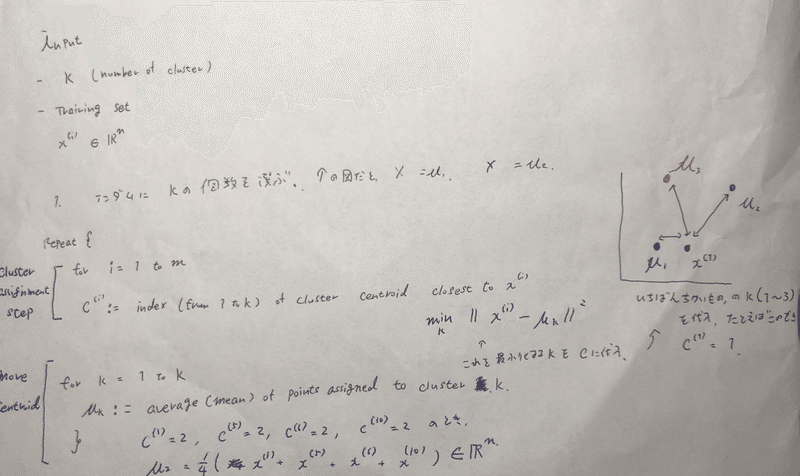
Cluster centroid を決めるアルコリズムは、、
repeat{
for i = 1 to m
c(i) <- index( from 1 to k ) of cluster centroid closest to x(i)
つまり、min || x(i) - μ(i)|| をとるKをCに代入する。(K= μ(1),μ(2),,,,,,)
上の右の方の図だと、x(1)は、μ(1)にもっとも近いので、c(1) = 1 となる。
また、cluster centroid を動かすステップのアルコリズムは、、
for k = 1 to k
μ(n) <- average(mean) of points assigned to cluster K.
}
たとえば、C(1)=2, C(5)=2, C(6)=2, C(10)=2 だった時、
μ(2) <- 1/4 *( x(1) + x(5) + x(6) + x(10) )

ちなみに、
C(i) = x(i)がどのクラスターに分けられているか表す。
μk = クラスターKの場所を表す。
μc(i) = x(i)に割り振られている(x(i)を囲んでいる)クラスターを表す。
また、目的関数は、
J = ( C(1)~C(m), μ(1)~μ(k)) = 1/m * || x(i) - μc(i)||^2
であり、c (1~m), μ(1~k)をトレーニングする。(出す)
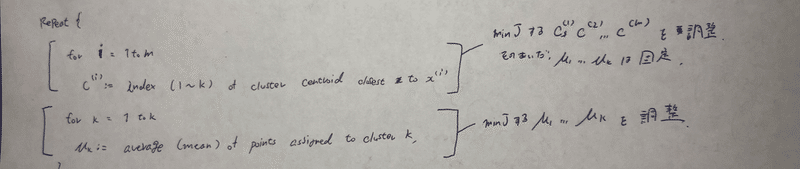
アルコリズムの中で同時にCとμを計算するのではない。
repeat{
for i = 1 to m
c(i) <- index(1 ~ k) of cluster centroid closest to x(i)
Cluster centroid を移動させるステップで、minJするc(1~m)を調節。その間μ(1~k)は固定。
for k = 1 to k
μk <- average (mean) of points assigned to closest k
Cluster centroid をクラスターの平均に移動させるステップでminJするμ(1~k)を調節。
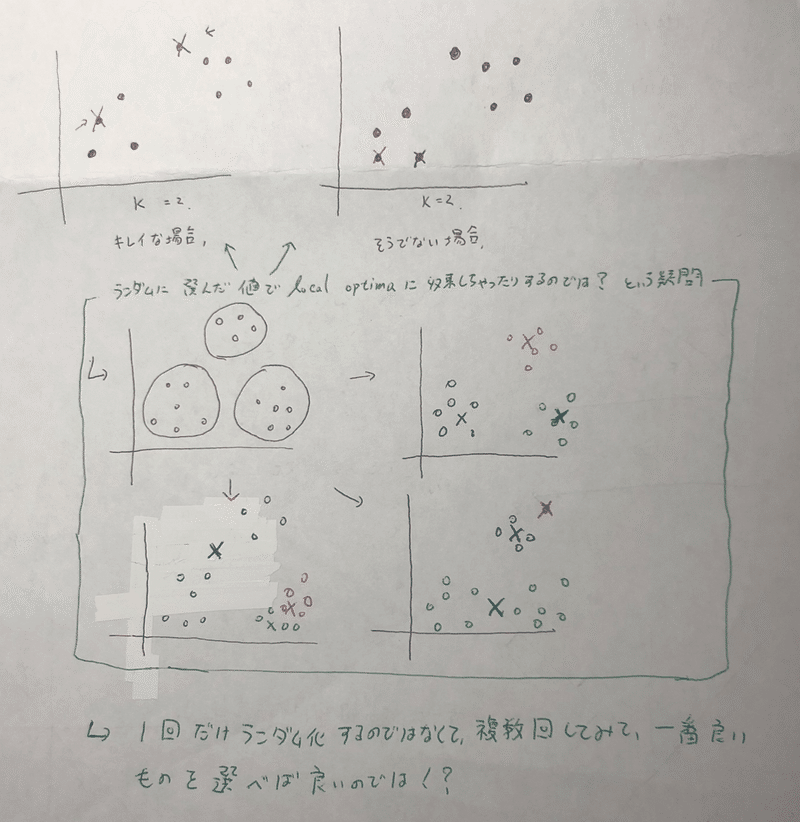
てか、キレーに点が分かれていたらいいけど、そうでない場合は Local optimaに収束しちゃわないか?という疑問が出る。実際ローカルな値に収束してしまうことがある。
→では、initialの値を一回だけランダム化してアルコリズムを回すのではなく、複数のinitialの値でアルコリズムを回してみて、一番良いものを採用しようではないか。

これがそのアルコリズム。
for i = 1 to 100 {
Randomly initialize K-means,
Run K-means , Get C(1~m), μ(1~K),
Compute cost function J
}
100回、回しているから、100個のJが得られる。その後一番小さい値のJを選ぶ。
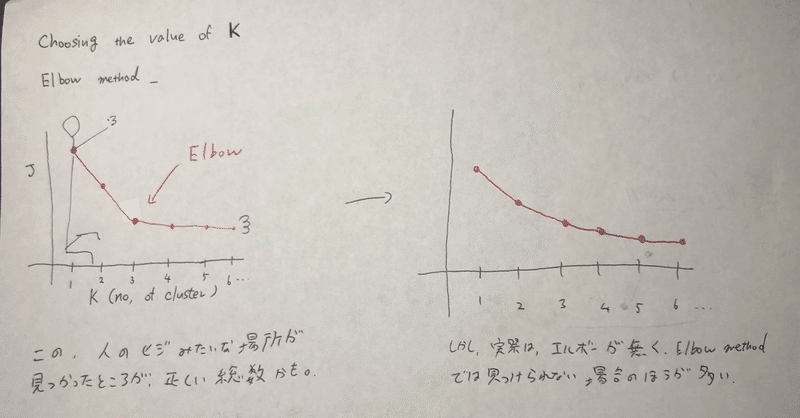
で、クラスター数(number of k) どうするよという疑問が生まれる。
Elbow method というものが存在する。Kの個数をx軸、Jをy軸にプロットして、ニンゲンの肘レベルで折れ曲がっているところがあれば、そこが最適なkの個数カモね。
・・だけど、現実にはエルボーが存在することは稀で、あまりElbow method は使われない。
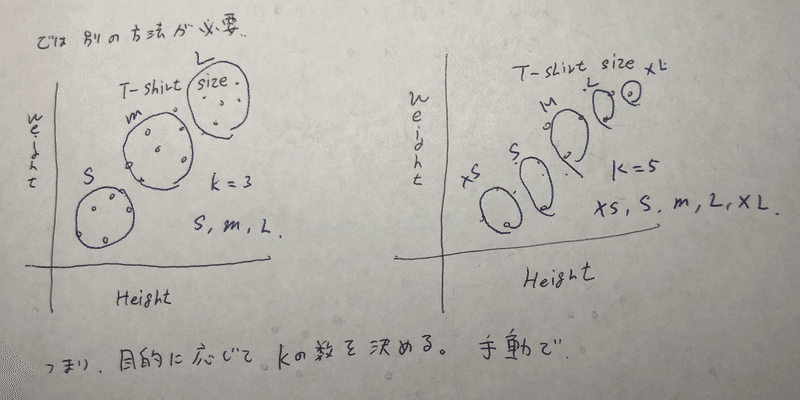
じゃあ実際どうやってkの個数を決めているか。
現在のところ、高度なアルコリズムはなく、実際の(ビジネスなどの)目的によって決めている。
たとえば、TシャツのサイズをS、M、Lと三分割したければ、kの個数は3だし、xs、s、m、l、xlとしたければkは5つ必要である。
→目的に応じて主観的にkの個数を決める
PCA_____
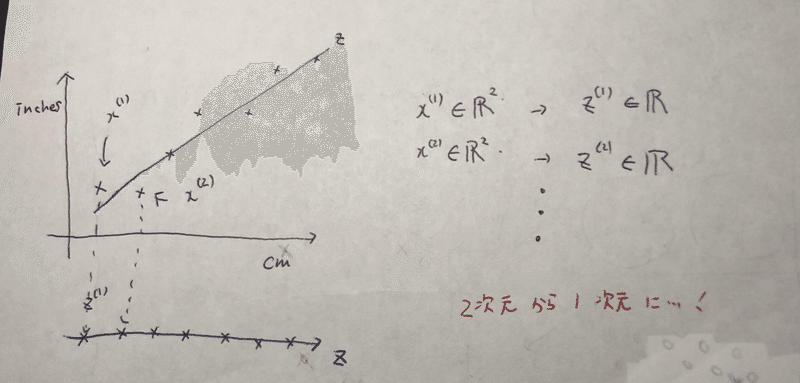
PCA とは、次元を下げるメソッド。
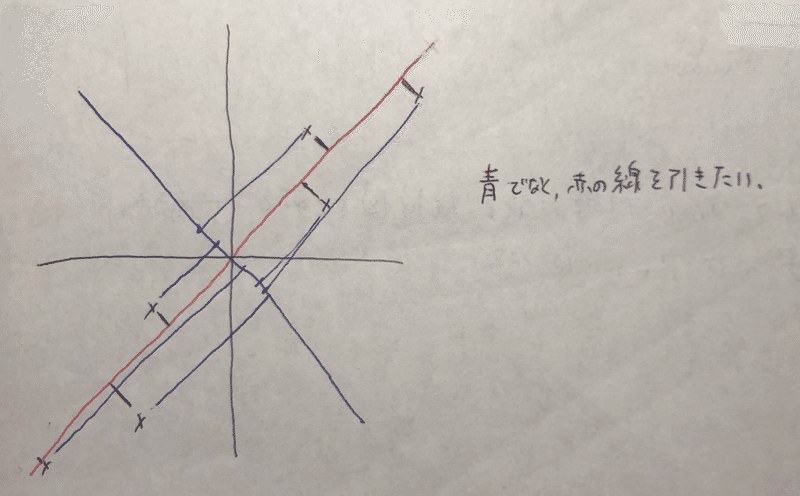
青の線でなく、赤の線を引きたい。つまり、それぞれの距離がminとなるような線を選びたい(最小二乗法的な・・?)
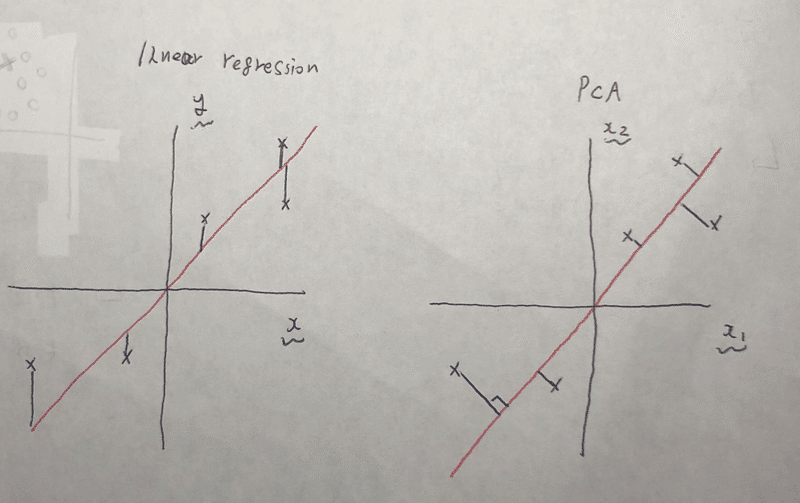
ただ、最小二乗法とは全く違うアルコリズムである。
図的に説明すると、左が最小二乗法で右がPCA。
また、最小二乗法ではx、y軸があるのに対して、PCAはx1、x2軸しかない。つまり、最小二乗法はyの予測を念頭に置いているが、PCAは目的変数などない。根本から違うアルコリズムである。
また、PCAを行う際は、必ずヒューチャースケーリングを行う必要がある。
PCAの流れとしては(ノート取ってない・・)
1.Σ(共分散行列)を計算する (sumではない、注意)
2.Σ(共分散行列)の固有ベクトルを計算する。
3.計算した固有ベクトルを用いてn次元からk次元に圧縮したベクトルzを計算する。
1.Σ(共分散行列)の計算。
Σ = 1/m * Σ(1~m) {x(i) * x(i)'}
vector ver..
Sigma = (1/m) * X' * X
2.共分散行列Σの固有ベクトルを計算する。
[U, S, V] =svd(Sigma)
ちなみにUが固有ベクトル。Σと同様n*n行列。
3.Uの最初のk列を取り、n次元からk次元に圧縮したベクトルkを計算する。
z(i) = U_reduce' * x(i)
U_reduce'はk*n行列で、xがn*1行列だから、zはk*1行列になる。
コードだと、
Ureduce = U( : , 1:k )
Z = X * Ureduce
ここまで一気にまとめると、、
Sigma = (1/m) * X' * X 共分散行列を求める
[U, S, V] = U( : , 1:k ) 固有ベクトルを求める
z = X * Ureduce 圧縮したzを出す。
圧縮したk次元からn次元に戻すには__
z(i) = Ureduce' * x(i) だから
x(l)_approx = Ureduce * z(l)
いくつの次元まで圧縮するか__圧縮する次元数の決め方
二乗射影誤差と全体の分散の比率を取って何%未満とする(たとえば、1%未満なら、99%の主成分が保持されていることになる)
1.1/m Σ(1~m) { || x(i) - x_approx(i) ||^2} ...① 二乗射影誤差を求める
2.1/m Σ(1~m) { || x(i) || ^2 } ...② 全体の分散を求める。
3.① / ② <= 0.01 を満たす最小のk次元を求める。
具体的には、
[ U, S, V ] = svd(Sigma) を呼び出し、Sを用いる。
Σ(1~k) Sij / Σ(1~n) Sij >= 0.99 を満たす最小のkを求める。当てはまらなければ少しずつkの値を増やしていく。
PCAを使うときに気を付けること__
PCAはトレーニングセットにのみ行う。PCAの主な用途は、次元を減らすことでのスピードアップ、メモリの削除、データの可視化(2、3次元のグラフを描くため)である。したがって、次元を減らすことによる over fitting を均すために使用するのは間違った使い方。あくまでもover fitting を防ぐなら正則化を使う。
課題__
1.findClosestCentroids
FINDCLOSESTCENTROIDS computes the centroid memberships for every example
idx = FINDCLOSESTCENTROIDS (X, centroids) returns the closest centroids
in idx for a dataset X where each row is a single example. idx = m x 1
vector of centroid assignments (i.e. each entry in range [1..K]) らしいです。
idx にそれぞれのデータに最も近いcentroidを保存させるらしいです。
function idx = findClosestCentroids(X, centroids)
K = size(centroids, 1)
idx = zeros(size(X,1),1)
for i = 1 : size (X, 1)
min_c = inf
for j =1 : K
c =(X( i ,: ) - centroids( j ,: )) * (X( i , : ) - centroids( j , : ))'
if c <= min_c
min_C =c
idx(i) = j
end
end
end
2.computeCentroids
それぞれのクラスターの平均値を出し、centroidsを移動させていくステップです。
function centroids = computeCentroids(X, idx, K)
[m n] =size(X)
cemtrpods = zeros(K, n)
---- your code here----
for k = 1:K
sum_X = zeros(1,n);
count = 0;
for i = 1:m
if idx(i) == k
sum_X(1,:) = sum_X(1,:) + X(i,:);
count = count + 1;
end
end
mean_X = 1/count*sum_X;
centroids(k,:) = mean_X;
end
3.kMeansInitCentroids
最初のcentroidsを決めます。
1.training exampleをランダムに並び替え
2.最初のk個をinitial centroids に
function centroids = kMeansInitCentroids(X, K)
centroids = zeros ( K, size(X,2))
---your code here---
random_idx = randperm(size(X, 1)) 1.ランダムに並び替え
centroids = X(random_idx(1 : k ), :) 2.最初のk個をとる
4.pca
XでPCAを回す課題です。そのまま。
function [U, S] = pca(X)
[m , n] = size(X)
U = zeros(n)
S = zeros(n)
---your code here---
Sigma = 1/m * (X)' * X
[U, S, V] =svd(Sigma)
5.project data
Uの最初のk個を取ってPCAを回します。
function Z = projectData( X, U, K)
Z = zeros (size(X, 1), K)
--- your code here----
U_reduce = U( : , 1 : K ):
Z = X * U_reduce:
end
6.recoverData
5で作ったzをもとに戻します。
function X_rec - recoverData (Z, U, K)
X_rec = zeros(size(Z, 1), size(U, 1))
---your code here--
for i = 1 : size(U,1)
X_rec( : , i ) = Z * U( i , 1:K )'
end
この記事が気に入ったらサポートをしてみませんか?
