
【ImageJ】画像ファイルの形式を意識して開く
生命科学系研究者のえいこです。「画像ファイルの形式」って意識したことありますか?画像ファイルにはtifやjpg、pngなど様々な種類があることはご存知だと思います。でも、それぞれどう言う風に違うのか?と言われたら曖昧な方も多いのではないかと思います。
私は、とりあえず画像がキレイだから(ファイルサイズが大きいから)、顕微鏡で撮影すると大抵このファイル形式で保存されるからと言う理由で"tif"ファイルで保存していました。最近は、どの会社の顕微鏡で撮影するかによってファイル形式が異なっていることがよくあります。(Licaの顕微鏡で撮影すると"lif"、CarlZeissの顕微鏡で撮影すると"czi"など...)
今回は、画像のファイル形式が違うと何が違うのか?を中心にまとめておこうと思います。参考にした教科書はこちら。
tif, jpg, pngって何が違うの?
まずは、基本的な画像形式について押さえておきましょう。画像データは基本的には「数値データ」なので、ファイル形式を意識するのは画像を保存する時だと思います。
よく使われるtif, jpg, pngは「数値データ」なのは一緒なのですが、保存する時に「圧縮されるかどうか」が異なると言うことを覚えておきましょう。
画像を圧縮するとは?
簡単に言うと、画像を表示させるときの情報を減らすと言うこと。JPEGでは画像の情報を、「8画素×8画素」のブロックに小分けにして数値の並びをどう言う風に並んでいるかと言う情報に変換(離散コサイン変換)します。ブロックごとになるのでその境目がノイズとして検出されることもあるようです。写真などでは色の情報を落とすことで圧縮されます。
「ファイル形式」が重要だと言う具体的な例を見てみましょう。学会発表用の資料を作る時、共同研究者の先生にデータを報告するときなどはファイルサイズが小さく、一般的なソフトで開ける画像形式が良いです。逆にデータ解析をする時には圧縮されない・可逆的な圧縮形式のファイルの方が正確に解析ができます。ファイル形式と圧縮、データサイズに関しては下の表にまとめてみました。

顕微鏡で写真を撮ると大抵"tif"ファイルで保存されますよね。そして上の表には「種類が多い」と書きました。tifファイルは"TIFF(Tagged Image File Format)"で"タグ"を付け加えることで様々な情報を盛り込むことができるようになっています。
顕微鏡会社によってファイル形式が異なるワケ
顕微鏡で撮影するとまずは"tif"ファイルで保存されます。同じ"tif"の一種なのですが、冒頭で出てきた通り顕微鏡会社によってファイル形式が異なっています。これは、画像の数値マトリックス以外の情報が違うからファイル形式が異なるのです。
tifファイルはタグを付け加えることで様々な情報を盛り込むことができると言うことでした。最近では、顕微鏡で撮影すると画像データの他に、「データに関する高次情報(メタデータ)」も保存されるようになっているのです。メタデータの例としては、スケール、露光時間、撮影したレンズの種類、チャネルなど。
ImageJで画像のメタデータを確認する
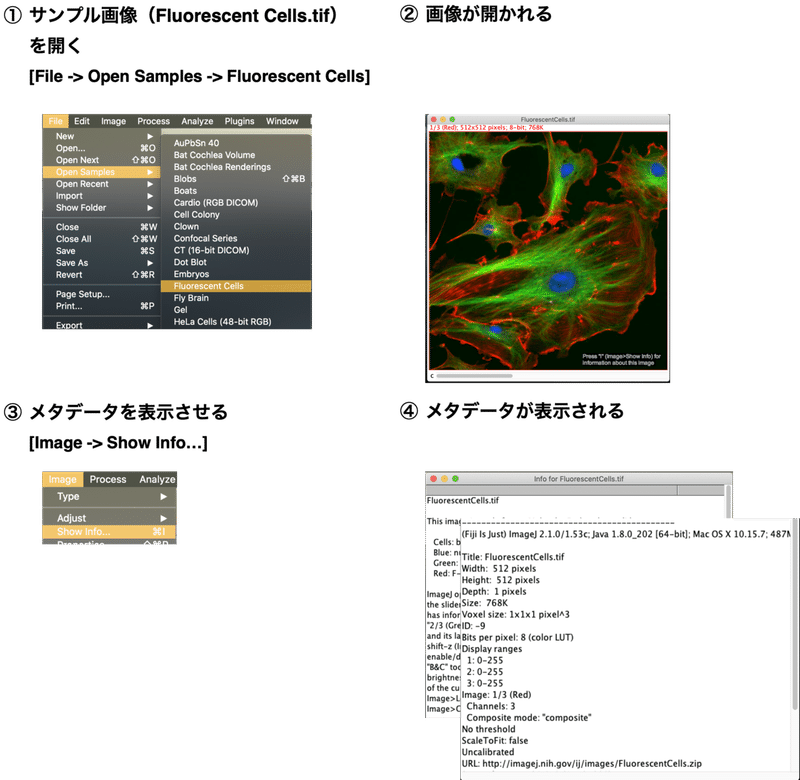
このメタデータの書き込み方の違いで顕微鏡会社ごとにファイル形式が異なるのです。でも、それぞれの顕微鏡で撮影したものを撮影した機械のところへ行って解析しなければいけないのは大変ですよね。
ImageJでは異なるファイル形式のデータを統一されたフォーマット"OME-tiff"で表示できる"LOCIツール"があります。"LOCIツール"で画像を開いてみましょう。
"LOCIツール"を使って顕微鏡会社独自のファイル形式の画像を開く
先ほどさらっと書いてしまいましたが、顕微鏡会社ごとの独自フォーマットを統一する"OME-tiff (Open Microscopy Environment)"と言う規格が作られました。ImageJに入っている"LOCI(Laboratory for Optional and Computational Instrumention)ツール"はOME形式で開くためのツールです。
LOCIツールを使えば、顕微鏡会社ごとに設定されている独自のパラメータを統一し、メタデータの構造を標準化された構造に変換して表示することができます。
実際にImageJでLOCIツールを使ってみましょう。
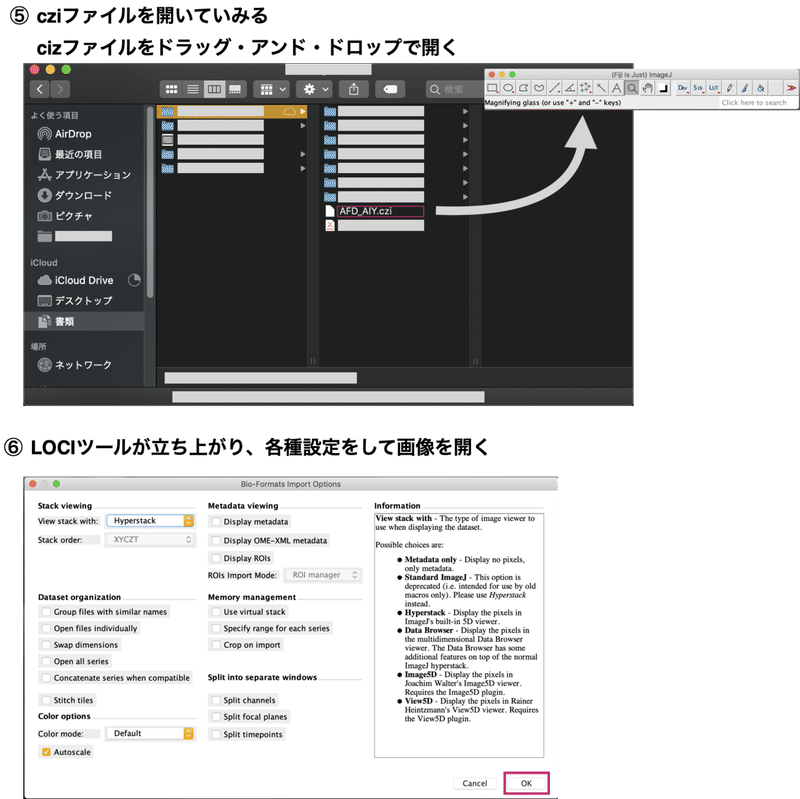
このように、ファイルをドラッグ・アンド・ドロップすると自動的にLOCIツールが表示されますが、自動的に表示されない場合は[Plugins]からも起動できます。
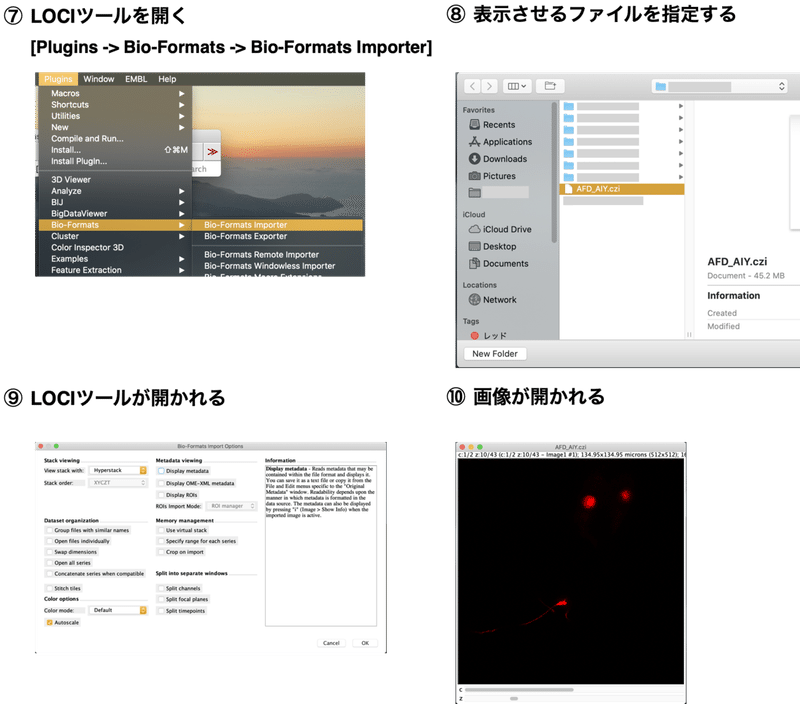
LOCIツールの画面を少し補足しておきましょう。
Stack viewing
開いた多次元データをどのように表示するかを選択できる。Z軸方向(深度)、t軸方向(時間軸)、c軸方向(色軸)がどの順番で並んでいるかを指定することができる。
Dataset organization
1枚ずつ保存されている画像多次元データをどのように扱うかを選択できる。
Color options
色情報の選択肢。Autoscaleのチェックを入れておくと、表示するときに自動的に輝度を調整して表示してくれる。(元の数値データは変更されない)
Metada viewing
メタデータを表示する選択肢。
Memory management
多次元データはメモリを消費しやすい。メモリを節約する時にはこちらを選択する。"Use virtual stack" はメモリサイズを超えるような大きなファイルを開くときに便利な機能。画像全体ではなく、一部がメモリに読み込まれ、操作に応じて自動的にハードディスクから読み込まれる。Tiffファイルに関してはもともと設定されている。
Split into separate windows
特定の次元で別々にデータを開くことができる。
cziファイル以外にもLicaで取得できるlifファイルも同様にLOCIツールを使って開くことができます。
今回使用したcziファイルはCMCI-EMBLプラグインに入っているサンプル画像を使用しました。サンプル画像の取得の仕方はこちら(事前にCMCI-EMBLプラグインを入れておきます)↓
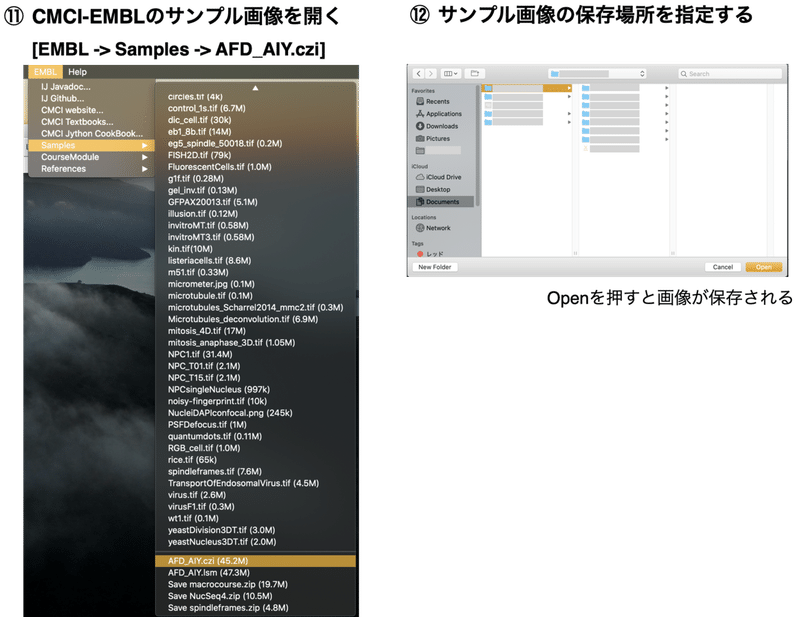
実際に自分で撮影した画像で、LOCIツールを使って画像を開いてみてください。LOCIツールの存在を知っておけば、顕微鏡で撮影した画像の保存時にメーカーフォーマット+汎用画像データ(.tifや.jpgなど)を保存する必要がなくなりPCもスッキリするのではないでしょうか?解析もできるようになれば自分のPCでImageJ一つあれば作業ができるというありがたさ!
と言うことで、今回はLOCIツールを使って画像を開いていくことをしてきました。が、画像の形式を意識するのは「画像を保存する時」でしたね。解析が終わった画像をファイル形式を指定して保存する場合は、[File -> Save As...]でファイル形式を選んで保存するだけ。これでPCに保存されるデータ量がグッと減るのではないでしょうか。
次回は、多次元画像データについて勉強していきたいと思います。
それでは、また!
最後までお読みいただきありがとうございます。よろしければ「スキ」していただけると嬉しいです。 いただいたサポートはNGS解析をするための個人用Macを買うのに使いたいと思います。これからもRの勉強過程やワーママ研究者目線のリアルな現実を発信していきます。
